限制性片段长度多态性实验——
- 格式:docx
- 大小:45.90 KB
- 文档页数:2
限制性片段长度多态性(Restriction Fragment of Length Polymorphism,RFLP)一基本原理各种限制性酶能识别特定的碱基序列,并将其切开。
碱基的变异可能导致切点的消失或新切点的出现,从而引起DNA片段长度和数量的差异。
用特定的限制性内切酶消化目标DNA并通过电泳将长度不同的片断分开,并印记于硝酸纤维滤膜上,再与相应的探针杂交,就可以检测限制性片段长度多态性(RFLP).二主要材料DNA抽提用试剂,限制性内切酶,dNTP及Taq聚合酶电泳用琼脂糖或聚丙烯酰胺配制试剂,PCR扩增仪,水浴锅,电泳仪和电泳槽,硝酸纤维素滤膜或尼龙膜,探针标记物等。
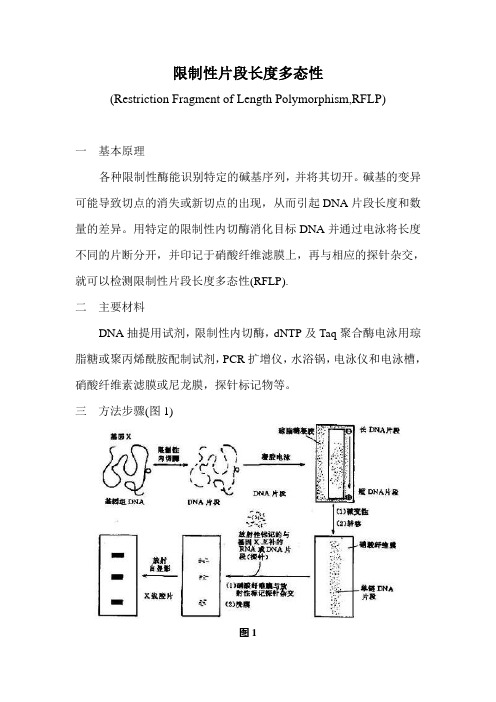
三方法步骤(图1)图1(一) 样本DNA制备采用常规DNA抽提的方法或DNA抽提试剂盒提取样本DNA,置低温下保存。
对大多数样本而言,用于分析的样本DNA片段须先从总DNA中分离获取,并制备足够的量。
为此,RFLP分析常先用PCR方法扩增目标片段。
无论怎么做,必须保证DNA样本的纯度,这一点是非常重要的。
(二) 限制性内切酶降解样本DNA根据不同的目标DAN,选择合适的限制性内切酶。
目前常用的限制性内切酶有EcoRⅠ和HindⅢ等。
该步骤必须保证酶解完全。
如果有必要,可以用琼脂糖凝胶电泳溴化乙锭分析酶解结果。
酶解的时间根据实际情况而定。
(三) 电泳电泳的主要目的是把DNA片段按大小(长短)分离开来,得到一个根据分子量排列的连续带谱。
电泳可采用琼脂糖凝胶电泳,也可采用聚丙烯酰胺凝胶电泳。
时间由几小时到24小时不等。
(四) 转印所谓转印,就是将已经电泳的DNA片段通过一定的方法转到固相支持物上。
常用的固相支持物有硝酸纤维素滤膜或尼龙膜。
转印前需经过碱变性溶液处理,将双链DNA变性为单链DNA。
转印的方法一般有三种。
根据DNA分子的复杂度转移2―12小时。
1 盐桥法;也叫毛细管转移法。
先把硝酸纤维素滤膜放在20×SSC 溶液中浸透,然后把滤膜平铺在凝胶上,再在滤膜上放上浸过20×SSC溶液的滤纸3张,再在该滤纸层上铺上干燥的滤纸3张,由于滤纸的吸附作用,胶下的缓冲液透过凝胶被吸附上来,同时胶中的DNA 分子就转移到滤膜上。

中国农业大学学报 2009,14(4):1-9Journal o f China A g ricultur al U niv ersity末端限制性片段长度多态性技术(T -RFLP)在微生物群体分析上的应用与技术优化李献梅 王小芬 崔宗均*(中国农业大学农学与生物技术学院/中国农业大学生物质工程中心,北京100193)摘 要 末端限制性片段长度多态性技术(T-RFL P)是以荧光标记引物P CR 为基础,根据末端限制性片段长度区分出微生物群体组成的一种微生物群体图谱法。
本文综述了该方法在群体微生物分析上的应用及DN A 提取、PCR 扩增、酶切和数据分析等步骤的技巧与优化,总结了科学应用该技术的要求以及和多重PCR 、gy r B 基因等相结合的观点。
关键词 T -RF LP ;微生物群体;图谱分析;PCR;克隆中图分类号 Q 936 文章编号 1007-4333(2009)04-0001-09 文献标志码 A收稿日期:2008-10-27基金项目:国家/十一五0科技支撑计划(2006BAD07A 01;2006BA D25B04)第一作者:李献梅,博士研究生,E -mail:staro fchina@g 通讯作者:崔宗均,教授,主要从事生物质资源利用与微生物生态研究,E -mail:acuizj@Applications and optimizations of T -RFLP in fingerprintingenvironm ental microbial populationsLI Xian -mei,WANG Xiao -fen ,CUI Zong -jun *(College of Agronomy and Biotechnology/Center of Biomas s Engineeri ng,Chi na Agricul tural U nivers i ty,Bei jing,100193,China)Abstract Terminal re stric tio n fra gment leng th polymorphis m (T -RFLP)is one o f micro bia l popula tio n fing erprinting me tho ds tha t is ba sed o n labele d primers -PCR to ide ntify microbial co mposition a ccording to diffe re nt terminal re stric tio n frag me nt leng ths.The applic ations and te chnica l o ptimizations we re o ve rv iew ed o f T -RFLP thro ugh the pro cedures of DNA preparation,PCR,enzyme dig estion and da ta analysis.The sc ientific applic ation re quire me nts and new view s of combination with multiple -PCR o r g yr B g e ne we re dra wn.Key words T -RFLP;microbial po pulation;fing erprinting ana lysis;PCR;clone libra ry当前环境微生物组成及生态研究已经成为微生物领域的一大热点[1]。

第9章 动物DNA限制性片段长度多态性分析9.1 动物DNA限制性片段长度多态性基本概况DNA限制性片段长度多态性(restriction fragment length polymorphism,简称RFLP)是进行动物物种分化和群体遗传多样性研究的一项重要经典技术,而线粒体DNA和核糖体DNA 则是动物RFLP研究中最为常用的两种遗传标记(Nei,Tajima 1981)。
动物线粒体DNA是一种共价闭环的环状双链DNA,具有严格的母系遗传方式,每个细胞中有 1 000~10 000个拷贝,容易从组织中分离、提纯(Brown等 1979;Avise等 1983;Lansman 1983)。
提取线粒体DNA的实验技术比较简单,且重复性好(王文,施立明 1993)。
mtDNA的基因结构比较简单、稳定,分子量小(15.7~19.5kb),处于限制性内切酶分析范围,因此易于进行结果分析(Brown 1979)。
在脊椎动物中,mtDNA无组织特异性,即个体内的线粒体DNA具有高度的均一性(Avise等 1983),这就有利于限制性内切酶分析。
更为重要的是,mtDNA进化速度快,是单拷贝核DNA的5~10倍,因而是进行近缘种间和种内群体间分化关系研究的良好遗传标记(Brown等 1979,1983;Wilson 1985;Avise 1986;Harrison 1989)。
生物多样性研究中基本保护单元----进化显著性单元(evolutionary significant units,简称ESUs)(Ryder 1989)的确定,就极大地依赖于不同动物群体线粒体DNA单倍型的系统发育关系(Moritz 1994)。
ESUs的定义为:在mtDNA单倍型上互为单系群(monophyly)、在核基因座位上等位基因频率有显著差别的群体即称为ESUs。
强调mtDNA的互为单系群,不仅因为它在进化上的重要性,而且因为理论上和模拟实验均说明隔离群体在一定时间后能达到这种状态。


作物基因定位的途径一、引言作物基因定位是指确定作物基因在染色体上的位置和位置与性状之间的关系。
它是作物遗传学和分子育种的基础,为作物改良提供了重要的理论和技术支持。
本文将从不同角度探讨作物基因定位的途径,并介绍它们的优缺点和应用情况。
二、作物基因定位的途径1.传统遗传学方法•遗传连锁图法:通过分析遗传连锁图,确定染色体上基因的相对位置。
主要方法有三点配子试验法、二点试验法和多点试验法。
•遗传映射法:利用遗传连锁图和群体杂种图,确定基因的绝对位置。
主要方法有连锁不平衡分析和融合连锁分析。
2.分子标记辅助选育方法•RFLP(限制性片段长度多态性):通过分离、纯化和测定限制性酶切片段,将其与遗传连锁图相结合,实现基因定位。
•SSR(微卫星标记):通过PCR扩增微卫星DNA片段,检测其在不同位点的长度变异,确定基因位置。
•SNP(单核苷酸多态性):通过测定基因组中特定位置上的单核苷酸变异,将其与遗传连锁图相结合,实现基因定位。
•AFLP(扩增片段长度多态性):通过PCR扩增剪切DNA片段,将其与遗传连锁图相结合,实现基因定位。
3.基因组学方法•位置克隆法:将感兴趣的基因从新引入敲除构建中,通过其对敲除的表型进行定位。
•QTL(数量性状位点)定位法:通过连锁分析和基因表达研究,确定数量性状与基因之间的关系。
•基因组关联分析法:通过分析大量群体的基因组序列和表型数据,确定基因组区域与性状相关的位点。
三、作物基因定位的优缺点1.传统遗传学方法的优缺点•优点:简单易行,不需要复杂的实验设备和技术。
•缺点:分辨率较低,需要大量的试验材料和时间。
2.分子标记辅助选育方法的优缺点•优点:高分辨率,可以精确定位基因。
•缺点:成本较高,需要复杂的实验设备和技术。
3.基因组学方法的优缺点•优点:高通量,可以同时分析大量基因和性状。
•缺点:分析结果复杂,需要强大的数据分析能力。
四、作物基因定位的应用情况作物基因定位的途径已经在作物改良中得到了广泛应用。
基因多态性的检测方法多态性(polymorphism)是指处于随机婚配的群体中,同一基因位点可存在2种以上的基因型。
在人群中,个体间基因的核苷酸序列存在着差异性称为基因(DNA)的多态性(gene polymorphism)。
这种多态性可以分为两类,即DNA位点多态性(site polymorphism)和长度多态性(longth polymorphism)。
基因多态性的主要检测方法简述如下:1.限制性片段长度多态性(Restriction Fragment Length Polymorphism,RFLP):由DNA 的多态性,致使DNA 分子的限制酶切位点及数目发生改变,用限制酶切割基因组时,所产生的片段数目和每个片段的长度就不同,即所谓的限制性片段长度多态性,导致限制片段长度发生改变的酶切位点,又称为多态性位点。
最早是用Southern Blot/RFLP方法检测,后来采用聚合酶链反应(PCR)与限制酶酶切相结合的方法。
现在多采用PCR-RFLP法进行研究基因的限制性片段长度多态性。
2.单链构象多态性(SSCP):是一种基于单链DNA构象差别的点突变检测方法。
相同长度的单链DNA如果顺序不同,甚至单个碱基不同,就会形成不同的构象。
在电泳时泳动的速度不同。
将PCR产物经变性后,进行单链DNA凝胶电泳时,靶DNA中若发生单个碱基替换等改变时,就会出现泳动变位(mobility shift),多用于鉴定是否存在突变及诊断未知突变。
3.PCR-ASO探针法(PCR-allele specific oligonucleotide, ASO):即等位基因特异性寡核苷酸探针法。
在PCR扩增DNA片段后,直接与相应的寡核苷酸探杂交,即可明确诊断是否有突变及突变是纯合子还是杂合子。
其原理是:用PCR扩增后,产物进行斑点杂交或狭缝杂交,针对每种突变分别合成一对寡核苷酸片段作为探针,其中一个具有正常序列,另一个则具有突变碱基。
医学分子生物学简答题基因与基因组真核生物基因组特点(1)分子量很大的DNA与蛋白质形成染色体存在于核内。
约30亿bp(2)转录与翻译不同步,转录产物为单顺反子,功能相关基因分散排布,无操纵子结构。
(3)基因组存在高比例的非码顺序(non coding sepuence, NCS)(4)大量重复序列(repeat sequence)存在。
(5)基因多为不连续的,被插入序列(IS)所分隔,这种现象称为断裂基因(6)功能相关基因构成各种基因家族2.评判蛋白质编码基因的五项标准(1)开放阅读框(open reading frame, ORF):是始于起始密码子并终于终止密码子的一串密码子。
;(2)序列特征——密码子偏爱和剪接点;(3)序列保守性:一个功能在不同物种内是保守的基因,大都表现出外显子的保守性和内含子的多变性;最好是对适当进化距离的物种间序列进行比较,但保守性序列也可能是非转录的调控单元。
(4)转录产物:寻找基因的表达产物——RNA或蛋白质.;(5)基因失活。
3.HGP的概念及主要内容、遗传标记答:HGP:测定人类基因组全序列为目标的巨大工程主要内容:(1)人类基因组作图及序列分析(22+X+Y, 30亿bp);(2)基因的鉴定与定位(约5万个基因);(3)基因组研究技术的建立和创新;(4)模式生物基因组作图及序列分析;(5)信息系统的建立、储存及相应软件的开发;(6)相关产业的开发。
遗传标记:第一代遗传标记是RFLP(restriction fragment length polymorphism,限制性酶切片段多态性)第二代遗传标记是STR(short tandem repeat,简短串连重复序列),6000个标记,96年已全部完成第三代遗传标记是SNP(restriction fragment length polymorphism,单核苷酸多态性),不再是分析长度,而是直接测序4.基因组学主要研究亚领域和主要研究内容答:5.DNA多态性的种类,并熟悉其应用答:1)DNA位点多态性(DNA site polymorphism):这种多态性是由控制某些性状的DNA碱基差异造成的。
实验六■限制性片段长度多态性
(Restriction Fragment Length Polymorphism, RFLP)
—、实验目的
学习和掌握限制性片段长度多态性(RFLP )遗传标记的基本原理和检测方法。
二、实验原理
第一代分子遗传标记RFLP是根据不同品种(个体)基因组的限制性内切酶的酶切位点碱基发生突变,或酶切位点之间发生了碱基的插入、缺失、重排等,导致酶切片段大"俊生了变化,这种变化可以通过
PCR ,酶切及琼脂糖凝胶电泳逬行检测,从而可比较不同品种(个体)的DNA水平的差异(即多态性),RFLP已被广泛用于基因组遗传图谱构建、基因走位以及生物进化和分类、遗传多样性等研究。
三、仪器、材料与试別
(-)仪器:电热恒温水浴锅,琼脂糖凝胶电泳及检测系统
(二)材料与试列:Hind JU限制性内切酶,10xM Buffer,待分析的PCR产物,DL2000 DNA Marker, 6xLoading Buffer (上样缓冲液),灭菌双蒸水,1.5%琼脂糖凝胶(注意:含溟化乙锭EB ,致癌,请带手套操作),0.5%TBE (电泳缓冲液1
四、实验步骤
1、Hind m限制性内切酶酶切
反应体积(10pl )f在0.2ml Eppendorf管中依次加样:
10xM buffer i ML
ddH2O 5.5 pL
Hind 皿0.5 pL
PCR产物 3 ML
37°C水浴消化2h ,消化产物全部用于琼脂糖检i则。
2、琼脂糖凝胶电泳
配置1.5%琼脂糖凝胶,取10pl酶切产物+1川上样缓冲液280V恒压电泳。
DNA带负电荷, 电泳时
DNA从负极向正极泳动。
待泳动至凝胶的1/2-2/3位置时,停止电泳,紫外检测仪下观察。
五、作业
写出本实验的具体操作过程并描述你所观察到的结果。
(画图)
M12 34 5678 9 10
Hind HI内切酶识别位点:AIAGCTT
TTCGATA。