基因敲除小鼠的制作方法
- 格式:doc
- 大小:27.00 KB
- 文档页数:1

基因敲除小鼠(Knockoutmice)制备技术方法基因敲除小鼠,人们使用复杂的方法使小鼠体内的某一个基因不表达,从而使小鼠呈现这个基因缺失的状态,可用于研究这个基因的功能。
但如果某个基因功能特别重要,这个基因缺失可能具有胚胎致死性,那我们就无法得到这种基因敲除的小鼠了,于是人们发明了条件性基因敲除技术。
这一技术可以实现在特定的时间、特定的细胞或组织内使某个基因沉默。
方法是首先在目的基因(就是打算敲除的那个基因)的两侧分别插入一段名为LoxP的DNA序列(LoxP序列是一段34bp的DNA序列,两端的13个碱基为回文序列,中间的8个碱基决定LoxP的方向。
然后我们需要用到一种带有Cre酶的转基因小鼠了。
Cre重组酶于1981年从P1噬菌体中发现,属于λ Int酶超基因家族。
Cre重组酶基因编码区序列全长1029bp(EMBL数据库登录号X03453),编码38kDa蛋白质。
是一种位点特异性重组酶,能介导两个LoxP位点(序列)之间的特异性重组,使LoxP位点间的基因序列被删除或重组。
LoxP(locus of X-over P1)序列来源于P1噬菌体,是有两个13bp反向重复序列和中间间隔的8bp序列共同组成,8bp的间隔序列同时也确定了LoxP的方向。
Cre 在催化DNA链交换过程中与DNA共价结合,13bp的反向重复序列是Cre酶的结合域。
其序列如下:5' - ATAACTTCGTATA - ATGTATGC - TATACGAAGTTAT - 3'3' - TATTGAAGCATAT - TACATACG - ATATGCTTCAATA - 5'Cre-LoxP系统的特性Cre重组酶介导两个LoxP位点间的重组是一个动态、可逆的过程,可以分成三种情况:1、如果两个LoxP位点位于一条DNA链上,且方向相同,Cre重组酶能有效切除两个LoxP位点间的序列;2、如果两个LoxP位点位于一条DNA链上,但方向相反,Cre重组酶能导致两个LoxP位点间的序列倒位;3、如果两个LoxP位点分别位于两条不同的DNA链或染色体上,Cre酶能介导两条DNA链的交换或染色体易位。

基因敲除小鼠的制作方法基因敲除小鼠是一种常用的遗传工具,在科学研究中被广泛应用于功能基因组学和疾病模型研究。
基因敲除是指通过特定技术手段,将小鼠体内的目标基因完全沉默或失活,从而研究该基因在发育、生理以及疾病机制中的功能。
本文将介绍基因敲除小鼠的制作方法,包括设计目标基因的敲除载体、胚胎干细胞的筛选和注射、外显子敲除策略的选择等。
1.设计目标基因的敲除载体敲除载体是嵌入目标基因的重要工具。
它通常包含正向与反向的同源臂(homology arms)以及选择标记(如抗生素抗性基因)。
同源臂的长度通常在2-5 kb之间,确保在同源重组时准确而有效地替代目标基因。
此外,敲除载体中还应该包含可诱导甲基化的Cre-loxP重组体系或者FLP-FRT重组体系,以用于后续的基因定向敲除或基因重新组装。
2.筛选胚胎干细胞胚胎干细胞是从内胚层发育而来的多潜能细胞,可以分化为整个鼠体的各种组织和器官。
敲除载体首先需要通过电转或霰粒枪等手段转染到胚胎干细胞系中。
转染后,胚胎干细胞需要进行抗生素筛选,以过滤未转染的细胞。
为了确保目标基因的敲除率,可以使用增强绿色荧光蛋白(eGFP)等标记基因,通过荧光显微镜观察转染细胞的表达情况。
3.敲除载体注射到小鼠受精卵中一旦确认胚胎干细胞中存在敲除载体,接下来就是将胚胎干细胞植入小鼠受精卵。
这个步骤一般由经验丰富的研究人员或者专业公司进行。
首先,选择合适的受精卵(通常为C57BL/6J小鼠品系),然后利用显微操作技术,将敲除载体注射到受精卵的核酸注入腔。
注射后,将受精卵转入对应营养液中培养一定时间,以期达到最佳着床率。
4.敲除鼠胚移植到配子体内经过培养后,将敲除的胚胎植入雌性激素准备好的代孕小鼠(通常为白色的株系,如ICR)。
移植后,将代孕小鼠继续养育,直至分娩。
5.验证敲除小鼠的敲除效果通过提取敲除小鼠的DNA,可以利用PCR、Southern blot和DNA测序等技术验证敲除效果。

基因敲除小鼠的实验流程
一、前期准备
1、检索标记基因:采用全基因组测序技术或大规模基因组关联分析法筛选出敲除对研究有重要作用的基因;
2、设计敲除构建:根据筛选出的基因特异性序列,对基因进行深入分析,结合已有研究成果,根据基因的功能和结构确定可有效敲除的基因结构模型;
3、制备修饰质粒:根据设计模型,制备适当的质粒,使其具有足够的重组能力和具有全套的特异性对象;
4、选择载体:选择合适的载体(含有敲除的质粒),使敲除的基因更容易被载入小鼠细胞中进行修饰;
二、基因敲除实验
1、小鼠胚胎动物模型:小鼠胚胎是敲除小鼠研究的传统动物模型,采用小鼠母体体外受精,利用载体质粒将敲除基因引入胚胎,敲除的基因将被遗传给后代小鼠;
2、小鼠嵌合体模型:采用基因修饰技术将敲除基因嵌入小鼠细胞的质粒,多功能的抗体定位蛋白可以用来将质粒载入小鼠细胞,利用抗体定位系统,将修饰的嵌入小鼠胚胎,诱导而成嵌合体,使敲除的基因能够传递给后代;
3、选择敲除后的小鼠:将敲除实验的小鼠孵化。

一、常规基因敲除鼠( Conventional Knockout )常规基因敲除是通过基因打靶,把需要敲除的基因的几个重要的外显子或者功能区域用 Neo Cassette 替换掉。
这样的小鼠其全身所有的组织和细胞中都不表达该基因产物。
此类基因敲除鼠一般用于研究某个基因在对小鼠全身生理病理的影响,而且这个基因没有胚胎致死性。
二、条件性基因敲除小鼠( Conditional Knockout )条件性基因敲除小鼠是通过基因打靶,把两个 loxP 位点放到目的基因一个或几个重要的外显子的两边。
该小鼠和表达 Cre 酶小鼠杂交之前,其目的基因表达完全正常。
当和组织特异性表达 Cre 酶的小鼠进行杂交后,可以在特定的组织或细胞中敲除该基因,而该基因在其他组织或细胞表达正常。
条件性基因敲除鼠适用范围为:( 1 )该基因有胚胎致死性;( 2 )用于研究该基因在特定的组织或细胞中的生理病理功能。
三、基因敲入小鼠( Knockin )基因敲入小鼠是通过基因打靶,把目的基因序列敲入到小鼠的相应基因位点,使用小鼠的表达调控元件指导目的基因表达。
此类基因敲入鼠一般用于药物的筛选,信号通路的研究等。
一、 ZFN 技术制作基因敲除鼠ZFN 能够识别并结合指定的基因序列位点,并高效精确地切断。
随后细胞利用天然的DNA 修复过程来实现 DNA 的插入、删除和修改,这样研究人员就能够随心所欲地进行基因组编辑。
这在过去是无法想象的,传统的基因敲除技术依赖细胞内自然发生的同源重组,其效率只有百万分之一,而 ZFN 的基因敲除效率能达到 10% 。
利用这些技术进行小鼠基因的定点敲除和敲入,可以把时间从一年缩短到几个月。
这项技术中设计特异性的 ZFN 是最关键的环节,目前研究者采用计算生物学方法设计高特异性的 ZFN,但 ZFN的脱靶( off target ),也就是把不该切的地方切了的问题仍是一个挑战。
也正因为这个原因,利用 ZFN 技术进行小鼠的基因修饰还无法完全取代传统技术。


基于CRISPR Cas9技术基因敲除小鼠(Cas9-KO)的制作方法一、CRISPR/Cas9靶向基因敲除小鼠制作的基本技术原理:通过CRISPR/Cas9基因敲除技术,crRNA通过碱基配对与tracrRNA(trans-activating RNA)结合,形成双链RNA。
这一tracrRNA:crRNA二元复合体指导Cas9蛋白在crRNA引导序列靶标的特定位点剪切双链DNA。
在与crRNA引导序列互补的位点,Cas9蛋白的HNH核酸酶结构域剪切互补链而Cas9 RuvC-like 结构域剪切非互补链,实现敲除目的基因的功能,制备基因敲除小鼠模型。
二、具体步骤如下:一)模型制作策略制作:利用生物信息学手段(NCBI&IMPC&MGI),分别仔细分析目的基因敲除后小鼠的生存能力及繁育能力,并结合邻近基因的影响,最终选择合适的敲除区域进行敲除方案的设计,出具相应的制作策略。
二)载体的设计和构建:使用麻省理工学院的CRISPR Design工具(/),依据中靶Score的高低及脱靶Score的高低设计一对长度为20bp的针对靶标DNA的寡聚核苷酸链序列用于制备sgRNA,并在该靶区域设计引物用于后续阳性小鼠的基因鉴定。
1、制备sgRNA的实验方法步骤:1)线性化pUC57-GDNA-T7载体中提pUC57-GDNA-T7载体,用BsaI线性化过夜。
胶回收保存备用。
2)引物退火及加磷酸将上下游引物(干粉)稀释,再进行引物退火及加磷酸。
3)连接&阳性菌落筛选取步骤二中的加磷酸产物与线性化载体pUC57-GDNA-T7进行连接,该连接反应在干式恒温器中进行。
对连接产物进行转化,涂板,37°C培养箱过夜培养。
再用PCR&测序的方法筛选阳性克隆,再将测序正确的克隆进行甘油菌保种,-80°C保存备用4)制备转录模板以构建好的sgRNA载体为模板进行PCR扩增,将PCR产物切胶回收,回收产物离心后倒掉上清留DNA沉淀,再溶解DNA。


基因敲除小鼠的制备流程基因敲除小鼠已经成为现代生命科学基础研究和药物研发领域不可或缺的实验动物模型,在生命科学、人类医药和健康研究领域中发挥着重要的作用。
基于胚胎干细胞的基因打靶技术、EGE技术(基于Crispr cas9技术)是当下比较火热的基因敲除小鼠制备技术。
利用这两种技术制备基因敲除小鼠的流程是什么样的?一、基于胚胎干细胞的基因打靶技术制备基因敲除小鼠的流程:1.课题设计,订购课题BAC菌;2.按照课题设计,完成打靶载体设计和构建;3.将重组载体电转到胚胎干细胞中,用G418筛选转染后的胚胎干细胞,得到阳性克隆;4.进一步通过PCR和southern blot杂交技术(基因敲除小鼠检测金标准)对上一步得到的阳性克隆进行筛选,得到稳定整合外源基因的胚胎干细胞阳性克隆;5.将胚胎干细胞阳性克隆注射到小鼠囊胚中,并植入到假孕小鼠的子宫内;6.得到嵌合鼠,并获得F1阳性杂合子小鼠。
基于胚胎干细胞的基因打靶技术制备基因敲除小鼠是目前为止唯一一个可以满足几乎所有基因组修饰要求的打靶技术,但目前只应用在小鼠的基因敲除上,而且其周期长工作量大。
二、利用EGE技术(基于Crispr cas9技术)制备基因敲除小鼠的流程1.设计构建识别靶序列的sgRNA;2.设计构建致靶基因切割的EGE系统载体质粒;3.利用百奥赛图自主开发的UCA试剂盒对sgRNA/Cas9进行活性检测;4.设计构建打靶载体;5.体外转录sgRNA/Cas9 mRNA;6.小鼠受精卵原核注射sgRNA/Cas9 mRNA和打靶载体;7.获得Fo代小鼠,利用PCR对Fo代小鼠进行基因型鉴定;8.获得F1代小鼠,利用PCR和southern blot杂交技术(基因敲除小鼠检测金标准)对F1代小鼠进行基因型鉴定。
虽然EGE技术(基于Crispr cas9技术)制备基因敲除小鼠看似比基于胚胎干细胞的基因打靶技术制备基因敲除小鼠流程繁琐,其实不然,EGE技术(基于Crispr cas9技术)系统构建简单,基因敲除/敲入效率高,速度快,可实现多基因、多物种基因敲除/敲入,最快2个月即可得到F0代阳性鼠,5个月得到F1F1代杂合子小鼠。

基因敲除:我的老鼠只有我懂作者:遥遥(转载请注:解螺旋·医生科研助手)如果是在做动物实验,而且是基因敲除鼠的实验,那么就会做大批量的小鼠基因型鉴定试验,来确定自己小鼠的基因型,然而,再做实验的过程中往往会出现一些意想不到的结果,我作为一只实验狗在这里为大家提一点点小建议。
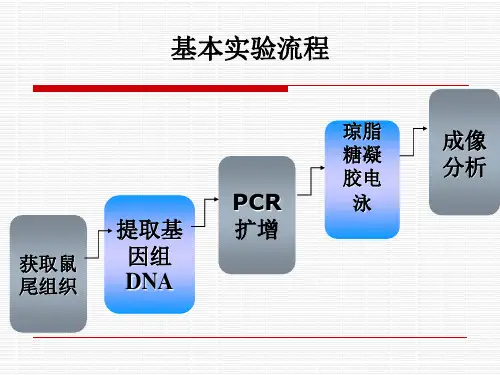
提取DNA1、组织提取DNA 剪下老鼠0.5-1.2cm尾巴或者耳朵或者称取20mg组织样本,将样本剪碎放在1.5ml的EP管中。
2、在每个EP管中加入275微升的裂解液(我们实验室使用的是promega的试剂盒)将加入裂解液的组织放在55摄氏度的水浴箱中过夜(16—18h)裂解完全的标志是组织完全溶解。
3、第二天振荡每一个EP管,混匀后再离心,转速13000rmp时间是3分钟,取上清分离毛发,将取的上清加到柱型管中(有滤网的小柱型管,柱型管套装在1.5的EP管中)。
4、在每个柱型管中加入250微升的Wizard SV lysis Buffer,此裂解液需要提前水浴30min,55摄氏度。
5、离心3min 转速13000rpm ,倒掉滤液。
6、在每个管中加入650微升wash solution 离心1min转速13000rpm,这一步骤重复四遍,每一次都弃去管底的废液。
7、4次洗完之后,什么都不加再离心一次,1min 转速13000rmp。
8、离心之后取出,放入到新的EP管中,每一个EP管中加入50微升Nuclease-free-water(无核酸酶水)需要提前水浴30min中55摄氏度。
加的过程中要充分覆盖管底,静置2min,离心2min,保留EP 管中的液体,此步骤重复两次。
(如果要搁置需要保存在-20冰箱中)。
自此DNA已经提取完成。
显影1、将获得的液体加入PCR混合液中,凝胶电泳,鉴定基因型。
PCR混合液的配制方法ddH2O 9.5微升,Taq12.5微升,上游引物0.5微升,下游引物0.5微升。
每一个EP管中的总量是24微升,提取的DNA 只加1微升。
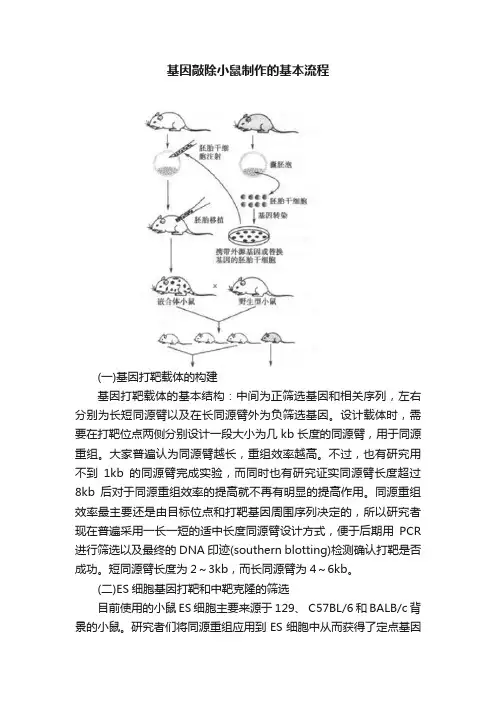
基因敲除小鼠制作的基本流程(一)基因打靶载体的构建基因打靶载体的基本结构:中间为正筛选基因和相关序列,左右分别为长短同源臂以及在长同源臂外为负筛选基因。
设计载体时,需要在打靶位点两侧分别设计一段大小为几kb长度的同源臂,用于同源重组。
大家普遍认为同源臂越长,重组效率越高。
不过,也有研究用不到1kb的同源臂完成实验,而同时也有研究证实同源臂长度超过8kb后对于同源重组效率的提高就不再有明显的提高作用。
同源重组效率最主要还是由目标位点和打靶基因周围序列决定的,所以研究者现在普遍采用一长一短的适中长度同源臂设计方式,便于后期用PCR 进行筛选以及最终的DNA印迹(southern blotting)检测确认打靶是否成功。
短同源臂长度为2~3kb,而长同源臂为4~6kb。
(二)ES细胞基因打靶和中靶克隆的筛选目前使用的小鼠ES细胞主要来源于129、 C57BL/6和BALB/c背景的小鼠。
研究者们将同源重组应用到ES细胞中从而获得了定点基因修饰的目的,通过将DNA片段导入细胞中,利用片段上的宿主细胞同源臂进行同源重组,将目的基因置换插入细胞基因组中整合表达。
在ES细胞中进行同源重组需要将打靶载体进行线性化后,通过诸如电转染(electroporation)、核转染等手段导入细胞中,研究已经证明线性化载体更有利于同源重组的发生。
目前,基因打靶事件的确定通常是首先用PCR反应筛选中靶的ES 细胞克隆。
PCR引物的设计原则是一个引物位于同源臂外,另一个引物位于载体内。
用PCR扩增同源臂短臂,成功的基因打靶克隆会有扩增产物出现。
阳性克隆还需要Southem blotting分析进一步验证。
确定正确后,用于下一步的ES细胞显微注射,一体以产生嵌合体小鼠。
(三)ES细胞克隆的胚胎显微注射和胚眙移植筛选得到的中靶细胞通过显微注射的方式注入到囊胚期胚胎的囊胚腔中,然后将囊胚移植到如假孕母鼠体内,从而产生子代嵌合小鼠。
(四)基因敲除小鼠培育嵌合小鼠需与野生型小鼠交配,以实现基因修饰生殖系传递。
转基因、基因敲入/敲除动物技术已经成为现代生命科学基础研究和药物研发领域不可或缺的重要技术,该技术从上世纪七八十年代诞生以来,已有近四十年的历史,经典技术如DNA原核显微注射、胚胎干细胞显微注射技术一直以来经久不衰,并逐渐从基础研究实验室转向商业模式,成为一项高度标准化的新兴产业一、技术介绍与研究进展转基因、基因敲入/敲除动物技术已经成为现代生命科学基础研究和药物研发领域不可或缺的重要技术,该技术从上世纪七八十年代诞生以来,至今已有近四十年的历史,经典技术如DNA原核显微注射、胚胎干细胞显微注射技术一直以来经久不衰,在小鼠模型构建方面日趋完善,并且如同剪切酶和抗体等常规分子生物学试剂的制备技术一样,逐渐从基础研究实验室转向商业模式,成为一项高度标准化的新兴产业,催生了数以百计的创新药物和数以千计的优秀文章。
尽管如此,传统技术仍然存在一些难以克服的缺陷,如步骤繁琐、周期漫长、成功率低、费用高昂等,而ZFN和TALEN等新技术的出现,或有可能将这一局面彻底改变。
二、同源重组技术原理基因敲除鼠技术是上世纪80年代中后期基于DNA同源重组的原理发展起来的,Capecchi和Smithies在1987年根据同源重组(homologous recombination)的原理,首次实现了ES的外源基因的定点整合(targeted integration),这一技术称为"基因打靶"(gene targeting)或"基因敲除"(gene knockout),利用这种ES的显微注射就可以制作出基因敲出小鼠(KO Mice: knockout mice);由于这一工作,Capecchi和Smithies于2007年与Evans分享了诺贝尔医学奖。
同源重组(homologous recombination)定义:是指发生在姐妹染色单体(sister chromatin) 之间或同一染色体上含有同源序列的DNA分子之间或分子之内的重新组合。
基因敲除小鼠的方法
1. CRISPR/Cas9基因编辑技术,CRISPR/Cas9技术是一种高效的基因编辑工具,可以用来精确地敲除小鼠基因。
首先,科学家设计合成一段RNA序列,使其与目标基因序列相匹配,然后将这段RNA和Cas9蛋白复合体导入小鼠胚胎内。
复合体会通过识别并切割目标基因,导致基因敲除。
2. 胚胎干细胞技术,另一种常见的基因敲除小鼠方法是利用胚胎干细胞。
科学家可以将设计好的基因敲除载体导入小鼠胚胎干细胞中,使其发生基因敲除。
然后,这些修改过的干细胞可以被植入小鼠胚胎内,从而产生基因敲除小鼠。
3. 遗传改造小鼠技术,除了CRISPR/Cas9和胚胎干细胞技术,科学家还可以利用遗传改造技术来实现基因敲除。
这种方法涉及到选择性育种和杂交,通过选择性地交配和繁殖,最终得到具有特定基因敲除的小鼠品系。
总的来说,基因敲除小鼠的方法主要包括CRISPR/Cas9基因编辑技术、胚胎干细胞技术和遗传改造小鼠技术。
这些方法都是在实验室条件下进行的,需要经过严格的实验设计和操作流程,以确保
基因敲除的准确性和有效性。
同时,这些方法也为科学家提供了强大的工具,用于研究基因在生物体内的功能和作用机制。
T7E1酶切法进⾏基因敲除⼩⿏的基因型鉴定
T7E1酶切法进⾏基因敲除⼩⿏的基因型鉴定:
下⾯是举例:
T7E1酶切法检测突变体⼩⿏实验步骤:
1、PCR扩增target site周围序列(⼀般设计500bp左右, target site 最
好不位于中央,这样将酶切出两条⼤⼩不同的带)。
2、将实验组与对照的PCR产物按如下体系进⾏退⽕处理(95℃5min,⾃然
冷却⾄室温)
3、
理中,37℃反应30min后跑2%的琼脂糖凝胶电泳检测分析突变效果。
根据TALEN形成的突变位置和PCR引物的位置,PCR产物为642bp, 若发⽣突变后,⽤T7E1酶切,将产⽣约为:520bp和120bp的两条突变型的带。
下⾯三种情况来判断⼩⿏或细胞为野⽣型、杂合⼦、纯合⼦
1)纯合⼦⼩⿏:样品⼩⿏的PCR产物⾃我杂交,经T7E1酶切,只有⼀种带型(与野⽣型带⼤差不多);⽽样品⼩⿏的PCR 产物与野⽣型⿏PCR产物杂交,出现两种带型(突变体带+野⽣型带)。
2)杂合⼦⼩⿏:样品⼩⿏的PCR产物⾃我杂交,经T7E1酶切,出现两种带型(突变体带+野⽣型带);⽽样品⼩⿏的PCR产物与野⽣型⿏PCR产物杂交,也出现两种带型(突变体带+野⽣型带)。
3)野⽣型⼩⿏:样品⼩⿏的PCR产物⾃我杂交,经T7E1酶切,只有⼀条带;⽽样品⼩⿏的PCR产物与野⽣型⿏PCR产物杂交也出现⼀种带。
基因敲除小鼠的方法基因敲除小鼠模型是生物医学研究领域中常用的实验动物模型之一。
通过对特定基因进行敲除,科研人员可以研究该基因在生物体内的功能和影响,从而深入了解该基因对生物体的生理和病理过程的调控作用,为人类疾病治疗和药物研发提供重要的实验基础。
下面我们将介绍关于基因敲除小鼠的方法。
一、基因敲除小鼠的原理和意义1.1 基因敲除原理基因敲除是指通过人工手段破坏特定基因的DNA序列,使其失去功能。
在小鼠模型中,通常利用基因敲除技术将目标基因进行突变或删除,从而观察小鼠在不同生理状态下的表型变化,探索目标基因在生物体内的功能和作用机制。
1.2 基因敲除的意义基因敲除小鼠模型可以帮助科研人员研究特定基因的生物学功能,了解其在生物体内的作用机制。
基因敲除小鼠模型也能够为疾病研究和药物开发提供重要的实验依据,有助于发现新的治疗靶点和疾病治疗方法。
二、基因敲除小鼠的制备方法2.1 基因敲除小鼠的选择在进行基因敲除小鼠实验前,首先需要选择合适的小鼠品系和基因靶向。
常用的小鼠品系包括C57BL/6、BALB/c等,而基因敲除的选择通常基于目标基因在疾病或生理过程中的重要性。
2.2 敲除载体的构建和筛选制备基因敲除小鼠需要先构建敲除载体,通常采用基因工程技术将靶向基因进行突变或删除,然后将这些构建好的载体导入至小鼠胚胎干细胞中,进行筛选和培养。
2.3 胚胎干细胞的培养与筛选在培养和筛选过程中,科研人员需要将导入敲除载体的胚胎干细胞引入小鼠胚胎中,然后进行体外培养和筛选,以筛选出正常的敲除基因小鼠。
2.4 敲除小鼠的鉴定和繁殖成功培育出的敲除小鼠需要进行PCR鉴定和繁殖,以得到稳定传代的敲除小鼠品系。
对敲除小鼠进行系统的表型观察和分析,以确定目标基因敲除后的表型变化。
三、基因敲除小鼠的应用和前景3.1 基因敲除小鼠在生物学研究中的应用基因敲除小鼠模型广泛应用于生物学研究领域,包括生理学、免疫学、神经科学、遗传学等各个领域。
基因敲除小鼠的实验流程1.设计基因敲除小鼠实验方案在开始实验之前,需要明确研究目的,确定需要敲除的基因,并设计相应的实验方案。
一般可以使用 CRISPR-Cas9 系统来实现基因敲除,在设计基因敲除实验方案时,需要选择合适的 sgRNA 序列,以及设计恰当的引物用于检测突变。
2.获得基因敲除小鼠的胚胎干细胞为了实现基因敲除,需要获得基因敲除小鼠的胚胎干细胞。
一种常用的方法是利用胚胎干细胞对外源DNA的高度易感性,将敲除基因的质粒DNA转染到小鼠胚胎干细胞中。
3.筛选敲除基因的胚胎干细胞株系将转染了敲除基因的胚胎干细胞以悬浮培养的方式进行培养,培养一段时间后,利用一定的筛选条件来筛选出含有敲除基因的胚胎干细胞株系。
筛选条件可包括对抗生素的使用或筛选标记基因的表达。
4.制备敲除基因小鼠的固定胚胎干细胞系通过体外培养,将敲除基因的胚胎干细胞系定植在培养皿上,培养数代以后,将其冻存,以备后续的实验使用。
5.实施敲除基因小鼠的胚胎干细胞基因改造将固定的胚胎干细胞系重新激活,转染 Cas9 和 sgRNA,利用CRISPR-Cas9 系统使这些细胞具有敲除基因的突变。
6.识别敲除基因的胚胎干细胞阳性克隆株对转染了 Cas9 和 sgRNA 的胚胎干细胞进行筛选,通过 PCR、Western blot、Southern blot等技术方法识别出敲除了目标基因的阳性克隆株。
7.将敲除基因的胚胎干细胞注入小鼠的早期胚胎取出已受精的小鼠卵母细胞,利用显微操作将敲除基因的胚胎干细胞注入到小鼠的早期胚胎中。
利用体外受精或者通过体内或体外的胚胎移植方式将基因敲除干细胞注入受体小鼠。
8.制备基因敲除小鼠的嵌合小鼠将已注入敲除基因的胚胎干细胞的受体小鼠进行嵌合以产生基因敲除小鼠。
嵌合可以通过体内或体外的胚胎移植方式进行。
9.筛选识别基因敲除小鼠对产生的嵌合小鼠进行筛选,确认敲除基因是否成功。
可以通过 PCR、Western blot、Southern blot等技术方法对小鼠体细胞或组织进行分析。
基因敲除鼠的构建方法
基因敲除鼠是一种重要的遗传工具,它们能够帮助科学家们研究基因在生物学过程中的作用。
基因敲除鼠构建方法主要包括以下步骤: 1. 设计基因敲除鼠的目标基因序列,选择合适的外显子或内含
子进行靶向敲除。
2. 制备CRISPR/Cas9系统,包括Cas9蛋白、sgRNA以及质粒载体等。
3. 将CRISPR/Cas9系统导入到鼠胚胎干细胞中,使用
CRISPR/Cas9系统导致目标基因的敲除。
4. 鉴定敲除鼠胚胎干细胞中基因敲除的效率,通过PCR、Western blot等方法验证敲除效果。
5. 将基因敲除鼠胚胎干细胞注入到新生小鼠的内脏器官进行移植,培养出基因敲除小鼠。
基因敲除鼠的构建方法是一项复杂的工程,需要科学家们对基因编辑技术的熟练掌握和实验经验。
通过基因敲除鼠的研究,科学家们能够更加深入地了解基因在生物体内的作用,为疾病治疗和新药研发提供更为有效的手段。
- 1 -。
..
一、常规基因敲除鼠(Conventional Knockout)
常规基因敲除是通过基因打靶,把需要敲除的基因的几个重要的外显子或者功能区域用Neo Cassette 替换掉。
这样的小鼠其全身所有的组织和细胞中都不表达该基因产物。
此类基因敲除鼠一般用于研究某个基因在对小鼠全身生理病理的影响,而且这个基因没有胚胎致死性。
二、条件性基因敲除小鼠(Conditional Knockout)
条件性基因敲除小鼠是通过基因打靶,把两个loxP 位点放到目的基因一个或几个重要的外显子的两边。
该小鼠和表达Cre酶小鼠杂交之前,其目的基因表达完全正常。
当和组织特异性表达Cre酶的小鼠进行杂交后,可以在特定的组织或细胞中敲除该基因,而该基因在其他组织或细胞表达正常。
条件性基因敲除鼠适用范围为:(1)该基因有胚胎致死性;(2)用于研究该基因在特定的组织或细胞中的生理病理功能。
三、基因敲入小鼠(Knockin)
基因敲入小鼠是通过基因打靶,把目的基因序列敲入到小鼠的相应基因位点,使用小鼠的表达调控元件指导目的基因表达。
此类基因敲入鼠一般用于药物的筛选,信号通路的研究等。
获得嵌合体及之后品系纯化详细流程:
基因敲除其他方法:
一、ZFN技术制作基因敲除鼠
ZFN能够识别并结合指定的基因序列位点,并高效精确地切断。
随后细胞利用天然的DNA 修复过程来实现DNA的插入、删除和修改,这样研究人员就能够随心所欲地进行基因组编辑。
这在过去是无法想象的,传统的基因敲除技术依赖细胞内自然发生的同源重组,其效率只有百万分之一,而ZFN的基因敲除效率能达到10%。
利用这些技术进行小鼠基因的定点敲除和敲入,可以把时间从一年缩短到几个月。
这项技术中设计特异性的ZFN是最关键的环节,目前研究者采用计算生物学方法设计高特异性的ZFN,但ZFN的脱靶(off target),也就是把不该切的地方切了的问题仍是一个挑战。
也正因为这个原因,利用ZFN技术进行小鼠的基因修饰还无法完全取代传统技术。
二、TALEN技术制作基因敲除鼠
TALEN 技术是一种崭新的分子生物学工具。
科学家发现,来自一种植物细菌的TAL蛋白的核酸结合域的氨基酸序列与其靶位点的核酸序列有恒定的对应关系。
利用TAL的序列模块,可组装成特异结合任意DNA序列的模块化蛋白,从而达到靶向操作内源性基因的目的,它克服了ZFN方法不能识别任意目标基因序列,以及识别序列经常受上下游序列影响等问题,而具有ZFN相等或更好的灵活性,使基因操作变得更加简单方便。
然而同样因为脱靶的问题,利用TALEN技术进行小鼠的基因修饰仍然无法取代传统技术。
;.。