硕士研究生分子生物学实验步骤
- 格式:ppt
- 大小:44.00 KB
- 文档页数:9

可编辑修改精选全文完整版分子生物学实验方法与步骤表达蛋白的SDS-聚丙烯酰胺凝胶电泳分析一、原理细菌体中含有大量蛋白质,具有不同的电荷和分子量。
强阴离子去污剂SDS与某一还原剂并用,通过加热使蛋白质解离,大量的SDS 结合蛋白质,使其带相同密度的负电荷,在聚丙烯酰胺凝胶电泳(PAGE)上,不同蛋白质的迁移率仅取决于分子量。
采用考马斯亮兰快速染色,可及时观察电泳分离效果。
因而根据预计表达蛋白的分子量,可筛选阳性表达的重组体。
二、试剂准备1、30%储备胶溶液:丙烯酰胺(Acr)29.0g,亚甲双丙烯酰胺(Bis)1.0g,混匀后加ddH2O,37O C溶解,定容至100ml, 棕色瓶存于室温。
2、1.5M Tris-HCl(pH 8.0:Tris 18.17g加ddH2O溶解, 浓盐酸调pH至8.0,定容至100ml。
3、1M Tris-HCl(pH 6.8:Tris 12.11 g加ddH2O溶解, 浓盐酸调pH至6.8,定容至100ml。
4、10% SDS:电泳级SDS 10.0 g加ddH2O 68℃助溶,浓盐酸调至pH 7.2,定容至100ml。
5、10电泳缓冲液(pH 8.3:Tris 3.02 g,甘氨酸18.8 g,10% SDS 10ml加ddH2O溶解, 定容至100ml。
6、10%过硫酸铵(AP): 1gAP加ddH2O至10ml。
7、2SDS电泳上样缓冲液:1M Tris-HCl (pH 6.82.5ml,-巯基乙醇1.0ml,SDS 0.6 g,甘油2.0ml,0.1%溴酚兰1.0ml,ddH2O 3.5ml。
8、考马斯亮兰染色液:考马斯亮兰0.25 g,甲醇225ml,冰醋酸 46ml,ddH2O 225ml。
9、脱色液:甲醇、冰醋酸、ddH2O以3∶1∶6配制而成。
二、操作步骤采用垂直式电泳槽装置(一)聚丙烯酰胺凝胶的配制1、分离胶(10%的配制:ddH2O 4.0ml30%储备胶 3.3ml1.5M Tris-HCl2.5ml10% SDS 0.1ml10% AP 0.1ml取1ml上述混合液,加TEMED(N,N,N’,N’-四甲基乙二胺10μl 封底,余加TEMED4μl ,混匀后灌入玻璃板间,以水封顶,注意使液面平。

DNA分子的限制性内切酶消化限制性内切酶可特异地结合于一段被称为限制酶识别序列的DNA序列位点上并在此切割双链DNA。
绝大多数限制性内切酶识别长度为4、5或6个核苷酸且呈二重对称的特异序列,切割位点相对于二重对称轴的位置因酶而异。
一些酶恰在对称轴处同时切割D NA双链而产生带平端的D NA片段,另一些酶则在对称轴两侧相对的位置上分别切断两条链,产生带有单链突出端(即粘端)的DNA片段。
1个单位限制性内切酶是指在最适条件下,在50μl体积1小时内完全切开1μgλ噬菌体DNA所需的酶量。
不同的限制性内切酶生产厂家往往推荐使用截然不同的反应条件,甚至对同一种酶也如此。
但是,几乎所有的生产厂家都对其生产的酶制剂优化过反应条件,因此购买的内切酶说明书上均有其识别序列和切割位点,同时提供有酶切缓冲液(buffer,10×、5×)和最适条件,使得酶切反应变得日益简单。
一、限制性内切酶对D NA消化的一般方案1.限制性内切酶反应一般在灭菌的0.5ml离心管中进行。
2.20μl体积反应体系如下:DNA0.2-1 μg10×酶切buffe r 2.0 μl限制性内切酶1-2 u(单位)加ddH2O至20 μl限制性内切酶最后加入,轻轻混匀,稍加离心,放置于最适温度水浴并按所需的时间温育,一般为37℃水浴消化1hr。
3.用于回收酶切片段时,反应总体积可达50-200μl,各反应组分需相应增加。
4.多种酶消化时若缓冲液条件相同,可同时加入;否则,先做低温或低盐的酶消化,再做高温或高盐的酶消化。
5.酶切结束时,加入0.5M EDTA(pH 8.0)使终浓度达10mM,以终止反应。
或将反应管在65℃水浴放置10m i n以灭活限制性内切酶活性。
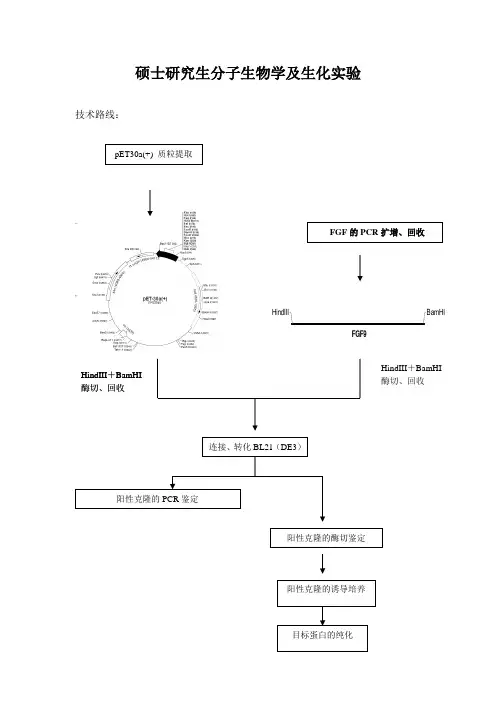
硕士研究生分子生物学及生化实验技术路线:HindIIIBamHIFGF9FGF 的PCR 扩增、回收HindIII 连接、转化BL21(DE3)阳性克隆的PCR 鉴定+BamHI 酶切、回收HindIII +BamHI 酶切、回收pET30a(+) 质粒提取阳性克隆的酶切鉴定阳性克隆的诱导培养 目标蛋白的纯化一、感受态细胞的制备1.实验材料及试剂受体菌:BL21(DE3)、质粒pET-30a+转化液:50mM CaCl2培养基:LB培养基(pH7.0) 121℃,20min 高压灭菌NaCl 5g/L酵母提取物5g/L胰蛋白胨10g/L2..实验步骤1. 感受态细胞的制备1)活化的BL21(DE3)平板上挑取一单菌落,接种于100mL LB液体培养基中37℃振荡培养过夜。
2)取培养过夜的菌液100μL接种于100ml新鲜的LB培养基中,37℃振荡培养约5h。
3)每组取2支10ml离心管,各转入8ml菌液,4000r/min离心3min,收集菌体。
4)弃去上清液,在涡旋器上将细胞悬浮于冰冷的2mL 50mmol/L CaCl2溶液中,将2管合并为1管,并在冰上放置15-30min。
4000r/min离心3min。
5)弃去上清液,加入1ml冰冷的50mmol/L CaCl2溶液,小心悬浮细胞,置冰上,即制成了感受态细胞悬液,可直接用于转化实验,也可加入占总体积20%左右高压灭菌过的甘油,混合后分装于Eppendorf管中,置于-70℃条件下,可保存6-12个月。
二、FGF的PCR扩增与回收(一)试剂电泳缓冲液(50×TAE)Tris 242g冰醋酸 57.1mlEDTA(0.5mol/L pH8.0) 100ml使用时用蒸馏水稀释50倍。
DNA快速纯化/回收试剂盒(二)方法1.PCR反应条件目的基因模板来源于质粒pcDNA3.1-FGF,将质粒稀释100倍,PCR反应采用50μl反应体系。

细菌的活化及培养1.将实验室用到的烧杯、锥形瓶、培养皿等实验器皿用超声机超声20min,然后用自来水冲洗,再用蒸馏水冲洗干净,放入烘箱中烘干。
按照培养基成分配置液体培养基,分别调好pH值后,然后进行灭菌30min。
牛肉膏蛋白胨培养基配方接种:在超净工作台中,取1 mL的菌种加入液体培养基中,混匀,37℃恒温振荡培养12h。
2.菌种的保存(采用甘油保藏法)先取800uL的菌悬液于1.5mL的无菌离心管中,再加入200uL的灭菌甘油,用封口膜将离心管口封号,以上操作均在超净工作台中完成,然后将装有甘油和菌悬液的混合液在漩涡震荡以上混匀,保存至-78℃。
细菌DNA的提取采用TIANamp Bacteria DNA kit试剂盒对单增李斯特菌进行基因组DNA抽提。
操作步骤如下:1)取细菌培养液1ml,1000rpm离心一分钟,尽量吸净上清。
2)向菌体沉淀中加入200μl缓冲液GA,振荡至菌体彻底悬浮。
注意:对于较难破壁的革兰氏阳性菌,可略过第二步,加入溶菌酶进行破壁处理,具体方法为:加入180μl缓冲液(110μl TE缓冲液,70μl溶菌酶),37℃处理30min以上。
3)向管中加入20μl蛋白酶K溶液,混匀。
4)加入220μl缓冲液GB(改良磷酸盐缓冲液),振荡15s,70℃放置10min,溶液应变清亮,简短离心以去除管盖内壁的水珠。
注意:加入缓冲液GB时可能会产生白色沉淀,一般70℃放置时会消失,不会影响后续实验。
如溶液未变清亮,说明细胞裂解不彻底,可能导致提取DNA 量少和提取出的DNA不纯。
5)加220μl无水乙醇,充分振荡混匀15s,此时可能出现絮状沉淀,简短离心以去除管盖内壁的水珠。
6)将上一步所得溶液和絮状沉淀都加入一个吸附柱CB3中(吸附柱放入收集管中),12000rpm离心30s,倒掉废液,将吸附柱CB3放入收集管中。
7)向吸附柱CB3中加入500μl缓冲液GD(使用前请先检查是否已加入无水乙醇),12000rpm离心30s,倒掉废液,将吸附柱CB3放入收集管中。

分子生物学实验方案设计(五篇模版)第一篇:分子生物学实验方案设计梅衣属ITS条形码物种的快速鉴定实验目的:一、学习并熟练地衣DNA提取技术二、掌握PCR技术的原理及操作三、DNA条形码技术的应用实验技术路线:1、地衣总DNA的提取2、PCR扩增3、基因测序4、基因序列对比地衣总DNA提取:1.取带有子囊盘或未有子囊盘的带藻地衣体30~300mg,用无菌水冲洗,除去表面杂质,再用DNA提取液浸泡2~3h(4℃)。
2.将地衣体取出,滤纸吸干表面液体,剪碎放于预冷的研钵中,加入液氮研磨至粉末状。
3.将粉末小心、全部转入1.5ml离心管中,加入400μL DNA提取液,混匀。
4.加入10% SDS 50μL、氯化苄150μL,振荡混合,于50℃保温1h,每隔10min振荡混合一次。
5.保温1h后,每管加入3mol/L NaAc 50μL,混匀冰浴15min.6.用等体积酚:氯仿:异戊醇(25:24:1)和等体积酚:氯仿(1:1)各抽提一次,直至无白色沉淀为止。
7.移上清液于另一离心管,加入2倍体积无水乙醇,4℃放置2~3h,15000r/min,离心10min(4℃),弃上清液。
8.将沉淀用70%乙醇冲洗2次后,真空干燥,再加入50~80μL TE 缓冲液,保存于冰箱(4℃)PCR扩增:稀释50 倍用于后续 PCR 操作。
真菌 ITS rDNA 由通用引物 ITS5(5′-GGAAGTAAAAGTCGTAACAAGG-3′)、ITS4(5′-TCCTCCGCTTATTGATATGC-3′)。
PCR 反应条件:95℃预变性5min;95℃变性30s,52℃退火30s,72℃延伸 2min,反应 32 个循环;72℃延伸 10min,4℃保存。
反应结束后,PCR 产物通过0.8%的琼脂糖凝胶电泳检验,ABI3730DNA 分析仪测序。
序列比对:所获测序结果序列包含小亚基部分结尾序列、ITS1-5.8S-ITS2、大亚基部分起始序列,去除大小亚基部分序列后所得片段大小约500bp,即为所需的序列利用 DNAMAN(Lynnon Biosoft)软件将测序结果同 GenBank 下载数据进行比对分析,如果序列长度不同,则只保留共有区段。


一目的基因的获得细菌基因组DNA的提取(一)仪器1)台式高速离心机2)恒温水浴箱3)枪式移液器4)灭菌的EP管和Tip头(二)试剂1)溶液1:25%蔗糖50mmol/LTris-Hcl,pH8.050mmol/LEDTA,pH8.0500ug/ml溶菌酶(现用现配)100ug/mlRNase2)溶液Ⅱ:100mmol/LTris-Hcl,pH8.01%SDS400ug/ml蛋白酶K3)酚:氯仿:异戊醇(25:24:1)4)氯仿:异戊醇(24:1)5)3mol/LNaAc(pH5.2)6)异丙醇或无水乙醇7)70%乙醇8)TE缓冲液:10mmol/LTris-Hcl,pH8.01mmol/LEDTA,pH8.0(三)实验步骤1)5mL细菌过夜培养,5000rpm离心10分钟,去上清液。
2)将菌体细胞悬于250uL溶液1中,37°C水浴保温过夜。
3)加250uL溶液Ⅱ,55°C水浴保温4小时。
4)加0.9倍体积的酚/氯仿/异戊醇(25:24:1),轻轻混匀,于14000rpm离心10分钟,转移水相至新的Ep管,重复步骤4一次。
5)向转移的水相中加入0,9倍体积的氯仿/异戊醇(24:1),轻轻混匀,于14000rpm离心10分钟,转移水相至新的Ep管,重复步骤5一次。
6)向转移的水相中加入0.1倍体积的3mol/LNaAc和0.7倍体积的异丙醇(或2.5倍体积的无水乙醇),冰上放置30分钟。
7)14000rpm离心20分钟,弃上清,用0.5mL70%乙醇洗涤沉淀一次,弃上清,沉淀于室温干燥。
8)将DNA沉淀溶解于15ulTE缓冲液中,-20°C保存。
9)进行DNA含量和纯度检测,琼脂糖凝胶电泳鉴定。
植物DNA的SDS提取法:(一)试验试剂:1)研磨缓冲液:称取59.63gNaCl,13.25g柠檬酸三钠,37.2gEDTA-Na分别溶解后合并为一,用0.2mol/L的NaOH调至pH7.0,并定容至1000ml。


《分子生物学实验技术》实验报告实验一碱裂解法小量提取质粒DNA一、实验原理二、在碱性环境中, 细菌膜破裂释放内容物。
染色体DNA变性分开, 而质粒DNA虽变性但仍处于缠绕状态。
将pH调至中性并有高盐存在及低温的条件下, 染色体DNA.大分子量的RNA和蛋白质在SDS的作用下形成沉淀, 而质粒DNA仍然为可溶状态。
通过离心, 可除去大部分细胞碎片、染色体DNA.RNA及蛋白质, 最后用酚、氯仿抽提纯化得到质粒DNA。
三、操作步骤主要步骤1.细菌的培养2.细菌的收集和裂解3.质粒DNA的分离和纯化4.质粒的鉴定具体操作如下:1.取1.5 ml过夜菌液于EP管中, 12 000 r/min离心1min, 弃去上清液。
2.加100 μl 溶液Ⅰ, 剧烈震荡使菌体悬浮均匀, 室温放置5min 。
3.加200 μl 溶液Ⅱ(现配现用), 轻柔颠倒混匀4~6次, 室温放置5min 。
4.加150μl预冷溶液Ⅲ, 轻柔颠倒混匀4~6次, 冰上放置10分钟。
5. 12 000 r/m离心10 分钟(如果上清液仍然浑浊, 可将上清液移出, 再次离心5分钟)。
6.吸出上清液,加2.5倍体积的无水乙醇,混匀,室温放置10分钟,12 000r/min离心10分钟,沉淀DNA 。
7.弃上清,挥干,所得DNA溶于30μlddH2O中,用于后续试验。
三、实验结果获取极少量白色固体, 主要为DNA, 也不排除有蛋白质等杂质, 但是不影响后续实验的继续进行。
实验二氯化钙法进行质粒转化及筛选一、实验原理细菌处于0 ℃, 在CaCl2低渗溶液中, 菌体细胞膨胀成球形, DNA则与CaCl2形成抗Dnase的羟基-磷酸钙复合物粘附于细胞表面, 经42 ℃短时间热冲击处理, 促进细胞吸收DNA复合物, 在丰富培养基上生长数小时后, 球状细胞复原并分裂增殖, 重组子的基因在被转化的细菌中得到表达, 在选择性培养基平板上, 可选出所需的转化子。

《分子生物学实验技术实验操作指南》目录1 植物基因序列分析 (3)2 引物设计的原则 (4)3 试剂的配置和灭菌 (5)4 植物基因组DNA的提取 (6)5 DNA的定量 (8)6 DNA的电泳 (9)8 PCR产物的回收 (12)9 PCR产物连接T载体和感受态细胞的转化 (14)10 菌落(液)PCR (17)11 质粒DNA的提取 (18)12 质粒DNA的酶切 (20)1 植物基因序列分析1.基因组DNA(genomic DNA, gDNA):功能基因包括三个基本序列:5’上游区,转录区和3’下游区。
2.5’上游区和3’下游区:转录起始位点上游和转录终止位点下游的序列,主要起到基因表达调控作用。
3.转录区:转录起始位点和转录终止位点之间的序列,经核不均一RNA最终被加工为成熟的mRNA。
4.信使RNA(messenger RNA, mRNA):由DNA的一条链作为模板转录而来的,携带遗传信息的能指导蛋白合成的一类单链核糖核酸。
由编码区、上游的5’非编码区和下游3’非编码区组成,5’端带有7-甲基鸟苷-三磷酸帽子结构,3’端有多腺苷酸尾巴。
5.互补DNA(complementary DNA, cDNA):具有与某m链呈互补的序列的。
6.蛋白质编码区(sequence coding for amino acids in protein, CDS):与蛋白质序列一一对应的DNA序列,且该序列中间不含其它非该蛋白质对应的序列。
只有外显子,不含内含子。
7.5’非翻译区(5’-untranslated region, 5’UTR):mRNA位于基因转录起始位点至编码区翻译起始密码子之间顺序。
8.3’非翻译区(3’-untranslated region, 3’UTR):mRNA位于编码区翻译终止密码子下游一段不被翻译的序列。
2 引物设计的原则1.引物长度:一般为20-25bp。
若产物长度等于或小于500bp,可选用16-18bp的引物;若产物长达5kb,则需要用更长的引物。

DNA分子的限制性内切酶消化限制性内切酶可特异地结合于一段被称为限制酶识别序列的DNA序列位点上并在此切割双链DNA。
绝大多数限制性内切酶识别长度为4、5或6个核苷酸且呈二重对称的特异序列,切割位点相对于二重对称轴的位置因酶而异。
一些酶恰在对称轴处同时切割DNA双链而产生带平端的DNA片段,另一些酶则在对称轴两侧相对的位置上分别切断两条链,产生带有单链突出端(即粘端)的DNA片段。
1个单位限制性内切酶是指在最适条件下,在50μl体积1小时内完全切开1μgλ噬菌体DNA所需的酶量。
不同的限制性内切酶生产厂家往往推荐使用截然不同的反应条件,甚至对同一种酶也如此。
但是,几乎所有的生产厂家都对其生产的酶制剂优化过反应条件,因此购买的内切酶说明书上均有其识别序列和切割位点,同时提供有酶切缓冲液(buffer,10×、5×)和最适条件,使得酶切反应变得日益简单。
一、限制性内切酶对DNA消化的一般方案1.限制性内切酶反应一般在灭菌的0.5ml离心管中进行。
2.20μl体积反应体系如下:DNA 0.2-1 μg10×酶切buffer 2.0 μl限制性内切酶1-2 u(单位)加ddH2O 至20 μl限制性内切酶最后加入,轻轻混匀,稍加离心,放置于最适温度水浴并按所需的时间温育,一般为37℃水浴消化1hr。
3.用于回收酶切片段时,反应总体积可达50-200μl,各反应组分需相应增加。
4.多种酶消化时若缓冲液条件相同,可同时加入;否则,先做低温或低盐的酶消化,再做高温或高盐的酶消化。
5.酶切结束时,加入0.5M EDTA(pH 8.0)使终浓度达10mM,以终止反应。
或将反应管在65℃水浴放置10min以灭活限制性内切酶活性。
6.如消化反应体积过大,电泳时加样孔盛不下,可加以浓缩:终止反应后,加入0.6体积的5M乙酸铵(或1/10体积3M NaAc)和2倍体积无水乙醇,在冰上放置5min,然后于4℃离心12000g×5min。
分⼦⽣物学实验技术实验内容概要2006年《分⼦⽣物学实验技术》实验内容⼀、RT-PCR(⼀)总RNA的提取实验安排:每两⼈抽提⼀管。
为了使操作同步以节省时间,各组样品请⼀起离⼼。
操作步骤:1、将100µl液体样品加⼊1.5ml Ep管中,再加⼊900µl冰预冷的LS-Biotragents TM(苯酚和异硫氰酸胍的混合物)。
2、将样品剧烈混合后,在室温放置5min。
3、加⼊200µl氯仿,颠倒Ep管混和两次,并剧烈振荡混和10s。
4、在4℃条件下,以10000×g离⼼15min。
5、将上层⽔相转移到⼀个新的Ep管中,加⼊等体积的异丙醇(Isopropanol)并混匀,然后在4℃放置⾄少10min。
6、在4℃条件下,以10000×g离⼼15min后,⼩⼼并尽可能地去除全部上清夜。
7、⽤1ml 75%⼄醇洗涤RNA沉淀和管壁。
8、将RNA沉淀进⾏⼲燥(不能完全⼲燥)处理后,⽤10µl⽆RNase污染的⽔(RNase-Free Water)将RNA溶解并于-20℃保存。
注意事项:1、所有的玻璃器⽫均应在使⽤前于180℃的⾼温下⼲烤6hr或更长时间。
2、所⽤的塑料材料,如吸头、离⼼管等需⽤0.1% DEPC⽔浸泡过夜。
3、配制的溶液应尽可能⽤0.1% DEPC,在37℃处理12hr以上。
然后⽤⾼压灭菌除去残留的DEPC。
不能⾼压灭菌的试剂,应当⽤DEPC处理过的⽆菌双蒸⽔配制,然后经0.22µm滤膜过滤除菌。
4、操作⼈员需在超净⼯作台上操作,并戴⼀次性⼝罩、⼿套,实验过程中⼿套要勤换。
(⼆)反转录实验安排:每⼈做⼀管。
反应体系(20µl):按下列顺序加样反应条件:42℃ 1h1、加样时,⼀般从体积⼤的开始加,样品最后加。
如在⼀般的PCR反应体系中,应先加⽔、Buffer、dNTPs、引物,最后加酶和模板。
2、液体应直接加到管底,且每加⼀种试剂后应更换新的吸头。
重组转化与阳性克隆筛选标准操作程序一、准备材料材料清单:1.1塑料制品培养皿(6cm),离心管(1.5ml,2ml),96孔板,枪头和枪头盒,离心管架,胶塞1.2玻璃器皿蓝盖瓶(100ml,250ml),锥形瓶(100ml)1.3试剂胶回收试剂盒, takara PMD TM 19 cloning试剂盒(包括PMD19,solution I),Amp 溶液,胰蛋白胨, 酵母粉, NaCl, 琼脂粉,E. col i DH5α菌株,,60%无菌甘油,引物,PCR mix,感受态细胞1.4设备离心机,超净工作台,纯水机,培养箱,漩涡震荡器,移液器,灭菌锅,烘箱微波炉,pcr仪,恒温摇床。
1.5 杂物记号笔,废液缸,封口膜,酒精灯二、灭菌枪头灭菌1.5ml和2.0ml的离心管灭菌。
灭菌后,取出以上物品放入烘箱中烘干三、各种试剂的配制1 、LB固体培养基100ml(无抗)组成成分:方法:将LB 培养基全部加入自来水中,定容至100ml,搅拌使其充分溶解,调节PH 值到7.2 左右。
可分装,121℃高压灭菌15min,倒平板。
若需倒制含抗生素的平板,灭菌后培养基冷至不烫手时再加入抗生素,摇匀倒板。
倒好的含抗生素的平板4℃保存,2 周内用完2、LB液体培养基100ml(无抗)组成成分:将LB 培养基全部加入自来水中,定容至100ml,搅拌使其充分溶解,调节PH 值到7.2 左右。
121℃高压灭菌15min。
四、实验步骤1、扩增目的序列 PCR(50ul)2、目的片段胶回收1)准备实验服,手套,1.5ml离心管2)当所需DNA片段完全分离时,转移凝胶至紫外灯上,尽可能快地把所需的DNA 片段切下来.注:切胶时尽量把多余的凝胶切去,DNA曝露在紫外灯下不能超过30秒.3)将带有目的片段的凝胶块转移至1.5ml离心管(离心管已经称重了)中,称重得出凝胶块的重量加入三倍体积的ADB,于55-65℃水浴中温浴7min或至凝胶完全融化,每2-3 min振荡或涡旋混合物.4)取一个干净的柱子装在一个干净的2ml收集管内(已备好).5)将第三步获得的DNA/熔胶液全部转移至柱子中.室温下10,000 g离心1分钟.弃收集管中的滤液,将柱子套回2ml收集管内.6).如果DNA/凝胶熔液的体积超过700ul,一次只能转移700ul至柱子中,余下的可继续重复第5步至所有的溶液都经过柱子.每7)弃收集管中的滤液,将柱子套回收集管内.移700ul Wash buffer(已用无水乙醇稀释的)至柱子中.室温下10,000g离心1min.注意:浓缩的Wash Buffer 在使用之前必须按标签的提示用乙醇稀释.如果DNA 洗涤缓冲液在使用之前是置于冰箱中的,须将其拿出置于室温下。