两分钟学习凝胶分析软件BandScan
- 格式:doc
- 大小:1.22 MB
- 文档页数:12

凝胶定量软件Quantity One使用简介1、内容简介凝胶电泳是每个做分子生物学的同学天天都要打交道的基本技术。
电泳之后的信息处理与电泳本身同样重要。
目前有大量软件可以用于分析电泳结果,比较有名的比如BandScan、BandLeader、Sigma Gel等等。
今天要向大家介绍的是来自Bio-Rad的1D凝胶定量软件Quantity One(Bio-Rad还有一个做2D凝胶分析的软件PDQuest)。
这个软件的最新版本可以在Bio-Rad的主页免费下载。
目前的最新版本是4.52版2、Quantity One的定量方法Quantity One的分析功能顾名思义主要用来进行凝胶或者培养皿的荧光定量分析。
它的分析功能或者说分析方式主要有4种:泳道/条带轨迹定量法;等高线直接定量法;菌落计数;分子量测定这三种方法中使用最为方便也是最为广泛的应该是等高线定量法(Volumn Contour)。
它通过半自动描绘电泳条带的等高线边缘来得到等高线区域内部面积,再将该面积乘以区域内平均光密度值得到条带内部总的信号量。
当然这种分析方法的弊病显而易见:无法同等得排除不同泳道的背景亮度;等高线的绘制处于“半自动”状态,即需要人为判断作为等高线标准的电泳条带的边缘;最致命的是在几个电泳条带距离十分接近的时候几乎无法绘制单一条带的轮廓(常出现连续的几个条带等高线相连而无法分离出单独条带的轮廓)。
三种方法中个人感觉最为科学和严谨的应该是泳道/条带轨迹定量法(Trace Tracking)。
这种方法使用起来步骤较为繁琐,必须通过泳道识别---电泳条带识别两个连续的步骤才能进行定量。
然而这种方式的最大优点在于它可以完全抛弃人为主观因素进行全自动定量。
他的定量方式为:首先根据不同电泳条带的光密度值绘制光密度曲线,然后计算光密度曲线下面积作为电泳条带的定量根据。
大家可能会问他能不能排除泳道背景?答案是肯定的,它能够最大程度的排除不同泳道之间的背景差异,让各个泳道上的不同电泳条带在一条几乎相同的起跑线上进行对比。

三维分子类RASMOL 2.7.2.1 观看生物分子3D微观立体结构的软件。
非常有名,巨棒!RasTop 2.03 为RasMol 2.7.1的图形用户界面软件CHIME 2.6 SP5 直接在浏览器中观看3D分子。
MolMol 2k.2 将pdb等格式的蛋白文件通过微调,存成普通的图形文件。
CrystInfo 1.0 用来快速、容易地构建、观察与检查晶体3d结构。
PDViewer PDB格式文件的查看程序。
DS ViewerPro 5.0 trail 3维分子浏览工具。
ICMLite 2.8 3维分子浏览工具,有一些其他软件没有的功能。
VMD 1.82 3维分子浏览工具,可以进行动态显示。
CN3D 4.1 3D分子结构观察软件。
WPDB 2.2 PDB文件检索显示分析软件。
DTMM 4.1 Demo 3维分子模型显示、编辑与构建程序。
gopenmol2.32 显示并分析分子结构及其特性的软件。
POV-Ray 3.6b3 生成三维图像工具软件。
WinMegaPov 1.0 3D渲染软件POV-Ray非官方编译软件。
MolPOV 2.0.8 将PDB文件转化为POV格式文件的软件。
Mol2Mol 5.2.1Demo 分子文件格式转换软件。
PovChem 2.1.1 将PDB文件转化为POV格式文件的软件。
Ortep-3 for Windows 1.076 生成分子的热椭圆形点图软件。
PLATON1.07 通用结晶学软件工具。
Mage 6.35 读取并演示Kinemage格式文件的专用软件。
Prekin 6.35 将PDB格式文件转换为Kinemage格式文件的软件。
Swiss-PdbViewer 3.7 sp5 PDB文件显示与分析软件。
DINAMO 蛋白序列排队比较编辑与三维模型构建工具软件。
PCMolecule2 Lite 查看PDB格式文件的免费软件。
StrukEd Demo 化学分子编辑与三维模型生成软件。

序列分析软件DNAMAN的使用方法中文演示文稿第一部分:软件介绍1.DNAMAN是什么?-DNAMAN是一款用于DNA和蛋白质序列分析的软件。
-它提供多种功能,包括序列比对、引物设计、限制酶分析和进化树构建等。
2.DNAMAN的应用领域-DNAMAN广泛应用于生物学、生物技术和医药领域。
-它可以帮助研究人员进行序列分析、设计实验方案和解读实验结果。
第二部分:基本序列比对1.创建新项目-打开DNAMAN软件,点击“新建”按钮创建新项目。
-输入项目名称和序列信息,保存并打开该项目。
2.导入序列-点击“导入”按钮,选择需要比对的序列文件,点击“确定”导入。
-系统会自动将序列导入到项目中。
3.序列比对-选择需要比对的序列,点击“比对”按钮进行序列比对。
-系统会自动比对序列并生成比对结果。
4.结果解读-比对结果以图形和文本形式展示。
-可以通过选择不同的比对算法和调整参数来优化比对结果。
第三部分:引物设计1.创建新项目-打开DNAMAN软件,点击“新建”按钮创建新项目。
-输入项目名称和序列信息,保存并打开该项目。
2.导入标记序列-点击“导入”按钮,选择需要设计引物的标记序列文件,点击“确定”导入。
-系统会自动将标记序列导入到项目中。
3.引物设计-点击“引物设计”按钮,选择设计引物的参数和算法。
-系统会根据所选参数和算法自动生成引物设计结果。
4.结果解读-引物设计结果以图形和文本形式展示。
-可以通过选择不同的参数和算法来优化引物设计结果。
第四部分:限制酶分析1.创建新项目-打开DNAMAN软件,点击“新建”按钮创建新项目。
-输入项目名称和序列信息,保存并打开该项目。
2.导入限制酶序列-点击“导入”按钮,选择需要分析的限制酶序列文件,点击“确定”导入。
-系统会自动将限制酶序列导入到项目中。
3.限制酶分析-点击“限制酶分析”按钮,选择分析参数和算法。
-系统会根据所选参数和算法自动进行限制酶分析。
4.结果解读-限制酶分析结果以图形和文本形式展示。

电泳条带的光密度分析原理电泳图⽚光密度分析原理与⽅法⽆论是DNA电泳图⽚还是RNA电泳图⽚,或者是蛋⽩的凝胶电泳图⽚包括Western blot膜、或者是化学发光膜图⽚,在图像分析的⾓度来看,都是同样的⼀种条带光密度分析的问题。
分析电泳条带有专门的⼀类图像分析软件。
常见的是Bandscan, Quantity One, 由各个凝胶电泳成像系统⼚商⾃⼰编制的软件就更多了。
我喜欢⽤的是Labworks, 是UVP公司⽣产的凝胶电泳成像系统上配⽤的拍摄与分析软件,实际上就是传说中⼤名⿍⿍的gel-pro。
这个软件可以看作是Image-pro plus应⽤在电泳条带分析上的专业版。
所以⽤惯了IPP后对gel-pro就会感觉很熟悉。
很快就能学会使⽤。
现在UVP已经不使⽤labworks了,另弄了⼀个visionworks 软件,⽤起来就是没gel-pro舒服。
实际上所有的电泳条带分析软件在分析原理与功能上都没什么差别。
只是软件界⾯与操作程序上各有不同。
所以,了解了条带的分析原理后也就能⾃⼰学会各种软件的使⽤了。
分析电泳条带也是从拍摄电泳照⽚开始的。
⽤平时玩的数码相机拍摄的电泳条带⼀般不能⽤来进⾏定量的分析。
⽽必须使⽤专门的拍摄设备。
⼤家⼀般称它为“凝胶成像系统”。
它集中了观察,拍摄与分析凝胶的所有功能。
⼀个凝胶成像系统包括了三个部分,暗箱,相机与电脑上的控制分析程序。
让我们从使⽤成像系统的操作⽅法中来了解如何正确地拍摄与分析凝胶吧。
⼀、暗箱⼀个最简单的暗箱就是个不透光的箱⼦⾥⾯放了⼀个紫外灯箱,⾼级⼀点的暗箱则有好⼏组灯光照明。
当然,在箱⼦上⾯会有⼀个安放相机的地⽅。
紫外灯箱是观察与拍摄凝胶的必需照明设备。
它是⽤来观察EB染⾊的凝胶条带的。
⾥⾯的紫外灯光能提供256nm,302nm及365nm的紫外光。
在观察EB的红⾊荧光条带时,⼀般⽤302nm。
在观察与拍摄蛋⽩凝胶的时候,必须使⽤透射⽩光照明。
有的暗箱⾥会有⽩板照明灯,平时向上掀起靠在后壁上,使⽤时拉下来。

凝胶成像系统的使用方法
本系统属全自动电脑控制影像分析系统,主要拍摄核酸的agarose电泳胶片,及蛋白质的acrylamide电泳胶片。
在与markers比较后,可精确计算样本的分子量,对核酸电泳也可准确定量,是分子生物学及生化蛋白试验的重要检测工具。
本系统包括暗箱,紫外透射仪,摄像头,变焦镜,电脑以及专业凝胶图象采集及分析.软件(3.3版)。
该软件提供对电泳凝胶、斑点测量、PCR密度的定量分析等。
此外,还提供图像采集、标注、打印、数据和图像发送到Word、Excel、图像处理等功能。
1.使用方法
(1) 打开电脑,启动凝胶分析软件,进入用户界面。
(2)打开采集凝胶图像的暗箱装置电源开关,放入凝胶,打开紫外光源或白光光源,将采集装置与电脑上采集卡相连,然后选择“文件”菜单中的“图像采集”或工具栏的“图像采集”按钮,出现图像窗口:“开始采集”、“停止采集”、“采集图像”等。
如果图像不清晰,可以调节暗箱上的变焦镜,使之清晰。
可直接将图像保存起来,也可通过“图像处理”对图像进行旋转、裁剪、滤波、调节对比度……方面的处理。
(3)对读入的电泳凝胶图像,可以启动电泳凝胶分析系统。
在“功能选择”菜单中选择“电泳凝胶”或在工具栏上的“电泳凝胶”工具,将出现泳道分析工具栏,并在“图像显示”子窗口中出现一个红色的矩形框。
还可进入“条带分析”系统,对条带进行编号,对二条以上的条带进行比较。
(4)采集图像结束后,关闭暗箱电源开关,从暗箱中取出凝胶,并将玻璃板清洗干净,晾干。

凝胶定量软件Quantity One使用简介1 内容简介凝胶电泳是每个做分子生物学的同学天天都要打交道的基本技术。
电泳之后的信息处理与电泳本身同样重要。
目前有大量软件可以用于分析电泳结果,比较有名的比如BandScan、BandLeader、Sigma Gel等等。
今天要向大家介绍的是来自Bio-Rad的1D凝胶定量软件Quantity One(Bio-Rad还有一个做2D凝胶分析的软件PDQuest)。
这个软件的最新版本可以在Bio-Rad的主页免费下载。
2 Quantity One的定量方法Quantity One的分析功能顾名思义主要用来进行凝胶或者培养皿的荧光定量分析。
它的分析功能或者说分析方式主要有4种:泳道/条带轨迹定量法;等高线直接定量法;菌落计数;分子量测定--------------------------------------------------------------------------------这三种方法中使用最为方便也是最为广泛的应该是等高线定量法(Volumn Contour)。
它通过半自动描绘电泳条带的等高线边缘来得到等高线区域内部面积,再将该面积乘以区域内平均光密度值得到条带内部总的信号量。
当然这种分析方法的弊病显而易见:无法同等得排除不同泳道的背景亮度;等高线的绘制处于“半自动”状态,即需要人为判断作为等高线标准的电泳条带的边缘;最致命的是在几个电泳条带距离十分接近的时候几乎无法绘制单一条带的轮廓(常出现连续的几个条带等高线相连而无法分离出单独条带的轮廓)。
三种方法中个人感觉最为科学和严谨的应该是泳道/条带轨迹定量法(Trace Tracking)。
这种方法使用起来步骤较为繁琐,必须通过泳道识别---电泳条带识别两个连续的步骤才能进行定量。
然而这种方式的最大优点在于它可以完全抛弃人为主观因素进行全自动定量。
他的定量方式为:首先根据不同电泳条带的光密度值绘制光密度曲线,然后计算光密度曲线下面积作为电泳条带的定量根据。

生物软件汇总,强烈推荐!质粒绘图Plasmid Processor 1.02 绘制质粒图软件。
Plasmid Processor Pro 绘制质粒图软件,与Plasmid Processor是同一个作者。
WinPlas 2.7 demo 质粒绘图软件商业版。
DMUP beta 环状质粒绘图软件测试版。
Plasmid Toolkit 1.4s 质粒绘制软件。
pDRAW 1.1.60 DNA分析与绘图软件,可绘制线性或环形DNA图。
Redasoft Visual Cloning 2000 Demo 是有名的绘制质粒图Redasoft Plasmid 1.1软件的升级版。
SimVector 2.01 Demo 质粒图绘制软件。
CloneMap 2.11 Demo 质粒作图软件演示版。
pCIRCLE 2nd Flash MX 2004 制作的用于绘制环状质粒的软件图像处理Image Tool 3.0 科学用途的处理图像软件。
Image J 1.31 用Java语言写成的科学用途的处理图像软件。
Cross Checker 2.91 基因指纹图分析软件。
ALFmap 1.22 ALF (Amersham Pharmacia) 图像格式转换软件。
Band Leader 3.0 凝胶图像处理软件。
Scion Image 4.12 图像处理与分析工具。
OSIRIS 4.18 通用医学图像处理与分析软件。
Melanie 4 Viewer 免费Melanie图像查看器。
ChromoZoom 1.1 比较两个图像的相同与不同之处软件。
bandscan 5.0 Demo 蛋白凝胶电泳图像分析软件。
SigmaScan Pro 5.0 Demo 图像分析软件30天全功能演示版。
SigmaGel 1.0 Demo 凝胶图像分析软件。
TotalLab 2.0 评估版图像分析软件。
LabImage 2.71 评估版凝胶图像分析软件。

Oligo 6 Tour 主要功能介绍Oligo是一种多功能的程序,通过从一个序列中搜索、选择寡核苷酸而广泛运用于PCR、DNA 测序、定向诱变及各种杂交中。
它采用nearest neighbor thermodynamic values的方法计算出杂交的温度及寡核苷酸的二级结构。
Oligo软件已经被认可作为一种选择及分析寡核苷酸的工业软件,运用于各种分子生物学中。
最早的商业化的软件在1989年被开发出来。
本文描述了Oligo软件的最重要的特征及性能。
1、主窗口当你运行Oligo、并输入序列之后,Oligo出现了两个窗口:上面的一个为Tm窗口(The Melting Temperature window),下面的一个为内部稳定性窗口(五聚体的DG),还有第三个窗口,即寡核苷酸频率窗口,隐藏在内部稳定性窗口之后。
--图1,2Tm窗口显示了一部分的DNA/RNA的活性片段,Tm的散点图显示了在这个片段中的每20个碱基的Tm值。
分析的片段的长度是可变的,取决于monitor resolution。
圈出来的序列部分及黄色的bar即代表当前分析的20个碱基的Tm值【注:Olig6.71的版本为20个碱基,与原文的21个碱基不同】.可通过点击窗口的左下角的Upper、Lower按钮选择上游引物、下游引物。
划分Tm图二等分的水平线代表了这个序列的所有的21个碱基的寡核苷酸的平均Tm值(or free energy or degeneracy or %GC)。
在Tm图的分别为双链的核苷酸序列及相应的氨基酸【彩色的代表使用的密码子】--图3内部稳定性窗口显示了寡核苷酸的内部稳定性(五具体的自有能)。
可被用于预测用于PCR 或测序反应特异性的把握度2. Analyze - Key Info显示寡核苷酸的基本信息。
--图4,53. Analyze - Duplex Formation显示了上游引物、下游引物的潜在的二级结构的形成。

凝胶定量软件Quantity One使用简介1 内容简介凝胶电泳是每个做分子生物学的同学天天都要打交道的基本技术。
电泳之后的信息处理与电泳本身同样重要。
目前有大量软件可以用于分析电泳结果,比较有名的比如BandScan、BandLeader、Sigma Gel等等。
今天要向大家介绍的是来自Bio-Rad的1D凝胶定量软件Quantity One(Bio-Rad还有一个做2D凝胶分析的软件PDQuest)。
这个软件的最新版本可以在Bio-Rad的主页免费下载。
目前的最新版本是4.52版2 Quantity One的定量方法Quantity One的分析功能顾名思义主要用来进行凝胶或者培养皿的荧光定量分析。
它的分析功能或者说分析方式主要有4种:泳道/条带轨迹定量法;等高线直接定量法;菌落计数;分子量测定这三种方法中使用最为方便也是最为广泛的应该是等高线定量法(V olumn Contour)。
它通过半自动描绘电泳条带的等高线边缘来得到等高线区域内部面积,再将该面积乘以区域内平均光密度值得到条带内部总的信号量。
当然这种分析方法的弊病显而易见:无法同等得排除不同泳道的背景亮度;等高线的绘制处于“半自动”状态,即需要人为判断作为等高线标准的电泳条带的边缘;最致命的是在几个电泳条带距离十分接近的时候几乎无法绘制单一条带的轮廓(常出现连续的几个条带等高线相连而无法分离出单独条带的轮廓)。
三种方法中个人感觉最为科学和严谨的应该是泳道/条带轨迹定量法(Trace Tracking)。
这种方法使用起来步骤较为繁琐,必须通过泳道识别---电泳条带识别两个连续的步骤才能进行定量。
然而这种方式的最大优点在于它可以完全抛弃人为主观因素进行全自动定量。
他的定量方式为:首先根据不同电泳条带的光密度值绘制光密度曲线,然后计算光密度曲线下面积作为电泳条带的定量根据。
大家可能会问他能不能排除泳道背景?答案是肯定的,它能够最大程度的排除不同泳道之间的背景差异,让各个泳道上的不同电泳条带在一条几乎相同的起跑线上进行对比。

半定量RT-PCR小结(一)试验原理半定量PCR是用于确定目的基因mRNA表达的量,通常以一个管家基因作为参照,传统的方法是将RNA逆转录获得cDNA,然后使用等量的cDNA作为PCR模板,用特异引物进行扩增得到目的基因和管家基因的片段,由于不同组织或细胞等来源的目的基因表达量不同,在同一个PCR体系条件下扩增得到的产物不同,而管家基因的表达则相同(理论上,实际可能在不同组织,细胞周期或者外界因素如药物处理后也会有差异),然后使用灰度扫描软件(如Totallab 或Bandscan)对目的基因的扩增条带进行灰度扫描,得到各条带的灰度,经过内参矫正后,得到一个相对值。
用凝胶成像系统自带的分析软件分析目的条带和内参照的平均密度值,也称为灰度值。
用目的条带的平均密度值除以内参照的平均密度值,即所需要的RATIO,看density 与area 的乘积。
而两个基因可以进行同管扩增,有时无法同管扩增,则分管扩增。
同一次实验中,这个相对值理论上与其对应RNA的量成正比。
这个方法是比较粗糙的,结果也受多方面的影响,可信度并不高。
如果有条件的话建议做Northern blot 或Realtime PCR。
首先要保证PCR过程处在它对数期,第二要保证每管扩增的效率相同,第三电泳点样、凝胶成像的分辨率等等。
与经典的检测基因的表达方法如Northern 印迹、RNase 保护分析、原位杂交及S1 核酸酶分析等相比,半定量RT-PCR 法具有灵敏度高(比杂交法高1 000~10 000 倍) 、专一性好、快速简便、所需样品量少及耗费较低等诸多优点。
然而,已有的研究表明,由于用半定量RT-PCR 法分析基因表达水平的影响因素较多,导致该法目前只能用于粗略比较样品间的基因表达水平。
(二)试验步骤<1>总RNA抽提总RNA提取时,根据大鼠发育时期及总RNA量,研磨小脑组织加入适量Sol D;总RNA沉淀过夜后,用适量Sol D重悬并加入等体积异丙醇保存于-80度;<2>模板cDNA制备取出总RNA用于制备cDNA时,可以根据EP管内沉淀量判断总RNA量的多寡,视反应需要沉淀适合体积总RNA,如果沉淀量较多,一般吸取100 ul其内即含数ug 总RNA即可进行一两个反转录反应;多余的总RNA重悬加入适量Sol D重悬并加入等体积异丙醇保存于-80度;注:RT反应体系为Takara公司推荐体系,本次RT-PCR试验中,由于天气热/冰箱有点问题、试剂盒用了许久,造成其中的dNTP、酶、oligoDT降解严重,以至于严重地影响试验进行;王老师让我将dNTP、酶、oligoDT的反应量均增加为原来的1.5倍;这样使总RNA 能更充分地用于反转录为cDNA。
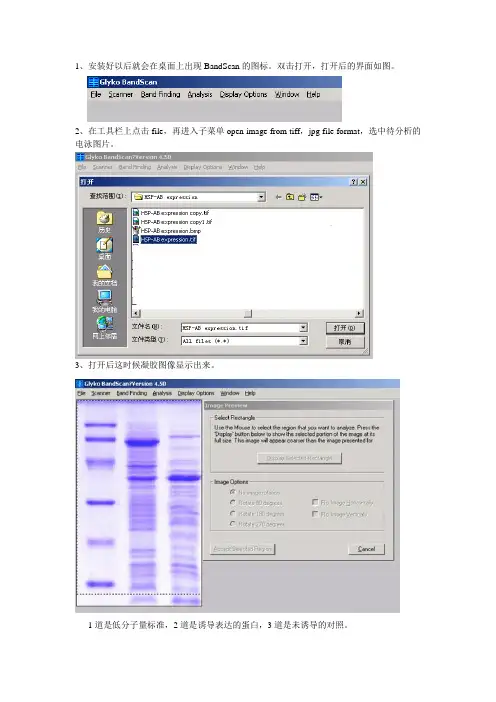
1、安装好以后就会在桌面上出现BandScan的图标。
双击打开,打开后的界面如图。
2、在工具栏上点击file,再进入子菜单open image from tiff,jpg file format,选中待分析的电泳图片。
3、打开后这时候凝胶图像显示出来。
1道是低分子量标准,2道是诱导表达的蛋白,3道是未诱导的对照。
4、按住鼠标左键从凝胶图左上角划到右下角,松开鼠标,划出的长方形框包扩了整个待分析的范围。
这时,命令框内均变成黑色的(可以操作)。
上面那个undorect可以取消选框,下面的image options可以选择是否和如何旋转图像。
如不改动,直接单击accept selected region。
4、继续会出现color to signal selections复选框。
这个是选择信号检测方式直接用默认的original image,或选择gray scale(灰阶)。
如不改动,单击use current image。
7、这时每一个条带都自动被框起来了:)红色+记号是条带的中心位置,兰色竖线是泳道位置。
同时出现了一个复选框select more or select few bands。
可以根据总灰度、最大灰度和大小为标准来自动选择蛋白条带。
拉动左边的滚动条,可以增加或减少条带的数量。
选好后单击ok。
9、现在就可以看看蛋白的表达量了。
打开window/band table。
出现了一个bandlocation的表格。
把它最大化,放到右边。
这里的数据是每个条带的中心位置(红色+的位置)。
10、在band location的数据类型data type中,选择percent signal,就会出现各个条带灰度值占本泳道条带总灰度值的百分比。
单击自己的表达条带,则条带中心的+和相应的百分数的背景都会变成绿色。
我表达的融合蛋白约占细菌总蛋白的29.8%。
我们要的数据就得到了。
应用16S rRNA 高通量测序法比较人与常用实验动物口腔菌群的异同顾东曙;陈傍柱;江霞;刘海月;那顺巴雅尔;周宏伟;顾为望【摘要】Objective To provide original reference data for oral ecosystem research, Tibet minipigs, beagle dogs, rhesus monkey, New Zealand white rabbits and Wistar rats were selected to study their respective characteristics of oral microbial mmunities and compared with normal data of humans.Methods Total DNA was extracted from the specimens of oral microbial communities of Tibet minipigs, beagle dogs, rhesus monkey, New Zealand white rabbits and Wistar rats, and used to amplify 16S rRNA V4 fragments with labeled universal primers.The diversity and structure of microbial communities from those animals were compared with that of humans using BIPES and QIIME analysis after Illumina sequencing of16S rRNA V4 fragments.Results The richness of the oral microbial communities of humans and the five species of laboratory animals was significantly different (P <0.05).Different species of animals have their own unique oral flora, among which the oral flora of the monkey is the most similar to that of humans.Conclusions Among the five species of laboratory animals, the oral microbial communities of rhesus monkeys and humans have highest similarity. Specifically, the Fusobacterium and Porphyromonas levels of rhesus monkeys is most similar to those of humans.Our findings indicate that rhesus monkeys may be suitable animal model for studies of human oral microbial communities.Tibet minipigsmay be suitable animal model for Proteobacteria studies, while beagle dogs may be appropriate for modeling of diseases related to Spirochaetes.%目的:应用16S rRNA 高通量测序法测定几种常用实验动物(西藏小型猪、比格犬、猕猴、新西兰兔、Wistar 大鼠)的口腔菌群,并和人的口腔菌群进行对比分析,为口腔微生态的动物模型研究提供基础性资料。
分子生物学软件简要功能介绍在进展分子生物学研究时,常需要用到各种计算机软件。
这些软件种类不少,功能大致一样,到底使用哪个,其实可根据各人的使用习惯来选择。
如下的软件,大局部可在网上找到全功能的版本或各种演示版或试用版,只要以软件名为关键词用搜索引擎搜索就可以了。
网上也有此类各种软件的总结。
下面为此类软件的简单介绍:1.三维分子类2.DNA分析3.RNA分析4.蛋白质分析5.生化教学6.生化工程7.序列格式转换8.引物分析9.序列综合分析10.进化树分析11.质粒绘图12.图像处理13.数据处理14.检索与阅读15.基因芯片16.其他功能软件17.化学绘图18.化学应用19.在线综合工具20.在线蛋白工具21.RNA analysis22.在线引物设计RASMOL:观看生物分子3D微观立体结构RasTop:为RasMol 的图形用户界面软件CHIME:直接在浏览器中观看3D分子MolMol:将pdb等格式的蛋白文件通过微调,存成普通的图形文件raswin.exe.gz:rasmol(win).1 rasmol新版本与汉化版本CrystInfo:用来快速、容易地构建、观察与检查晶体3D结构PDViewer:PDB格式文件的查看程序Weblab Viewlite:三维分子浏览工具与大量分子文件例子Weblab ViewerPro:三维分子浏览工具ICMLite:三维分子浏览工具,有一些其他软件没有的功能VMD:三维分子浏览工具,可以进展动态显示3D:3D分子结构观察软件WPDB:PDB文件检索显示分析软件DTMM:三维分子模型显示、编辑与构建程序Mole:高性能的大分子三维图形显示计算工具gopenmol:显示并分析分子结构与其特性POV-Rayv:生成三维图像工具软件MolPOV:将PDB文件转化为POV格式文件的软件Mol2Mol:分子文件格式转换软件PovChem:将PDB文件转化为POV格式的文件Ortep-3 for Windows:生成分子的热椭圆形点图PLATON:通用结晶学软件工具Mage:读取并演示Kinemage格式文件的专用软件 Prekin :将PDB格式文件转换为Kinemage格式文件Swiss-Pdb Viewer:PDB文件显示与分析软件DINAMO:蛋白序列排队比拟编辑与三维模型构建工具PCMoleeule2 Lite:查看PDB格式文件的免费软件StrukEd :化学分子编辑与三维模型生成软件JMVC:使用JAVA技术编写的三维分子查看器ReView:读取与分析XYZ格式三维分子文件Oscail:用来处理、定义与检查小分子单晶的软件包Moilin:分子构建与观察软件Tinker:与Moilin配套的DOS下的分子设计建模Biodesigner:免费的分子建模与显示软件MoluCAD:全功能的分子建模与显示工具软件 Viewer Activex Control:三维分子显示控件MarvinView:JAVA语言编写的化学分子二维与三维显示程序ACD/3D Viewer for ISIS:免费的ISISDraw三维显示插件Amira:高等三维显示建模系统AmiraMol:Amira 2.3 相应的显示三维分子的增强工具Visualize:分子建模和研究软件包ScientificGL:C++OpenGLAPI三维分子开发工具Sojourner:找出小蛋白的最小能量构形并实时演示的软件DNAClub:DNA处理软件JaMBW:分子生物学软件包DNATool:功能很全面的DNA序列分析工具包pDRAW:DNA分析与绘图软件,可绘制线性或环形DNA图ANNHYB:用来帮助进展PCR引物设计与基因探针设计的软件RESTRICTION ANALYSIS:限制酶消化工具ABIView:ABI格式文件显示与编辑软件Chromas:ABI格式文件显示与编辑软件Sequence viewer:获取与观察从GSDB获取的DNA序列数据与其关联特性的工具DNAssist:DNA序列分析工具DNAProbe:核苷酸序列设计工具DnaSP:DNA序列种群遗传学分析软件DFW:DNA分析软件Artemis R4:以Java语言写成的序列查看工具’ACT R1:以Java语言写成的序列比拟查看器GDA:主要用来进展不连续基因数据的统计分析 RDP:从一组排队比拟(Align)的核酸序列中查找潜在的重组体软件Sequencher:装配DNA小片段为大的连续序列或毗连(序列)群"Contig"软件MehCalc:自动计算DNA序列热力学数据的Excel电子表格宏软件基因探索者:中文界面的功能集成、高效、快捷的基因分析软件ConsInspector:DNA蛋白结合位点预测识别软件MatInd与Matlnspector:快速匹配DNA序列与共有序列的软件工具GBuilder:JAVA语言编制的用来分析与显示DNA序列的软件GenomePixelizer:帮助理解基因组中的簇基因(C1ustermggene)之间的相互关系的软件LabBook Genomic XML Viewer:图形化显示并处理GenBank序列数据的免费软件Gene Construetion Kit 2 :管理并显示克隆策略中的分子构建过程软件Genalysis:比拟基因组或大量基因序列的工具软件RNAdraw:RNA二级结构分析软件RNAstructure:预测RNA 级结构图RnaViz:RNA二级结构图绘制程序ANTHEPROT:蛋白序列分析软件包pSAAM:蛋白序列分析软件包VHMPT:螺旋状膜蛋白拓扑结构观察与编辑软件aminoXpress:免费的多功能蛋白分析软件包mmp.zip:将生化代谢中的各种途径用图表的形式表示出来linpath.zip:线性酶反响模拟软件protlab:蛋白质纯化仿真软件MOLMED.ZIP:生化根底概念演示教学程序Biochem:生化教学文件photo:光合作用教学程序Adrenalin:肾上腺素在肝糖原代谢中的作用演示Virtual Cell Lab:多媒体细胞生物学教学程序brd.zip:生物反响器(发酵罐)设计软件BioStat:BioStatB发酵罐控制程序PenSimv:青霉素发酵模拟软件BioProSim:发酵实时模拟软件vised:序列输入分析和格式转换软件ForCon:多序列文件格式转换软件SeqVerter:序列格式转换软件GeneStudio LE Version:序列格式显示、编辑与转换工具软件FASTA/BLAST SCAN:FASTA与BLAST查询输出文件的处理软件Revp:序列格式转换软件primer Premier 5.0:引物设计工具Oligo:引物分析著名软件Primer Designer:专门用 pASK-IBA~pPR-IBA表达载体免费的引物设计辅助软件Array Designer:批量设计DNA和寡核苷酸引物工具Beacon Designer:PCR定量分析分子信标(Molecularbeacon)设计软件NetPrimer:基于WEB界面的引物设计程序pcgene:分子生物应用软件 MACAW:多序列构建与分析软件Clustal W:用来对核酸与蛋白序列进展多序列比拟的软件Clustal X:ClustalWWindows界面程序FASTA:数据库中查找同源序列软件GeneDoc:对序列进展相关分析等操作BLAST与Blastcl3:数据库中查找类似序列的软件与客户端软件SeqPup 0.9:生物分子序列编辑与分析软件K-Estimator 5.5:进化基因学研究软件,评估两条核酸序列核苷酸替代数BioEdit:序列编辑器与分析工具软件DAMBE:综合性序列工具软件LaserGene:综合性序列工具软件SeaView:图形化多序列队列编辑器 Jalview:用Java语言写的多序列队列编辑器DNASIS:序列综合分析工具Genamics Expression:是一个DNA与蛋白序列分析工具Vector NTIViewer:载体查看软件Jellyfish:多功能序列分析软件ProSeq:核酸序列编辑与种群遗传学分析软件Gap4 database viewer:Gap4基因装配数据库读取显示软件SMS:DNA与蛋白序列分析与格式化在线工具集合Omiga:核酸与蛋白序列综合性分析软件Staden:综合序列分析工具软件包Vector NTISuite:综合性蛋白核酸分析工具包INCA:Java脚本语言写成的BLAST服务器客户端程序ISYS:NCGR开发的用JAVA语言写成的数个不同类生物信息软件与数据库的软件集合平台DNA Scriptor:DNA与蛋白序列综合分析软件Sequence Quickie-Calc:非常紧凑的分子生物学工具软件PhyloGrapher:用来显示与研究相类似的基因与蛋白序列之间的进化关系的软件phylip:进化树分析软件,并可绘制进化树TreeView:进化树处理软件GeneTree:比拟基因与种系进化树的程序NDE:用来编辑NEXUS格式文件的程序TreeMap:用来可视化地比拟主、从进化树的程序Spectrum:分析进化信息而不用将之转化为进化树的软件Phyltools:计算与处理进化树数据的软件tree-puzzle:核酸序列、蛋白序列相似性分析与进化树构建工具ATV:JAVA语言编写的显示"New Hampshire"与NHX格式的进化树文件软件TREECON:构建和绘制进化树的软件包ProBiosys比拟表现型分类法数据和分析计算核酸序列数据距离值的软件Plasmid Processor:绘制质粒图软件Plasmid ProcessorPro:绘制质粒图软件,与Plasmid Processor是同一个作者WinPlas:质粒绘图软件商业版DMUP:环状质粒绘图软件测试版Plasmid Toolkit:质粒绘制软件pDRAW:DNA分析与绘图软件,可绘制线性或环形DNA图SimVector:质粒图绘制软件Image Tool:科学用途的处理图像软件Image J:用Java语言写成的科学用途的处理图像软件Cross Checker:基因指纹图分析软件ALFmap:ALF(Amersham Pharmacia)图像格式转换软件Band Leader:凝胶图像处理软件Scion lmage:图像处理与分析工具OSIRIS:通用医学图像处理与分析软件Melanie 3 Viewer:免费Melanie图像查看器Smart Draw:流程图绘制软件演示版GIMPWin:图像处理自由软件ChromoZoom:比拟两个图像的一样与不同之处软件bandscan:蛋白凝胶电泳图像分析软件SigmaScanPro:图像分析软件30天全功能演示版SigmaGel:凝胶图像分析软件TotalLab:图像分析软件Lablmage:凝胶图像分析软件GelDiff:定量比拟两个2D凝胶图像的不同之处的JAVA软件Timediff:分析蛋白/基因表达图谱时间序列的JAVA软件QuantiScan:使用简单功能专一的凝胶扫描、分析软件PDQuest:分析2维凝胶并生成数据库的标准软件CurveExpert:用于ELISA标准曲线拟合的软件Cliekh Graph:实验数据作图软件Statistica:专业统计软件GraphPad PRISM:著名的数据处理软件NoSA:中文非典型数据统计分析系统CrossGraphs:多变量数据库图形显示软件SigmaPlot:绘图和数据分析软件包30天全功能评估版SYSTAT:数据统计分析与作图的利器30天全功能演示版SigmaStat:智能统计软件30天全功能演示版PeakFit:自动别离、拟合与分析非线性数据软件TableCurve:自动两维曲线拟合与经验公式查找软件TableCurve :自动三维曲面拟合与经验公式查找软件SPSS:非常权威且有名的数据统计处理软件30天全功能演示版Origin:易于使用的科学用途数据绘图与数据分析处理工具软件DATb:进展生物曲线拟合与数据分析的免费软件数据作图助手:对实验结果进展数据分析和作图的专业软件PatentIn:用于将序列专利提交给美国国家专利与商标局的辅助软件Checker:用于将序列专利提交给美国国家专利与商标局的辅助软件ica32t.exe:中国生物学文献数据库检索客户端软件PathDB检索程序:(代谢途径数据库)检索程序PubCrawler:Medline文献库与GenBank核酸序列库检索软件NetRoseBrowser:PDG格式浏览器Book Express:专门用于超星数字图书下载的工具软件EndNote:专业参考文献查询软件Reference Manager:专业参考文献查询软件Procite:参考资料检索管理软件Sequin:数据库GenBank,EMBL,DDBJ查询软件MiniViewer:数图阅览器presslt:G(NLC)〞JPG转换功能软件Refs:参考文献管理软件Scholars Aid:文献参考资料等日常资料的整理软件paperworks:免费的参考文献管理软件KD:知识仓库建库管理系统AMADA:用来组织、研究、显示、分析微数组(Microarray)数据软件ScanAlyze:进展微矩阵荧光图像分析软件Cluster:对大量微矩阵数据组进展分析处理的软件TreeView:用图形来显示Cluster软件分析的结果软件AMAD:微数组数据库ArrayMakerv:Stanford大学Brown实验室提供的基因芯片研究全套设备相配套的软件与文档J-Express:分析微矩阵(Microarray)实验获得的基因表达数据的软件digitizer:图形数字化软件,可以将曲线图转化为数据与等式Graph Paper:坐标纸打印软件DynaFit:酶动力学数据或配体—受体结合数据处理软件Migrate:从遗传学角度,估算人口移民率程序arlequin:人口遗传学软件正交设计助手:正交实验设计辅助工具软件FBAT:家族遗传相联检验的统计程序GGT32:图形化基因型表示软件CERVUS:使用共显性标记数据推断亲缘关系的软件Jarnac:代谢过程模拟软件包Gepasi:化学与生物化学反响动力学仿真与优化软件BCT:微生物趋向性模拟程序StochSim:随机生物化学反响模拟软件Bio_MW:生物化学分子量计算软件MatchCode:将蛋白和核酸序列进展简单匹配和格式化输出的中文软件Map Manager:回交、杂交与重组自交系分析遗传作图实验结果的软件MolEco:以遗传学方法评估杂乱交配频率的软件Canvas:绘图软件免疫室管理系统:中文免疫室综合软件Cyrillic:家谱绘图软件Frozen Cell Stock Monitor:用来管理储存在液氮容器中的生物样品(例如细胞系、血清等等)的程序MICE:虚拟动物饲养设施,用来帮助管理饲养设施中的实验动物软件DBsolve:代谢与酶—受体结合模拟软件boxit:管理生物样品的数据库系统GRR:检测系谱误差(pedigreeerrors)的应用软件健康药霸:药典类的软件AceDB:基因组数据库软件MAPL98、DIAL98与GEST98:El本学者编制的几个统计基因学(StatisticalGenetics)软件PED:系谱(Pedigree)绘制软件PEDRAW:系谱(Pedigree)绘制软件quantiRT:内置宏程序,用来辅助定量RT-PCR实验的Excel文件BateView:管理与追踪小型的实验鼠生殖群体的Excel文件MassXpert:帮助科学家预测与分析从蛋白组学研究中获得的蛋白质质谱数据的软件MestRe-C与MestRe-D:分析、显示与仿真1D与2D磁共振图谱的软件ACD/SpecViewer:免费光谱数据显示软件ACD/MR Viewer:ACD/HNMR用来显示ACD/NMR Predictors预测的所绘化学结构式Viewer:磁共振图谱文件的免费软件WinMDIver:分析流式细胞仪数据文件的免费软件TestDNA:根据成分值生成细胞周期FCS文件的免费工具软件Cylchred:细胞周期分析(CELLCYCLEANALYSIS)软件gX-Path Vision:生成、编辑与显示生物代谢途径的工具软件生物五笔:生物医学专业输入法ACD/CHEMSKETCH:绘制分子结构的免费软件与其汉化版Chemfont:化学符号与TureType字体,可以在Word中直接插入文章中clip.zip:化学图片集,含有近400幅与化学有关的GIF图片ISIS DRAW:绘制化学结构式的免费软件AutoNom:ISIS/Draw软件的插件(自动生成符合IUPAC命名规如此的化合物名称) ChemWindows:绘制化学结构式的免费软件MarvinSketch:JAVA语言编写的小巧好用的化学结构式绘制程序ACD/Structure Drawing Applet:绘制化学结构式免费JAVA小程序ChemPen:绘制化学结构式软件ChemPen+:绘制化学结构式软件ChemPen:绘制化学结构式软件mmcalc.exe:分子量计算器hxfc.zip:化学方程式配平软件cmcalc10.exe:化学试剂制备计算器alkne.exe:有机化学命名练习程序chembl32.zip:Windows95下的免费化学方程式配平程序 periodic.zip:小巧的元素周期表ptab32.zip:高级元素周期表CAF:计算机辅助配方软件CFT:化学式教师元素屋:查询112种元素的各个信息的中文软件化学反响方程式编辑器:制作化学反响方程式、离子方程式、分子式、离子式等CRS:化学反响方程式配平器Model ChemLabv:化学实验教育软件与其汉化版periodic.exe:免费的元素周期表化学品电子手册:一个综合性的化学品手册Biology Workbench:基于WEB的生物学综合工具sewer:网上常用在线工具集合,本地版BCM Search Launcher:蛋白序列二级结构预测综合站点DAS:蛋白跨膜预测服务器、输入蛋白序列,预测跨膜区域TopPred:蛋白预测服务器提供的膜蛋白拓扑学预测在线工具SOSUI:膜蛋白分类和二级结构预测在线工具PSIpred-MEMSAT:进展二级结构预测与跨膜拓朴结构预测HMMTOP:预测蛋白序列的跨膜螺旋与拓扑结构服务器SMART:提供蛋白序列,在结构域数据库中查询/显示出其结构域与跨膜区等TMpred:预测蛋白序列跨膜区TMHMM:预测蛋白的跨膜螺旋The PredictProtein server:提供蛋白数据库查询,预测蛋白各种结构的服务SPLIT:膜蛋白二级结构预测服务器PRED-TMR:提供基于SwissProt数据库统计分析的预测蛋白跨膜片段的服务CoPreThi:基于INTERNET的JAVA程序,预测蛋白的跨膜区TMAP:提供预测蛋白跨膜片段的服务21.RNA analysisPattern Search and Discovery:巴斯德研究所提供的常用RNA在线分析工具DNA sequence analysis:巴斯德研究所提供的常用特征序列查询工具Search Genes and Coding Regions:巴斯德研究所提供的常用DNA在线分析工具Oligonucleotide Calculator:巴斯德研究所提供的基因与编码区查找工具解链温度计算器:JAVA语言写的寡核苷酸计算器,给出核酸序列,计算GC百分比、解链温度、长度、分子量。
【关键字】文件Band Leader凝胶电泳图象分析工具,共享软件,45天之内得向Magnitec公司注册,无注册费。
帮助文件有该软件所有功能的指南。
提供处理DNA或蛋白分子凝胶电泳图象和从凝胶电泳图象获得相关数据的工具。
对于那些进行遗传学和分析生物学研究和开发等涉及到凝胶电泳及相关技术的人员是一个非常有帮助的软件.从扫描仪获得凝胶电泳的图象之后,Band Leader可做如下操作:1,将图象显示在计算机屏幕上2,对图象作一些处理使感兴趣的信号更明显3,分析图象,通过对所选择区域进行密度分析来获得相关数据4,将获得的数据存盘系统要求:硬件:386或更高配置,鼠标,软件:Windows 操作系统软件安装:解开BandLeader.zip,执行install.exe文件,会在选定的路径下看到包含以下文件的文件夹BANDLEADER(缺省路径C:\):1. LEADER.EXE (executable)2. LEADER.HLP (help file)2. DIB.DRV (driver)3. README.WRI (This file)4. *.BMP (various sample images)5. REGISTER.WRI (registration form)运行:双击leader.exe即可运行。
出现如图主窗口,用法:点击下拉菜单file, 选取load, 即可载入相应的图形文件,可识别bmp或tif格式的灰阶图形,彩色图形将被转化为灰阶,图形文件由扫描仪等得到。
载入图形后,成如下界面:在VIEW→ZOOM IN (ZOOM OUT)中,可对载入的图形放大、缩小,在PROCESS 中,可转换前景色和背景色(REVERSE),还可以对图形进行降噪处理(CLEAR NOISE),即通过软件的运算使图形背景更干净一些。
OPTION,选择参数,如GRID网格行列各多少格,SPOT类型(方点或圆点,点的大小,等),如何判断BAND等。
两分钟学习凝胶分析软件BandScan(Step by step )
转载请注明来自丁香园
发布日期: 2005-07-02 12:14 文章作者: palmyard
文章编辑: admin
Step by step to BandScan
>>By Palmyard.
按:现在有很多常用的分子生物学软件,但对于初学者,尤其是不太擅长计算机者,使用起来有相当难度。
琢磨说明书,或者自己一点点试验,难免觉得头绪纷繁,乃至兴味索然。
后来我发现,其实用一个实例,从头到尾示范一遍,可以在几分钟内直观地学会这个软件的基本使用方法。
其他的一些情况就可以自己去摸索了,这样学起来很快。
因此我打算做一个常用分子生物学软件STEP BY STEP的系列。
这个想法在心里很久了,但是一直没有时间付诸实践,的确是因为没有较为空闲的时间。
今天就从BandScan这个软件开始。
由于不是很熟悉制作过程,奋战一整天,终于完成了第一个实例学习。
BandScan是一个很常用的凝胶图像分析软件,我们用它重要是为了分析蛋白表达量。
现在,我就以我自己表达的一个蛋白图,来示范这个软件分析的过程。
最后结果是得到目的蛋白占总蛋白的百分数。
1.安装软件BandScan5.0。
主程序可以在网上找到(请搜索以往的帖子或用google搜索BandScan_dl.exe)。
安装以后加个补丁(如果找不到补丁可以email我*****************,这有4.3、4.5和5.0的补丁,请尽量自己先找一下)。
2.安装好以后就会在桌面上出现BandScan的图标。
双击打开。
3.打开后的界面如图。
.打开一张扫描好的电泳图。
BandScan可以识别TIF和JPG格式的图片,很方便。
打开工具栏上的file/open tiff,jpg file format,选中待分析的图片。
5.打开。
这时候凝胶图像显示出来。
1道是低分子量标准,2道是诱导表达的蛋白,3道是未诱导的对照。
旁边有一个图像预览窗口Image Preview,可以看到,除了cancal,其它命令框都是灰色的(不可操作)。
其实这里是要你先选定一个图像处理的范围(select rectangle)。
6.从凝胶图左上角按住鼠标左键划到右下角,松开鼠标,划出的长方形框包扩了整个待分析的范围。
这时,命令框内均变成黑色的(可以操作)。
上面那个undo rect可以取消选框,下面的image options可以选择是否和如何旋转图像。
本例中均不改动,直接单击accept selected region。
7.出现color to signal selections复选框。
这个是选择信号检测方式,直接用默认的original image,或选择gray scale(灰阶)。
本例中不改动,单击use current image。
8.出现如下分析框。
它是自动转换成灰阶的。
其左上角的数字是鼠标所在的坐标和灰度值。
移动鼠标就可
以看到它的变化。
9.现在让BandScan来分析一下电泳条带吧:)打开工具栏上的band finding/find bands,出现一个让你选择有几条泳道的框selecting number of lanes,我们设置成3道。
10.看,每一个条带都自动被框起来了:)红色+记号是条带的中心位置,兰色竖线是泳道位置。
同时出现了一个复选框select more or select few bands。
可以根据总灰度、最大灰度和大小为标准来自动选择
蛋白条带。
拉动左边的滚动条,可以增加或减少条带的数量。
选好后单击ok。
11.现在就可以看看蛋白的表达量了。
打开window/band table。
出现了一个band location的表格。
把它最大化,放到右边。
这里的数据是每个条带的中心位置(红色+的位置)。
12.在band location的数据类型data type中,选择percent signal,就会出现各个条带灰度值占本泳道条带总灰度值的百分比。
13.单击自己的表达条带,则条带中心的+和相应的百分数的背景都会变成绿色。
我表达的融合蛋白约占细
菌总蛋白的29.8%。
我们要的数据就得到了。
自己再重复一下整个过程,会发现,原来是如此简单:)
总结:真的只是领进门。
因为BandScan绝不仅仅是这么简单,它还可以做很多事情。
比如通过设定一个条带的量(标准)来推断表达蛋白的量。
比如用手动方式加入未被自动选择的条带,等等。
举个例,我们发现我的目的条带上面有几个浅条带没有被选上,可以用band finding/manually insert a band命令把它们选上。
这时,条带百分数也会随之改变。
这样直观的学习想必是大家都喜欢的吧:一个例子学会一个软件。
我更加希望,有更多的高手可以用这种方式把初学者迅速领进门。
有什么问题,请大家跟帖子或联系我,也希望大家支持我,把这件事情坚持做
下去。