选择标记基因
- 格式:docx
- 大小:3.32 KB
- 文档页数:2


基因工程中标记基因的作用一、引言随着科技的不断进步,基因工程已经成为了一个备受关注的领域。
标记基因是其中一个重要的组成部分。
标记基因是指在目标基因上加入一段特定序列,以便于在后续的研究中对该基因进行追踪和分析。
本文将从多个方面介绍标记基因在基因工程中的作用。
二、标记基因的种类1. 绿色荧光蛋白(GFP):GFP是一种广泛应用于生物学研究中的标记基因,它可以发出绿色荧光,并且可以被很多生物体表达。
2. β-半乳糖苷酶(LacZ):LacZ是一种常用的β-半乳糖苷酶,它可以被转化为蓝色染料,在研究中常用于检测目标基因是否表达。
3. 抗生素抗性标记:抗生素抗性标记是通过将目标基因与抗生素抗性相关序列结合起来,使得目标细胞能够在含有相应抗生素的培养基上存活下来。
三、使用标记基因进行转化1. 选择正确的标记基因:在进行基因转化之前,需要选择一个合适的标记基因。
一般来说,应该选择能够被目标细胞表达的标记基因。
2. 将标记基因与目标基因结合:在进行转化之前,需要将目标基因与所选的标记基因结合起来。
这可以通过PCR、限制性内切酶等方法实现。
3. 将结合后的DNA导入到目标细胞中:使用适当的方法将结合后的DNA导入到目标细胞中。
这可以通过电穿孔、微注射等方法实现。
四、使用标记基因进行筛选1. 抗生素抗性筛选:将转化后的细胞培养在含有相应抗生素的培养基上,只有含有抗生素抗性序列的细胞才能够存活下来。
2. 荧光筛选:通过观察GFP或其他荧光蛋白发出的荧光信号,可以确定哪些细胞已经成功地表达了目标基因。
五、使用标记基因进行定位1. 确定目标细胞类型:在使用标记基因进行定位之前,需要确定所研究的细胞类型。
不同类型的细胞可能对同一标记基因的表达有不同的反应。
2. 观察标记基因的表达:通过观察标记基因的表达情况,可以确定目标基因在细胞中的位置。
例如,如果目标基因被与GFP结合,那么可以通过观察细胞中GFP的分布情况来确定目标基因在细胞中的位置。


名词解释汇总1.分子杂交不同的DNA 片段之间,DNA 片段与RNA 片段之间,如果彼此间的核苷酸排列顺序互补也可以复性,形成新的双螺旋结构。
这种按照互补碱基配对而使不完全互补的两条多核苷酸相互结合的过程称为分子杂交。
2.cDNA文库以mRNA为模板,经反转录酶催化,在体外反转录成cDNA,与适当的载体(常用噬菌体或质粒载体)连接后转化受体菌,则每个细菌含有一段cDNA,并能繁殖扩增,这样包含着细胞全部mRNA信息的cDNA克隆集合称为该组织细胞的cDNA文库。
cDNA文库特异地反映某种组织或细胞中,在特定发育阶段表达的蛋白质的编码基因,因此cDNA文库具有组织或细胞特异性。
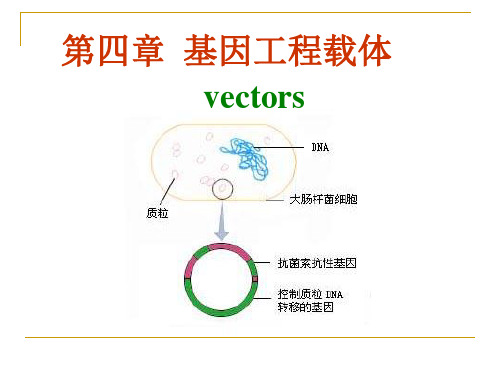
3.选择标记基因选择基因(又称选择标记基因),主要是一类编码可使抗生素或除草剂失活的蛋白酶基因,这种基因在执行其选择功能时,通常存在检测慢(蛋白酶作用需要时间)、依赖外界筛选压力(如抗生素、除草剂)等缺陷。
4.ORF开放阅读框(Open Reading Frame, ORF)从起始密码子开始,是DNA序列中具有编码蛋白质潜能,一段无终止密码子打断的碱基序列。
5.转化转化(transformation)是某一基因型的细胞从周围介质中吸收来自另一基因型的细胞的DNA 而使它的基因型和表现型发生相应变化的现象。
该现象首先发现于细菌。
6.转染专指感受态的大肠杆菌细胞捕获和表达噬菌体DNA分子的过程7.转导转导(transduction)由噬菌体将一个细胞的基因传递给另一细胞的过程。
它是细菌之间传递遗传物质的方式之一。
其具体含义是指一个细胞的DNA或RNA通过病毒载体的感染转移到另一个细胞中。
8.质粒不亲和性在没有选择压力下,两种不一样的质粒不能在同一宿主细胞系中稳定共存的现象9.RACERACE是基于PCR技术基础上由已知的一段cDNA片段,利用锚式PCR,快速扩增cDNA末端从而获得已知mRNA内一段小序列与3‘或5’的cDNA序列技术。

ura3筛选标记原理一、背景介绍URA3基因是酵母中的一种选择性标记,能够使酵母细胞在含有5-FOA(5-氟乙酸)的培养基上死亡。
因此,在URA3筛选标记下,只有不含URA3基因的酵母菌落能够生长,从而实现对含有特定表达物的酵母菌落的筛选。
二、URA3筛选标记原理1. URA3基因结构URA3基因位于酵母染色体Ⅴ号,包括一个启动子、一个编码区和一个终止子。
编码区共有7个外显子和6个内含子,编码了一个分子量为37kDa的蛋白质。
该蛋白质具有UMP合成酶活性,在细胞内合成UMP(尿苷一磷酸)。
2. 5-FOA代谢途径5-FOA是一种可以被细胞代谢成为有毒代谢产物5-FU(5-氟尿嘧啶)的化合物。
在细胞中,5-FOA首先被转化为FUR1底物FUMP(氟尿嘧啶核苷一磷酸),然后由FUR1酶催化进一步转化为5-FU,最终导致细胞死亡。
3. URA3筛选标记原理URA3基因的存在使得酵母菌落在含有尿苷的培养基上生长。
但是,在含有5-FOA的培养基上,只有不含URA3基因的酵母菌落能够生长。
这是因为,5-FOA可以被转化为有毒代谢产物5-FU,而URA3基因编码的UMP合成酶正是合成UMP的关键酶。
当酵母细胞中含有URA3基因时,细胞能够合成足量UMP来对抗5-FOA代谢产物5-FU的毒性,从而保证细胞存活;而当酵母细胞中不含URA3基因时,则无法合成足量UMP来对抗5-FU的毒性,最终导致细胞死亡。
三、应用URA3筛选标记广泛应用于酵母遗传学中。
利用该标记可以筛选出表达特定蛋白质或具有特定表型的单倍体或二倍体菌落,并进行后续研究。
此外,在重组DNA技术中,URA3筛选标记也被用于筛选重组酵母菌落。
四、总结URA3筛选标记是一种常用的酵母选择性标记,通过对5-FOA代谢途径的利用,实现了对含有特定表达物的酵母菌落的筛选。
该标记在酵母遗传学和重组DNA技术中得到广泛应用。
正负筛选标记基因【原创实用版】目录一、正负筛选标记基因的定义与作用二、正负筛选标记基因的应用实例三、正负筛选标记基因的优势与局限四、未来发展趋势与挑战正文一、正负筛选标记基因的定义与作用正负筛选标记基因,是指在基因工程中,用于筛选带有特定基因的细胞或生物的一对基因。
其中,正筛选标记基因能够使细胞或生物表现出某种特定性状,从而便于筛选;而负筛选标记基因则使细胞或生物无法表现出该特定性状,从而间接筛选出带有正筛选标记基因的细胞或生物。
这种标记基因的应用,极大地提高了基因工程的效率和准确性。
二、正负筛选标记基因的应用实例以抗药性基因为例,我们可以将抗药性基因与正筛选标记基因(如荧光蛋白基因)和负筛选标记基因(如抗生素抗性基因)同时导入目标细胞。
然后,通过荧光显微镜筛选出带有荧光蛋白的细胞,再通过抗生素筛选出带有抗生素抗性基因的细胞。
这样就可以得到同时带有抗药性基因、荧光蛋白基因和抗生素抗性基因的细胞。
三、正负筛选标记基因的优势与局限正负筛选标记基因技术的优势主要体现在其高效性和特异性上。
它可以在一次实验中筛选出大量带有目标基因的细胞或生物,大大提高了基因工程的效率。
同时,由于正负筛选标记基因的作用机制,使得筛选结果具有高度特异性,减少了假阳性和假阴性的可能性。
然而,正负筛选标记基因技术也存在一些局限。
首先,它需要引入两个以上的外源基因,可能会对宿主细胞或生物的基因组造成影响。
其次,对于某些特殊情况(如筛选标记基因本身与目标基因位于同一区域),正负筛选标记基因技术可能无法有效应用。
四、未来发展趋势与挑战随着基因编辑技术的不断发展,正负筛选标记基因技术也将迎来新的发展机遇。
一方面,研究人员将开发出更加高效、安全的筛选标记基因;另一方面,也将探索将正负筛选标记基因技术与其他基因编辑技术相结合,以提高基因编辑的效率和准确性。
然而,这也将带来新的挑战,如如何确保筛选标记基因的安全性、如何解决多重筛选带来的复杂性等问题。
puc57质粒序列一、前言pUC57质粒是一种常用的质粒,广泛应用于分子生物学实验中。
本文将从pUC57质粒的序列结构、特点、应用等方面进行详细介绍。
二、pUC57质粒的序列结构pUC57质粒是一种小型环状DNA分子,其大小约为2686bp。
它由多个不同功能的序列组成,包括起始位点(ori)、选择性标记基因(ampR)、多克隆位点(MCS)等。
1. 起始位点(ori)起始位点是DNA复制开始的地方。
在pUC57质粒中,起始位点为ColE1来源的ori,长度约为100bp。
该序列具有高度保守性,并且能够与DNA复制酶结合,启动DNA复制过程。
2. 选择性标记基因(ampR)选择性标记基因是一种能够使细菌对抗抗生素的基因,在pUC57质粒中,选择性标记基因为ampR。
当细菌含有该基因时,可以通过加入氨苄青霉素来筛选出含有该质粒的细菌。
3. 多克隆位点(MCS)多克隆位点又称为多重克隆位点或多克隆克隆位点,是一种能够容纳多个限制性内切酶切割位点的DNA序列。
在pUC57质粒中,MCS 的长度约为70bp,包含多个限制性内切酶切割位点,能够方便地进行DNA片段插入和克隆。
三、pUC57质粒的特点1. 高拷贝数pUC57质粒具有高拷贝数,即每个细胞中含有多个复制的质粒。
这一特点使得pUC57质粒在分子生物学实验中应用广泛。
2. 易于操作pUC57质粒具有较小的大小和简单的结构,使得它易于操作和改造。
同时,它还具有很高的转化效率和筛选效率。
3. 多克隆位点pUC57质粒中包含了多个限制性内切酶切割位点,方便进行DNA片段插入和克隆。
4. 选择性标记基因pUC57质粒中含有选择性标记基因ampR,可以通过加入抗生素来筛选出含有该质粒的细菌。
四、pUC57质粒的应用1. 克隆表达pUC57质粒中含有多克隆位点和选择性标记基因,可以方便地进行DNA片段插入和克隆。
同时,它还具有较小的大小和高拷贝数,使得它在表达载体中应用广泛。
筛选marker基因的方法在生物信息学和分子生物学领域,marker基因的筛选对于理解生物过程和疾病机制至关重要。
本文将详细介绍筛选marker基因的方法,以帮助研究人员在探索生物学奥秘的道路上迈出坚实的步伐。
一、什么是marker基因?Marker基因,即标记基因,是指在特定生物过程中具有代表性的基因,通常用于指示生物体的某种生理或病理状态。
通过研究marker基因,我们可以更好地理解生物过程,揭示疾病发生发展的奥秘。
二、筛选marker基因的方法1.数据库筛选法数据库筛选法是利用已有的生物信息学数据库,如GeneCards、OMIM、UniProt等,通过关键词搜索、功能注释和表达谱分析等方法,筛选出与特定生物过程或疾病相关的marker基因。
2.生物信息学分析(1)差异表达分析:通过高通量测序技术(如RNA-seq)获得不同样本的基因表达数据,利用DESeq2、EdgeR等差异表达分析软件,筛选出显著差异表达的基因。
(2)共表达网络分析:基于基因表达数据,构建共表达网络,利用Cytoscape等软件进行可视化分析,筛选出与目标基因紧密相关的核心基因。
(3)功能富集分析:利用DAVID、GOA等工具,对差异表达基因进行功能富集分析,找出与特定生物过程或通路相关的基因。
3.实验验证(1)qPCR:通过实时荧光定量PCR技术,验证候选marker基因在不同样本中的表达水平。
(2)免疫组化:利用免疫组化技术,检测候选marker基因在组织中的表达和分布情况。
(3)细胞功能实验:通过基因敲除或过表达技术,观察候选marker基因对细胞生物学功能的影响。
三、总结筛选marker基因的方法多种多样,研究者可以根据具体研究背景和需求,选择合适的方法进行筛选。
需要注意的是,筛选出的marker基因需经过严格的实验验证,以确保其可靠性和准确性。
选择标记基因
选择标记基因是一种常见的实验技术,它在基因组中定位和标记感兴趣的基因。
通过选择标记基因,研究人员能够更好地理解基因在细胞功能和生物过程中的作用。
本文将介绍选择标记基因的原理、方法和应用。
一、选择标记基因的原理
选择标记基因的原理是利用特定的标记物将感兴趣的基因与其他基因区分开来。
这些标记物可以是荧光蛋白、酶或其他易于检测的分子。
通过将标记物与目标基因连接在一起,研究人员可以追踪、定位和检测目标基因的表达。
1. 转基因技术:通过将标记基因导入到目标细胞或生物体中,使其表达。
这种方法常用于研究基因功能和表达调控机制。
2. 基因编辑技术:利用CRISPR/Cas9等基因编辑技术,直接修改基因组中的目标基因,使其带有标记物。
这种方法可以实现精确的基因编辑和标记。
三、选择标记基因的应用
1. 研究基因功能:通过标记特定基因,研究人员可以观察其在细胞生长、分化和发育等过程中的作用,揭示基因功能的机制。
2. 观察基因表达:标记基因可以用于研究基因的转录和翻译过程,帮助研究人员了解基因的表达调控机制。
3. 追踪细胞命运:通过标记基因,研究人员可以追踪细胞的分化、迁移和增殖等过程,研究细胞命运决定的机制。
4. 疾病研究:选择标记基因可以用于研究疾病的发病机制和治疗方法。
例如,标记癌基因可以帮助研究人员了解癌细胞的生物学特性和治疗靶点。
四、总结
选择标记基因是一种重要的实验技术,它在基因组研究中扮演着重要角色。
通过选择标记基因,研究人员可以更好地了解基因的功能、表达调控和细胞命运等方面。
选择标记基因的方法多种多样,包括转基因技术和基因编辑技术。
选择标记基因在基础研究和应用研究中具有广泛的应用前景,有助于推动基因组学和生物医学的发展。