全基因组选择及其在奶牛育种中的应用
- 格式:doc
- 大小:60.50 KB
- 文档页数:6

奶牛育种新技术近年来,随着科技的不断进步,奶牛育种也迎来了新的技术突破,这些新技术的应用不仅提高了奶牛的产奶量和品质,还有助于改善奶牛的健康状况和适应环境能力。
本文将介绍几种新的奶牛育种技术,并探讨其对农业生产的影响。
基因编辑技术是奶牛育种的一项重要突破。
通过基因编辑技术,科学家们可以精确地改变奶牛的基因组,以实现特定的育种目标。
例如,可以通过编辑奶牛的基因来增加其产奶量、改善乳脂肪和乳蛋白含量,甚至提高奶牛对特定疾病的抵抗力。
这种技术的应用,不仅可以提高奶牛的生产力,还有助于减少对抗生素的使用,从而降低了食品中抗生素残留的风险。
利用人工智能技术进行奶牛育种也成为一种热门趋势。
人工智能技术可以通过分析大量的数据,提供更准确的奶牛育种建议。
通过对奶牛的遗传信息、饲养环境和生产表现等方面进行综合分析,人工智能可以帮助农民更好地选择适合的交配组合,提高奶牛的遗传质量。
此外,人工智能还可以监测奶牛的行为和健康状况,及时发现异常情况并采取措施,提高奶牛的健康和生产效益。
除此之外,基于基因组选择的技术也在奶牛育种中得到了广泛应用。
基因组选择是通过测量奶牛基因组中的单核苷酸多态性(SNP)来评估奶牛的遗传价值。
通过分析SNP与特定性状之间的关系,可以选择具有优良遗传特性的奶牛进行繁殖,提高整个群体的遗传水平。
基因组选择不仅可以加速育种进程,还可以提高奶牛的遗传进展,从而提高产奶量和品质。
生殖技术的进步也为奶牛育种带来了新的机遇。
例如,体外受精技术可以将优良种牛的精子和卵子在实验室中结合,然后将受精卵移植到母牛的子宫中发育。
这种技术不仅可以提高种牛的繁殖效率,还可以加速优良基因的传播。
另外,胚胎冷冻技术也可以延长优良种牛的繁殖寿命,提高种牛的繁殖效益。
奶牛育种新技术的出现为农业生产带来了革命性的变化。
这些新技术不仅可以提高奶牛的产奶量和品质,还可以改善奶牛的健康状况和适应环境能力。
然而,我们也应该看到,新技术的应用还面临一些挑战和风险,如技术成本、道德伦理和食品安全等问题。

gwas 育种例子
全基因组关联分析(Genome-wide Association Study, GWAS)是一种用于识别与复杂性状或疾病相关的基因变异的方法。
在育种领域,GWAS可以用于鉴定与农艺性状、产量、抗病性等相关的基因变异,从而加速育种进程。
以下是一些使用GWAS进行育种的例子:
1. 玉米产量:研究人员使用GWAS技术,鉴定了与玉米产量相关的多个基因变异。
这些变异可以作为育种标记,用于培育高产量的玉米品种。
2. 小麦抗病性:通过GWAS,科学家们发现了与小麦抗病性相关的基因变异。
这些变异可以用于培育抗病性强的小麦品种,提高作物的抗病能力。
3. 水稻耐旱性:在水稻中,研究人员利用GWAS技术,鉴定了与耐旱性相关的基因变异。
这些变异可以为培育耐旱的水稻品种提供指导。
4. 奶牛产奶量:在奶牛育种中,GWAS被用于识别与产奶量相关的基因变异。
这些变异可以作为选择育种的依据,提高奶牛的产奶性能。
5. 苹果品质:在苹果育种中,研究人员利用GWAS技术,鉴定了与苹果品质相关的基因变异。
这些变异可以为培育优质苹果品种提供帮助。
需要注意的是,GWAS虽然能够提供与复杂性状相关的基因变异的信息,但不能直接用于育种。
在实际育种过程中,还需要结合传统的育种方法和分子标记辅助选择等技术,以提高育种效率和准确性。

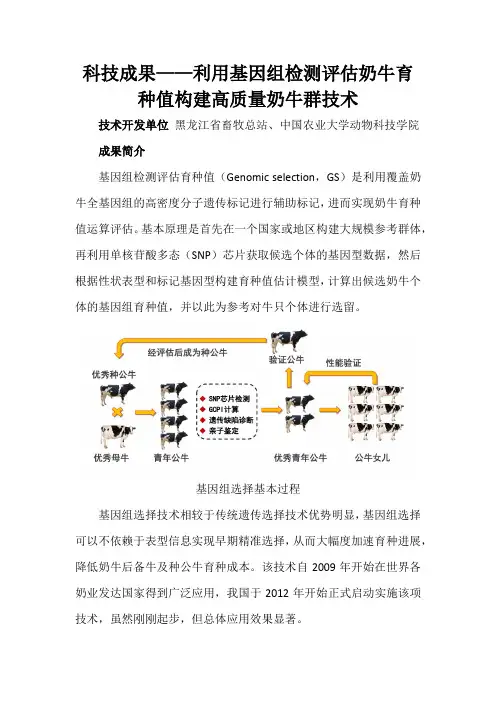
科技成果——利用基因组检测评估奶牛育种值构建高质量奶牛群技术技术开发单位黑龙江省畜牧总站、中国农业大学动物科技学院成果简介基因组检测评估育种值(Genomic selection,GS)是利用覆盖奶牛全基因组的高密度分子遗传标记进行辅助标记,进而实现奶牛育种值运算评估。
基本原理是首先在一个国家或地区构建大规模参考群体,再利用单核苷酸多态(SNP)芯片获取候选个体的基因型数据,然后根据性状表型和标记基因型构建育种值估计模型,计算出候选奶牛个体的基因组育种值,并以此为参考对牛只个体进行选留。
基因组选择基本过程基因组选择技术相较于传统遗传选择技术优势明显,基因组选择可以不依赖于表型信息实现早期精准选择,从而大幅度加速育种进展,降低奶牛后备牛及种公牛育种成本。
该技术自2009年开始在世界各奶业发达国家得到广泛应用,我国于2012年开始正式启动实施该项技术,虽然刚刚起步,但总体应用效果显著。
基因组选择基本原理应用情况目前基因组选择技术已经得到广泛应用,奶业发达国家的青年公牛选择已经100%应用基因组选择技术,截止到2019年美国和加拿大基因组参考群体已达到448007头荷斯坦牛(其中公牛群体56970头)。
荷兰、德国和北欧三国也相继成立了基因组选择技术平台,参考群体规模也在不断扩大。
我国奶牛基因组选择技术平台是从2008年开始研发,中国农业大学奶牛分子育种团队于2012年申请《中国荷斯坦牛基因组选择分子育种技术体系的建立与应用》项目,已通过教育部科技成果鉴定,开始在全国推广应用,实现了青年公牛基因组选择全覆盖。
截至2020年12月,中国荷斯坦牛基因组选择参考群体累计3497头,荷斯坦青年公牛全部完成了基因组检测和评估。
技术效果2009年我国奶牛育种开始应用基因组选择技术,该技术显著提高了奶牛遗传世代进展,降低种公牛及母牛核心群培育成本。
参测牛群主要经济性状的遗传进展得到加速提升,产奶量的平均遗传进展提升58.1kg/年;乳脂量的平均遗传进展提升1.42kg/年;乳蛋白量的平均遗传进展提升 1.76kg/年,在产奶性能遗传进展方面增加直接效益4.6亿/年。

全基因组选择育种值估计是一种利用覆盖全基因组的高密度分子标记进行选择育种的方法。
其原理是通过构建预测模型,根据基因组估计育种值(Genomic Estimated Breeding Value,GEBV)进行早期个体的预测和选择,从而缩短世代间隔,加快育种进程,节约大量成本。
统计模型是全基因组选择的核心,影响着全基因组预测的准确度和效率。
传统预测方法基于线性回归模型,但难以捕捉基因型和表型间的复杂关系。
相较于传统模型,非线性模型(如深度网络神经)具备分析复杂非加性效应的能力,人工智能和深度学习算法为解决大数据分析和高性能并行运算等难题提供了新的契机,深度学习算法的优化将会提高全基因组选择的预测能力。
全基因组选择已应用于奶牛、生猪的品系选育中,但在家禽育种方面的研究和应用相对较少。
随着分子标记检测技术不断发展,分子育种进入了全基因组选择时代,这将推动现代育种向精准化和高效化方向发展。


中国乳用种公牛遗传评估概要随着中国乳制品消费的不断增加,乳用种公牛的遗传评估变得越来越重要。
乳用种公牛的遗传评估是指对公牛的遗传背景和潜在遗传能力进行全面评估,以确定其在育种计划中的适用性和贡献。
本文将概述中国乳用种公牛遗传评估的主要内容和方法。
乳用种公牛的遗传评估需要考虑的主要指标包括产奶量、蛋白质含量、乳脂含量、体型特征等。
这些指标是评估公牛育种潜力的重要依据。
产奶量是指公牛后代在特定条件下的平均每天产奶量,是乳制品生产的关键指标之一。
蛋白质含量和乳脂含量则决定了乳制品的质量和价值。
体型特征是指公牛的身体大小、体重等特征,也与产奶潜力密切相关。
乳用种公牛的遗传评估方法主要包括遗传参数估计、基因组选择和遗传评价三个方面。
遗传参数估计是通过对公牛后代数据的分析,计算得出公牛遗传指标的相关参数,如遗传方差、遗传相关等。
这些参数可以用来评估公牛的遗传潜力和遗传背景。
基因组选择是指利用现代遗传学技术,通过测序和分析公牛基因组的方法,筛选出具有优良遗传特征的个体,以提高育种效果。
遗传评价是指根据公牛后代的表现和遗传背景,对公牛进行综合评价,确定其育种价值和使用范围。
乳用种公牛的遗传评估还需要考虑到环境因素的影响。
环境因素对公牛后代的生长和表现有一定影响,因此在遗传评估中需要进行环境校正。
环境校正是通过对公牛后代数据进行统计分析,排除环境因素的干扰,更准确地评估公牛的遗传能力。
乳用种公牛的遗传评估是育种工作中的重要环节,对于改良乳牛品种、提高乳制品产量和质量具有重要作用。
通过遗传评估,可以筛选出具有优良遗传特征的公牛,作为育种计划的重要资源。
遗传评估还可以为乳制品生产提供科学依据,指导养殖户选择适合的公牛种源,提高养殖效益。
中国乳用种公牛的遗传评估是乳制品生产和育种工作中的重要环节。
通过对公牛遗传背景和潜在遗传能力的评估,可以筛选出具有优良遗传特征的个体,提高育种效果和乳制品质量。
乳用种公牛的遗传评估需要考虑产奶量、蛋白质含量、乳脂含量、体型特征等指标,采用遗传参数估计、基因组选择和遗传评价等方法进行评估。

奶牛tpi指标解读一、引言奶牛是现代畜牧业中最重要的家畜之一,其产奶量、奶品质和繁殖性能直接关系到乳品产业的发展。
随着科技的进步和育种技术的不断创新,奶牛育种逐渐成为提高奶牛生产性能的关键。
在众多的奶牛品种中,荷斯坦奶牛以其高产的奶量、良好的综合生产性能和较强的适应性而受到广泛关注。
为了评估荷斯坦奶牛的遗传性能,美国奶牛育种协会采用了基因组综合选择指数(TPI)这一指标。
本文将对奶牛TPI指标进行详细解读,以期为广大奶牛育种从业者提供参考。
二、TPI指标的定义及作用1. 定义TPI(Total Performance Index)是指奶牛育种中对荷斯坦牛进行遗传评估的综合选择指数。
它是一种基于基因组测序技术的遗传评估方法,可以全面、准确地评估奶牛的遗传性能。
2. 作用TPI指标主要用于衡量荷斯坦奶牛在生产性能、体型和繁殖等方面的遗传优势。
TPI值越高,表示奶牛的遗传性能越好。
通过比较不同奶牛的TPI值,可以筛选出具有较高遗传潜力的优秀奶牛,为奶牛育种提供科学依据。
三、TPI指标的计算方法TPI指标的计算涉及到多个性状的遗传评估,包括产奶量、乳脂量、蛋白量、体型评分和繁殖性能等。
计算方法如下:1. 首先,对每个性状进行遗传评估,得到相应的育种值。
2. 然后,根据各性状的育种值,计算奶牛的净价值(NM)。
净价值是衡量奶牛在生产性能、体型和繁殖等方面综合效益的指标。
3. 最后,将奶牛的净价值与体型评分相乘,得到TPI值。
四、TPI指标在奶牛育种中的应用1. 筛选优秀奶牛通过比较奶牛的TPI值,可以筛选出具有较高遗传潜力的优秀奶牛。
在国内外的奶牛育种实践中,TPI值已成为选拔优秀奶牛的重要依据。
2. 指导奶牛繁殖TPI指标可以反映奶牛的繁殖性能。
在育种过程中,可以根据TPI值选择具有较高繁殖潜力的奶牛进行繁殖,以提高繁殖效率和后代品质。
3. 优化奶牛品种结构通过比较不同奶牛品种的TPI值,可以发现品种间的遗传差异。

“全基因组关联分析”资料合集目录一、全基因组关联分析在作物农艺性状研究中的应用二、玉米12个农艺性状的全基因组关联分析及玉米氮响应相关基因的鉴定三、全基因组关联分析在水稻遗传育种中的应用和研究进展四、支气管哮喘的全基因组关联分析研究进展五、水稻苗期稻瘟病抗性的全基因组关联分析六、全基因组关联分析的进展与反思七、甘蓝型油菜分枝角度和株高全基因组关联分析八、基于SNP芯片和全测序数据的奶牛全基因组关联分析和基因组选择研究九、桃基因组及全基因组关联分析研究进展全基因组关联分析在作物农艺性状研究中的应用一、引言在过去的十年中,随着基因测序技术的飞速发展,全基因组关联分析(Genome-wide Association Study,GWAS)已成为研究作物农艺性状的重要工具。
作物农艺性状是指作物在生长发育过程中表现出的形态、生理和产量等特征,这些性状通常受到多个基因的控制,并且会受到环境因素的影响。
通过GWAS,我们可以识别与特定农艺性状相关的基因变异,进一步理解作物生长发育的规律,并为作物育种提供重要的指导。
二、全基因组关联分析的原理和方法GWAS的基本原理是利用单核苷酸多态性(SNP)作为分子标记,通过比较不同品种或群体中SNP位点的差异,来寻找与特定农艺性状相关的基因变异。
在作物研究中,常用的方法包括基因组重测序和基因组扫描。
基因组重测序是对作物种质资源进行全基因组测序,以获取高精度的基因型信息。
基因组扫描则是利用已发表的SNP数据和农艺性状数据,进行大规模的关联分析。
三、全基因组关联分析在作物农艺性状研究中的应用1、作物产量:通过GWAS,研究者已经识别了许多与作物产量相关的基因变异。
例如,在玉米中,与产量相关的基因变异被发现与植物生长和发育的多个阶段有关,包括叶片大小、节间长度和花粉传播等。
这些发现为提高作物产量提供了重要的理论依据。
2、作物抗病性:GWAS也被广泛应用于研究作物的抗病性。
例如,在小麦中,研究者发现了一种与对白粉病抗性相关的基因变异。

奶牛繁殖性能的遗传分析与改良研究随着人类对食物需求的增加和肉类产品的消费量不断攀升,对于畜牧业的发展也提出了更高的要求。
在现代畜牧养殖业中,奶牛可谓是最重要的生产动物之一。
然而,奶牛在繁殖方面存在着一些问题,如产后发情、优生、难产等问题,给畜牧养殖业的发展带来了很大的困扰。
因此,探索奶牛繁殖性能的遗传分析和改良研究非常必要。
一、奶牛繁殖性能的遗传分析奶牛繁殖性能是指奶牛在繁殖生殖方面所表现出的性能。
这一属性具有较强的遗传性,因此对其遗传机理进行分析和研究是非常必要的。
奶牛繁殖性能的遗传分析主要包括四个方面:1. 血统分析血统分析是奶牛繁殖性能遗传分析的基础,通过此项技术可以追踪分析牛只的血统关系,并为后续的研究提供参考。
2. 遗传指标分析遗传指标分析是通过检测奶牛在繁殖方面表现出的性能并对其进行测定,以判断牛只在此方面的遗传特点以及传递方式。
这项技术的主要难点在于如何对繁殖性能进行定量评估和准确测量。
3. 遗传连锁图谱分析遗传连锁图谱分析主要是用于研究奶牛繁殖性能的细微遗传特征和染色体上的遗传基因,为相关的遗传搭配和改良提供科学数据。
4. 基因组测序技术基因组测序技术是当前比较先进的遗传分析技术之一,它可以全面解析奶牛的基因组序列,确定其遗传基因和功能参数,为奶牛遗传性能改良提供重要的参考。
二、奶牛繁殖性能的改良研究除了遗传分析,改良奶牛的繁殖性能也是重要的一部分。
目前,主要采用的改良研究方法有以下几种:1. 选育优良品种优良品种是繁殖改良的重要手段之一。
当前,国内外研究机构已经培育出了许多性能优良的奶牛品种,如荷斯坦奶牛、杰尔西奶牛、布朗奶牛等。
在选育过程中,要注意保持品种纯正,避免品系交叉。
2. 遗传资源保护保护遗传资源是保障奶牛繁殖性能稳定的重要步骤。
要加强对优良品种的保护和管理,进行优生优育规划,避免过度开发过度利用导致遗传资源的流失。
3. 遗传改良技术遗传改良技术是目前世界范围内应用广泛的奶牛繁殖性能改良技术。

基因组学技术在动物育种中的应用与进展随着科技的迅速发展,人们的生活水平也得到了极大的提高。
在农业领域,一系列技术的推广和应用,为动物育种带来了革命性的变化。
而其中,基因组学技术的发展和应用,更是让育种领域逐渐变得更加高效、精准和可持续。
在本文中,我们将探讨基因组学技术在动物育种中的应用与进展。
一、基因组学技术的发展在分子生物学的基础上,基因组学技术的迅速发展,为研究动物的基因结构及其功能带来了更广泛更深入的视角。
随着全基因组测序技术和其他高通量测序技术的逐渐发展和普及,我们可以更加深入的研究生物体内部的基因表达调控机制,为动物育种的优化和改良提供有力支持。
二、基因组学技术在动物育种中的应用1. 基因检测和分型技术基因组学技术可以用于检测动物的基因多样性和表型多样性,为育种和遗传改良提供有力支持。
通过基因分型技术,我们可以了解动物基因组的组成结构、基因复制数和多态性等情况,为选择最优的亲本提供科学依据。
2. 基因组选择和淘汰基因组学技术可以有效地辅助育种师进行基因组选择和淘汰。
基于多态性位点的分析方法能够更精确地确定个体基因组的遗传基础,发现存在隐性遗传缺陷的生物,将其淘汰并选择优良个体作为下一代的亲本,可以显著提高后代基因的纯化度,实现育种目标的高效达成。
3. 基因编辑技术基因组学技术还可以通过基因敲除和基因编辑技术,对动物基因进行精准调控和修改,实现育种和遗传改良的目标。
例如,在奶牛育种中,可以利用基因编程技术,删去一些不良的基因,增强奶牛乳品的营养指标。
对于其他动物,这种技术也可以被广泛应用,以对其肉质、毛皮、体型和产量等方面进行改良。
三、基因组学技术在动物育种中的进展随着基因组学技术的不断革新,育种师和科学家们开始不断尝试创新性地将其应用于育种领域,推动育种进程的高效、精准和可持续发展。
在这方面,基因组编辑和人工智能技术的结合,将是未来育种领域的方向之一。
四、基因编辑和人工智能技术结合基因编辑技术相比基因改良技术,能够做到更加精准地选择一个目标基因,并删去或插入一个特定片段或者功能,从而对目标生物的基因组进行修饰,以实现指定的生物学功能的变化。
首页 科技服务 测序指南 基因课堂 市场活动与进展 文章成果 关于我们全基因组选择1. Meuwissen T H, Hayes B J, Goddard M E.Prediction of total genetic value using genome-wide dense marker maps[J]. Genetics, 2001, 157(4): 1819 1829. 阅读原文>>2. Haberland A M, Pimentel E C G, Ytournel F, et al. Interplay between heritability, genetic correlation and economic weighting in a selection index with and without genomic information[J]. Journal of Animal Breeding and Genetics, 2013, 130(6): 456-467. 阅读原文>>3. Wu X, Lund M S, Sun D, et al. Impact of relationships between test and training animals and among training animals on reliability of genomic prediction[J]. Journal of Animal Breeding and Genetics, 2015, 132(5): 366-375. 阅读原文>>4. Goddard M E ,Hayes BJ. Genomic selection [J]. Journal of Animal Breeding and Genetics,2007,124:323:330. 阅读原文>>5. Heffner E L, Sorrells M E, Jannink J L. Genomic selection for crop improvement [J]. Crop Science, 2009, 49(1): 1-12. 阅读原文>>参考文献全基因组选择简介Meuwissen等[1]在2001年首次提出了基因组选择理论(Genomic selection , GS),即利用具有表型和基因型的个体来预测只具有基因型不具有表型值动植物的基因组育种值(GEBV)。
中国荷斯坦牛基因组选择分子育种技术体系的建立与应用项目简介项目名称:中国荷斯坦牛基因组选择分子育种技术体系的建立与应用推荐单位意见:中国农业大学等单位针对我国奶牛育种的落后状况和我国奶业由数量增长型向质量效益型转型的迫切需求,瞄准现代动物基因组学技术的发展趋势及其在奶牛育种中的巨大应用价值,历时19年,对奶牛基因组选择分子育种技术开展了系统全面的研究,在奶牛重要性状功能基因挖掘、基因组参考群体构建、基因组育种值预测方法、奶牛主要遗传缺陷基因诊断等方面取得了创新性的研究成果,建立了完整的中国荷斯坦牛基因组选择分子育种技术体系。
该技术体系2012年被农业部指定为我国荷斯坦牛青年公牛的遗传评估方法,并在全国推广应用,极大提升了我国奶牛育种的技术水平,显著提高了我国自主培育种公牛的能力,大大加快了我国奶牛群体的遗传改良速度,缩短了与发达国家的差距,取得了显著的经济效益和社会效益。
经多方客观评价,该成果的整体技术水平达到国际先进,部分达到国际领先。
该项目获国家授权发明专利15项、软件著作权14项,发表学术论文94篇,其中SCI 收录期刊论文62篇,获2015年北京市科学技术一等奖。
该项成果科技支撑体系完整,创新明显,技术先进成熟,经济效益显著。
推荐该项目为国家科学技术进步奖一等奖。
推荐单位:北京市2015年12月29日项目简介:奶业是国民经济的重要组成部分。
以选育优秀种公牛为核心的奶牛育种,是实现群体遗传改良、提高奶业生产水平和效率的关键。
我国奶业生产水平与发达国家有较大差距,根本原因是我国奶牛群体的遗传水平低,依靠传统的育种技术难以改变这种状况。
以基因组选择为核心的分子育种技术提供了机遇,与传统育种技术相比,该技术可大大提高群体遗传改良速率。
项目组自1996年起,系统开展了中国荷斯坦牛基因组选择分子育种技术研究,取得了一系列重要创新性研究成果,建立了完善的技术体系,并大规模产业化应用。
主要技术创新点为:(1)挖掘了奶牛重要经济性状功能基因。
·2011·20·科技Research on X-sex Control of Frozen Semen for Jersey and Holstein-FriesianWEI Huan 1,LI Ming 2,LI Xiu-liang 2,LIU Rui-xin 2,LUO Meng-huo 2(1.Technical Extension Station of Animal Production in Hechi City,Hechi,quangxi547000;2.Guangxi Institute of Animal Science,Nanning,Guangxi 530001)Abstract :A trial of artificial insemination(AI)has been carried out with frozen semen of X-sex control for the cows of Jersey andHolstein -Friesian,as well as the donators of embryo transfer to evaluate the effect on this AI research work.Results from the synchronous estrus,superoulation,AI and embryo collection in this trial showed that the conception rate and the amount and rate of available embryo from the donators were affected by the frozen semen of X-sex control;higher conception rate was found in heifers other than delivered cows and Holstein -Friesian other than Jersey cows in different breeds.However,the results of amount of available embryo and rate of available embryo from the younger donators were much better than the delivered ones in the same breed,and the better result also occurred in Holstein-Friesian when the comparison was done in the same age of different breed cows.Key words :Jersey ;Holstein-Friesian ;Frozen semen of X-sex control ;Evaluation of results在生产上采用CIDR+PG+LHRH-A 3的方法更经济实惠。
中国水产科学 2011年7月, 18(4: 936−943 Journal of Fishery Sciences of China综述收稿日期: 2011−03−14; 修订日期: 2011−04−10.基金项目: 国家自然基金资助项目(30730071; 30972245; 农业科技成果转化资金项目(2010GB24910700. 作者简介: 于洋(1987−, 硕士研究生. E-mail:***************通信作者: 张晓军, 副研究员.E-mail:*************** DOI: 10.3724/SP.J.1118.2011.00935全基因组选择育种策略及在水产动物育种中的应用前景于洋1,2 , 张晓军1 , 李富花1 , 相建海11. 中国科学院海洋研究所实验海洋生物学重点实验室, 山东青岛266071;2. 中国科学院研究生院, 北京 100049摘要: 全基因组选择的概念自2001年由Meuwissen 等提出后便引起了动物育种工作者的广泛关注。
目前, 澳大利亚、新西兰、荷兰、美国的研究小组已经应用该方法进行了优质种牛的选择育种, 并取得了很好的效果。
此外在鸡和猪的选择育种中也有该方法的应用, 但在水产动物选育中尚未见该方法使用的报道。
本文对“全基因组选择育种”的概念和提出背景进行了归纳, 对全基因组选择育种的优势进行了阐述, 并详细介绍了其具体的策略, 总结了目前全基因组育种所广泛采用的方法以及取得的成果, 旨在为该方法在水产动物育种方面的应用研究提供科学参考。
关键词: 全基因组选择; 水产动物育种; SNP; QTL; 全基因组育种值估计中图分类号: S96 文献标志码: A 文章编号: 1005−8737−(201104−0935−08人类对于动物的选择育种由来已久, 最初所进行的只是简单的人工驯化。
随着遗传学研究的发展, 尤其是“数量遗传学理论”的提出, 动物育种技术进入快速发展时期。
基因组选择技术在奶牛育种规划中的应用
由于奶牛育种规划在当今牛奶生产中的重要作用,基因组选择技术成为改善奶牛品质和产量的重要手段之一。
基因组选择技术是一种强大的遗传技术,可以将几个世纪以来积累在牛群中的有价值的基因组优势传递给后代,从而使牛群的进化更快、更稳定。
目前,基因组选择技术经过不断的发展,已经在改善奶牛表型,增加牛牛产量上受到越来越多的应用。
尤其是基于可生物学利用性的选择标准,可以有效地指导和优化奶牛育种规划,从而帮助改善牛群的竞争力。
首先,基因组选择技术可以通过找出有价值的基因从牛群中筛选出显著优异的物种。
然后,基于基因水平的分子育种技术可以根据牛群中的不同基因水平,将不同的基因类型归类,从而有效地改善牛群的表现和产量。
其次,基因组选择技术可以对奶牛的遗传背景进行分析,分析牛群的表现优势和表型缺陷。
接下来,结合可物理可用性的牛群分子育种规划,可以有效地改进和提升牛群的表现和产量状况。
最后,可以利用这些表现数据,进行准确的牛群育种规划,以获得更高更稳定的牛群性状和产量,最大限度地发挥牛群的潜在价值。
总之,基因组选择技术是一种强大的遗传技术,能够有效控制、调节和优化奶牛育种规划,提升奶牛的表现和产量,为现代牛奶生产和奶牛育种增添一份新的强项。
国内外奶牛基因组选择技术应用效果及策略孙东晓(中国农业大学北京100000)一、如何判断优秀奶牛个体?奶牛的生产性状、功能性状具有复杂的遗传基础。
成母牛可通过个体本身表型值判断是否优秀,公牛、犊牛和青年牛无表型,只能通过系谱利用个体及亲属表型进行判断,可靠性较低;或通过后裔测定进行判断,虽然准确性高,但世代间隔长、成本高。
基因组选择技术(GS),可以不依赖表型信息,从遗传上实现早期选择,从而加快遗传进展,有效降低育种成本,其突出优势是“快、准、高”。
基因组选择的特点:与传统育种方法相比,GS 最突出的优势是:快、准、高。
快:生产性能提升更快,尤其对于世代间隔较长的奶牛。
准:所选群体中的优秀个体选得更准,因为这种技术利用了基因组信息,遗传评估准确性更高,给待选个体的“好与坏”排队排得更准。
高:育种收益高,即育种收益与育种投入比例,因为选得准、进展快等,GS 可更多地降低育种成本,实际育种收益要远高于传统育种收益。
二、基因组选择在奶牛育种中的应用效果自2009年起,世界各国陆续将GS 技术应用在公牛育种中,成效显著;近几年在母牛群体中的应用也逐渐普及。
截止2020年12月,美国已累计对300多万头母牛进行了基因组检测,各国基因组遗传评估基本情况如下图所示:各国基因组遗传评估情况《奶牛》2021年01期54三、基因组选择在母牛群体中的具体应用策略通过基因组选择技术对母牛从遗传上进行早期评定,可为牧场的育种决策提供科学依据,具体应用策略包括以下几个方面:1.犊牛、青年牛早期准确选择:早期选择遗传上优秀的犊牛、青年牛,淘汰差牛。
2.青年牛价值评定。
3.为配种决策提供科学指导:①顶级母牛选作核心群,优秀母牛选作胚胎供体母牛;②中上等母牛使用性控冻精配种,中下等母牛使用常规冻精配种;③低等牛使用肉牛冻精配种。
4.精确计算近交系数,进行精准选种选配。
5.亲子鉴定、遗传缺陷有害基因检测。
来源:动物分子数量遗传与奶牛育种注:引自《McNeel AK,Reiter BC,Weigel D,Osterstock J,Di Croce FA.Validation of genomic predictions for well⁃ness traits in US Holstein cows.J Dairy Sci.2017;100:9115–9124.》案例:美国随机选择11个商业牧场的3400头怀孕母牛(69%经产牛,31%头胎牛),对基因组遗传评估的有效性进行分析。
发表于《中国奶牛》,2011全基因组选择育种技术及在奶牛育种中应用进展范翌鹏1孙东晓1* 张勤1张胜利1张沅1刘林2(1.中国农业大学动物科技学院,北京,100193; 2.北京奶牛中心. 北京. 100085)摘要:全基因组选择是指基于基因组育种值(GEBV)的选择方法,指通过检测覆盖全基因组的分子标记,利用基因组水平的遗传信息对个体进行遗传评估,以期获得更高的育种值估计准确度。
由于可显著缩短世代间隔,全基因组选择作为一种育种新技术在奶牛育种中具有广阔的应用前景,目前已经成为各国的研究热点。
不同国家的试验结果表明,在奶牛育种工作,基于GEBV 的遗传评估可靠性在20-67%之间,如果代替常规后裔测定体系,可节省92%的育种成本。
本文综述了全基因组选择的基本原理及其在各国奶牛育种中的应用现状和所面临的问题。
关键词:全基因组选择,奶牛育种Genome-Wide Selection and its Application in Dairy Cattle FAN YiPeng, SUN Dongxiao, ZHANG Qin, ZHANG Shangli, ZHANG Yuan, LIU Lin (College of Animal Science Technology, China Agricultural University, Beijing, 100193) Abstract: Genomic selection refers to selection decisions based on genomic breeding values (GEBV). The GEBV are calculated as the sum of the effects of dense genetic markers, or haplotypes of these markers, across the entire genome, thereby potentially capturing all the quantitative trait loci (QTL) that contribute to variation in a trait. Genomic selection has become a focus of study in many countries as the new breeding method. Reliabilities of GEBV for young bulls without progeny test results in the reference population were between 20 and 67%. By avoiding progeny testing, bull breeding companies could save up to 92% of their costs [1]. In this paper, we first review the progress of genomic selection, including the principle, methods, accuracy and advantages of genomic selection. We then review the application of genomic selection in dairy cattle.Key words: Genomic Selection, Dairy Breeding全基因组选择(Genomic Selection,GS),即全基因组范围的标记辅助选择(Marker Assisted Selection, MAS),指通过检测覆盖全基因组的分子标记,利用基因组水平的遗传信息对个体进行遗传评估,以期获得更高的育种值估计准确度。
研究已表明,标记辅助选择可提高奶牛育种遗传进展[2][3],但是在目前奶牛育种工作中却无法大规模推广应用标记辅助选择。
因为奶牛的生产性状和健康性状均受大量基因座位共同影响,通过有限数量的已知标记无法大幅度加快遗传进展;其次,通过精细定位策略鉴定主效基因需花费大量人力物力和时间;而且利用标记信息估计育种值的计算方法也很复杂。
全基因组选择基于基因组育种值(Genomic Estimated Breeding Value, GEBV)进行选择,其实施包括两个步骤:首先在参考群体中使用基因型数据和表型数据估计每个染色体片段的效应;然后在候选群体中使用个体基因型数据估计基因组育种值(genomic breeding value,GEBV)[4],模拟研究证明,仅仅通过标记预测育种值的准确性可以达到0.85(指真实育种值与估计育种值之间的相关,而可靠性则指其平方)。
如果在犊牛刚出生时即可达到如此高的准确性,对奶牛育种工作则具有深远意义。
模拟研究表明:对于一头刚出生的公犊牛而言,如果其GEBV的估计准确性可以达到经过后裔测定估计得到的EBV准确性同样高的程度,相当于可以利用2岁公牛代替5岁乃至更老的公牛作为种用,遗传进展率将提高一倍。
与奶牛常规后裔测定体系相比,可节省92%的育种成本[1]。
1.不同国家的全基因组选择实施情况及GEBV估计准确性1.1澳大利亚的研究结果在澳大利亚后裔测定体系中选择出生于1998~2003年的共计798头荷斯坦公牛(Genetics Australia 测定),利用Bovine SNP50芯片对56,947个SNP标记进行了个体基因型测定。
使用两种方法计算GEBV:第一种方法为Meuwissen等提出的BLUP方法[4],假设所有的SNP效应均来自于同一个正态总体,即假设所有的SNP效应很小,且σgi2相同。
第二种方法为BayesA方法,认为影响一个性状的大部分SNP标记具有微小的独立效应,但一小部分SNPs具有相对较大效应(即不同SNP的效应不同)。
计算结果表明,使用GEBV预测TBV的可靠性高于利用系谱指数对公牛进行预测的可靠性,而后者正是目前后裔测定体系中青年公牛育种值预测的主要方法(表1)。
表1 公牛出生时不同育种计划中育种值估计的可靠性性状参考群体中表型记录条数SNP数目EBV可靠性GEBV可靠性(BLUP)GEBV可靠性(BayesA)ASI 637 3,889 0.38 0.44 0.48APR 635 3,414 0.35 0.53 0.55 乳蛋白量637 4,055 0.28 0.45 0.48乳蛋白率637 4,369 0.20 0.29 0.36繁殖性状332 3,090 0.16 0.18 0.14此外,繁殖性能的GEBV可靠性远低于生产性状GEBV。
可能是由于繁殖性能为低遗传力性状,需要更多的表型记录才能预测得到更准确的GEBV。
在澳大利亚的研究中,相对于其他性状而言,只有少部分公牛(332头)具有繁殖性能记录,因此降低了数据分析的统计效力。
除繁殖性能以外,其他性状通过Bayes方法得到的GEBV可靠性比BLUP方法提高了2%~7%。
目前,澳大利亚的全基因组选择工作主要由澳大利亚畜禽合作研究中心执行的,其参考群公牛规模已经达到了2000头,并于2010年正式发布了GEBV。
1.2 新西兰的研究结果新西兰家畜遗传改良公司(LIC)于2008年公布新西兰奶牛GEBV估计的可靠性[5]。
以4,500头左右后裔测定公牛为参考群体,公牛规模及其出生年度范围均远高于澳大利亚群体。
利用Bovine SNP50芯片对所有公牛进行基因分型。
采用BLUP, BayesA, BayesB(考虑了某些SNP效应为0的情况)[4],线性角回归[6]和贝叶斯回归[7]等方法估计GEBV并进行比较。
此外,在GEBV中还加入了系谱指数(加性育种值)信息。
LIC于2009年8月开始正式公布GEBV,每年评估两次。
对无表型数据的青年公牛产量性状、体重、繁殖性能、体细胞数和长寿性进行估计,其GEBV的估计可靠性在50~67%之间;而采用系谱指数的估计育种值可靠性仅为34%。
普遍高于澳大利亚所得的数据结果,其原因可能是由于新西兰采用的参考群体规模远高于澳大利亚。
通过贝叶斯方法得到的GEBV估计可靠性比BLUP方法高2~3%,而回归方法得到的GEBV可靠性较低。
LIC已经于2009年8月开始正式公布GEBV,每年评估两次。
1.3美国的研究结果美国组建的参考群体包括3,576头荷斯坦公牛,共计38,416个有效SNP用于预测方程,SNP检测同样采用Bovine SNP50芯片。
预测方法与Meuwissen等提出的BLUP方法相似,不同之处为假定所有标记效应符合正态分布;另外也采用贝叶斯方法,考虑到有些基因具有较大效应,因此在分布中加入了一个较大的尾(与1.2中的BayesA方法相似)。
与澳大利亚和新西兰研究结果一致的是,也将基于系谱的多基因效应或父母育种值平均值整合到预测方程中,拟合选择指数得到GEBV。
针对所有性状,GEBV的平均估计可靠性达到50%,而仅仅利用系谱指数对青年公牛进行预测的平均可靠性仅为27%。
BLUP方法仅比贝叶斯方法的可靠性低1%,这一结果与澳新两国的结果一致。
目前,美国和加拿大的参考群公牛规模合计已达到18000头以上(美国9300头,加拿大8800头),并分别于2009年1月、8月在官方正式颁布的公牛育种值中包含基因组育种值,称为GPTA(Genomic Predicted Transmitting Ability);1.4 荷兰的研究结果荷兰的基因组选择计划试验由CRV公司组织执行,其参考群体包括了1,583头公牛,有46,529条有效SNP数据用于估计GEBV。
在计算GEBV可靠性时,挑选出生于1999~2003年间的429头公牛,随机抽取其中5%的公牛计算GEBV,然后与后裔测定EBV进行相关分析。
随机抽取过程重复20次,使得每头公牛均有一次机会作为参考公牛。
SNP效应的计算方法参考Meuwissen和Goddard (2004)提出的吉布斯抽样[8],只是用SNP代替了单倍型[9]。
结果显示,对于刚出生的公犊牛,GEBV 的估计可靠性比系谱指数均有所提高,对于不同性状提高的程度不同:乳蛋白率提高33%,乳蛋白量提高19%,肢蹄性状提高13%,乳房深度以及体细胞数提高13%,受精能力提高9%。
欧洲有多个国家开展了Eurogenomics的项目,截止去年,包括丹麦/芬兰/瑞典/挪威,法国,荷兰和德国在内的7个国家,参考群体公牛规模已经达到了16000头,除荷兰每年评估次数为24次外,其余国家评估次数均为12次,这些国家分别于2009年6~8月公布了其各自的官方GEBV。