MicroRNA Inhibitor产品使用手册
- 格式:pdf
- 大小:166.16 KB
- 文档页数:3

快速参考手册TaqMan MicroRNA Assays简要操作说明(Rev.A.0)本操作说明提供了TaqMan MicroRNA Assays的简要操作指南。
更详细信息,请至赛默飞世尔官方网站下载英文版说明书:https:///content/sfs/manuals/cms_042167.pdf一.配制反转录体系1.准备总RNA(1)注意:应选择适当的提取方法,避免提取过程中丢失microRNA(2)RNA定量2.准备反转录预混液(1)将Taqman MicroRNA反转录试剂盒中的组分放在冰上融化后涡旋混匀。
(2)准备一支无RNA酶的离心管,参照下表,在冰上配制反转录预混液。
组成成分反应体系100mM dNTPs(with dTTP)0.15µl反转录酶 1.00µl10X反转录buffer 1.50µlRNase抑制剂0.19µl无核酸酶H2O 4.16µl总体积7.00µl(3)涡旋混匀,离心。
(4)将反转录预混液放置在冰上备用。
3.配制反转录体系(1)在冰上将5x反转录引物和RNA模板融化。
涡旋混匀。
(2)15µl的反转录体系中,加入1-10ng RNA(5µl),7µl反转录预混液,3µl反转录引物,涡旋混匀,离心。
(3)将反应管放在冰上放置5分钟,然后进行反转录实验。
4.设置反转录步骤将反应管放入PCR仪内,按照下列的反应程序进行设置:模式:标准模式,反应体积:15µl阶段时间温度退火30分钟16°C延伸30分钟42°C失活5分钟85°C∞4°C2二.定量PCR 扩增1.准备定量PCR 反应体系(1)将反应用到的组分放在冰上融化,混匀后放置冰上备用。
(2)按照20µl 反应体系计算所需要的试剂量。
将各组分加入离心管中,涡旋混匀,离心。

Step 1: Sample Preparation & Nucleic Acid IsolationFor great results, use (click product names to learn more):Roche High Pure RNA Isolation KitRoche High Pure FFPET RNA Isolation KitRoche High Pure miRNA Isolation KitRoche RealTime ready Cell Lysis KitFrom which source (animal, organ, tissue) does the examined material originally come from? Which volume or mass or cell number was used for nucleic acid preparation?My MIQE Guide*Empowering results that matter Sponsored by Roche Applied Science Experiment title:Performed by:Date:Institution:Experimental design: How did you choose and set up your study (number of treated samplesand controls)Handling: Which tools or methods were used to obtain and process the primary samples (e.g., micro-dissection, macrodissection)?Method of processing and preservation: How was the sample treated and stored?If frozen – how and how quickly?If fixed – with what, and how quickly?If stored for longer: how and how long? (especially for FFPE samples)Extraction method:Which kit or instrument was used to extract/isolate the DNA/RNA from the starting material? Roche High Pure RNA Isolation Kit, High Pure FFPET RNA Isolation Kit, High Pure miRNA Isolation Kit, RealTime ready Cell Lysis Kit, or other (Please specify)Was the vendor’s protocol modified (If Yes, when, and how? e.g. by using additives)Did you do a DNAse or RNAse treatment? (If Yes, when?)Did you check for nucleic acid purity and integrity? If Yes: By using which instrument and method? What was the resulting purity (A260/A280)? What was the resulting yield? If No: Why not?Did you check for the presence of PCR inhibitors? If Yes: By using what (e.g. Cq dilutions, spike or other (please specify)If No: Why not?Final storage solution (e.g., buffer, H2O) for the purified total RNA:Storage time and temperature of the purified total RNA before use in RT-qPCR:Step 2:Reverse TranscriptionFor optimal results, use:Roche Transcriptor First Strand cDNA Synthesis KitRoche Transcriptor Universal cDNA MasterAmount of RNA and reaction volume:Priming oligonucleotide (if using gene specific primers) and concentration: Reaction temperature and time:Manufacturer of reverse transcription reagent(s) and catalogue number(s): Reverse transcriptase type and used concentration:Storage conditions of cDNA:Step 3:PCR Amplification and AnalysisFor best results, use:LightCycler® 480 Probes MasterFastStart Essential DNA Probes MasterFastStart Universal Probe Master (Rox)Target sequence and amplicon information: Target gene database sequence accession number:Location of amplicon:Amplicon length:Result of in silico specificity screen (BLAST, etc.):Information on pseudogenes, retropseudogenes or other homologs: Secondary structure analysis of amplicon:Determined by which method?Location of each primer relative to exons or introns (if applicable): Targeted splice variants:RTPrimerDB Identification Numbers: Manufacturer of oligonucleotides: Purification method:For probe-based assays: Probe type:qPCR reaction conditionsReaction volume and amount of cDNA/DNA per reaction: Primer, (probe), Mg2+ and dNTP concentrations: Polymerase identity:Buffer/kit manufacturer and identity (e.g., catalog number)Manufacturer and catalog number of plates or tubes and catalog number:Complete thermocycling parameters:Reaction setup: Was it manual or robotic? If robotic: Using which robot?Equipment: Which Real-Time PCR instrument was used? (Which Roche LightCycler® System or other (please specify)?)Validation of qPCR runs:Are you running a multiplex assay? If yes, please describe efficiency and limit of detection foreach assay:How did you check for specificity of amplification for each target (e.g., on a gel, by sequencing, melt-ing curve analysis or digest):For SYBR Green I assays: Cq of the non-template control reaction:Standard curve characteristics (slope and y-intercept):How many replicates did you use to establish the standard curve?(xx replicates per standard concentration)What was the lower and the upper limit of the standard curve?PCR efficiency calculated from slope:Confidence interval for PCR efficiency or standard error:r2 of standard curve:Information on linear dynamic range:Cq variation at lower limit: Confidence intervals throughout range:Evidence for limit of detection:How many reactions per run were used for controls? (please specify positive and negative controls, controls without template and No RT controls, e.g. Positive controls: 3 reactions in 5 replicates per 96 well plate)Data analysis:Vendor software: Which software type, version and algorithm provided by the PCR machine supplier was used to analyze the data?Specialist software: Which (if any) additional software was used? Self-developed algorithms,or other (please specify)Normalisation: Which reference gene(s) were used to calculate the relative expression of the studied genes?What was the reason for choosing these particular genes?Which algorithm (e.g., geNorm, bestkeeper, normfinder) was used to normalize for reference gene(s)Which principle was used for Cq calling?What was the number and of biological replicates used?How was their concordance?How many technical replicates were used, and at which step (RT or qPCR)? What was the observed repeatability (intra-assay variation)?What was the observed reproducibility (inter-assay variation, %CV)The MIQE guidelines empower results that truly matter. And so does Roche.Visit to discover all the materials you need for truly remarkable research results.* modified based on the list in the original MIQE guidelines publication with permission of the MIQE authors.For life science research only. Not for use in diagnostic procedures. LIGHTCYCLER and FASTSTART are trademarks of Roche.All other product names and trademarks are the property of their respective owners. NOTICE: This product may be subject to certain use restrictions. Before using this product, please refer to the Online Technical Support page () and search under the product number or the product name, whether this product is subject to a license disclaimer containing use restrictions.Published byRoche Diagnostics GmbH Sandhofer Straße 116 68305 Mannheim Germany© 2013 Roche Diagnostics. All rights reserved.*********** 1012。

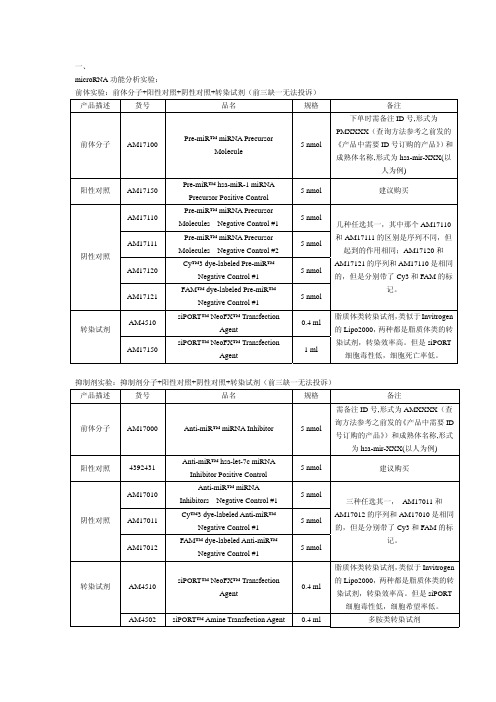
miRNA产品使用说明RN:R10034.5 产品简介micr ON™ miRNA mimic是miRNA模拟物,化学合成的成熟miRNA双链,即用型;micr OFF™ miRNA inhibitor是miRNA抑制物,化学修饰的成熟miRNA互补单链,即用型;micr ON™ miRNA agomir是特殊化学修饰的miRNA 激动剂,适用于细胞实验、动物实验,即用型;micr OFF™ miRNA antagomir是特殊化学修饰的miRNA 拮抗剂,适用于细胞实验、动物实验,即用型。
运输保存产品以冻干粉的形式,常温运输。
收到产品后,请于-20℃~-80℃保存,冻干粉可以稳定保存一年。
使用前瞬时离心,用RNase-free HO或灭菌ddH2O,配制成20μM储存液,分装保存,避免反复冻融(不超过5次)。
2表1 20μM储存液的配置参考0.25 0.5 1 2 5 20溶解体积(μl) 12.5 25 50 100 250 1000 注:如需进行高内涵筛选试验,可选择miRNA Library。
使用前须知为避免外界因素(包括酶,极端pH或者温度条件等)导致产品降解,所有操作请严格遵循RNA操作规则。
实验过程中,产品最好于冰上放置,使用完毕后请于-20℃~-80℃小心保存。
细胞实验方法:为了降低细胞密度、试剂用量,转染效率等因素导致的孔间差异,保证实验的可靠性和可重复性,一般建议:1)转染实验中每个转染样品至少设置3个复孔;2)接种细胞时,每孔接种的细胞数量尽量保持一致,且细胞在各孔的表面平均分布。
1. 转染浓度miRNA产品最佳工作浓度因不同的细胞类型及研究目的而异。
锐博生物推荐的miRNA mimic初始浓度为50nM,miRNA inhibitor浓度为100nM,客户可根据实验具体情况优化转染浓度,优化的范围建议为10~200nM。
注:miRNA inhibitor往往需要用到较大的用量才能观察到较好的抑制效果,相当于miRNA mimic的几倍用量,这可能与miRNA inhibitor竞争性抑制的作用机制及作用效率有关。

Real-time qPCR 检测操作手册1/ 6一、实验概述Real-time Quantitative PCR Detecting System ,即实时荧光定量核酸扩增检测系统,也叫实时定量基因扩增荧光检测系统,简称qPCR 。
是一种在PCR 反应体系中加入荧光基团,利用荧光信号积累实时监测整个PCR 进程,最后通过特定数学原理对未知模板进行定量分析的方法,实现了PCR 从定性到定量的飞跃。
二、实验流程三、实验材料: 1.主要试剂试剂名称 试剂来源 Cat.No. Trizol 上海普飞 3101-100 M-MLV promega M1705 dNTPs promega U1240 oligo dT 上海生工 B0205 Bulge-LoopTM miRNA广州锐博 详见实验报告 qPCR Primer Set Rnase Inhibitor promega N2115 Primer(R&F) 上海生工 详见实验报告 SYBR Master MixtureTAKARADRR041B2/ 6样本收集Trizol 法抽取RNARNA 反转录获取cDNAReal-time qPCR2.主要器材器材名称来源Cat.NoNanodrop 分光光度计Thermo 2000/2000C稳压电泳仪上海天能EPS-600超细匀浆机FLUKO公司F6/10Real time PCR 仪器Agilent公司MX3000p反转录耗材Axygen四、实验步骤:1.总RNA 抽提(1)收取样品,Trizol 裂解。
细胞样品:收集细胞(6 孔板 80%细胞密度),2000 rpm 离心5min,去上清,细胞沉淀中加入1mL Trizol,充分混匀后室温静置 5 min,然后转移至新的 1.5 mL EP 管中;组织样品:将待研磨的组织样品从液氮或者-80℃冰箱中取出,无菌刀片于干冰上将组织样品切割成约 3 mm×3 mm×3 mm 大小,置于装有 1 mL Trizol 裂解液的 1.5mL EP 管中。

血清microrna 检测方法以BIOG 试剂盒为例,进行介绍产品特点 试剂盒组成 组分 50次 100次 吸附柱和收集管各50个 各100个 溶液E 6mL 12mL 溶液Q 42mL 84mL 裂解液 12 mL 24 mL 析出液 4.5 mL 9 mL 洗涤液 9 mL 18 mL 洗脱液 3mL 6 mL 消化液 1.2 mL 2.4 mL RNA Carrier 0.24 mL 0.48 mL 说明书1份1份操作步骤1. 请自行准备:无水乙醇、1.5mL 离心管、2.0mL 离心管。
2. 取出析出液和洗涤液,按以下操作:a) 析出液:4.5mL 加入25.5mL 无水乙醇;9mL 加入51mL 无水乙醇。
b) 洗涤液:9mL 加入21mL 无水乙醇;18mL 加入42mL 无水乙醇。
c) 配制好的析出液及洗涤液如出现沉淀,可在37℃溶解,摇匀后使用。
3. 取2.0mL 离心管1支,依次加入1mL 血清/血浆样本、100μL 溶液E 、700μL 溶液Q ,上下颠倒混匀,室温/4℃静置20分钟。
4. 12,000 rpm 4℃离心5分钟,弃上清,收集离心管内沉淀。
5. 向沉淀中依次加入200μL 裂解液、4μL RNA Carrier 、及20μL 消化液,漩涡震荡至沉淀完全分散,56℃水浴10 分钟。
6. 加入500μL 析出液,轻轻颠倒混匀,如有半透明悬浮物,不影响RNA 的提取与后续实验。
7. 将吸附柱放入收集管内,将上述溶液转入吸附柱内,静置2 min ,12,000 rpm 4℃离心1 min ,弃收集管内废液。
8. 将吸附柱放回收集管内,加500μL 洗涤液至吸附柱内,12,000 rpm 4℃离心1 min ,弃收集管内废液。
9. 将吸附柱放回收集管内,12,000 rpm 4℃离心2 min ,离去残留的洗涤液。
10. 取出吸附柱,放入新的1.5 mL 离心管内,加入30-50 μL 洗脱液,静置3 min ,12,000 rpm 4℃ 离心2 min ,收集RNA 溶液。
miRNA腺病毒操作手册miRNA简介MicroRNAs(miRNAs)是一类长度为18-24个核苷酸的非编码RNA分子。
MiRNA通过与靶基因mRNA上的互补序列结合,降解mRNA或抑制mRNA的翻译。
MiRNAs在细胞分化、增殖、凋亡和癌细胞发生中发挥重要的调控作用。
MiRNAs 来源于具有60-80 个核苷酸茎环结构的microRNA 前体(premir)和序列更长一些的microRNA初级转录产物(primir)。
MiRNA在核内由RNA聚合酶II(polII)转录生成,最初产物primir具有帽子结构和多聚腺苷酸尾巴。
ViGene生物的mirAD-腺病毒microRNA前体表达系统包括三个相关产品:microRNA前体穿梭载体、microRNA前体腺病毒载体和预制microRNA前体腺病毒。
microRNA前体腺病毒简介重组腺病毒是进行基因转移和表达的工具,功能强大且易于操作。
腺病毒独特的生物学特征使它成为“载体的首选”,被科研工作者广泛应用。
首先,它能够感染包括分裂、非分裂细胞和干细胞在内的多种细胞。
第二,病毒滴度高。
第三,高滴度的病毒可获得高感染效率和高表达量。
第四,病毒进入细胞后,病毒基因组不整合到细胞染色体上,因此瞬时表达外源基因的重组腺病毒不会诱导宿主细胞中染色体的变化。
ViGene生物选用的是应用最广泛的复制缺陷型的人类血清5型腺病毒,该腺病毒载体缺失E1和E3基因。
E1 基因在组装感染性病毒颗粒时必不可少,可在HEK293T细胞病毒包装过程中得到补充,而E3基因可有可无。
由于E1和E3基因的缺失,腺病毒载体可插入高达7.5kb的外源基因。
pMir-microRNA precursor 是基于microRNA 前体表达系统的质粒。
microRNA前体天然的茎环结构被克隆到质粒的SgfI和MluI双酶切位点。
为了保持推测的发卡结构和诱导正确的内源性反应,miRNA茎环结构的两侧具有它的150-200bp的天然序列。
非编码RNA相关产品介绍miRNA相关产品MicroRNA (miRNA)是在真核生物中发现的一类内源性的具有调控功能的非编码RNA,其大小长约20~25个核苷酸, miRNA进化保守数量种类丰富,而且不被翻译成蛋白质,但具有调控基因表达的重要功能。
miRNA能特异地结合到信使RNA(mRNA)内的互补区域以促进其降解或抑制翻译,miRNA广泛参与细胞增殖、分化、凋亡及细胞周期调控等过程,在基因实验中起着关键作用。
miRNA的过表达和封闭miRNA的过表达可以使用miRNA的模拟物,或者将pri-mir构建至表达载体上。
构建miRNA的下调即封闭,主要的技术是sponges技术或者miRNA inhibitor (miRNA互补单链的模拟物)来实现。
sponges技术是通过将若干段连续miRNA成熟序列互补序列串联,使之在细胞内表达,吸附目标microRNA,达到下调miRNA的目的。
miRNA靶基因双荧光素酶检测服务荧光素(Luciferase)是自然界中能够产生生物发光的酶的统称,其中最有代表性的是来自萤火虫体内(Firefly)和海肾(Renilla)体内的两类萤光素酶。
在荧光素酶的催化下,荧光素底物可以高效的转变成氧萤光素,同时发出强烈的光。
鉴于此,研究者利用荧光素酶开发出一套极其灵敏且使用方便的基因报告系统。
目前这一系统被广泛用于miRNA靶基因的验证、转录因子结合位点与启动子活性分析、RNA降解、信号转导、药物筛选、动物活体成像等领域。
技术特点Hanbio能为您提供强大的学术支持团队协助设计科学合理的实验方案、荧光素酶检测报告实验相关的载体构建过程及测序的原始报告、原始数据和数据的整理过程,完整的microRNA靶基因双荧光素酶检测报告以及报告质粒。
LncRNA相关产品长链非编码RNA(Long non-coding RNA, lncRNA)是一类长度超过200nt的长链非编码RNA分子,是RNA聚合酶II转录的副产物,它可在多种层面上(表观遗传调控、转录调控以及转录后调控等)调控基因的表达,是遗传学的研究热点。
MicroRNA Inhibitor产品使用手册
规格:5nmol*2(对照为2.5nmol*4)
纯化方式:HPLC 纯化
产品形式:干粉
贮存条件:在-20℃或者-80℃
保存期:1 年,在-20℃或者-80℃
质量控制:PAGE 检测确定产品是准确分子量大小的单链的MicroRNA Inhibitor HPLC 纯化并分析单链的MicroRNA Inhibitor 纯度>95%
●注意事项
RNA inhibitors 在操作过程,如果有外源的核酸酶存在,RNA inhibitors容易发生降解。
在进行相关试验中,请带手套进行操作,尽量用无RNAase污染的试剂,试管,移液枪和枪头。
收到产品后尽快贮存在–20°C 或–80°C环境中。
●MicroRNA Inhibitor的重悬
在最大转速为4,000 X g 的低速条件下离心EP 管,让MicroRNA Inhibitor 聚集在试管的底部。
1.轻轻的打开管盖.
2. 5nmol加入DEPC水250 ul,配成20uM的储备液
3.柔和的用移液枪吹打储备液5 次
4.根据具体用量情况分装,避免多次冻融
5.在重新贮存的时候注意密封好EP 管
6.贮存在-80°C,以备使用
●转染程序
转染效率对不同的细胞株和不同的转染试剂是不同的。
我们建议miRNA inhibitor 的终浓度为15-100nM最优化的转染浓度最终还是需要通过试验来确定。
我们发现典型的试验中最佳的浓度范围是15-100nM,但是优化的浓度范围我们设定在1-100nM 之间。
A:转染试剂的推荐量,根据您订购的试剂不同应做适当的调整
B:所显示的添加量是miRNA inhibitor 终浓度为30nM 的量。
由于最大miRNA inhibitor 活性的量在不同的细胞类型是有差异的,所以推荐您自行优化。
C 对细胞密度只是推荐值,不同的细胞株有一定的变化,主要看细胞的大小和生长的状况,
一般来说我们推荐细胞融合度在30-70%为佳。
转染优化
优化转染效率是使得miRNA inhibitor 活性最大化的的最关键的一个因素之一,对每种转染试剂而言,首先要确定一款最合适的转染试剂,主要看从以下几点着手处理:
转染试剂的量
MicroRNA inhibitor 的量
转染时的细胞密度
转染时候的操作顺序
细胞与转染试剂/siRNA 复合物的接触时间。