转录组数据分析解读及实例操作
- 格式:pdf
- 大小:4.30 MB
- 文档页数:48

单细胞转录组数据实验设计与分析方法总结概述:单细胞转录组数据实验设计与分析是一种高通量技术,可以深入研究单个细胞的转录水平,揭示细胞间的异质性。
本文将就单细胞转录组数据实验设计与分析的方法进行总结和说明。
一、单细胞转录组数据实验设计:单细胞转录组数据实验设计的关键是确保高质量的单细胞RNA测序数据,并能够反映细胞群体的真实情况。
以下是一些常用的实验设计方法:1. 单细胞分离与捕获:单细胞分离是单细胞转录组分析中的第一步,关乎到实验的准确性和可靠性。
常见的单细胞分离方法有流式细胞仪、微操作、雾化等。
在选择单细胞捕获方法时,要考虑到细胞的完整性、RNA的保存情况以及细胞数量等因素。
2. 补体DNA合成(cDNA)与等位酶链式反应(LAMP):将单细胞提取的RNA反转录成cDNA是单细胞转录组实验的关键步骤。
LAMP是一种常用的cDNA合成方法,可以通过等温放大反应来避免RNA的选择性放大,减少偏差的引入。
3. 序列库构建:在构建转录组测序库的过程中,需要将cDNA样品连接到测序适配体上,然后进行PCR扩增等处理。
这一步骤中的实验设计包括确定PCR循环数、适配体的选择、文库的质检等。
二、单细胞转录组数据分析方法:单细胞转录组数据分析是从测序得到的海量数据中提取有用信息的过程。
以下是一些常用的数据分析方法:1. 数据质量控制与预处理:在进行数据分析之前,需要对原始数据进行质量控制与预处理。
这包括去除低质量序列、去除污染序列以及进行质量评估等步骤。
常用的质量控制工具包括FastQC、Trimmomatic等。
2. 数据降维与聚类分析:数据降维是将高维的单细胞转录组数据映射到低维空间的过程。
常用的降维方法有主成分分析(PCA)、流行学习(UMAP)和t分布随机近邻嵌入(t-SNE)等。
降维后的数据可以用于聚类分析,将细胞按照相似性进行分组。
3. 差异表达基因分析:差异表达基因分析是指对不同类型的单细胞进行比较,找出差异表达的基因。

转录组测序数据分析及其应用基因组学研究一直是生命科学领域的重要分支,而随着高通量测序技术的发展,转录组学研究也变得越来越重要。
转录组测序是一种高通量的测序技术,可用于分析RNA的产生和使用。
它可以用于解析基因表达调控机制、鉴定新的转录产物、发现新的基因及其功能以及研究基因表达的变化。
而分析和处理转录组测序数据则是实现这些研究目标的关键步骤。
本文将介绍转录组测序数据分析的流程以及其在生命科学研究中的应用。
1. 转录组测序数据分析的流程转录组测序数据分析的流程包括测序数据质量控制、选用合适的参考基因组进行比对、基因表达量计算、差异表达基因筛选、功能注释及通路分析等步骤。
(1)数据质量控制在数据分析之前,需要对原始测序数据进行质量控制。
质量控制通常包括检查样品的测序深度和比对率,检测是否存在序列重复和序列污染,并通过统计和图形化分析来评估测序数据的准确性和一致性。
一些常用的工具如FastQC和Trimmomatic可以用于数据质量控制。
(2)比对与注释转录组测序数据的比对和注释是数据分析过程中的关键步骤,它可以帮助我们理解基因组中那些区域正在表达这些转录物,并且可以使下游分析过程更加准确和可靠。
常用的比对软件有TopHat和STAR等,同时,基于火山图和MA-plot等绘图技术,对比对结果进行筛选与统计分析,即可确定差异表达的基因。
(3)差异表达基因筛选通过比对和注释分析后,我们可以通过基因表达量的计算来确定哪些基因在不同的实验条件下差异表达。
常用的对基因表达量计算的方法有FPKM和TPM等,同时也适用于多样品比较的统计方法如edgeR和DESeq2等,以筛选差异表达的基因。
(4)功能注释及通路分析结合差异表达基因的结果进行进一步的功能注释和通路分析,通过各种生物信息学工具对其进行KEGG、GO、Cytoscape等分析,以便确定关键的基因、分子和通路在生物学过程中的作用。
2. 转录组测序数据的应用转录组测序数据被广泛应用于生命科学领域中的多种研究,如基因组结构与表达、药物研发、癌症研究、农业作物育种、蛋白质组学、环境科学等等。

有参考基因组的转录组生物信息分析模板转录组是指一些特定生物体在特定时期和特定环境下,在其中一种特定的组织或细胞中所表达的所有基因的mRNA的总和。
转录组测序技术的发展使得我们能够全面了解基因的表达水平和差异,并帮助我们深入探索特定生物体的功能和特性。
本文将为您提供一个转录组生物信息分析的模板,以帮助研究者进行转录组数据分析。
一、质检与预处理1. 检查转录组测序数据的质量,使用FastQC等工具查看测序质量报告。
2. 根据报告,去除测序中存在的接头污染、低质量碱基,以及过短或过长的reads。
3. 使用Trimmomatic等工具进行reads修剪和过滤,保留高质量的reads。
二、比对到参考基因组2. 使用比对软件如Bowtie2、STAR等将reads比对到参考基因组上。
3. 根据比对结果生成BAM/SAM文件,并使用Samtools等工具对文件进行排序和索引。
三、基因表达量估计1. 使用HTSeq、featureCounts等软件对比对结果进行基因表达量估计,生成基因计数矩阵。
2. 将基因计数矩阵导入R或Python环境,进行表达量分析和统计。
3. 使用DESeq2、edgeR等软件对不同样本之间的差异表达基因进行筛选和统计。
四、差异表达基因分析1. 使用DESeq2、edgeR等软件进行差异表达基因分析,确定在不同条件下表达显著变化的基因。
2.使用热图、散点图、MA图等工具可视化差异表达基因的分布和表达模式。
五、注释分析1. 使用生物信息学工具如DAVID、enrichR等进行功能富集和通路分析,找出差异表达基因所涉及的生物学过程和通路。
2. 利用基因本体论(Gene Ontology)和KEGG数据库等进行差异表达基因的功能注释。
六、蛋白质互作网络分析1.将差异表达基因输入蛋白质互作数据库如STRING等,构建差异表达基因的蛋白质互作网络。
2. 使用Cytoscape等工具进行蛋白质互作网络的可视化和分析。

生物信息学中的转录组测序数据分析与应用转录组测序数据是生物信息学领域中的重要数据资源,它能够揭示生物体在特定条件下所表达的基因信息。
通过对转录组测序数据的分析和应用,我们能够深入了解基因的表达模式、功能和调控机制,从而进一步研究生物体在不同生理、病理条件下的变化。
一、转录组测序数据分析1. 数据质控在进行转录组测序数据分析前,首先需要对数据进行质控。
这一步骤主要包括测序质量评估、去除接头序列和低质量读段、去除PCR扩增重复序列等。
通过对数据进行质控,可以减少后续分析中的错误和偏差,确保得到可靠的结果。
2. 数据预处理数据预处理包括基因组比对和转录本定量。
基因组比对将测序数据与参考基因组进行比对,确定基因的位置信息。
转录本定量则是通过统计测序reads在基因上的分布情况,计算基因的表达水平。
常用的工具包括Bowtie、HISAT2、TopHat2等。
3. 差异表达基因分析差异表达基因分析是通过对不同样本之间的转录组数据进行比较,筛选出在各组样本中表达差异显著的基因。
可以采用一般线性模型(generalized linear model,GLM)或非参数方法(如Wilcoxon秩和检验)进行差异分析。
差异表达基因分析还可以进行聚类分析、GO富集分析、通路分析等,进一步了解差异基因的功能和调控机制。
4. 合成基因和调控区域分析通过转录组测序数据,可以预测合成基因和调控区域。
合成基因是由多个基因重组而成的新的基因,在生物体的进化和发育过程中发挥重要作用。
调控区域则是基因的上游区域,通常包含启动子、增强子等,对基因的转录水平和调控具有重要影响。
合成基因和调控区域的分析可以通过利用转录组测序数据进行RNA-Seq、ChIP-Seq等实验方法实现。
二、转录组测序数据应用1. 发现新的转录本转录组测序数据可以揭示未知的转录本,即那些在已知基因组注释中没有被发现的转录本。
通过对转录组测序数据的分析,可以通过构建转录本的转录本组装(transcriptome assembly)来鉴定并发现新的转录本。

使用生物大数据技术进行转录组分析的实用指南转录组分析是研究细胞内转录过程的重要手段,它可以帮助我们全面了解基因表达的调控机制以及某一生物条件下的基因表达模式。
随着生物大数据技术的快速发展,转录组分析已经成为了生物学研究中的一个关键领域。
本文旨在提供一份使用生物大数据技术进行转录组分析的实用指南,帮助读者快速掌握这一技术。
1. 数据获取转录组分析的第一步是获取所需的转录组数据。
目前,公共数据库如NCBI Gene Expression Omnibus (GEO)、European Nucleotide Archive (ENA) 和 Sequence Read Archive (SRA)等提供了大量的转录组数据。
可以通过这些数据库获取到不同物种、不同组织和不同条件下的转录组数据。
2. 数据预处理获得转录组数据后,需要进行一系列的预处理步骤。
这些步骤通常包括去除低质量序列、去除接头序列、去除污染序列、质量修剪、序列比对等。
常用的预处理工具有Trimmomatic、FastQC和Bowtie等。
3. 数据分析在数据预处理完成后,接下来进行转录组数据的分析。
转录组数据分析的目标是确定差异表达基因、寻找调控因子以及功能注释等。
下面是常见的转录组数据分析方法:3.1 差异表达基因分析差异表达分析是转录组分析中的关键环节,它可以帮助识别在不同条件下表达水平差异明显的基因。
常用的方法有DESeq2、edgeR和limma等。
这些方法可以通过统计学模型来确定差异表达基因,并生成差异表达基因列表。
3.2 富集分析富集分析是转录组数据分析的一个重要环节,它可以帮助确定差异表达基因的富集功能及通路。
常用的富集分析工具包括GOseq、KEGG和GSEA等。
这些工具可以根据差异表达基因列表,查询数据库中所包含的功能注释信息并进行富集分析。
3.3 基因网络分析基因网络分析可以帮助研究人员从整体上了解基因间的相互作用关系。
常用的基因网络分析工具有STRING、Cytoscape和GeneMANIA等。

转录组数据分析引言:转录组数据分析是研究生物体在特定条件下细胞内mRNA的表达情况的一种方法。
转录组数据分析的目的是识别差异表达的基因,揭示其在生物学过程中的功能和调控机制。
随着高通量测序技术的发展,转录组数据的获取变得更加容易和可行,因此转录组数据分析也成为生命科学研究中的重要工具之一。
本文将介绍转录组数据分析的基本流程和常用的分析方法。
一、转录组数据的获取转录组数据的获取通常使用高通量测序技术,例如RNA-Seq和microarray。
RNA-Seq是一种基于测序的转录组分析技术,可以直接测定细胞或组织中的mRNA的序列,无需参考基因组序列,因此能够检测到新的转录本和未注释的基因。
Microarray是一种基于杂交的转录组分析技术,通过固定的探针阵列检测RNA样本中的mRNA的水平。
两种技术各有优劣,研究人员可以根据自己的研究目的选择适合的方法。
二、转录组数据分析的基本流程转录组数据分析的基本流程包括数据预处理、差异表达分析和功能注释。
数据预处理是对原始转录组数据进行筛选、过滤和归一化处理,以得到可靠的表达量信息。
差异表达分析通过统计学方法找出在不同条件下差异表达的基因。
功能注释则针对差异表达基因进行功能分析,识别其参与的生物学过程和通路。
三、数据预处理数据预处理是转录组数据分析的第一步,其目的是筛选和过滤掉噪声数据,并对数据进行归一化处理。
常见的数据预处理步骤包括:读取原始数据、去除低质量读段、去除接头序列和低质量碱基、去除rRNA序列、对碱基进行质量修剪、进行序列比对和表达量计算、基因表达量归一化等。
数据预处理的目的是为后续的差异表达分析做好准备。
四、差异表达分析差异表达分析是转录组数据分析的核心步骤,它通过统计学方法找出在不同条件下差异表达的基因。
常见的差异表达分析方法有DESeq2、edgeR和limma等。
这些方法利用统计学模型和假设检验来计算基因表达的显著性差异,并生成差异表达基因列表。

生物信息学中的转录组测序数据分析流程解析转录组测序是一种基于高通量测序技术的生物学研究方法,用于研究特定物种在特定生理或环境条件下所产生的所有转录本(mRNA)。
转录组测序数据分析是将原始的测序数据转化为有意义的生物学信息的过程。
本文将解析转录组测序数据分析的基本流程。
1. 数据质量控制(Quality Control,QC)数据质量控制是在转录组测序数据分析中非常重要的一步,它能够及早发现并剔除测序过程中产生的低质量测序数据,保证后续分析的准确性。
常用的QC工具包括FastQC和Trimmomatic。
FastQC用于检查测序数据的质量分布情况,发现可能存在的测序错误和污染问题。
Trimmomatic则用于去除低质量的测序片段和接头,提高数据的质量。
2. 数据比对数据比对是将测序数据与参考基因组进行比对的过程。
比对的目的是将测序片段精确地定位到基因组上,并获得每个基因组区域的覆盖度和深度等信息。
常用的比对工具包括Bowtie2和TopHat。
Bowtie2是一种基于Burrows-Wheeler Transform的短序列比对工具,适用于低错配率的比对。
TopHat则是一种用于对转录组数据进行比对和注释的工具,可以检测新基因和外显子剪接事件。
3. 定量分析定量分析是研究不同转录本在不同条件下的表达水平差异的过程。
常用的定量工具包括Cufflinks和HTSeq。
Cufflinks是一种用于估计转录本表达水平和发现新的转录本的工具。
它可以根据RNA-Seq数据拼接转录本,并计算不同基因或转录本的表达水平。
HTSeq则是一种用于计算不同基因的读数的工具,读数可以用来估计基因的表达水平。
4. 差异分析差异分析是研究在不同处理条件下,基因或转录本的表达水平是否存在显著差异的过程。
常用的差异分析工具包括DESeq2和edgeR。
DESeq2是一种基于负二项分布模型的差异表达分析工具,它可以对转录本进行差异分析,并计算基因的表达水平在不同条件下的折叠变化。

生物信息学中的转录组数据分析教程转录组数据分析是生物信息学中的重要领域,它研究基因组水平上的基因在不同组织、不同条件下的表达差异。
本文将为您介绍如何进行转录组数据分析的一般步骤和常见方法。
一、数据获取与预处理转录组数据通常以FASTQ格式存储,其中包含了测序机器输出的原始测序数据。
在进行数据分析之前,首先需要获取适当的转录组数据和对其进行预处理。
一般步骤包括质量控制、去除低质量序列、去除接头序列、过滤低质量碱基等。
在质量控制阶段,我们可以使用一些工具如FastQC来检查数据的质量,确保后续分析的准确性。
二、比对与基因表达量计算在转录组数据分析中,比对是寻找转录组数据对应于基因组的位置信息。
比对过程一般分为两个步骤:第一步是将转录组数据映射到参考基因组上,这可以使用一些工具如Bowtie、STAR和HISAT等进行;第二步是通过统计转录组数据在每一个基因的表达量,这可以使用工具如HTSeq和featureCounts等进行。
三、数据标准化与差异表达分析转录组数据的表达量通常具有较大的差异性,为了在样本间进行比较,需要对数据进行标准化。
在标准化过程中,我们可以采用一些方法如RPKM、TPM和FPKM等,将转录组数据的表达量进行归一化。
差异表达分析是比较不同组间的基因表达差异,通常使用一些统计学方法如DESeq2、edgeR和limma等。
四、功能注释和富集分析功能注释与富集分析是转录组数据分析的重要环节。
功能注释通过对差异表达基因进行生物学功能和通路的注释,可以帮助我们了解差异表达基因的潜在功能和作用机制。
富集分析则是用来确定基因表达差异是否与特定的生物过程或通路相关联。
常用的功能注释和富集分析工具有DAVID、GOseq和KEGG等。
五、绘图与可视化分析可视化分析是转录组数据分析中的重要环节,它通过图表和图像等形式展示数据结果,帮助我们更好地理解和解释数据。
在转录组数据分析中,可以使用一些工具如R包的ggplot2和pheatmap等进行数据可视化。
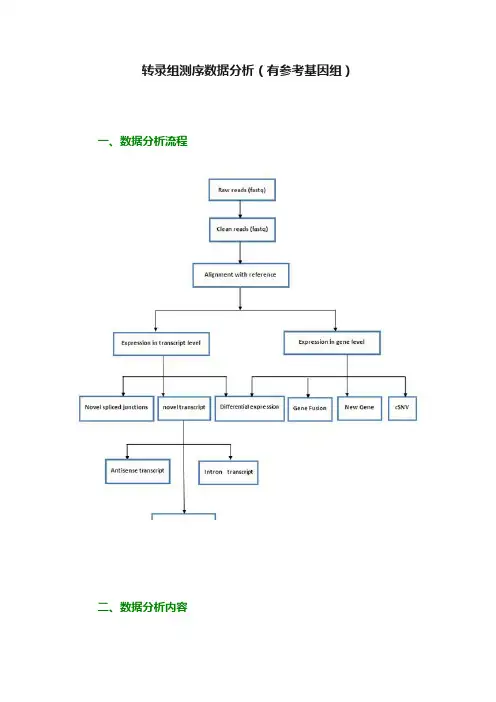
转录组测序数据分析(有参考基因组)一、数据分析流程二、数据分析内容1. 数据预处理目的:对原始测序数据进行一定程度的过滤。
原理:根据测序接头以及测序质量对原始的测序数据进行预处理,其中,测序质量Q与测序错误E之间的关系如下:结果:对预处理后质量以及碱基分布统计进行统计2. 比对基因组目的:将经过预处理的测序数据与参考基因组进行相似性比对。
原理:Burrower-Wheeler转换算法与splicing比对算法。
1)Burrower-Wheeler转换算法:由于测序数据量非常大,与整条基因组比对所需资源与时间是较为巨大的。
目前,我们采用Burrower-Wheeler(BWT)算法对基因进行建立索引、碱基压缩等过程,这样可以很大程度上加快比对速度,减少比对过程中所需资源。
2)splicing比对算法:即分段比对算法,当某条测序序列位于转录本剪切位点时,也就是这条序列同时属于两个外显子,如果将它与参考基因组进行比对,由于基因组两个外显子之间含有intron区,那么它将无法找到它合适的位置;但是应用分段比对算法就可以将这条测序序列分割变成多段子序列,然后应用这些段子序列与基因组进行比对,这样就可以找到它们真正的位置。
Vps28基因的一个分段比对的结果,蓝线连接的两端即为被分割的子序列,可见此种算法非常的适用于转录组测序。
结果展示:应用比对结果进行一些相关mapping统计,测序饱和度及测序5’,3’ bias统计。
Multi mapping,Unique mapping及Unique gene-body mapping统计。
饱和度分析,当reads达到一定测序量后,基因覆盖率基本达到饱和。
测序3’,5’偏好性统计,测序主要集中于基因bady区,两端偏向性较轻。
3. 基因表达水平研究目的:应用基因组比对结果进行基因定量。
原理:从指定物种基因模型(基因结构)中得到gene、exon、intron以及UTR等位置信息,通过基因组比对结果计算出在不用区域富集片段数目,然后应用RPKM/FPKM标准化公式对富集片段的数量进行归一化。

使用生物大数据技术分析转录组数据的步骤解析转录组学是研究特定生物体内转录(基因表达)的技术和方法。
随着生物技术的发展,转录组数据的获取变得越来越容易。
生物大数据技术则为我们提供了分析这些转录组数据的工具和方法。
本文将介绍使用生物大数据技术分析转录组数据的步骤,并解析每个步骤的内容和目的。
第一步:序列清理和质量控制在转录组测序之后,首先需要对原始序列数据进行清理和质量控制。
清理过程主要包括去除低质量的碱基、去除过度复制的序列以及去除连接序列等。
质量控制则用于评估数据质量,检测测序错误和污染。
第二步:序列比对和基因定位清理后的序列将被比对到参考基因组上,以确定每个序列的来源和位置。
序列比对的目的是将测序reads精确地映射到参考基因组上,并计算每个读数在基因组上的覆盖度和多样性。
第三步:表达量估计和差异分析通过将每个读数映射到基因上,可以估计每个基因的表达量。
表达量估计是通过算法分析每个基因的覆盖度和多样性来衡量基因表达水平。
在此基础上,可以进行差异分析,即比较不同条件下基因的表达量差异,以发现关键调控基因或差异表达基因。
第四步:功能注释和通路分析差异表达基因的功能注释是指对这些基因进行生物学功能和通路的解读。
通过与公共数据库中的基因注释信息进行比对,可以了解差异表达基因所涉及的生物学过程和功能。
通路分析则通过将差异表达基因映射到特定的代谢途径或信号传导通路上,来揭示基因在生物过程中的作用和相互关系。
第五步:功能验证和实验验证对于发现的差异表达基因,需要进行功能验证和实验验证。
功能验证包括体外和体内实验,通常使用基因敲除、过表达或RNA干扰等技术来验证基因对特定生物学现象的影响。
实验验证则可以通过PCR、Western blotting等实验方法来验证分析结果的可靠性。
第六步:结果解读和报告编写最后一步是结果解读和报告编写。
在完成转录组数据分析后,需要将结果解读并编写成报告,以便于研究者和其他学术群体对研究成果进行理解和应用。

转录组数据分析解读及实例操作1.数据预处理:对原始转录组数据进行质控和过滤,如去除低质量的序列或测序错误,去除接头序列等。
3.表达量估计:通过计算与每个基因相关的转录本数量,估计每个基因的表达水平。
4.差异表达分析:比较不同条件下的基因表达水平,寻找差异表达的基因。
5.功能注释:对差异表达的基因进行功能注释,探究其在生物过程中的作用和相关的信号通路。
6.富集分析:通过富集分析,找出差异表达的基因是否在特定的功能类别或通路中过度表示。
7.数据可视化:将分析结果可视化展示,以便更好地理解和解读转录组数据。
以下是一个转录组数据分析的实例操作:1. 数据准备:从高通量测序平台获取转录组数据(如RNA-seq数据)。
2.数据预处理:使用适当的软件对原始数据进行质量控制和过滤,去除低质量序列和接头序列等。
3. 数据对齐:将预处理后的转录组数据与参考基因组或转录组进行比对,可以使用Bowtie、STAR等工具进行比对。
4. 表达量估计:使用软件(如HTSeq、featureCounts等)统计每个基因的转录本数量,得到基因的表达矩阵。
5. 差异表达分析:使用DESeq2、edgeR等工具,比较不同条件下的基因表达水平差异,并筛选出显著差异表达的基因。
6.功能注释:通过使用数据库和工具(如DAVID、GO、KEGG等),对差异表达基因进行功能注释,分析其在生物过程中的作用和相关的信号通路。
7. 富集分析:使用富集分析工具(如GSEA、clusterProfiler等),将差异表达基因与已知的功能类别或通路进行比较,寻找显著富集的类别或通路。
8. 数据可视化:使用R语言中的ggplot2、heatmap等包,将转录组数据的分析结果可视化展示,以便更好地理解和解读分析结果。
在实际应用中,转录组数据分析可以应用于许多领域,如生物医学研究、生物进化、植物育种等。
通过分析转录组数据,可以揭示生物体的分子机制和疾病发生发展的模式,有助于发现新的治疗靶点和药物开发。
生物信息学的转录组数据分析一、引言转录组是一个生物组织或细胞中所有转录的RNA分子的总和,它反映了基因在特定条件下的表达水平。
转录组数据分析是生物信息学中的一个重要领域,它通过对转录组数据的处理和解读,可以揭示基因的功能和调控机制,以及在疾病发生发展中的作用。
本文将介绍转录组数据分析的基本步骤和方法。
二、数据预处理转录组数据通常以测序的形式存在,因此首先需要进行数据质控和预处理。
数据质控主要包括去除接头序列、低质量序列过滤、去除待测序列污染等步骤,以保证后续分析的准确性和可靠性。
预处理包括剔除低质量碱基、去除接头序列、剪切序列、质量修剪、构建序列库等步骤,以准备分析所需的干净数据。
三、基因表达分析基因表达分析是转录组数据分析的核心内容之一。
它通过比较不同条件下的基因表达水平,揭示基因的差异表达情况。
基因表达分析方法包括差异基因表达分析、基因聚类分析和基因富集分析等。
差异基因表达分析可以筛选出在不同条件下表达显著差异的基因,通过Gene Ontology(GO)和通路富集分析可以进一步了解这些差异基因的功能和相关通路。
四、基因调控网络分析基因调控网络分析是转录组数据分析的另一个重要方面。
它通过挖掘转录因子和靶基因之间的关系,揭示基因调控网络的结构和功能。
基因调控网络分析方法包括共表达网络分析和转录因子-靶基因分析等。
共表达网络分析可以用来发现与特定条件相关的基因模块,而转录因子-靶基因分析可以用来确定重要的转录因子并预测其功能。
五、功能注释与通路分析功能注释和通路分析是转录组数据分析的重要环节。
功能注释用于对差异表达基因进行功能注释,以了解其可能的生物学功能和参与的调控通路。
通路分析则是将差异基因映射到特定通路中,以揭示基因在特定生物学过程中的功能和相互作用关系。
功能注释和通路分析可以辅助我们理解基因调控网络的功能和调控机制。
六、数据可视化数据可视化是转录组数据分析的一个重要环节,它通过图表、散点图、热图等形式展示转录组数据的信息,增强数据分析结果的直观性和可解释性。
生物信息学中的转录组数据分析方法与工具研究转录组数据分析是生物信息学领域的重要研究方向,它对于理解基因表达调控、发现新的转录本、预测基因功能等具有重要意义。
在本篇文章中,我们将详细介绍生物信息学中的转录组数据分析方法与常用的分析工具。
转录组是特定细胞或组织中所有mRNA的集合。
通过转录组数据分析,可以了解细胞或组织中所有基因的表达水平,从而揭示细胞功能和生物过程的调控机制。
下面我们将介绍转录组数据分析的常见步骤及相关的分析方法与工具。
第一步是数据预处理。
转录组数据通常是通过RNA测序技术获得的,因此需要进行质控和清洗,去除低质量的测序reads、适配体和重复序列等。
常用的数据预处理工具包括Trimmomatic、FastQC等。
第二步是序列比对。
将清洗后的 reads 与参考基因组进行比对,得到每个 reads 的位置信息。
比对结果可以用于计算基因的表达量以及检测新的转录本。
常见的比对工具有Bowtie、HISAT2、STAR等。
第三步是基因表达量的计算。
通过将测序 reads 映射到参考基因组的基因区域,可以计算出每个基因的表达量。
常用的工具有HTSeq、FeatureCounts等。
第四步是差异表达分析。
差异表达分析可以用来寻找在不同条件下表达水平发生显著变化的基因。
常用的差异表达分析工具有DESeq2、edgeR等。
第五步是功能注释和富集分析。
对差异表达基因进行功能注释和富集分析可以帮助我们理解这些基因在生物过程中的功能和调控机制。
常用的工具有DAVID、GSEA等。
除了上述基本步骤外,还有一些高级的转录组数据分析方法和工具,可以进一步挖掘和解析转录组数据的信息。
例如,可以通过融合多种类型的数据,如基因表达、蛋白质互作和代谢通路等,来构建转录组的整体网络。
常用的工具有Cytoscape。
此外,还有一些专门用于分析非编码RNA的工具,例如miRNA和lncRNA。
对于miRNA数据的分析,常用的工具有miRDeep2、miRanda等。
转录组数据分析方法与应用随着高通量测序技术的广泛应用,越来越多的转录组数据被生成和收集,这些数据对于研究基因表达调控、发现新基因和新剪接变体、诊断疾病等方面具有重要意义。
因此,转录组数据分析已成为当前生物学研究中的重要方向之一。
而转录组数据分析的常用方法之一就是基于RNA-Seq技术的差异表达分析。
本文将从数据获取、数据预处理、差异表达分析、生物信息学工具及其应用等方面,探讨转录组数据分析的方法和应用。
一、数据获取转录组数据获取的最重要的技术手段是RNA-Seq。
RNA-Seq是一项革命性的技术,通过将参考基因组上的所有转录本进行定量分析,可以获得全转录组的基因表达谱,还可以识别新的基因、新的剪接变异、可变剪接事件等。
RNA-Seq技术最重要的是能够获得高通量的转录本序列信息,但与其他高通量技术相比,不同转录物的表达水平测定的相关性较弱,需要更多的样本。
除了RNA-Seq,核酸芯片技术也被广泛应用于转录组数据的获取。
二、数据预处理在进行差异表达分析之前,对于原始的转录组数据进行完整的质量评估和处理将产生重要的影响。
由于RNA-Seq是一种PCR扩增技术,测序错误率是非常高的,为了避免这些错误对研究的影响,需要对原始数据进行筛选和过滤。
主要的处理过程包括:去除低质量序列、去除未被测到的序列、去除重复序列、去除核酸污染物等。
三、差异表达分析差异表达分析是转录组研究的核心部分,通过比较样本之间的差异来对基因表达谱进行解释。
RNA-Seq数据的分析流程通常包含数据预处理、对数据进行比对和定量、根据基因显著性筛选差异表达基因、寻找差异表达基因与通路、功能的相关性及其下游效应等。
在对数据进行分析之前,需要首先对数据进行标准化处理,以消除影响分析结果的不必要因素。
标准化处理通常包括对数据进行正则化、去批次效应、去OTU效应等。
差异表达分析的步骤:1. 表达定量。
标准化RNA-Seq中的表达量是FPKM或者RPKM。
真核转录组讲解及数据解读PPT转录组结果解读转录调控研究部北京诺禾致源科技股份有限公司OUTLINE简介实验部分生物信息分析概述1转录组是指特定组织或细胞在某个时间或某个状态下转录出来的所有RNA的总和,主要包括mRNA和非编码RNA。
转录组研究是研究基因功能和结构的基础,对生物体的发育和疾病的发生具有重要作用。
RNA-seq技术流程主要包含两个部分,建库测序和数据分析。
2实验部分(RNA检测、建库、测序))琼脂糖凝胶电泳:分析样品RNA完整性及是否存在杂质污染。
NanoPhotometerspectrophotometer:检测RNA纯度(OD260/280及OD260/230比值)。
Agilent 2100 bioanalyzer:精确检测RNA完整性。
链特异性文库优势:相同数据量下可获取更多有效信息;能获得更精准的基因定量、定位与注释信息51、一般动物样品会有三条带:28S 、18S 、5S ,如果提取过程经过过柱处理或者利用CTAB+LiCl 方法提取,5S 可能较暗或者没有。
昆虫或者软体动物等样品只有1条比较明显的带,例如:牡蛎、果蝇、螨虫、蝗虫、蚊、蚕等2、植物样品有三条带:25S 、18S 、5S ,有些特殊物种或部位可能本身含条带比较多,如果条带清晰,也可初步判定合格3.原核生物中主要有5S 、16S 、23S rRNA叶片小鼠蚊动物植物原核RIN 5RIN 7RIN 8RIN 9RIN 4RIN 6RIN 10RIN 2RIN 1RIN 值范围示意图问与答文献要求OD260/OD230≥1.8,OD260/OD230如果小于2.0,则表明样品中被碳水化合物、盐类或有机溶剂污染;OD260/OD230合格的标准是多少呢?答:OD260/OD230≥2.0,且OD260/OD280≥2.0这说明RNA 提取结果是相当好的,一般在1.8-2.1之间就说明RNA结果十分好,但是nanodrop的灵敏度没有2100好,因此我们主要根据2100检测结果来判定RNA是否合格,一般只要RIN值和RNA总量达到我们的判定标准的话,我们就会判为合格。
转录组学数据分析方法的研究及其应用从基因组学到转录组学,高通量技术的发展推动了分子生物学领域的发展。
在转录组学领域中,RNA测序技术已成为研究人员探究生命多样性、研究基因表达变化和功能的关键工具。
但是,数据分析是RNA测序研究中面临的挑战之一。
随着RNA测序数据的数量急剧增加,转录组学数据分析方法的研究及其应用变得至关重要。
本文将着重讨论RNA测序数据的分析方法及其应用。
一、RNA测序数据处理1.1 数据预处理RNA测序技术生成的数据具有一定的噪音和偏差。
因此,在进行RNA测序数据分析之前,需要对原始数据进行预处理。
预处理包括去除低质量序列、去除接头序列、去除重复序列和过滤低表达基因等步骤。
RNA测序数据预处理的目的是减小对下游分析的影响,同时提高后续数据分析的准确性和可靠性。
1.2 基因表达量分析在RNA测序数据分析中,基因表达量分析是最常见的应用之一。
基因的表达量是指RNA分子的数量,通常使用reads per kilobase of exon model per million mapped reads (RPKM)或fragments per kilobase of transcript per million mapped reads (FPKM)作为基因表达的度量。
基因表达量分析可以研究不同样本或不同组织之间的基因表达变化。
二、差异基因分析差异表达分析可以帮助我们比较不同样本或组织中基因表达的变化。
差异基因分析可以发现可能涉及疾病和生理过程的基因和通路。
在差异基因分析中,通常会将RNA测序数据分为不同的组,例如病人组和对照组。
然后,可以使用不同的统计方法来确定不同组之间的基因表达差异是否具有统计学意义。
差异表达分析还可以发现新的基因或表达物,以及预测可能的分子机制。
三、富集分析富集分析可以解释在差异基因分析中发现的有意义的表达差异。
通常会使用基因集富集分析来鉴定差异表达基因的生物学功能和通路。