microRNA反转和定量引物设计原理、实验方法
- 格式:doc
- 大小:244.00 KB
- 文档页数:5

microRNA qRT-PCR引物套装是包含了miRNA特异的逆转录引物,以及PCR正反向引物。
通常样本采用U6作为miRNA检测的内参,适用于单个和多个样本不同miRNA的定量分析。
根据目的miRNA设计独特茎环和加poly(A)结构,提供人源、大鼠源以及小鼠源miRNA的逆转录引物及定量PCR 引物,引物具有灵敏度高,可特异地结合miRNA分子并且具有更好的稳定性,检测范围广,能有效地扩增待检测样本。
(1) Stem-loop miRNA qRT-PCR
利用特殊的茎环结构反转引物在SYBR Green荧光染料或TaqMan探针的作用
下检测miRNA。
图1.Stem-loop miRNA qRT-PCR检测试剂的实验流程图。
利用特殊的茎环结构反转引物
在SYBR Green 荧光染料或TaqMan探针的作用下检测miRNA
(2) 加Poly(A)尾miRNA qRT-PCR
在成熟的miRNA的3‘端加poly(A)尾后,通过带通用序列的poly(T)引物反
转延伸,在SYBR Green 荧光染料的作用下PCR检测反转录形成的miRNA。
图2.加Poly(A)尾miRNA qRT-PCR检测试剂盒的实验流程图
在成熟的miRNA的3’端加poly(A)尾后
通过poly(T)引物反转延伸,在SYBR Green荧光染料的作用下PCR检测反转录形成的miRNA
miRNA引物验证
1、反应条件均一,灵敏度高。
2、可检测少量样本中的成熟的miRNA。
3、方便,经济,覆盖面广,重复性好。
4、高特异性和灵敏度确保了结果的可靠性和重复性。

microRNA反转和定量引物设计microRNA(miRNA)是一类长度约为18-25个核苷酸的小分子非编码RNA,它们能够通过与靶向mRNA配对而引起转录后基因沉默、翻译后调控等作用。
miRNA在生物体内广泛存在,对于肿瘤发生和发展、免疫应答、细胞分化等生物学过程起着重要作用。
首先,让我们来了解一下miRNA反转的原理。
miRNA反转是将miRNA通过逆转录酶酶(Reverse Transcriptase)转录成DNA的过程。
反转录过程中需要两个基本核酸片段:miRNA的反向亚基(RT primer)和反转录引物(RT primer)。
RT基质是由具有未匹配的末端,可以与miRNA互补配对的引物分子。
反转录过程通过与RT引物的匹配,引发RNA链延长的启动,使得miRNA转录成DNA。
这个反应需要RNA酶H(RNase H)的参与来降解RNA模板。
最终通过PCR扩增获得足够的DNA产物用于后续实验。
然后,我们来了解一下miRNA定量引物设计的步骤。
miRNA定量引物设计主要包括两个部分:上游引物和下游引物。
上游引物被设计成能够特异性结合到miRNA的3’端,而下游引物结合到miRNA的5’端。
这样,当PCR扩增时,上下游引物与miRNA的结合使得DNA在目标区域扩增。
设计定量引物需要考虑以下几个因素:1.引物的长度:通常上游引物长度为19-25个核苷酸,下游引物长度为18-22个核苷酸。
引物长度的选择应遵循引物结合的特异性和扩增效率。
2. 引物的Tm值:Tm值是引物与模板的熔解温度。
建议设计上游引物和下游引物的Tm值在55-65℃之间,以提高引物与miRNA的结合特异性。
3. 引物的序列特异性:为了确保引物能够特异性地结合于目标miRNA,应尽量避免引物与其他miRNA或基因组的序列匹配。
4. 引物的其他特性:引物的GC含量、自身二聚体形成和hairpin结构等也需要考虑。
在设计引物时,可以借助一些在线工具和软件,如Primer3、miRprimer和OligoAnalyzer等,这些工具可以帮助用户进行引物设计和评估引物的特性、特异性和二聚体形成等。

microRNA(miRNA)引物设计及过程原理说明编辑整理:尊敬的读者朋友们:这里是精品文档编辑中心,本文档内容是由我和我的同事精心编辑整理后发布的,发布之前我们对文中内容进行仔细校对,但是难免会有疏漏的地方,但是任然希望(microRNA(miRNA)引物设计及过程原理说明)的内容能够给您的工作和学习带来便利。
同时也真诚的希望收到您的建议和反馈,这将是我们进步的源泉,前进的动力。
本文可编辑可修改,如果觉得对您有帮助请收藏以便随时查阅,最后祝您生活愉快业绩进步,以下为microRNA(miRNA)引物设计及过程原理说明的全部内容。
microRNA(miRNA)引物设计及过程原理说明microRNAs的平均长度23nt左右,所以miRNA引物设计与常规引物设计存在很大差别,以下讲解一下整个miRNA引物设计的过程加上实验流程,以帮助大家对各方面的学习。
首先,引物设计之前先介绍miRNA反转录合成cDNA的过程。
我们拿经典颈环序列GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGAC作介绍。
打开DNAstar软件的PrimerSelect;file打开下拉菜单;打开Enter New Primer…;粘贴颈环序列;点击OK。
颈环结构已经输入,下面查看颈环结构回形成的那些发夹结构。
选择颈环结构(鼠标点一下);点击Report下拉菜单;选择Primer Hairpins。
第一个发夹结构是实验所需的结构(dG=—20.4kc/m).下面结合has—miR—122—5P合成cDNA的具体过程讲解has—miR-122—5P:UGGAGUGUGACAAUGGUGUUUGU将U转T,方便后面使用:TGGAGTGTGACAATGGTGTTTGTmiRNAs的颈环结构引物:是将miRNAs的3’端后6位碱基反向互补添加到经典颈环结构的3’端形成的结构.(自己根据后面图片想一下原因,加6个碱基为经验所授)has—miR-122-5P的后6位:GTTTGThas-miR-122-5P的后6位的反向互补序列:ACAAAC颈环引物序列:5’—GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACACAAAC -3’查看形成的发夹结构(方法前面有讲)看一下miRNA和颈环引物在一起会是怎么样子:在含反转录酶及适当的条件下,miRNA和其颈环引物将会合成cDNA链:GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACACAAACACCATTGTCA CACTCCA查看cDNA结构:我们知道了cDNA的合成过程和序列,同时学习了miRNA 的反转录过程。
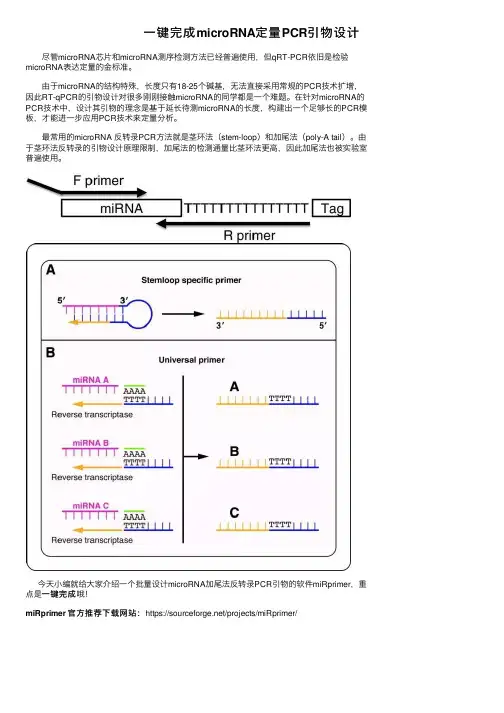
⼀键完成microRNA定量PCR引物设计尽管microRNA芯⽚和microRNA测序检测⽅法已经普遍使⽤,但qRT-PCR依旧是检验microRNA表达定量的⾦标准。
由于microRNA的结构特殊,长度只有18-25个碱基,⽆法直接采⽤常规的PCR技术扩增,因此RT-qPCR的引物设计对很多刚刚接触microRNA的同学都是⼀个难题。
在针对microRNA的PCR技术中,设计其引物的理念是基于延长待测microRNA的长度,构建出⼀个⾜够长的PCR模板,才能进⼀步应⽤PCR技术来定量分析。
最常⽤的microRNA 反转录PCR⽅法就是茎环法(stem-loop)和加尾法(poly-A tail)。
由于茎环法反转录的引物设计原理限制,加尾法的检测通量⽐茎环法更⾼,因此加尾法也被实验室普遍使⽤。
今天⼩编就给⼤家介绍⼀个批量设计microRNA加尾法反转录PCR引物的软件miRprimer,重⼀键完成哦!点是⼀键完成miRprimer 官⽅推荐下载⽹站:https:///projects/miRprimer/⽹站后台⽂件直接下载miRprimer地址:https:///project/miRprimer/miRprimer2_installer.zipmiRprimer2_installer.zip,整个软件的压缩包只有2.68M (2848kb)⼤⼩。
提醒:提醒1. 软件⽀持在Windows XP或更⾼系统中运⾏,还没在苹果电脑Mac OS系统测试(原因是⼩编的钱包羞涩~~)2. 经⼩编测试,最新版本miRprimer顺利运⾏,不需在电脑中安装Ruby脚本环境。
第⼀步miRprimer2_installer.zip压缩包2.68M⼤⼩,下载后解压缩⽣成同名⽂件夹,内有三个⽂件:input_miRs.txt、miRprimer2.exe、README.txt。
特别提⽰:不要更改这些⽂件的⽂件名。
特别提⽰:第⼆步input_miRs.txt,fasta格式储存的是需要设计引物的microRNA名字和序列。
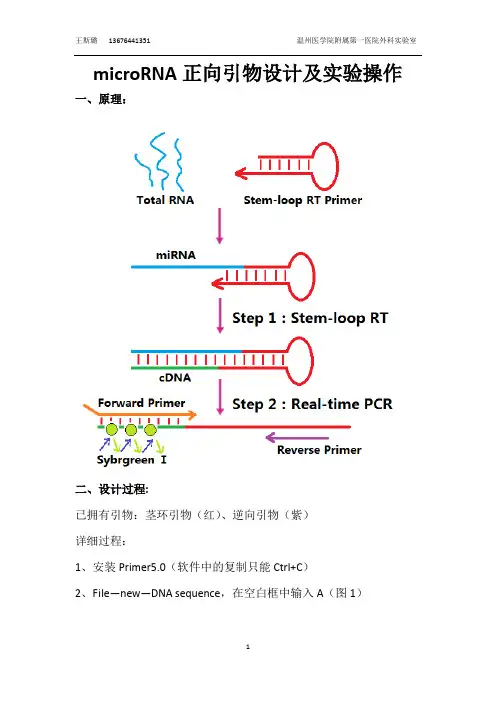
microRNA正向引物设计及实验操作一、原理:二、设计过程:已拥有引物:茎环引物(红)、逆向引物(紫)详细过程:1、安装Primer5.0(软件中的复制只能Ctrl+C)2、File—new—DNA sequence,在空白框中输入A(图1)(图1)3、复制要设计的microRNA(let7b:tgaggtagtaggttgtgtggtt),跳出一个选择框,选择Reverse Complemented(图2)(图3)4、在输入的序列后输入几个G,为正链(橙)5’端突出的互补链(图4)5、点击Primer,跳出图5(图5)6、选择Edit Primers,跳出图6(图6)7、删去空白框中的引物,键盘输入Ctrl+C(即复制要设计的microRNA 序列),在跳出的对话框中选择Reversed,随即在False Priming框下出现红色的Found,点击Found,再点击对话框靠右的Primer,出现图7图7 8、在出现的空白框中调节引物的长度和Tm温度:长度通过删除3’端的碱基控制在15-18个,但其Tm值不得少于35度;Tm温度通过5’端加入G或C来调节,尽量控制在56-59度。
9、调节好后的引物点击对话框中的Analyze,分析修改后的引物。
尽量避免引物二聚体(dimer)的3’端的互补。
10、将修改好的引物复制到目标文档中时,注意引物的方向,一般默认为5’端在前,3’端在后。
三、实验操作1、引物原液稀释:(1)拿到引物原液后,先点离,将引物管中的干粉贴到管壁上。
(2)加入相当于引物管中的nmol值的10倍的水(ddH2O或去离子水),例如nmol=24.456,则加入245ul的水。
(3)在引物管上标记引物的名称和稀释后的浓度,按以上方法稀释后的浓度为100umol。
(4)将稀释好的引物储存液混匀,放置一个上午或颠倒混匀一个小时,再将储存液稀释100倍到1umol,即取1ul的储存液加入到99ul 的ddH2O或去离子水中,并做好引物的标记。

microRNA引物设计及过程原理说明MicroRNAs (miRNAs) are a class of small non-coding RNA molecules, typically about 22 nucleotides in length, that play a crucial role in regulating gene expression. They bind tospecific messenger RNA (mRNA) molecules and interfere with their translation or promote their degradation, thereby controlling protein production.The process of designing miRNA primers involves several steps and considerations. Firstly, it is important to identify the target miRNA sequence that you want to amplify. This can be done by mapping the known miRNA sequences to the genome or by searching public miRNA databases. Once the target miRNA sequence is identified, a primer design software or algorithm can be used to generate the primers.There are several criteria that need to be considered when designing miRNA primers. Firstly, the primers should be specific to the target miRNA and should not have significant homology to other miRNAs or non-target RNA molecules. This can be achieved by checking the primer sequence against existing miRNA databases or by performing a BLAST search to identify potential off-target binding sites.Secondly, the primers should have a melting temperature (Tm) within a specific range to ensure optimal binding to the target miRNA. A Tm around 60-70°C is generally preferred. The Tm canbe calculated using thermodynamic parameters, such as the primer sequence and length.Thirdly, the primers should be designed in such a way that they can be easily synthesized and labeled if necessary. This involves selecting appropriate primer lengths and avoiding secondary structures or hairpins that can interfere with primer synthesis or efficiency.Once the primers have been designed, they can be synthesized using standard oligonucleotide synthesis techniques. If necessary, the primers can be labeled with fluorescent or radioisotopic tags for downstream applications, such as quantitative PCR or miRNA expression profiling.In summary, the process of miRNA primer design involves identifying the target miRNA sequence, designing primers that are specific to the target sequence, considering factors such as melting temperature and ease of synthesis, and validating the primers for their effectiveness in specifically amplifying the target miRNA. This process allows researchers to study the expression and function of miRNAs, which can provide valuable insights into gene regulation and disease mechanisms.。

microRNA实验方法作者:Mary Johnson如果您想将您的microRNA实验方法,或想更新这里microRNA实验方法信息. 请与我们联系。
概观miRNAMicroRNAs(miRNAs)是一类非编码的小RNA分子,通过与靶RNA的3´UTR互补或部分互补结合,使其降解或介导其翻译抑制,参与细胞增殖、凋亡、分化、代谢、发育、肿瘤转移等多种生物学过程.miRNAs 基因通常位于基因间或编码蛋白基因的内含子中,在核内由RNA聚合酶II或III转录产生具有特征性茎环结构的pri-miRNA,然后在Drosha-DGCR8复合体的作用下,剪接成70nt的pre-miRNA,它由exportin5由核内运到胞浆。
在胞浆内,pre-miRNA在Dicer酶作用下剪切成22bp的成熟双链miRNA,其中的一条链与RISC结合而参与基因转录后水平的调控。
根据miRBase数据库,目前所发现的miRNAs已超过700个,随着高通量测序的应用,将会有更多的新的miRNAs被发现。
可能近90%的人类基因受到miRNAs调控,然而,当过表达或抑制某一个miRNA时,在发生调变的众多基因当中寻找并鉴定其中起关键作用的靶基因仍然具有相当大的挑战。
目前鉴定miRNA 靶基因的常用策略是利用生物信息学软件预测,结合基因芯片分析以及生物学实验方法来研究miRNA的功能及寻找其中起重要作用的靶基因。
此外,利用蛋白质质谱来寻找miRNA靶基因也成为一种新的途径。
microRNA的筛选在上千个miRNA中,哪些miRNA在特定的生物学功能起着关键的作用呢?这是研究miRNA功能所面临的首要问题。
miRNA基因技术可以快速有效的提供miRNA表达图谱。
通过比较正常样本与疾病样本中miRNA表达图谱的差异,寻找在生物学功能上起作用的miRNA。
该技术为临床肿瘤诊断提供了新的思路,也为miRNA作为肿瘤的标志物提供了依据。

百奥迈科生物技术有限公司B 电话:地址:江苏省南通市经济与技术开发区常兴路miRNA Q-PCR Detection Primer Setz 概述:microRNA(miRNA)是一类有大约22 个核苷酸组成的非编码小分子RNA ,其广泛存在于真核生物中。
miRNA 在个体发育的不同时期及不同组织中有不同的表达模式,都表明了其在发育和分化中起有重大的调控作用。
迄今为此,对miRNA 的检测方法主要有Northern Blot 等基于分子杂交的方法,这些方法敏感度低、耗时长、RNA 的用量较大。
miRNA Q-PCR Detection Kit 采用了国际上公认的核酸检测标准技术――Real-Time PCR 技术来对miRNA 进行检测,其具有快速、特异性强、灵敏度高等优点。
z 实验原理:z 产品内容:ID Description Qty. RT Primer miRNA Reverse Transcription Primer 2 nmolForward Primer miRNA Forward Primer 2 nmolReverse Primer Universal Reverse Primer 2 nmolz 注意事项:引物为干粉形式,需先离心,后每管加入200 μL Nuclease-Free H 2O 稀释至10μM ,混匀后于-20o C 冻存备用;干粉形式引物可在室温下稳定保存1年。
稀释后溶液在-20o C ,避免在4o C 或室温长期存放;稀释后的溶液可先分装成小份冻存,避免多次反复冻融。
miRNA Q-PCR Detection Primer SetFor the detection and quantification of miRNA using real-time PCR.Catalog No.BK1010一、组成和储存:miRNA 定量检测引物包括以下组分,产品应在-20℃储存,避免在4℃或室温长期存放。
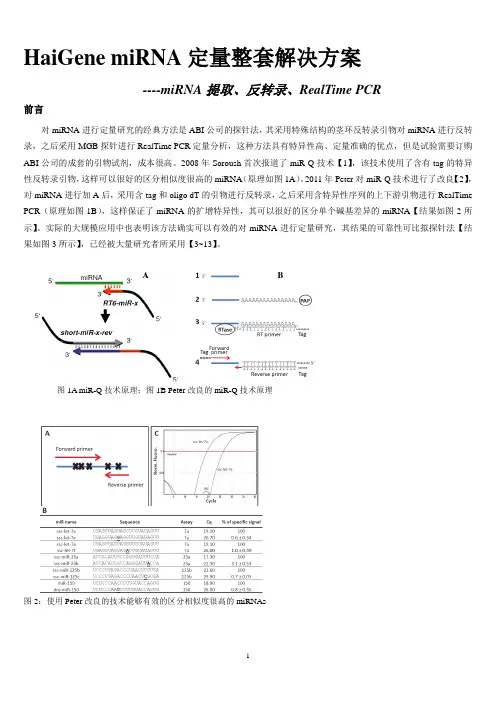
HaiGene miRNA 定量整套解决方案----miRNA 提取、反转录、RealTime PCR前言对miRNA 进行定量研究的经典方法是ABI 公司的探针法,其采用特殊结构的茎环反转录引物对miRNA 进行反转录,之后采用MGB 探针进行RealTime PCR 定量分析,这种方法具有特异性高、定量准确的优点,但是试验需要订购ABI 公司的成套的引物试剂,成本很高。
2008年Soroush 首次报道了miR-Q 技术【1】,该技术使用了含有tag 的特异性反转录引物,这样可以很好的区分相似度很高的miRNA (原理如图1A )。
2011年Peter 对miR-Q 技术进行了改良【2】,对miRNA 进行加A 后,采用含tag 和oligo dT 的引物进行反转录,之后采用含特异性序列的上下游引物进行RealTime PCR (原理如图1B ),这样保证了miRNA 的扩增特异性,其可以很好的区分单个碱基差异的miRNA 【结果如图2所示】。
实际的大规模应用中也表明该方法确实可以有效的对miRNA 进行定量研究,其结果的可靠性可比拟探针法【结果如图3所示】,已经被大量研究者所采用【3~13】。
图1A miR-Q 技术原理;图1B Peter 改良的miR-Q 技术原理图2:使用Peter 改良的技术能够有效的区分相似度很高的miRNAs图3:使用Peter改良的技术能够高效的扩增miRNAs海基优化的miRNA定量方案是在Peter改良的技术方法基础上建立起来的,包括三部分:高效提取miRNA、通过加A法反转录获得miRNA的cDNA、应用SYBR Green染料法对获得的cDNA产物进行RealTime PCR扩增。
采用该实验方案进行miRNA表达研究结果可靠、操作简便、重复性高、耗时短、费用低,操作流程如图4所示。
下面将该方案的使用特点和方法进行详细阐述。
图4 HaiGene优化的miRNA qRealTime PCR定量方案第一步miRNA的提取核酸的提取往往是研究过程的第一步、是实验的基础。
miRNA研究方法尽管早在1993年,科学家就发现了第一个microRNA,但直到2003年以后,这种小RNA分子的大作用才不断被发现。
研究显示:miRNA通过与转录本的相互作用,关闭或抑制基因的表达,影响了30%的基因。
miRNA在多个组织中,例如正常和肿瘤组织中差异表达。
因此,通过表达谱分析寻找疾病相关miRNA并进行发病机理研究,最终应用于肿瘤诊断和治疗,已经成为目前miRNA研究的重要方向。
在研究中,从深度测序、miRNA芯片到通过导入化学合成的mimics/antagomirs 等实现miRNA过表达和抑制,都是研究中常用的方法。
同时,在miRNA研究中,生物信息学分析扮演着越来越重要的角色。
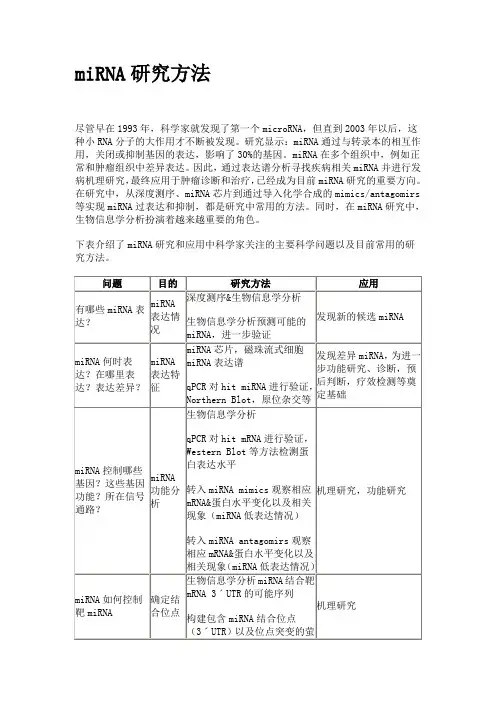
下表介绍了miRNA研究和应用中科学家关注的主要科学问题以及目前常用的研究方法。
miRNA主要研究技术深度测序抽提分离小分子(例如18-30nt)RNA,通过RT-PCR扩增之后,利用solexa深度测序,并进行生物信息学分析,获得miRNA表达谱。
深度测序结合生物信息学分析手段,可以对海量数据进行分析,分别统计出已知的miRNA (miRNA-known)、新的miRNA(miRNA-new)以及可能的新miRNA(miRNA-cadidate new),并对新发现的miRNA进行靶基因分析,功能预测等。
通过深度测序的方法,可以发现新的miRNA,为进一步的深入研究奠定基础。
miRNA芯片利用miRNA芯片(例如Agilent miRNA芯片),可以高通量分析miRNA表达的时空特异性、不同样本(例如癌组织和癌旁组织)中miRNA的差异表达,进而进行把基因分析,功能注释、通路分析,网络分析等,以了解miRNA在疾病发生中的作用。
与深度测序不同,miRNA芯片针对已知miRNA进行研究。
筛选到的差异miRNA可以利用q-pCR进行验证。
q-PCR q-PCR技术可以用来检测miRNA以及其靶mRNA和相关mRNA等的检测,主要方法有茎环法和加尾法等。

miRNA定量PCR1 miRNA 茎环反转录合成cDNA(1)反转录引物设计:对成熟miRNA采用茎环反转录合成cDNA,在成熟miRNA的3’端使用一条50nt 可自行折叠成茎环结构的特异性RT 引物,序列如下:5`-GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACNN-NNNN-3`,3’端的6个N代表与成熟miRNA 3’端反相互补的6个碱基。
(2)反应体系:在0.2mlPCR反应管中加入,总反应体系为20μL:5× First Strand synthesis Buffer 4μLdNTP Mix 1μLRNase Inhibitor(40 units/μl)1μLReverse Transcriptase(M-MLV)1μLStem-loop RT Primer 1μLTotal RNA (终浓度500 ng)XμLRNase-free ddH2O 补至20 μLTotal volume 20 μL(3)反应条件:Stage 1:将混匀的反应溶液置于65℃加热5min,之后放入冰中2min。
Stage 2:放置PCR仪里16℃热激30min,之后42℃60min,85℃,5min 使酶失活停止反转录。
2 miRNA实时荧光定量qRT-PCR(1)定量引物设计:反转录后cDNA产物的引物由一条特异性引物和一条通用引物组成,特异性引物基础序列于目标miRNA成熟序列互补,根据对引物的长度,Tm 值及GC含量的要求再用软件进行调整,可在5’端加2-3个GC以增加退火温度,平衡GC含量,U6被作为内参序列。
所有使用的引物序列被列在表3-1。
(2)应用SYBR染料法,对miRNA采用实时定量检测。
反应体系参照SYBR® Premix Ex TaqTMⅡ(Perfect Real Time)说明书,(反应液配制均在冰上进行)。
总反应体系为25μL:SYBR® Premix Ex TaqTMⅡ 12.5μLSpecific Forward primer (10μM)1μLUniversal Reverse primer (10μM)1μLcDNA 1st Strand 2μLddH2O 8.5μLTotal volume 25μL(3)Real Time PCR 反应程序:Stage 1:预变性95℃5min;Stage 2:40循环:95℃5s,60℃30s以上循环结束后进行65℃~95℃的融解曲线分析。
miRNA常⽤实验⽅法miRNA常⽤实验⽅法⼀、miRNA的检测⽅法miRNA的realtime-PCR检测⽅法1、realtime-PCR引物设计miRNA realtime-PCR引物设计⽅法:1)stem-loop RT引物设计:基于通⽤的茎环结构,只需要按照不同的miRNA序列修改最末端6个碱基即可。
通⽤茎环结构序列为:GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGAC 例如设计miR-1(UGGAAUGUAAAGAAGUAUGUAU)的RT引物,只需在通⽤茎环序列后架上mi RNA3’末端的6个碱基的反向互补序列,即GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGAC ATACAT2)realtime 上游引物设计:miRNA序列除去3’端6个碱基的剩余部分作为上游引物,如miR-1的上游引物为(注意把U改为T):TGGAATGTAAAGAAGT.检查引物的Tm值(⼀般参考DNAMAN),如果Tm值较低,则在5’端加GC使Tm值接近60度。
因此miR-1的上游引物可设计为:GCGCTGGAATGTAAAGAAGT,61.4度。
3)下游引物是通⽤的,序列为GTGCAGGGTCCGAGGT。
4)引物设计好后,需要通过预试验检测引物的特异性。
⼀般需要做溶解曲线来检测引物的特异性;同时最好将PCR产物进⾏电泳检测产物是否单⼀(因产物长度很⼩,需要3%以上的琼脂糖胶)。
2、miRNA反转录miRNA的反转录与⼀般基因的反转录过程基本相同。
因为其产物很短,⽤最普通的逆转录酶即可。
我们⼀般使⽤的是TIANGEN的MLV,逆转录体系为:RNA 500ng~2ug5xbuffer 2uldNTP 0.25ulDDT 0.25ulRRI 0.25ulMLV 0.25ulRT primer 0.5ulH2O(RNase free) 补⾄10ul程序为(PCR仪中通常命名为CTFRT)16度,30min;42度,60min;85度,5min;4度,hold。
教你设计miRNA引物近年来测序很火,很多实验室正在或已经完成了sRNA的测序工作,miRNA研究越来越火,然而,找公司设计引物合成引物真心贵(对于我们穷屌丝来说……)。
本人自己动手琢磨了会,哎哟喂~效果良好!好了不闲扯了,下面给大家分享一下我的方法。
miRNA引物设计(茎环法)原理:由于miRNA在20bp左右,没法根据其设计引物检测出来,于是,人们就先将其延长,也就是先加一个环状片段,这样就使得原来20bp左右的延伸为80bp左右了,然后就可以根据这个80bp片段设计引物啦。
具体操作:需要三种引物:逆转录引物(RT-primer,用于延长miRNA的)、实时定量PCR上游引物、实时定量PCR通用下游引物。
1逆转录引物逆转录引物序列由5’端茎环结构引物和3’端miRNA特异性序列组成。
“特异的反向引物约42-44个核苷酸,其5’端的36个核苷酸序列是固定的,形成一个8核苷酸环和20核苷酸茎的结构,其3’端的6-8个核苷酸就与microRNA互补。
”5’端茎环结构通常固定,如5’-CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAG-3’ ;3’端特异性序列由与成熟的miRNA 3’端若干寡核苷酸反向互补配对所得。
即:逆转录引物为“5‘-CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGNNNNNNNN-3’(N 为miRNA3’端反向互补的6-8个碱基)。
通俗地说就是,固定的颈环序列(网上很多或者用我这个也行)+miRNA末端6-8个反向互补碱基。
以mmu-[size=14.6666669845581px]miR-16-5p ([size=13.3333330154419px]UAGCAGCACGUAAAUAUUGGCG) 颈环序列+末尾 8个碱基反向互补。
即为:CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGcgccaata[size=13.3333330154419px]2实时定量PCR上游引物上游引物包括5’端通用序列和3‘端miRNA特异序列,5’端通用序列为:5’-GCCGAG-3’或5’-TCGGCAGG-3’,用于延伸实时定量PCR产物的长度;3’端为跟据成熟miRNA自身碱基组成及GC含量适当删减几个碱基所得的寡核苷酸或完整的成熟miRNA。
只需三步micRNA定量引物设计法(加尾法)引物设计应该是科研汪都需要掌握的一项基本技能吧,我们在之前讲过如何现成拿验证过的PCR引物,也有涉及到如何做进一步的引物修饰,那么如何对MicroRNA进行引物设计呢,作为20世纪最火的科研对象之一,本引物设计你不能不回啊。
MicroRNA一般由20多个碱基组成,引物位置基本固定。
下面给大家介绍简单的加尾法,只需设计正向引物,反向引物试剂盒自带。
另外还有茎环法,方法复杂,暂时不做介绍。
第一步转换序列,将原始序列中的U全部转化为TATGCGTAGCTTCGTACGTAGCTGT(原始序列为:AUGCGUAGCUUCGUACGUAGCUGU)第二步打开链接/support/techtools/cal c/将以上转化后的序列按下列方式输入,点击Submit出现以下页面,首先检验Tm值,62.2度,温度稍微高了些。
GC 含量正常。
第三步修改既然Tm值偏高,我们可以减少几个碱基,减少最后4个碱基(碱基只能去掉头或者尾,至于去掉几个,一般在保留19个以上碱基的情况下来确定,在此基础上保证GC含量和Tm值尽量满足要求。
以下,去掉4个尾部碱基,其他条件基本符合。
注意:首先得保证长度,不能太短。
作者琳琳猫----一个会烹饪会写古文的耿直女子,浙江大学食品工程学硕士,曾就职于华大基因7年。
有丰富的snp,甲基化,rna检测等方法学研究经验。
试剂测评专栏一定是进口试剂好么?国内同类产品哪个好?哪个性价比好?在这个鱼龙混杂的行业,一不留神就被坑。
我们是科学严谨的,光靠公司吹嘘我不干,在这个栏目,我们要拿实验数据来说话,猫姐在这个行业13年,乐于分享,勇于承担,Freescience将和猫姐一起,做一些事情,倒逼这个行业的质量能够越来越好。
自己动手microRNA引物设计miRNA是一类内源性非编码单链RNA,在体内以前体和成熟体的形式存在,而我们对某种的microRNA的表达检测的对象是成熟体,microRNA的成熟体长度约为19-25nt,由于其序列和性质的特殊性,我们需要使用与普通mRNA不同的方法对其进行引物的设计。
目前主流的方法有两种:茎环法和加尾法。
基本的原理图示如上,因为microRNA很短,引物为了方便接下来的检测我们可以通过具有颈环样结构的反转引物先将microRNA进行逆转录,然后再将用于表达检测的3'引物(即图中reverse)设计在反转引物上,最后结合mircoRNA的5'序列设计一个5’端的定量PCR引物(图中Forward)即可。
小结一下就是我们需要设计3条引物,一条做反转录的颈环引物,和2条用于做表达检测引物(Forward和reverse引物)。
茎环结构不但能有效地延长miRNA 的长度,同时它自身互补的构象可以避免与其他同源基因结合,减少了非特异性扩增的几率。
整个引物由 2 部分组成,一个通用的茎环结构和5 ~ 8 个与目的 miRNA 的3'端反向互补的碱基。
其序列通常为: 5'-GTCGTATCCAGTGCAGGGTC CGAGGTATTCGCACTGGATACGAC-3'。
其中下划线部分为茎环自身互补部分。
我们可以看看这个通用的茎环结构序列怎样互补成颈环的。
用DNAstar中的primer select点击report中的primer hairpins报告第一个就是我们通用的一个典型茎环结构了,接下来只需要根据我们的目标microRNA的序列在此序列后加入5-8个碱基与目的MicroRNA 的 3'端反向互补即可。
举个例子:hsa-miR-26a(注意大小写)首先需通过英国著名miRNA数据库网站miRBase (/)检索成熟miRNA序列。
在miRBase上可获得目前已经公布的各个物种的microRNA具体序列、基因分布、详细序列注册信息,可以通过浏览或搜索的方式查询目的miRNA。
mirRNA引物设计2miRNA常用实验方法;一、miRNA的检测方法;miRNA的realtime-PCR检测方法;1、realtime-PCR引物设计;miRNArealtime-PCR引物设计方法:;1)stem-loopRT引物设计:基于通用的茎;GTCGTATCCAGTGCAGGGTCCGAG;2)realtime上游引物设计:miRNA序列;3)下游引物是通用的,序列miRNA 常用实验方法一、miRNA的检测方法miRNA的realtime-PCR检测方法1、realtime-PCR引物设计miRNA realtime-PCR引物设计方法:1)stem-loop RT引物设计:基于通用的茎环结构,只需要按照不同的miRNA序列修改最末端6个碱基即可。
通用茎环结构序列为:GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGAC 例如设计miR-1(UGGAAUGUAAAGAAGUAUGUAU)的RT引物,只需在通用茎环序列后架上miRN A3’末端的6个碱基的反向互补序列,即GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACATA CAT 2)realtime 上游引物设计:miRNA序列除去3’端6个碱基的剩余部分作为上游引物,如miR-1的上游引物为(注意把U改为T):TGGAATGTAAAGAAGT.检查引物的Tm值(一般参考DNAMAN),如果Tm值较低,则在5’端加GC使Tm 值接近60度。
因此miR-1的上游引物可设计为:GCGCTGGAATGTAAAGAAGT,61.4度。
3)下游引物是通用的,序列为GTGCAGGGTCCGAGGT。
4)引物设计好后,需要通过预试验检测引物的特异性。
一般需要做溶解曲线来检测引物的特异性;同时最好将PCR产物进行电泳检测产物是否单一(因产物长度很小,需要3%以上的琼脂糖胶)。
microRNA_引物设计引言:microRNA是一类长度为21-25个核苷酸的小分子RNA,通过与靶mRNA结合,从而调控靶基因的表达。
microRNA的研究对于理解基因调控网络的功能和异常相关疾病的发生具有重要意义。
在研究microRNA功能和应用的过程中,引物设计是至关重要的步骤之一一、microRNA引物的特征1.引物长度:一般为18-24个核苷酸,确定引物长度要考虑到引物和目标mRNA的互补区域。
2.引物所在位置:应选择在microRNA序列的保守区域,以确保引物的特异性。
3.引物的GC含量:引物的GC含量最好在30%-60%之间,过高或过低的GC含量都会降低引物的特异性。
4.引物的Tm值:引物的Tm值应在50-75°C之间,以保证PCR反应的稳定性。
5.引物的3'端稳定性:引物的3'端最好是G或G/C碱基,以增加引物与靶mRNA的互补稳定性。
6.避免引物之间的重复:将已有的引物序列进行BLAST比对,避免引物之间的重复。
二、microRNA引物设计的步骤1.获取microRNA序列:可以通过数据库(如miRBase)查询或测序分析等方法获取所需的microRNA序列。
2.引物长度的确定:根据目的和实验需求,确定引物的长度,一般为18-24个核苷酸。
3.引物所在位置的选择:通过比对多个物种的microRNA序列,找到保守区域作为引物的设计区域。
4.计算引物的Tm值:根据引物长度、碱基组成等参数,计算引物的Tm值。
常用的公式有Wallace等提出的公式和Breslauer等提出的公式。
5.检查引物的互补性:使用工具(如UCSC Genome Browser或BLAST)检查引物与靶mRNA之间的互补性。
6.检查引物的特异性:使用工具(如miRWalk、miRTarBase等数据库)检查引物与其他非目标mRNA的互补性,以确保引物的特异性。
7.优化引物设计:根据实验需求和前期实验结果,对引物设计进行调整和优化。
microRNA的引物设计
以ssc-miR-222-3p为例设计引物,其成熟体序列为:AGCTACATCTGGCTACTGGGTCT
反向引物:每个反向引物的都带有一段固定的序列,可以形成一个茎环,
固定的序列为:5,-CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAG-3,
在这个序列后加上八个碱基,这八个碱基是ssc-miR-222-3p从后面数八个碱基的反向互补序列,就是CTGGGTCT的反向互补:AGACCCAG ,最后得到的反向引物的序列为
5,-CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAG AGACCCAG -3,
正向引物:每个正向引物也带有一段固定的序列,固定的序列为:ACACTCCAGCTGGG
在这个序列后加上于成熟体除后面六个外剩下的碱基序列,成熟体除掉后面六个碱基后序列为:AGCTACATCTGGCTACT
5,-ACACTCCAGCTGGG AGCTACATCTGGCTACT -3,
URP:统一反向引物,也是一段固定的序列,TGGTGTCGTGGAGTCG
U6引物F-CTCGCTTCGGCAGCACA ,R-AACGCTTCACGAATTTGCGT
使用方法:
1逆转的引物:所有要做的miRNA反向引物的混合,每个10微升
2PCR的引物:50微升的体系,30微升的水,10微升的正向引物,10微升的URP
2.hsa-miR-124(hsmq-0032 引物)
推荐退火温度:60℃
hsa-miR-124 扩增曲线示意图hsa-miR-124 融解曲线示意图
3.hsa-miR-125b(hsmq-0034 引物)
推荐退火温度:60℃
hsa-miR-125b 扩增曲线示意图hsa-miR-125b 融解曲线示意图
4.hsa-miR-137(hsmq-0035 引物)
推荐退火温度:60℃
hsa-miR-137 扩增曲线示意图hsa-miR-137 融解曲线示意图
5.hsnRNA U6 引物测试
推荐退火温度:60℃
hsnRNA U6 扩增曲线示意图hsnRNA U6 融解曲线示意图
6. 电泳结果
取5μl PCR 产物进行电泳,胶浓度(3%),所有检测都为两个阳性,一个NTC
其中泳道1 为hsa-miR-16、泳道2 为hsa-miR-124、泳道3 为hsa-miR-125b、泳道4 为
hsa-miR-137、泳道5 为hsnRNA-U6[img]。