lamp引物设计实例
- 格式:doc
- 大小:482.50 KB
- 文档页数:5

LAMP原理及引物设计与实例.LAMP引物的设计LAMP引物的设计主要是针对靶基因的六个不同的区域,基于靶基因3' 端的F3c、F2c和Flc区以及5' 端的Bl、B2和B3区等6个不同的位点设计4种引物。
FIP(Forward Inner Primer):上游内部引物,由F2区和F1C区域组成,F2区与靶基因3’端的F2c区域互补,F1C区与靶基因5' 端的Flc区域序列相同。
F3引物:上游外部引物(Forward Outer Primer),由F3区组成,并与靶基因的F3c区域互补。
BIP引物:下游内部引物(Backward Inner Primer ),由B1C和B2区域组成,B2区与靶基因3' 端的B2c区域互补,B1C域与靶基因5' 端的Blc区域序列相同.B3引物:下游外部引物(Backward Outer Primer ),由B3区域组成,和靶基因的B3c区域互补。
2.扩增原理60-65℃是双链DNA复性及延伸的中间温度,DNA在65℃左右处于动态平衡状态。
因此,DNA在此温度下合成是可能的。
利用4种特异引物依靠一种高活性链置换DNA聚合酶。
使得链置换DNA合成在不停地自我循环。
扩增分两个阶段。
第1阶段为起始阶段,任何一个引物向双链DNA的互补部位进行碱基配对延伸时,另一条链就会解离,变成单链。
上游内部引物FIP的F2序列首先与模板F2c结合(如图B所示),在链置换型DNA聚合酶的作用下向前延伸启动链置换合成。
外部引物F3与模板F3c结合并延伸,置换出完整的FIP连接的互补单链(如图C所示)。
FIP上的F1c与此单链上的Fl 为互补结构。
自我碱基配对形成环状结构(如图C所示)。
以此链为模板。
下游引物BIP与B3先后启动类似于FIP和F3的合成,形成哑铃状结构的单链。
迅速以3' 末端的Fl区段为起点。
以自身为模板,进行DNA合成延伸形成茎环状结构。


核酸环介导等温扩增技术(LAMP)引物设计与实例Time:2009-12-07 PM 14:52 Author:bioer Hits: 1459 times 烟头整理LAMP的特点LAMP与以往的核酸扩增方法相比具有如下优点:(1)操作简单LAMP核酸扩增是在等温条件下进行,对于中小医院只需要水浴锅即可,产物检测用肉眼观察或浊度仪检测沉淀浊度即可判断。
对于RNA的扩增只需要在反应体系中加入逆转录酶就可同步进行(RT-LAMP),不需要特殊的试剂及仪器。
(2)快速高效因为不需要预先的双链DNA热变性,避免了温度循环而造成的时间损失。
核酸扩增在l h内均可完成,添加环状引物后时间可以节省1/2,多数情况在20-30 rain均可检测到扩增产物。
且产物可以扩增至109倍,达0.5 mg/mL。
应用专门的浊度仪可以达到实时定量检测。
(3)高特异性由于是针对靶序列6个区域设计的4种特异性引物。
6个区域中任何区域与引物不匹配均不能进行核酸扩增。
故其特异性极高。
(4)高灵敏度对于病毒扩增模板可达几个拷贝,比PCR高出数量级的差异。
缺点:由于LAMP扩增是链置换合成,靶序列长度最好在300 bp以内。
>500 bp则较难扩增。
故不能进行长链DNA的扩增。
由于灵敏度高。
极易受到污染而产生假阳性结果。
故要特别注意严谨操作,以及在产物的回收鉴定、克隆、单链分离方面均逊色于传统的PCR 方法。
引物设计实例LAMP引物设计的在线网站(http://primerexplorer.jp/e/),只要导入靶基因就能自动生成成组引物。
以某一微生物的鞭毛基因为例讲解一下LAMP引物设计的过程:首先单击浏览按钮选择靶基因序列文件,靶序列默认的是小于22 kbp。
支持三个类型的文件,普通文本格式(仅含序列), FASTA格式和GenBank 格式文件。
第二,从下面三个选项中选择定参数设定(引物设计条件)条件。
基于GC含量的自动判断, 起始的参数是特定的:如果GC含量小于或等于45%.,则选取AT丰度高的区,如果GC含量高于60%,则选取GC丰度高的区,其它情况是标准设定状态。


金黄色葡萄球菌、大肠杆菌和表皮葡萄球菌LAMP检测方法的建立金黄色葡萄球菌、大肠杆菌和表皮葡萄球菌LAMP检测方法的建立引言近年来,食品安全问题引起了广泛关注。
细菌感染是食品中最常见的安全隐患之一,特别是金黄色葡萄球菌、大肠杆菌和表皮葡萄球菌的污染可能导致食品中毒事件。
因此,发展一种快速、准确、灵敏的检测方法对于食品安全具有重要意义。
本文旨在建立一种利用LAMP技术检测金黄色葡萄球菌、大肠杆菌和表皮葡萄球菌的方法。
材料与方法1. 实验样品准备:从市场上购买金黄色葡萄球菌、大肠杆菌和表皮葡萄球菌,采用传统培养方法培养,获得细菌培养物。
2. 提取细菌DNA:采用商业化DNA提取试剂盒,按照说明书操作,提取细菌DNA。
3. LAMP引物设计:根据金黄色葡萄球菌、大肠杆菌和表皮葡萄球菌的保守基因序列,设计与目标序列互补的引物。
4. LAMP反应体系:按照标准反应体系配制反应液,包括目标DNA、引物、酶和缓冲液。
将反应液加入LAMP反应管中。
5. LAMP反应条件:将LAMP反应管放入恒温水浴中,在特定的温度下反应一定时间。
6. LAMP产物分析:将LAMP反应产物进行凝胶电泳分析,观察是否有特异性扩增产物。
结果与讨论通过实验,我们成功建立了一种利用LAMP技术检测金黄色葡萄球菌、大肠杆菌和表皮葡萄球菌的方法。
首先,我们从市场上购买了金黄色葡萄球菌、大肠杆菌和表皮葡萄球菌的培养物,确保实验样品的来源真实性。
其次,我们采用商业化DNA提取试剂盒,成功提取了目标菌株的DNA,为后续的LAMP反应提供了可靠的实验材料。
接着,我们根据目标菌株的保守基因序列设计了特异性引物,确保检测方法的准确性和可靠性。
然后,我们按照标准反应体系配制了反应液,并将反应液加入LAMP反应管中。
通过恒温水浴,我们在特定的温度下进行了LAMP反应。
最后,我们将LAMP反应产物进行了凝胶电泳分析。
结果显示,在金黄色葡萄球菌、大肠杆菌和表皮葡萄球菌的目标序列位置,出现了特异性扩增产物。

LAMP技术原理和引物设计LAMP技术(Loop-mediated isothermal amplification),中文称为环介导等温扩增技术,是一种于2000年由Eiken Chemical Co. Ltd.日本公司开发的基于异十四链聚合酶反应(Bst聚合酶)的异源DNA快速扩增技术。
LAMP技术通过引物设计和反应条件的优化,实现在等温条件下对目标DNA的高效扩增。
下面将分别介绍LAMP技术的原理和引物设计。
LAMP技术的核心原理是通过酶的协同作用,在等温条件下进行DNA的扩增。
它利用一种特殊的DNA聚合酶(Bst聚合酶),能够在不需要高温退火的情况下,具有高度特异性和高效率地进行DNA合成。
LAMP技术本身具有极高的扩增速度,优势在于其在等温下,不需要复杂的设备和严格的实验条件,可以简化扩增过程。
同时采用特殊设计的引物组合,能够提高扩增特异性。
1.初始化反应:将反应体系中的DNA片段与引物(包括2个外端引物和2个补体引物)结合;2. 引物扩增:引物与Bst聚合酶作用,反应体系中的DNA得到扩增;3.聚合物合成:一种特殊的引物结合到目标DNA的5'末端,通过内端引物和内部位点进行扩增;4.循环放大:扩增产物作为新的模板参与反应,进行连续循环扩增。
通过这种等温扩增的方法,LAMP技术可以在短时间内获得大量的目标DNA,且具有很高的扩增特异性和灵敏度,可以用于分子生物学、诊断医学和病原检测等领域。
引物设计:引物设计是LAMP技术成功应用的重要因素之一、LAMP技术使用了4个单链引物,包括2个外端引物(forward outer primer,F3和reverse outer primer,B3)和2个内端引物(forward inner primer,FIP和reverse inner primer,BIP)。
外端引物负责扩增DNA的初始段,内端引物负责扩增DNA的中间段。
在引物设计中,需要注意以下几个方面:1.引物的特异性:要求引物能够有高度特异地结合到目标DNA的区域,确保扩增的目标是准确的;2.引物的长度和碱基组成:引物的长度通常为20-24个碱基,碱基组成要尽量避免重复序列和形成组内结构,以保证扩增效率和特异性;3.引物的位置和方向:合理选择引物的位置和方向,以确保扩增产物的特异性和有效性;4.引物的浓度:引物的浓度需要进行优化,以获得最佳的扩增效果。
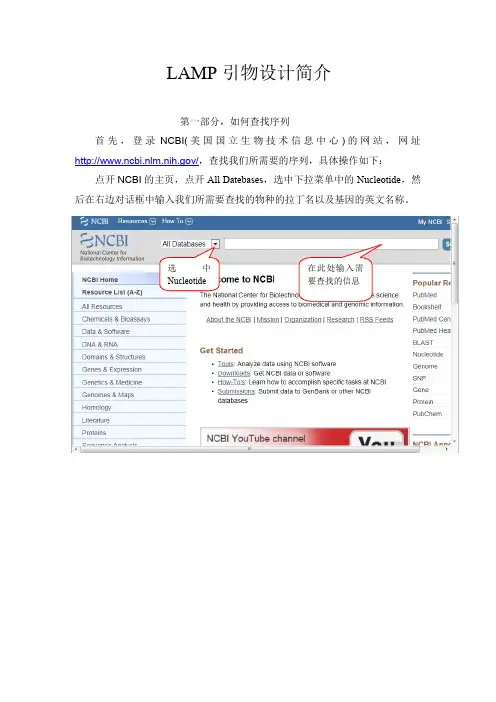
选中Nucleotide 在此处输入需要查找的信息LAMP引物设计简介第一部分,如何查找序列首先,登录NCBI(美国国立生物技术信息中心)的网站,网址/,查找我们所需要的序列,具体操作如下:点开NCBI的主页,点开All Datebases,选中下拉菜单中的Nucleotide,然后在右边对话框中输入我们所需要查找的物种的拉丁名以及基因的英文名称。
比如说我要查找大肠杆菌的16S rDNA的序列,那么只需要将大肠杆菌的拉丁名Escherichia coli以及16S rDNA输入即可,如下图所示,然后点击search。
如果NCBI的GenBank数据库有这些序列,那么我们就可以找到我们所需要的序列了。
当然,数据库中可能会出现很多相关的序列,这时我们需要自己慢慢查找自己需要的序列信息,如下图所示:接下来我们就浏览这些信息,然后将我们需要的信息复制下来,粘贴到TXT或者Word里。
这里我们示例选中的是第20条信息,当然你可以打开所有的你需要的信息。
如下图所示,我们可以得到基因的全部信息,包括来源、出自那片文献、核酸序列等等。
这样,我们的序列查找就完成了。
当然并不是所有物种的基因都可以在这里找到,在查不到的情况下,就需要我们自己通过PCR的方法将我们所需要的基因扩增出来,然后测序获得序列信息。
第二部分序列比对目前有一些分子生物学软件可以用于序列比对分析,像DNASTAR、ClustalW、MEGA等等,这里我们以MEGA5.0为例进行讲解。
首先,将目标序列和其它需要与其比对的序列放入一个文本文档中,以大肠杆菌的16S rDNA为例,格式如下图所示:打开MEGA5.0软件,点击Alignment的下拉菜单,选取Alignment的下拉菜单,选取Edit/Build Alignment,出现一个Alignment Editor的对话框,然后选择Creat a new alignment,点击OK。
出现下面的对话框,选择DNA软件就会出现下图的界面,单击Edit,选择下拉菜单中的Insert Sequence From File然后我们就把大肠杆菌的16S rDNA的序列及其他细菌的16S rDNA序列上传,出现下图的对话框,点击Alignment,选择下拉对话框的Align by ClustalW,对这个序列进行比对。

通过LAMP实现DNA的线性扩增一、LAMP 原理60-65℃是双链DNA复性及延伸的中间温度,DNA在65℃左右处于动态平衡状态。
因此,DNA在此温度下合成是可能的。
利用4种特异引物依靠一种高活性链置换DNA聚合酶。
使得链置换DNA合成在不停地自我循环。
扩增分两个阶段。
第1阶段为起始阶段,任何一个引物向双链DNA的互补部位进行碱基配对延伸时,另一条链就会解离,变成单链。
上游内部引物FIP的F2序列首先与模板F2c结合,在链置换型DNA聚合酶的作用下向前延伸启动链置换合成。
外部引物F3与模板F3c结合并延伸,置换出完整的FIP连接的互补单链。
FIP上的F1c与此单链上的Fl为互补结构。
自我碱基配对形成环状结构。
以此链为模板。
下游引物BIP与B3先后启动类似于FIP和F3的合成,形成哑铃状结构的单链。
迅速以3' 末端的Fl区段为起点。
以自身为模板,进行DNA合成延伸形成茎环状结构。
该结构是LAMP基因扩增循环的起始结构。
第2阶段是扩增循环阶段。
以茎环状结构为模板,FIP与茎环的F2c区结合。
开始链置换合成,解离出的单链核酸上也会形成环状结构。
迅速以3’末端的B1区段为起点,以自身为模板。
进行DNA合成延伸及链置换.形成长短不一的2条新茎环状结构的DNA,BIP引物上的B2与其杂交。
启动新一轮扩增。
且产物DNA长度增加一倍。
在反应体系中添加2条环状引物LF 和LB,它们也分别与茎环状结构结合启动链置换合成,周而复始。
扩增的最后产物是具有不同个数茎环结构、不同长度DNA的混合物。
且产物DNA为扩增靶序列的交替反向重复序列。
二、实验流程二、阶段实验及需要解决的问题(一) 实验准备阶段1、引物设计2、模板中引入合适的酶切位点3、不同梯度拷贝数模板的准备4、反应仪器的准备5、具有链置换活性的DNA聚合酶的选择6、扩增产物的检测方法7、扩增产物量的检测(二) 实验阶段1、反应体系的确定2、模板浓度的优化3、反应时间的优化4、扩增产物的检测5、以PCR的扩增作为对照三、具体实验方案(一) 引物的设计LAMP引物设计的在线网站(http://primerexplorer.jp/e/),只要导入靶基因就能自动生成成组引物。



LAMP引物设计LAMP(Loop-mediated isothermal amplification)是一种在等温条件下进行的核酸扩增技术。
与传统的PCR技术相比,LAMP技术具有操作简单、扩增速度快、特异性高以及不依赖于PCR仪等优点。
在LAMP技术中,引物的选择尤为关键。
合适的引物序列能够提高LAMP扩增的效率和特异性。
1.目标序列选择:首先根据所需检测的目标基因或序列,选择合适的目标序列。
该目标序列应该是特异的,并且在相关物种中高度保守。
2.引物设计:通常,LAMP反应需要4个引物,包括两个外部引物(F3和B3)、两个内部引物(FIP和BIP)以及一个环引物(LF和LB)。
FIP和BIP引物包含了一个共享的反向互补序列,以及两个非共享的序列。
外部引物和内部引物必须与目标序列匹配,并且具有适当的长度和GC含量。
3.引物序列分析:选择合适的引物序列后,使用生物信息学工具对其进行分析。
对于引物序列,可以使用BLAST等工具进行同源性比对,以确保其特异性。
4.引物序列优化:如果在初步设计的引物序列中存在问题(如二聚体、自相互作用等),则需要针对这些问题进行优化。
这可能包括引物序列的长度调整、GC含量的调整或引物序列的重新设计。
5.引物合成:最后,设计好的引物序列需要通过化学合成进行合成。
在合成过程中,引物的纯度和质量也需要进行检查,以确保引物的稳定性和有效性。
LAMP引物设计的关键是选择合适的引物序列。
合适的引物序列应该与目标序列完全匹配,并且能够提供高效的扩增,同时对非目标序列具有良好的特异性。
此外,引物序列的长度和GC含量也是重要的考虑因素。
过长或过短的引物序列可能导致扩增效率低下,而过高或过低的GC含量可能影响引物的稳定性和特异性。
总结起来,LAMP引物设计是一个复杂的过程,需要综合考虑引物序列的特异性、长度、GC含量等因素。
通过合理设计和优化,可以选择出高效、特异和敏感的引物序列,实现准确的荧光定量PCR检测。
环介导等温扩增引物设计环介导等温扩增(Loop-mediated isothermal amplification,LAMP)是一种常用的核酸扩增技术,其最大的优势是可以不受限地在恒定温度下进行反应。
为了实现快速、准确、可靠的DNA扩增,合理的引物设计是至关重要的。
下面就环介导等温扩增引物的设计进行详细介绍:一、引物长度引物长度对扩增效率和特异性有很大影响。
通常建议引物长度在18-24bp之间,太短的引物会导致扩增效率低下,太长的引物则容易出现多个峰或非特异性扩增。
因此,在长度的选择上需要平衡扩增效率和特异性,一般来说,20bp左右的引物是比较适合的选择。
二、引物序列选择引物序列选择需要保证具有较低的自身互补性和互补性。
通常情况下,引物中应避免出现多个重复序列,避免引物间形成二级结构或者嵌套回路,可以通过NCBI的Primer BLAST或DNAMAN等软件进行序列比对,评估引物设计的准确性。
三、环结构设计与PCR引物不同,LAMP引物一般会在5’端和3’端添加的特殊序列(FIP和BIP)和环的序列。
它们是设计LAMP的关键部分,必须确保它们的特异性和互补性。
FIP和BIP序列需要匹配到目标序列上,而F1c和B1c环序列主要用于在反应过程中拉开靶DNA,为DST和DHT提供起始端。
四、引物浓度引物浓度对LAMP反应影响也很大,它的作用是影响LAMP反应的灵敏度和特异性。
在引物浓度过高时,会出现非特异性扩增和产生大量的非特异性产物;而在引物浓度过低时,则可能影响LAMP反应的灵敏度。
因此,在引物浓度的选择上也需要进行优化。
综上所述,环介导等温扩增引物设计是一项较为关键的技术,涉及到引物长度、序列选择、环结构设计和引物浓度等方面。
在实际操作中,需要根据样品特点和实验要求来进行引物设计。
同时,还需要结合实验优化引物浓度以及其他实验条件,以获得高灵敏度和高特异性的扩增结果。
lamp环介导等温扩增实验步骤嘿,咱今儿个就来聊聊 lamp 环介导等温扩增实验那些事儿!你知道吗,做这个实验就像是一场奇妙的冒险!首先呢,咱得把各种“宝贝”准备齐全咯,这就好比战士上战场得有趁手的兵器呀!各种试剂啦、样本啦,一个都不能少。
然后呢,就该设计引物啦!这引物就像是给实验指明方向的指南针,可得精心设计,不然可就容易跑偏啦!选好合适的引物,那就是成功的一大半啦!接下来,就是把这些东西都放在一起,像是在搭积木一样,小心翼翼地搭建起我们的实验“大厦”。
反应体系的配制可不能马虎,一点点的差错都可能导致结果不理想哦。
配好体系后,就把它放进那个神奇的等温扩增仪器里啦,这仪器就像是一个魔法盒子,能让反应在合适的温度下悄悄进行。
在这个过程中,你可得有点耐心,就像等待花儿慢慢开放一样。
可别着急,着急可吃不了热豆腐哟!时间一点点过去,反应也在悄悄进行着。
你想象一下,那些小小的分子就像一群小精灵,在里面欢快地跳舞、变化。
等反应结束后,就该看看我们的成果啦!这时候就像是开奖一样,心里还有点小紧张呢。
看看是不是得到了我们想要的结果呀。
哎呀,这整个过程不就像是一场有趣的游戏嘛!只不过这个游戏需要我们特别认真、特别专注地去玩。
做lamp 环介导等温扩增实验,真的是一件既神奇又有趣的事情呀!它能让我们看到那些平时看不到的微小世界的变化,让我们对生命、对科学有了更深的理解和认识。
所以呀,如果你对这个实验感兴趣,那就大胆地去尝试吧!别怕困难,别怕失败,每一次的尝试都是一次成长,都是向成功迈进的一步呀!相信自己,你一定能在这个实验中找到属于自己的乐趣和收获!。
环介导等温扩增(LAMP)引物设计LAMP引物设计关键点:Tm值,引物末端稳定性,GC含量,二级结构,引物间距1.Tm值引物Tm值采用Nearest-Neighbor Method进行预测,该法所预测的值可能是最为接近真实情况。
通常情况下,Tm值会受实验条件所影响,例如盐浓度、寡核苷酸浓度等,因此需在实验条件下对Tm值进行预测。
●寡核苷酸0.1 μM;●Na+ 50 mM;●Mg2+ 4 mM;各个引物的最终Tm值需尽量满足以下条件:●F1c/B1c = 64-66 °C;●F2/B2、F3、B3 = 59-61 °C;●LoopF/LoopB = 64-66 °C;2.引物末端稳定性引物的末端是DNA复制的起点,因此需要一定的稳定性。
(通常情况下,引物需要一个稳定的5’端和一个相对不稳定的3’端,自由能绝对值越高,则形成的DNA双链越稳定,即越容易退火。
)●F2/B2,F3/B3,LoopF/LoopB的3’末端自由能≤-4 kcal/mol;●F1c/B1c的5’末端自由能≤-4 kcal/mol;此处的末端自由能指末端6个碱基的自由能。
3.GC含量引物的GC含量尽量在40-65%。
50-60%的GC含量可能会有更好效果。
4.二级结构需确保引物不会形成二级结构,例如二聚体,发卡结构。
5.引物间距●F2的5’末端到B2的5’末端约为120-160 bp;●F2的5’末端到F1c的5’末端约为40-60 bp;●F3的3’末端到F2的5’末端约为0-60 bp;1.上传序列打开PrimerExplorer V4网站:http://primerexplorer.jp/elamp4.0. 0/index.html上传所选的DNA特异序列(序列长度需小于2000 bp),参数选择Automatic Judgment,然后点击Primer Design。
系统自动对所上传的序列进行分析:●GC% ≤ 45%,则判定为AT Rich;●45% < GC% < 60%,则判定为正常;●GC% ≥ 60,则判定为GC Rich;不同情况对引物的要求不一。
核酸环介导等温扩增技术(LAMP)引物设计与实例Time:2009-12-07 PM 14:52 Author:bioer Hits: 1459 times 烟头整理
LAMP的特点
LAMP与以往的核酸扩增方法相比具有如下优点:
(1)操作简单
LAMP核酸扩增是在等温条件下进行,对于中小医院只需要水浴锅即可,产物检测用肉眼观察或浊度仪检测沉淀浊度即可判断。
对于RNA的扩增只需要在反应体系中加入逆转录酶就可同步进行(RT-LAMP),不需要特殊的试剂及仪器。
(2)快速高效
因为不需要预先的双链DNA热变性,避免了温度循环而造成的时间损失。
核酸扩增在l h内均可完成,添加环状引物后时间可以节省1/2,多数情况在20-30 rain均可检测到扩增产物。
且产物可以扩增至109倍,达0.5 mg/mL。
应用专门的浊度仪可以达到实时定量检测。
(3)高特异性
由于是针对靶序列6个区域设计的4种特异性引物。
6个区域中任何区域与引物不匹配均不能进行核酸扩增。
故其特异性极高。
(4)高灵敏度
对于病毒扩增模板可达几个拷贝,比PCR高出数量级的差异。
缺点:
由于LAMP扩增是链置换合成,靶序列长度最好在300 bp以内。
>500 bp则较难扩增。
故不能进行长链DNA的扩增。
由于灵敏度高。
极易受到污染而产生假阳性结果。
故要特别注意严谨操作,以及在产物的回收鉴定、克隆、单链分离方面均逊色于传统的PCR 方法。
引物设计实例
LAMP引物设计的在线网站(http://primerexplorer.jp/e/),只要导入靶基因就能自动生成成组引物。
以某一微生物的鞭毛基因为例讲解一下LAMP引物设计的过程:
首先单击浏览按钮选择靶基因序列文件,靶序列默认的是小于22 kbp。
支持三个类型的文件,普通文本格式(仅含序列), FASTA格式和GenBank 格式文件。
第二,从下面三个选项中选择定参数设定(引物设计条件)条件。
基于GC含量的自动判断, 起始的参数是特定的:如果GC含量小于或等于45%.,则选取AT丰度高的区,如果GC含量高于60%,则选取GC丰度高的区,其它情况是标准设定状态。
设计合适的引物是进行LAMP反应的关键,通过考虑碱基组成,GC含量,二级结构的形成,Tm值等因素可以通过Pimer Explore)(一种专门设计LAMP引物的软件)来设计
LAMP反应的引物。
在进行LAMP引物设计的时候有以下几个关键点需要考虑:
1. 引物之间的距离
F2区段的5’端到B2区段的5’端(LAMP反应扩增的区域)之间的距离建议是120~180bp。
F3区段的3’端到F2区段的5’端之间的距离是0~20bp(同理B2和B3之间的距离是0~20bp)。
F2区段的5’端到F1区段的5f端(形成环的部分)之间的距离是40~60bp。
2. 引物的Tm值
引物的Tm值采用近邻分析法(the nearest-neighbor method)来计算,这种方法是目前认为计算值最接近真实值的一种方法。
计算Tm值的时候会受到盐浓度(salt concentration)、寡核苷酸的浓度等实验条件的影响,所以最好是在确定的实验条件下来计算Tm值。
例如:寡核苷酸的浓度为0.1µmol,钠离子的浓度是50mM,镁离子的浓度4 mM等。
Tm(退火温度=△h*1000/[△S+Rln(C/4)]-273.15+16.6log[Na+]
式中,Tm为退火温度,℃;为摩尔气体常数,1. 987ca1/℃•mol ;△H为焓变;△s 为熵变;C为寡聚核苷酸的浓度;[Na+]为钠离子浓度。
对于GC含量正常或是GC含量富集的引物Tm值为60~65℃,而对于AT富集的引物Tm值为55~60℃。
在设计引物的时候,F1c和B1c的Tm值大概是65℃(64~66℃), F2,B2, B31的Tm值大概是60℃(59~61℃)。
3. 引物末端的稳定性
引物的末端作为DNA合成的起点必须有一定的稳定性,自由能改变值(△G)是指反应物的自由能与产物的自由能之差,反应朝着自由能减小的方向运行。
引物和目的基因之间的退火反应是一个动态平衡的反应,自由能改变值(△G)越小,引物与模板之间的退火反应越容易发生。
一般在进行引物设计的时候,F2/B2\F3/B3的3’端和F1 c/B1c的5’端的自由能改变值小于或等于-4Kcal/mol。
F1C的5’端扩增以后相当于F1的3’端,所以它的稳定性很重要。
4. GC含量
引物在设计的时候使其GC含量介于40%~65%之间,但是当引物的GC含量介于50%~60%时,引物的质量相对好一些。
5. 二级结构
引物在设计的时候要防止形成二级结构,这一点是十分重要的,特别是内引物。
本文来源于:生物问问博客,原文地址:/html/346.html。