引物设计1
- 格式:doc
- 大小:51.00 KB
- 文档页数:5

引物设计原理引物设计是一种在分子生物学中使用的技术,它可以用来控制基因的表达或用于获取某些基因组的一部分分子序列。
因此,引物设计的准确性和特异性非常重要,它可以满足分子生物学研究对高质量特异性DNA序列的需求。
引物设计原理是引物设计的基础,它涉及到基因组学、分子生物学、基因工程等科学领域,是研究引物设计的思路和方法标准。
分子生物学中的引物设计是基因组学研究的重要组成部分,通过建立特异性的引物,可以提高基因表达的特异性和准确性,从而加快对基因的研究和理解。
因此,引物设计的原理和方法是指其能够实现的标准,也是指DNA、RNA以及含有引物序列的模板的分子结构和表达特异性。
引物设计原理的基础是基因组学研究,由基因组学所获得的结果有助于分子生物学研究,特别是在揭示基因的功能和调控机制上。
利用基因组学获取的序列信息,可以设计具有特定功能的引物,如在载体上的引物序列设计、基因的PCR扩增、连锁反应以及基因编辑,都能够从引物设计获得有效的特异性和准确性。
生物信息学技术经常被用于引物设计,通过运用相关的软件和算法,结合基因组序列和已知的信息,可以进行引物设计。
这些软件有很多种,如OptiMAGE、OligoPerfect、PrimerSelect和GeneRunner 等,它们可以提供一些方便的工具来实现特定的引物设计。
此外,引物设计也应考虑到合成能力,即根据特定条件确定的引物的长度、GC含量和结构,以确保引物的有效性和可生物合成性。
一般而言,引物的最佳长度为18-25个核苷酸,其中包含一个或多个T节段,以简化引物合成过程。
引物的GC含量应保持在30-70%之间,并且不应含有太多的碱基重复(优先选择三个及以下的碱基重复),这有助于避免引物的自反应。
最后,引物设计应考虑引物的热稳定性,即引物在特定条件下的稳定性,引物的Tm值(融合温度)应保持在55-65℃,从而保证引物的有效性。
对于一号引物(环状),Tm值应高于3’端,以减少反应体系的复杂性;对于二号引物(键合),Tm值应与一号引物相同或略低,以减少反应体系的复杂性。

引物设计基本原则引物设计是指在引导读者进入文章主题或者吸引读者注意力的过程中,通过恰当选择和运用引言来达到预期效果的设计过程。
引物的设计不仅能够引导读者入文、稳定读者情绪,还能提高文章的可读性和吸引力。
下面是引物设计的几个基本原则:第一,引物设计要与文章主题相关。
引物在一定程度上是文章的立意之所在,它几乎决定了读者是否会继续阅读。
因此,引物与文章主题要密切相关,能够准确传达出文章的核心思想和信息。
引物与文章主题的相关性是一个引物是否成功吸引读者的关键因素。
第二,引物设计要具有足够的吸引力。
引物要能够立刻吸引读者的注意力,使其对文章产生兴趣。
可以通过新颖的观点、有趣的故事、强烈的情感或者悬疑的描述等方式来增加引物的吸引力。
同时,引物要注意与读者的背景和兴趣相关,尽量选取读者感兴趣的内容,以便更好地吸引读者。
第三,引物设计要具有引导性。
引物不仅要吸引读者,还要能够引导读者进入文章的主题或内容。
可以通过提出问题、引用权威观点、展示案例等方式来引导读者产生思考或者兴趣,从而使其进一步阅读下去。
引物的引导性在一定程度上决定了读者是否会对文章产生深入的理解和共鸣。
第四,引物设计要与写作目的相一致。
不同的写作目的对引物的要求也不同。
如果是科普文章,引物可以采用生活常识或者实验事实等方式;如果是讲故事的文章,可以使用引人入胜的故事片段或者引用名人的话语等方式。
引物与写作目的的一致性能够更好地达到写作的目的,并且使读者在读者的过程中不会产生迷惑或者矛盾感。
第五,引物设计要注意其长度和位置。
引物的长度要适当,过长容易让读者感到疲惫,过短则不足以吸引读者的注意力。
同时,引物一般放在文章的开头或者段落的开头,这样能够更好地引导读者进入文章,提高文章的连贯性和阅读流畅性。
综上所述,引物设计是一项重要的写作技巧,它能够引导读者进入文章主题,并且提高文章的可读性和吸引力。
在引物设计时,需要注意与文章主题相关、具有足够的吸引力、具有引导性、与写作目的相一致,以及注意引物长度和位置等方面的要求。


高中生物引物设计教案教学目标:
1. 了解引物在生物学研究中的重要性和应用;
2. 学习引物的设计原理和方法;
3. 能够根据实验需要设计合适的引物。
教学重点和难点:
重点:引物的设计原理和方法;
难点:引物的设计实例分析。
教学准备:
1. 讲师准备好课件和教学材料;
2. 学生准备好笔记本和课堂笔记;
3. 实验室准备好实验器材和试剂。
教学流程:
一、引言(5分钟)
1. 介绍引物在生物学研究中的重要性和应用;
2. 引导学生思考引物设计的意义。
二、引物的设计原理和方法(15分钟)
1. 介绍引物的概念和分类;
2. 讲解引物的设计原则和方法;
3. 展示引物设计实例,引导学生理解。
三、设计引物实践(20分钟)
1. 将学生分成小组,每组设计一个引物;
2. 讲师指导学生进行引物设计,解答问题。
四、引物设计实例分析(15分钟)
1. 学生汇报引物设计结果;
2. 讲师分析引物设计的优缺点,引导学生总结经验。
五、课堂讨论与总结(10分钟)
1. 学生分享设计引物的经验和体会;
2. 讲师总结课堂内容,引导学生思考。
六、作业布置(5分钟)
布置引物设计实践作业,要求学生设计一个引物并写一个简要报告。
教学评估:
学生完成作业并提交报告,评价引物设计的合理性和完整性。
教学延伸:
引导学生利用引物设计实现生物学实验和研究,培养其动手实践和创新能力。

引物设计(特别有用,良心推荐)引物篇1.引物是如何合成的?目前引物合成基本采用固相亚磷酰胺三酯法。
DNA合成仪有很多种, 主要都是由ABI/PE 公司生产,无论采用什么机器合成,合成的原理都相同,主要差别在于合成产率的高低,试剂消耗量的不同和单个循环用时的多少。
亚磷酰胺三酯法合成DNA片段,具有高效、快速的偶联以及起始反应物比较稳定的特点。
亚磷酰胺三酯法是将DNA固定在固相载体上完成DNA链的合成的,合成的方向是由待合成引物的3'端向5'端合成的,相邻的核苷酸通过3'→5'磷酸二酯键连接。
第一步是将预先连接在固相载体CPG上的活性基团被保护的核苷酸与三氯乙酸反应,脱去其5'-羟基的保护基团DMT,获得游离的5'-羟基;第二步,合成DNA的原料,亚磷酰胺保护核苷酸单体,与活化剂四氮唑混合,得到核苷亚磷酸活化中间体,它的3'端被活化,5'-羟基仍然被DMT保护,与溶液中游离的5'-羟基发生缩合反应。
第三步,带帽(capping)反应,缩合反应中可能有极少数5'-羟基没有参加反应(少于2%),用乙酸酐和1-甲基咪唑终止其后继续发生反应,这种短片段可以在纯化时分离掉。
第四步,在氧化剂碘的作用下,亚磷酰形式转变为更稳定的磷酸三酯。
经过以上四个步骤,一个脱氧核苷酸被连接到固相载体的核苷酸上。
再以三氯乙酸脱去它的5'-羟基上的保护基团DMT,重复以上步骤,直到所有要求合成的碱基被接上去。
合成过程中可以观察TCA处理阶段的颜色判定合成效率。
通过氨水高温处理,连接在CPG上的引物被切下来,通过OPC, PAGE等手段纯化引物,成品引物用C18浓缩,脱盐,沉淀。
沉淀后的引物用水悬浮,测定OD260定量,根据定单要求分装。
2.引物纯化方式有哪些,如何选择?◆C18柱脱盐:有人称其为简易反相柱,它对DNA有特异性的吸附,可以被有机溶解洗脱,但不会被水洗脱,所以能有效地去除盐分。

我收集的引物设计原则和方法~~~呵呵寡核苷酸的优化设计郑仲承( 中国科学院上海生命科学院生物化学和细胞生物学研究所,上海 200031 )在核酸分子杂交、DNA序列测定和通过PCR放大DNA片段等实验中,都需要使用寡核苷酸作为探针或引物,而对这些反应的质量起最重要影响作用的,就是这些寡核苷酸探针或引物。
用优化的寡核苷酸进行实验能够很快得到好的结果,而用不够合适的寡核苷酸时,常常得出似是而非的结果,不仅大大增加了后续实验的工作量,还可能一无所获。
怎样优化设计寡核苷酸呢?至少有下列几个方面的问题需要考虑。
1. 估测可能形成的DNA或RNA双链的稳定性寡核苷酸,无论是DNA的或者RNA的,都有形成双链结构的潜在可能性,正如下面反复提到的,这种结构对寡核苷酸的作用有很大影响。
所以,预测这种结构的稳定性对设计和优化寡核苷酸就很重要。
在一个双链结构中,碱基对的相对稳定性是由其邻近碱基决定的。
在热动力学中,这样的性质以双链形成时的自由能(ΔG)来表示。
现在,大多采用 Breslauer 等人提出的,以最接近的相邻核苷酸的动力学数值(自由能)来预测双链稳定性的方法。
为简化起见,所有的计算都在25 ℃条件下进行。
此时,最接近的相邻核苷酸的自由能是:此主题相关图片如下:ΔG(kcal/mol)例如,双链d(ACGG/CCGT)的ΔG是:ΔG(ACGG)=ΔG(AC)+ΔG(CG)+ΔG(GG)=-(1.3+3.6+3.1)=-8.0 kcal/mol此计算方法特别适用于测定其3′末端会形成双链的引物的相容性。
也可以用来计算发夹环结构的ΔG。
不过,这时需要根据环区内核苷酸的数量添加一定的数值。
如3个核苷酸时为5.2 kcal/mol;4个时为4.5;5个为4.4;6个是4.3;7和8个为4.1 kcal/mo1。
2. 选择引物的一般规则设计和选择引物时有5个要素必需注意。
2.1 引物的3′末端不互补引物的3′末端一定不能有很大的互补性,因为它们的互补会形成引物二聚体,这就会带来很大的问题,例如合成出非专一的产物,极大地减少所期望产物的得量。


引物设计程序(利用primer premier 5.0软件)1.从程序中打开primer premier 5.0软件,并将目的基因调出(可利用file 菜单中的子菜单,利用new菜单复制粘贴选择新的基因片段,或利用open菜单从程序中找出目的基因)。
2、设计前的准备工作1)进行引物设计时,一般有两种引物:一是不带酶切位点的引物;二是带酶切位点的引物。
一般从基因组中进行PCR扩增目的基因、而且不知道该目的基因扩增的难易程度时,建议设计不带酶切位点的引物。
如已知目的基因比较容易从基因组中扩增,则可以直接带酶切位点。
或如从质粒上扩增目的基因,则可以直接带酶切位点。
2)选择限制性酶切位点时,需考虑实验室已有的载体材料,即利用实验室的载体工具进行引物设计。
3)利用Primer中的enzyme菜单,判断目的基因中是否含有所选载体中可被限制性内切酶识别的位点;若存在,则这些酶不可用。
4)针对一个具体的载体确定可用的限制性酶种类后,还需考虑以下几方面:I、所选的两个酶的位置最好不要距离太近(特别是相邻的),否则加重错误引发的机率或是切割不完全;II、同时选择的两种酶最好有相同的Buffer(不同公司的产品,其Buffer会有所不一样,一般以宝生物公司的产品为准),以提高酶切效率、减少劳动量;III、尽可能地用具有粘性末端的限制性内切酶;IV、尽可能地减少载体上的基因序列连入目的基因上,即尽可能地减少工程蛋白中外来氨基酸的数量;V、利用蛋白纯化标签的位置是目的蛋白的前面还是后面,或前后均具有。
5)确定所用的限制性酶种类后,还需额外分析一下酶切位点的粘性末端是否会配对,如Sal I与Xho I酶切后的粘性末端能相互配对,则不可同时使用,否则导致载体自连。
6)确定所用的限制性酶种类后(结果记录到引物设计文档中),还需特别注意前面酶切位点的密码子阅读框架,尤其是后面一个酶切位点的。
如扩增的基因带终止密码子,则只需考虑前面的即可,后面的不需考虑;如不带终止密码子,则更需要特别注意后面酶切位点的阅读框架。
MicroRNAs (miRNAs) 是一类重要的非编码单链小RNA,主要调节蛋白质的翻译。
通常长度为21-23个核苷酸, 具有高度保守性,已成为疾病、组织重构和细胞分化等研究的热点。
因含量较低,miRNAs较难分离;一旦分离,这些18-24nt的RNAs经过纯化,加上5'和3'端接头,RT-PCR后才可克隆至载体并测序。
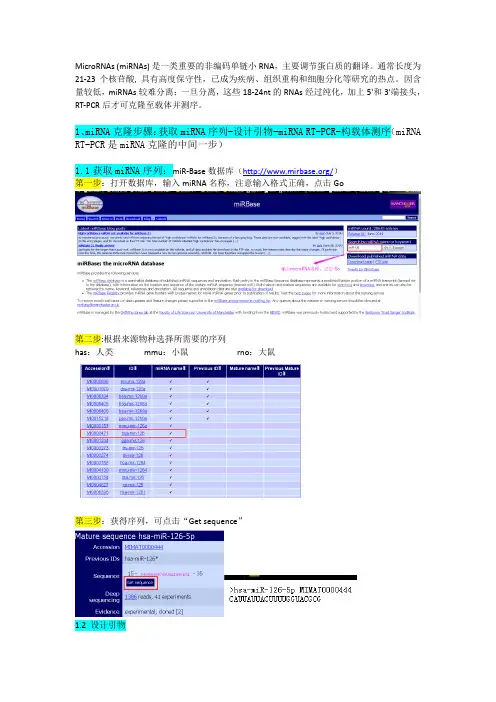
1、miRNA克隆步骤:获取miRNA序列-设计引物-miRNA RT-PCR-构载体测序(miRNA RT-PCR是miRNA克隆的中间一步)1.1获取miRNA序列:miR-Base数据库(/)第一步:打开数据库,输入miRNA名称,注意输入格式正确,点击Go第二步:根据来源物种选择所需要的序列has:人类mmu:小鼠rno:大鼠第三步:获得序列,可点击“Get sequence”1.2 设计引物以mmu-miR-30d-5p为例:1、获取miRNA成熟序列:UGUAAACAUCCCCGACUGGAAG将U全部改成T:TGTAAACATCCCCGACTGGAAG2、设计茎环引物,茎环引物由两部分构成,一部分是通用的序列(通用序列有很多种,这里只列举一种),如:CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGC另外一部分是针对某个miRNA特定序列,这一部分的序列是6个碱基,我们设计的是miR-30d-5p,从它的3‘端,开始数取6-8个序列,即GAAGGTCA,取它的互补序列,即为CTTCCAGT,将这段序列与茎环引物的通用序列,结合,就成了miR-30d-5p的反转录引物,如下所示:CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGCCTTCCAGT这个引物是在反转录的时候使用,它在反转录过程中的示意图如下所示:1.3构载体以自行设计的带有茎环结构的特异性反转录引物代替反转录试剂盒中的通用引物,按照试剂盒说明书,去除基因组DNA 污染后,合成cDNA 的第一条链。

定量pcr引物设计的详细步骤宝子,来给你唠唠定量PCR引物设计的步骤哈。
一、确定目的基因序列。
你得先知道你要研究的目的基因是啥呀。
这就好比你要找一个小伙伴,你得先知道他长啥样。
你可以去一些基因数据库,像NCBI这种超厉害的地方,找到你要的那个基因的序列。
这个序列就像是这个小伙伴的身份证号码,是独一无二的哦。
二、引物设计软件选择。
有好多好用的引物设计软件呢。
比如说Primer Premier,这个就像一个贴心的小助手。
你把目的基因序列输进去,它就能开始帮你设计引物啦。
还有Beacon Designer也很不错哦。
这些软件就像是魔法棒,能在基因的海洋里给你捞出合适的引物来。
三、引物设计参数设置。
这一步很关键哦。
一般来说呢,引物的长度大概在18 - 25个碱基左右就好啦。
太短了就像小短腿,不太稳;太长了又像大长脚,容易出问题。
还有引物的GC含量,最好在40% - 60%之间。
这就像是一个黄金比例,能让引物和模板结合得更牢。
另外,引物自身不能有太多互补的地方,不然它自己就抱成一团,不跟模板好好玩啦。
四、特异性检查。
设计好引物之后,可不能就这么不管了。
得检查一下它的特异性呢。
这就像是给引物做个忠诚度测试。
你可以用BLAST这个工具,把你设计的引物序列放进去,看看它是不是只和你的目的基因结合,要是和其他乱七八糟的基因也有结合,那可不行,就像找错小伙伴啦。
五、引物合成。
如果前面的步骤都没问题啦,那就要把引物合成出来。
这时候就可以找专门的公司啦,就像把设计图交给工匠,让他们做出真正的成品。
合成好的引物拿回来,就可以开始你的定量PCR之旅啦。
宝子,按照这些步骤来,设计定量PCR引物就不是啥难事啦。
加油哦! 。

primer 引物设计得分引物设计是分子生物学领域中一项重要的技术,它在PCR、DNA测序和基因克隆等实验中起着关键作用。
本文将从primer引物设计的原则、方法和应用等方面进行介绍。
引物设计的原则主要包括引物长度、碱基组成、GC含量和互补性等。
首先,引物长度一般在18-30个碱基对之间,过长的引物可能导致非特异性扩增,而过短的引物则可能无法特异性地结合靶序列。
其次,引物的碱基组成需要考虑到AT和GC的比例,过高或过低的GC含量都会影响引物的特异性。
此外,引物的互补性也是一个重要的考虑因素,引物之间应避免形成二聚体或内部自身互补结构。
在引物设计的方法方面,可以利用计算机软件进行辅助设计。
常用的软件包括Primer3、OligoAnalyzer和Primer-BLAST等。
这些软件可以根据用户输入的靶序列信息自动生成符合设计原则的引物序列,并给出引物的理论特异性评分。
设计完成后,还需要进行引物序列的BLAST比对,以确保引物只特异性地结合到目标序列上。
引物设计在分子生物学实验中有着广泛的应用。
在PCR反应中,引物的设计直接影响到扩增产物的特异性和效率。
在DNA测序中,引物的设计影响到测序反应的准确性和可靠性。
在基因克隆中,引物的设计决定着目标片段的扩增和连接效果。
因此,合理设计引物对于实验结果的可靠性和准确性至关重要。
总结起来,引物设计是分子生物学实验中不可或缺的一环。
通过遵循引物设计的原则和方法,科学家们可以设计出特异性好、效率高的引物,从而保证实验结果的准确性和可靠性。
引物设计的技术不断发展,将为分子生物学领域的研究提供更多可能性和机会。
引物的设计原则引物的设计原则是指在设计引物(primers)时需要考虑的一系列原则和规则,以确保引物在PCR(聚合酶链式反应)等实验中的准确性和有效性。
在分子生物学领域,引物是一种短小的DNA或RNA 片段,用于在DNA复制或扩增的过程中识别特定的DNA序列。
因此,在设计引物时需要遵循一定的原则,以确保实验结果的准确性和可靠性。
引物的设计应避免与非靶向DNA序列发生杂交。
这意味着引物的序列应具有足够的特异性,以确保其只与目标DNA序列结合,而不与其他非靶向的DNA序列结合。
为了实现这一点,设计引物时需要避免序列内部的重复性和互补性,以减少引物与非靶向序列的结合。
引物的设计应考虑引物的长度和GC含量。
引物的长度通常在18-25个碱基对之间,过长或过短的引物都会影响PCR的效果。
此外,引物的GC含量应在40-60%之间,过高或过低的GC含量也会影响引物的特异性和效率。
引物的设计还需要避免形成二聚体或自身结合。
引物之间不能形成二聚体,即引物与引物之间的相互结合,也不能自身形成二聚体,即引物的两端不能互相结合。
否则会影响引物的扩增效率和准确性。
引物的设计还需考虑引物的末端修饰。
引物的末端应避免含有碱基修饰或非特异性的序列,以确保引物的特异性和有效性。
在实际的引物设计过程中,通常会使用专门的引物设计软件来帮助设计符合上述原则的引物。
这些软件会根据目标DNA序列的特性和实验条件自动生成符合要求的引物序列,从而提高引物设计的效率和准确性。
总的来说,引物的设计原则包括避免与非靶向DNA序列发生杂交、考虑引物的长度和GC含量、避免形成二聚体或自身结合、以及考虑引物的末端修饰等方面。
遵循这些原则可以有效地设计出高效、特异性的引物,从而确保实验结果的准确性和可靠性。
引物设计原则
引物在分子生物学和遗传学领域中扮演着至关重要的角色。
引物是一种短小的DNA或RNA序列,用于识别和扩增目标DNA的特定区域。
在进行PCR、测序、杂交等实验中,引物的设计至关重要。
下面将介绍引物设计的基本原则。
引物设计的基本原则
1.引物长度
引物的长度通常在18-25个碱基对之间。
较长的引物可以提高特异性,但也增加了非特异性杂交的风险。
2.GC含量
引物的GC含量应在40-60%之间,过高或过低的GC含量都会降低引物的稳定性。
在引物设计时需要注意平衡GC含量,以确保引物的性能。
3.互补性
引物应该是互补的,即引物与靶标DNA的序列互补匹配。
在引物设计时,需要确保引物序列与靶标序列的互补性,以确保引物能够特异性地结合目标DNA。
4.避免重复序列和剪切位点
引物设计时需要避免引入重复序列和酶切位点,以防止引物在非特定区域结合或被酶降解。
5.避免自身和互相形成二聚体
引物设计时需要避免引物自身或与其他引物形成二聚体,以免影响引物扩增效率。
6.核苷酸组成
引物中尽量避免包含多个连续的相同核苷酸,例如连续的A或T会导致引物的非特异性结合。
7.无结构埃许曼结构
引物设计时需要避免引入结构埃许曼结构,以确保引物的特异性和扩增效率。
引物的设计是实验成功的关键因素之一,合理的引物设计可以提高实验结果的准确性和可靠性。
在进行引物设计时,需要根据目标序列的特点结合以上原则进行设计,从而确保引物的性能和效率。
摘要通过Beacon Designer v.8和Primers Blast 软件设计引物,使用PCR 扩增技术和琼脂糖凝胶电泳检测来筛选出目的基因片段,对目的片段序列进行Blast 比对,筛选出适合于鸭茅(L.)RPB1引物序列。
试验结果显示:设计的10对引物中有2对引物扩增条带唯一且清晰,扩增产物经比对后验证其为目的基因。
筛选出的RPB1引物对鸭茅物种起源进化及种质资源利用的研究具有重要的意义。
关键词鸭茅;RPB1;引物设计;PCR鸭茅(L.),又名果园草(Or -chardgrass )或鸡脚草(Cocksfoot ),是禾本科(Poac -cae )鸭茅属()的唯一物种[1]。
鸭茅的耐阴性较强、叶产量高[2],是畜牧业发展和改良环境的重要牧草草种之一,在青贮饲料的制备[3]、草地恢复[4]等方面广泛应用。
野生鸭茅的种质资源丰富,遗传多样性较高,是进行育种及物种保护的重要基因材料来源[2]。
目前,国内外对于鸭茅的起源及进化的研究集中于地理学数据和形态学标记[5]、分子标记[6]以及叶绿体和ITS 序列[7]数据之上。
随着鸭茅生存环境的变化,形态学分类和分子标记技术受外界影响较大,对物种分类会造成一定的误差[8]。
单拷贝核基因属于直系同源基因,继承双亲的遗传信息,没有核苷酸偏向性和内部重复,携带大量的信息位点,进化速率中等,更适合用于对物种进行系统发育分析[9]。
武泼泼等[10]基于单拷贝核基因1基因序列对中国短柄草种族进行分类研究,为其系统进化提供可靠证据。
胡晓艳等[11]利用单拷贝核基因对酸枣的遗传多样性进行研究,证明单拷贝适用于物种遗传多样性应用,这为后续的酸枣种质资源保护及利用奠定了基础。
刘舒宇等[12]通过设计并筛选出杨柳科单拷贝核基因引物,并对4个属的代表物种进行系统发育分析,为进一步深入了解杨柳科植物的遗传多样性提供数据支撑。
Joly 等[13]开发了单拷贝核基因作为核基因标记,重构了北美蔷薇属植物的多倍体起源。
引物设计注意事项嘿,朋友们!咱今儿来聊聊引物设计这档子事儿。
这可真是好比盖房子打地基,重要得很呐!你想啊,引物就像是一把钥匙,得合适才能把那扇基因的大门给打开呀。
要是引物设计得不好,那不就跟拿错钥匙开不了门一样嘛!那可就麻烦啦。
咱先说这特异性,这可是重中之重啊!你总不能让你的引物到处乱开锁吧,那不乱套啦!一定要保证它就对着咱想要的那个目标去,别瞎勾搭别的地方。
这就好比你去超市买东西,得认准了你要的那个牌子,别拿错啦。
还有长度,太短了可不行,那不就没劲儿了嘛,就像小矮子够不着高处的东西一样。
太长了也麻烦,太笨重啦,反应起来也不灵活呀。
所以得找个合适的长度,不短不长刚刚好。
GC 含量也得注意哦!就像炒菜放盐,放多了咸得慌,放少了没味道。
GC 含量不合适,那引物可能就不好使啦。
退火温度也很关键呀!温度高了,引物结合不上;温度低了,又容易出现非特异性结合。
这就跟洗澡水似的,太热了烫得慌,太冷了又冻得直哆嗦,得找到那个舒服的温度才行。
设计引物的时候,可别马马虎虎的,得仔细琢磨。
你想想,要是盖房子的时候地基没打好,那房子能结实吗?咱得对自己的实验负责呀!有时候我就想,这引物设计真的就像走钢丝,得小心翼翼,一步都不能错。
一个不小心,可能整个实验就白费啦。
咱可不能小瞧了这引物设计,它可是关乎着整个实验的成败呢!就像一场比赛,引物就是那个决定胜负的关键球员。
所以呀,咱得好好对待它,多花点心思,多尝试几次,总能找到最合适的那一对引物。
总之,引物设计可不是闹着玩的,得认真对待,仔细考量。
只有这样,咱才能在实验的道路上顺顺利利地走下去,得到咱想要的结果呀!这就是我关于引物设计的一些看法,大家可得记住咯!原创不易,请尊重原创,谢谢!。
自己动手microRNA引物设计miRNA是一类内源性非编码单链RNA,在体内以前体和成熟体的形式存在,而我们对某种的microRNA的表达检测的对象是成熟体,microRNA的成熟体长度约为19-25nt,由于其序列和性质的特殊性,我们需要使用与普通mRNA不同的方法对其进行引物的设计。
目前主流的方法有两种:茎环法和加尾法。
基本的原理图示如上,因为microRNA很短,引物为了方便接下来的检测我们可以通过具有颈环样结构的反转引物先将microRNA进行逆转录,然后再将用于表达检测的3'引物(即图中reverse)设计在反转引物上,最后结合mircoRNA的5'序列设计一个5’端的定量PCR引物(图中Forward)即可。
小结一下就是我们需要设计3条引物,一条做反转录的颈环引物,和2条用于做表达检测引物(Forward和reverse引物)。
茎环结构不但能有效地延长miRNA 的长度,同时它自身互补的构象可以避免与其他同源基因结合,减少了非特异性扩增的几率。
整个引物由 2 部分组成,一个通用的茎环结构和5 ~ 8 个与目的 miRNA 的3'端反向互补的碱基。
其序列通常为: 5'-GTCGTATCCAGTGCAGGGTC CGAGGTATTCGCACTGGATACGAC-3'。
其中下划线部分为茎环自身互补部分。
我们可以看看这个通用的茎环结构序列怎样互补成颈环的。
用DNAstar中的primer select点击report中的primer hairpins报告第一个就是我们通用的一个典型茎环结构了,接下来只需要根据我们的目标microRNA的序列在此序列后加入5-8个碱基与目的MicroRNA 的 3'端反向互补即可。
举个例子:hsa-miR-26a(注意大小写)首先需通过英国著名miRNA数据库网站miRBase (/)检索成熟miRNA序列。
在miRBase上可获得目前已经公布的各个物种的microRNA具体序列、基因分布、详细序列注册信息,可以通过浏览或搜索的方式查询目的miRNA。
引物设计原则引物设计是分子生物学实验中的关键步骤,特别是在PCR(聚合酶链反应)和测序等应用中。
引物的设计质量直接影响到实验的成败和结果的准确性。
本文将详细介绍引物设计的原则,并以实例说明这些原则的应用。
一、引物长度引物的理想长度一般在18-25个核苷酸之间。
过短的引物可能与非目标序列发生互补配对,导致非特异性扩增;而过长的引物则可能降低PCR效率,因为它们需要更高的温度才能完全熔解。
然而,在某些特殊情况下,比如GC含量极高或极低的情况下,可能需要调整引物长度。
二、Tm值Tm值是指DNA双链达到50%解离时的温度,它是衡量引物与模板结合强度的一个重要参数。
理想的Tm值应该在55-65℃之间。
如果Tm值过高,可能会导致引物无法有效退火;如果Tm值过低,则可能导致非特异性扩增。
三、GC含量GC含量也会影响引物的Tm值。
一般来说,GC含量越高,Tm值也越高。
理想的引物GC含量应在40%-60%之间。
过高或过低的GC含量都可能导致引物性能不佳。
四、引物二级结构引物不应含有自身互补的序列,否则会形成发夹结构,影响引物与模板的结合。
因此,应尽量避免引物内部的二级结构。
五、3'端稳定性引物的3'端决定了它是否能有效地与模板结合并进行延伸。
因此,3'端应尽可能稳定,避免存在弱的氢键或者错配。
六、避免跨外显子设计在设计用于检测基因表达的引物时,应避免跨外显子设计,因为这可能导致由于剪接变异而导致的扩增失败。
七、避开重复序列引物应避免包含重复序列,因为这可能导致非特异性扩增。
八、软件辅助设计现在有许多软件可以帮助我们设计引物,如Primer3、OligoAnalyzer等。
这些软件可以自动计算Tm值、GC含量、二级结构等参数,并帮助我们优化引物设计。
九、验证引物最后,无论我们多么小心地设计引物,都需要通过实验来验证其性能。
我们可以先用已知的目标序列进行PCR,看看引物是否能够有效地扩增出目标片段。
1-2890(引物1)#1: Product of length 640 (rating: 171)Contains region of the molecule from 1 to 640Tm: 72.1 C TaOpt: 48.8 C GC: 32.3Sense Primer:CCTGGTTAATCCAAATCACSimilarity: 100.0%Length: 19 Tm: 44.1 C GC: 42.1dH: -142.0 kcal/mol dS: -372.4 cal/mol dG: -29.2 kcal/molAntisense Primer:GACAGGCCCTAATTAAGTTSimilarity: 100.0%Length: 20 Tm: 45.0 C GC: 42.1dH: -158.0 kcal/mol dS: -418.4 cal/mol dG: -31.5 kcal/molTm Difference: 0.9GC Difference: 0#1: Product of length 540 (rating: 171)Contains region of the molecule from 1 to 540Tm: 72.2 C TaOpt: 49.4 C GC: 33.1Sense Primer:CCTGGTTAATCCAAATCACTSimilarity: 100.0%Length: 20 Tm: 45.8 C GC: 40.0dH: -149.8 kcal/mol dS: -393.2 cal/mol dG: -30.8 kcal/molAntisense Primer:ATAAGATTTGAGGTCAGCCASimilarity: 100.0%Length: 20 Tm: 46.4 C GC: 40.0dH: -147.7 kcal/mol dS: -386.3 cal/mol dG: -30.7 kcal/molTm Difference: 0.6GC Difference: 0.01-2890(引物2)#1: Product of length 603 (rating: 171)Contains region of the molecule from 514 to 1116Tm: 73.9 C TaOpt: 50.3 C GC: 37.0Sense Primer:TTGAAGATGGCTGACCTSimilarity: 100.0%Length: 18 Tm: 42 C GC: 47.1dH: -129.6 kcal/mol dS: -335.0 cal/mol dG: -27.9 kcal/molAntisense Primer:GGAGGCCCTTTAACTTAASimilarity: 100.0%Length: 20 Tm: 43.7 C GC: 44.4dH: -165.8 kcal/mol dS: -438.8 cal/mol dG: -33.2 kcal/molTm Difference: 2.0GC Difference: 10.01-2890(引物3)#1: Product of length 703 (rating: 171)Contains region of the molecule from 1063 to 1765Tm: 72.7 C TaOpt: 51.0 C GC: 33.6Sense Primer:GATGCAAATTCAAATCCAGTGGSimilarity: 100.0%Length: 22 Tm: 53.6 C GC: 40.9dH: -164.5 kcal/mol dS: -425.5 cal/mol dG: -35.9 kcal/molAntisense Primer:GGTATGCATGGAGGTGATTTATSimilarity: 100.0%Length: 22 Tm: 50.0 C GC: 40.9dH: -163.9 kcal/mol dS: -428.7 cal/mol dG: -34.3 kcal/molTm Difference: 3.6GC Difference: 0.01-2890(引物4)#1: Product of length 550 (rating: 171)Contains region of the molecule from 1644 to 2193Tm: 73.3 C TaOpt: 49.4 C GC: 35.6Sense Primer:GAAGGCAATCAATCTACTTGSimilarity: 100.0%Length: 20 Tm: 44.0 C GC: 40.0dH: -145.8 kcal/mol dS: -383.7 cal/mol dG: -29.6 kcal/molAntisense Primer:CTTAAGCCACTTTCTGCASimilarity: 100.0%Length: 18 Tm: 43.4 C GC: 44.4dH: -136.3 kcal/mol dS: -356.4 cal/mol dG: -28.3 kcal/molTm Difference: 0.6GC Difference: 4.41-2890(引物5)#1: Product of length 697 (rating: 171)Contains region of the molecule from 2134 to 2828Tm: 73.3 C TaOpt: 50.8 C GC: 35.2Sense Primer:CCACCTTTCAAAGAAAGCACSimilarity: 100.0%Length: 20 Tm: 49.0 C GC: 45.0dH: -156.0 kcal/mol dS: -408.5 cal/mol dG: -32.4 kcal/molAntisense Primer:a TTTCAGCTCGAAGACGCGSimilarity: 100.0%Length: 19 Tm: 54.5 C GC: 52.6dH: -147.1 kcal/mol dS: -378.6 cal/mol dG: -32.4 kcal/molTm Difference: 3.2GC Difference: 10.05070-5466#1: Product of length 455 (rating: 171)Contains region of the molecule from 20 to 474Tm: 79.8 C TaOpt: 55.0 C GC: 52.1Sense Primer:GGAATCTTACCTGCCAGTGSimilarity: 100.0%Length: 19 Tm: 46.8 C GC: 52.6dH: -141.9 kcal/mol dS: -368.8 cal/mol dG: -30.2 kcal/molAntisense Primer:CATTTCAACCCAGAGCCTCSimilarity: 100.0%Length: 19 Tm: 49.3 C GC: 52.6dH: -144.1 kcal/mol dS: -372.1 cal/mol dG: -31.4 kcal/molTm Difference: 2.6GC Difference: 0.041074-42159(引物1)Contains region of the molecule from 10 to 616Tm: 73.8 C TaOpt: 50.3 C GC: 36.6Sense Primer:CTGTGTGGCTATTATCTGCSimilarity: 100.0%Length: 20 Tm: 42.6 C GC: 47.4dH: -142.3 kcal/mol dS: -372.0 cal/mol dG: -29.6 kcal/molAntisense Primer:CACAAGCCCTCTTCAAAGCSimilarity: 100.0%Length: 19 Tm:46.6 GC: 50.0dH: -146.9 kcal/mol dS: -379.6 cal/mol dG: -31.9 kcal/molTm Difference: 4.0GC Difference: 2.641074-42159(引物2)#1: Product of length 504 (rating: 171)Contains region of the molecule from 598 to 1101Tm: 72.0 C TaOpt: 50.3 C GC: 32.7Sense Primer:GCTTTGAAGAGGGCTTGTGSimilarity: 100.0%Length: 19 Tm: 49.9 C GC: 52.6dH: -146.9 kcal/mol dS: -379.6 cal/mol dG: -31.9 kcal/molAntisense Primer:GCTGTTGGTTTTACCGACCSimilarity: 100.0%Length: 19 Tm: 49.5 C GC: 52.6dH: -149.4 kcal/mol dS: -387.3 cal/mol dG: -32.1 kcal/molTm Difference: 0.4GC Difference: 0.043500-44000#1: Product of length 540 (rating: 108)Contains region of the molecule from 1 to 540Tm: 74.4 C TaOpt: 47.6 C GC: 38.5Sense Primer:CGACCTAGAAGCTGTATTTCSimilarity: 100.0%Length: 20 Tm: 45.0 C GC: 43.6dH: -131.2 kcal/mol dS: -351.5 cal/mol dG: -24.6 kcal/molAntisense Primer:CTTCGCATGTTGCAAATGSimilarity: 100.0%Length: 18 Tm: 44.4 GC: 48.0dH: -144.9 kcal/mol dS: -367.8 cal/mol dG: -33.5 kcal/molTm Difference: 0.6GC Difference: 4.463962-664604#1: Product of length 601 (rating: 171)Contains region of the molecule from 7 to 607Tm: 75.9 C TaOpt: 53.1 C GC: 41.8Sense Primer:CATCCTTCTTGCTCGTTCCSimilarity: 100.0%Length: 19 Tm: 49.5 C GC: 52.6dH: -144.8 kcal/mol dS: -373.9 cal/mol dG: -31.5 kcal/molAntisense Primer:CATGACTCCAATGCAGCAGSimilarity: 100.0%Length: 19 Tm: 49.4 C GC: 52.6dH: -135.4 kcal/mol dS: -346.7 cal/mol dG: -30.3 kcal/molTm Difference: 0.1GC Difference: 0.0P21:10416-10865#1: Product of length 460 (rating: 162)Contains region of the molecule from 6 to 465Tm: 85.1 C TaOpt: 58.9 C GC: 65.0Sense Primer:ACCAGGGCCTTCCTTGTATSimilarity: 100.0%Length: 19 Tm: 50.1 C GC: 52.6dH: -152.5 kcal/mol dS: -395.6 cal/mol dG: -32.8 kcal/molAntisense Primer:CGTTTTCGACCCTGAGAGSimilarity: 100.0%Length: 18 Tm: 47.6 C GC: 55.6dH: -137.7 kcal/mol dS: -355.5 cal/mol dG: -29.9 kcal/molTm Difference: 2.4GC Difference: 2.9。