体外核酸诊断试剂盒引物探针研发的基本流程概要
- 格式:ppt
- 大小:1.10 MB
- 文档页数:23

荧光pcr探针法流程英文回答:Fluorescent PCR probe method is a widely used technique in molecular biology to detect and quantify specific DNA sequences. It involves the use of a fluorescently labeled probe that binds specifically to the target DNA sequence during the PCR amplification process. This method offers high specificity and sensitivity, making it ideal for various applications such as genotyping, mutation analysis, and gene expression studies.The workflow of the fluorescent PCR probe method typically involves the following steps:1. Primer design: Specific primers are designed toflank the target DNA sequence. These primers will be used to amplify the target sequence during the PCR reaction.2. Probe design: A fluorescently labeled probe isdesigned to specifically bind to the target DNA sequence. The probe contains a fluorescent dye at one end and a quencher molecule at the other end. When the probe is intact, the fluorescence of the dye is quenched by the proximity of the quencher.3. PCR reaction setup: The PCR reaction is set up with the DNA template, primers, and probe. The reaction mixture also contains dNTPs (deoxynucleotide triphosphates), Taq DNA polymerase, and buffer. The DNA template can be genomic DNA, cDNA, or plasmid DNA, depending on the specific experiment.4. Amplification: The PCR reaction goes through a series of temperature cycles, including denaturation, annealing, and extension. During the annealing step, the primers bind to the target DNA sequence, and the probe hybridizes to the complementary sequence within the amplicon. The polymerase then extends the primers, synthesizing new DNA strands.5. Fluorescence detection: As the PCR reactionprogresses, the probe is cleaved by the polymerase duringthe extension step. This leads to the physical separationof the fluorescent dye from the quencher, resulting in an increase in fluorescence signal. The fluorescence is measured in real-time using a fluorescence detection system, such as a real-time PCR instrument.6. Data analysis: The fluorescence data collectedduring the PCR reaction is analyzed using specific software. The software calculates the threshold cycle (Ct) value, which represents the cycle number at which the fluorescence signal reaches a detectable level. The Ct value can be used to quantify the initial amount of target DNA in the sample.Fluorescent PCR probe method is a powerful tool in molecular biology research and diagnostics. It allows forthe detection and quantification of specific DNA sequences with high sensitivity and specificity. This technique has revolutionized the field by enabling researchers to study gene expression, genetic variations, and disease-related mutations.中文回答:荧光PCR探针法是分子生物学中广泛应用的一种技术,用于检测和定量特定的DNA序列。
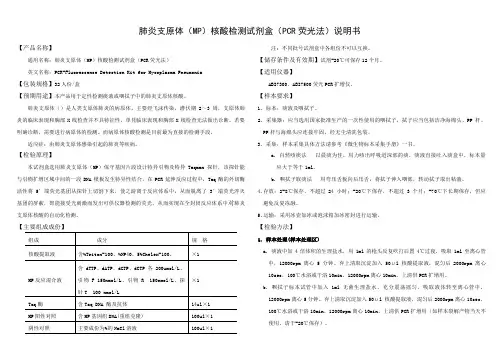
肺炎支原体(MP)核酸检测试剂盒(PCR荧光法)说明书【产品名称】通用名称:肺炎支原体(MP)核酸检测试剂盒(PCR荧光法)英文名称:PCR-Fluorescence Detection Kit for Mycoplasma Pneumonia【包装规格】32人份/盒【预期用途】本产品用于定性检测痰液或咽拭子中的肺炎支原体核酸。
肺炎支原体()是人类支原体肺炎的病原体,主要经飞沫传染,潜伏期2~3周,支原体肺炎的临床表现和胸部X线检查并不具特征性,单凭临床表现和胸部X线检查无法做出诊断。
若要明确诊断,需要进行病原体的检测。
而病原体核酸检测是目前最为直接的检测手段。
适应症:由肺炎支原体感染引起的肺炎等疾病。
【检验原理】本试剂盒选用肺炎支原体(MP)保守基因片段设计特异引物及特异Taqman 探针,该探针能与引物扩增区域中间的一段DNA模板发生特异性结合,在PCR延伸反应过程中,Taq酶的外切酶活性将5’端荧光基团从探针上切割下来,使之游离于反应体系中,从而脱离了3’端荧光淬灭基团的屏蔽,即能接受光刺激而发出可供仪器检测的荧光,从而实现在全封闭反应体系中对肺炎支原体核酸的自动化检测。
【主要组成成份】注: 不同批号试剂盒中各组份不可以互换。
【储存条件及有效期】试剂-20℃可保存12个月。
【适用仪器】ABI7300、ABI7500荧光PCR扩增仪。
【样本要求】1. 标本:痰液及咽拭子。
2. 采集器:应当选用国家批准生产的一次性使用的咽拭子,拭子应当包括洁净海绵头、PP杆、PP杆与海绵头应连接牢固,经无尘清洗包装。
3. 采集:样本采集具体方法请参考《微生物标本采集手册》一书。
a. 自然咳痰法以晨痰为佳,用力咳出呼吸道深部的痰,痰液直接吐入痰盒中,标本量应大于等于1ml。
b. 咽拭子取痰法用弯压舌板向后压舌,将拭子伸入咽部,转动拭子取出粘液。
4.存放:2-8℃保存,不超过24小时;-20℃下保存,不超过3个月;-70℃下长期保存,但应避免反复冻融。

引物设计与检测全过程引物设计是引物检测的第一步,目的是选择能够特异性地识别目标序列的引物。
引物通常由20-25个碱基组成,具有一定的GC含量,理论上GC含量应在40-60%之间。
引物设计时需要遵循以下几个原则:1.引物中不能出现序列重复或自身互补配对,以避免引物间产生二聚体或内聚体。
2.引物不能与非目标序列相互匹配,以避免非特异扩增。
3.引物的长度应适中,太短会导致非特异扩增,太长则会限制扩增效率。
4.引物的温度与Tm(解链温度)适配,以保证引物的特异性。
引物设计可以使用基于计算机的软件(如Primer3、OligoAnalyzer 等)或手动设计。
计算机软件会根据用户提供的目标序列,自动生成合适的引物。
引物设计完成后,接下来是引物的合成。
合成引物通常通过化学合成的方法,在合成过程中会使用特殊的保护基团来保护合成中间体,最后通过去保护反应来得到合成的引物。
得到合成的引物后,需要进行质量检测。
引物检测的主要目的是确保引物的纯度和正确性。
引物检测方法有多种,常用的包括聚丙烯酰胺凝胶电泳、毛细管电泳和高效液相色谱等。
在聚丙烯酰胺凝胶电泳中,引物经过电泳后,通过观察引物的迁移位置和带电量来判断引物的纯度和大小。
毛细管电泳是一种高效的引物检测方法,它能够提供引物的准确分离和确定大小。
高效液相色谱是一种基于溶液中组分在固定相表面的亲和作用分离的方法,可以快速准确地分析引物的纯度。
完成引物的质量检测后,即可用于目标序列的扩增。
引物的扩增可以使用PCR(聚合酶链反应)方法。
在PCR反应中,引物与模板DNA进行互补配对,通过引物自身的特异性,选择性地扩增目标序列。
PCR反应通常包含三个步骤:变性(使DNA解链),退火(引物与DNA模板互相结合)和延伸(通过DNA聚合酶合成新的DNA链)。
PCR反应需要使用特定的引物和一定的温度条件,以产生特异性扩增。
扩增后的目标片段可以通过凝胶电泳等方法进行分析和检测。
凝胶电泳会根据目标片段的大小和电荷量来进行分离和分析。

基因探针检测过程
基因探针(Gene Probe)检测是一种分子生物学技术,用于检测特定基因或DNA序列的存在和表达。
下面是基因探针检测的一般过程:
1.制备探针:
•选择目标基因或DNA序列:确定要检测的目标基因或DNA序列。
•合成或标记探针:探针可以通过合成DNA序列或标记已有的DNA序列制备。
标记通常使用放射性同位素(如32P或
35S)或非放射性标记物(如荧光染料、酶等)。
2.制备样品:
•提取DNA:从待测样本中提取目标DNA。
样本可以是血液、细胞、组织等。
•切割DNA:使用特定的酶酶解DNA,将其切割成适当的片段。
3.杂交反应:
•杂交液:将制备好的探针与目标DNA混合在一起,形成杂交液。
•条件设定:设定适当的温度和时间,使探针与目标DNA发生杂交。
这一步可以通过加热和冷却过程来模拟DNA的分离和
结合。
4.检测信号:
•放射性同位素标记:如果使用放射性同位素标记的探针,
通过暗室摄影或闪烁计数器等设备检测辐射信号。
•非放射性标记:如果使用非放射性标记物,比如荧光标记,通过荧光显微镜或流式细胞仪等设备检测信号。
5.结果分析:
•解释结果:根据信号的强弱和位置,确定目标基因或DNA 序列是否存在。
•结果验证:可以通过其他技术手段如聚合酶链反应
(PCR)、Southern印迹等来验证基因探针检测的结果。
基因探针检测是一种敏感、特异、高效的分子生物学技术,广泛应用于医学、生物学研究和疾病诊断等领域。


qPCR步骤及原理引言定量聚合酶链反应(quantitative polymerase chain reaction,qPCR)是一种广泛应用于分子生物学研究领域的技术。
它可以快速准确地定量检测样品中特定的DNA序列,对于基因表达研究、病原体检测以及遗传疾病的诊断等领域具有重要意义。
本文将介绍qPCR的步骤及其原理。
qPCR步骤qPCR的步骤主要包括以下几个方面:1. 模板DNA的提取或合成qPCR需要先从样品中提取或合成目标DNA序列。
对于复杂样品,提取DNA可以使用商用试剂盒或标准的DNA提取方法。
对于合成DNA,可以使用合成生物学技术进行人工合成。
2. 反应物的准备qPCR反应液的主要组成包括引物(primers)、探针(probes)、模板DNA、聚合酶(polymerase)以及缓冲液。
引物是特异性序列,用于扩增目标DNA。
探针通常是荧光探针,用于检测PCR扩增过程中的产物。
聚合酶是用于引物识别目标DNA、合成新链的酶。
缓冲液则提供稳定的pH环境和离子含量。
3. PCR扩增反应PCR扩增过程是利用聚合酶在一系列循环中,通过加热、退火和延伸的步骤,将目标DNA序列扩增成大量可检测的数量。
PCR扩增过程一般包括以下几个步骤:- 初始变性将PCR反应体系中的模板DNA变性至单链状态,使其成为反应的起点。
- 引物结合将反应体系降温,使引物与目标DNA序列的互补区域结合。
- DNA合成在适宜的温度下,聚合酶利用引物作为起始点,合成新链。
- 扩增循环通过一系列的加热、降温和延伸步骤,重复上述的引物结合和DNA合成,使DNA的数量呈指数增加。
4. 结果分析qPCR可以通过实时检测荧光信号的强度来定量测量PCR反应的结果。
通过计算荧光信号的CT值(threshold cycle),可以确定目标DNA的起始浓度。
CT值是指实时荧光信号达到阈值的循环数目。
qPCR原理qPCR的原理主要基于普通PCR反应的基础上加入了实时检测技术。

DNase检测试剂盒(荧光探针法)盒操作手册DNase检测试剂盒基于荧光基团标记的DNA探针,其一端标记有荧光报告基团分子(Fluor),另一端标记有淬灭基团。
当样本中不含DNase活性时,该探针稳定存在,淬灭基团的物理接近会将荧光报告基团中的荧光淬灭到极低水平,不会产生荧光信号;当样本中含有DNase活性时,探针被降解,荧光报告基团和淬灭基团在溶液中的空间上发生分离,产生逐渐增强的荧光信号;荧光信号增加的速率与酶的数量和活性成正相关。
一.主要组成成分及储存条件名称HBP002902(192T)HBP002903(48T)储存温度DNA探针1管1管-25~-15℃TE缓冲液 2.0mL0.5mL-25~-15℃DNase I标准品20μL10μL-25~-15℃(2U/μL)标准品稀释液12mL6mL-25~-15℃无DNase无DNase水25mL25mL-25~30℃二.预期用途可定量或定性检测单个环境,试剂或者耗材样品中的DNase,判断样本是否被DNase污染。
三.实验环境1.操作过程中需全程穿戴实验服,手套和口罩,严格控制DNase污染;2.为了防止操作过程中引入外源的DNase,前期加样请在超净工作台中进行,实验前用核酸酶清除剂清洁超净工作台中的操作表面,并打开超清洁工作平台紫外照射30分钟;3.其他工作区域在整个实验开始前,也先使用核酸酶清除剂对实验环境进行处理。
四.客户自备耗材仪器210μL移液器桌面离心机3100μL移液器qPCR仪/酶标仪4200μL移液器5DNase DNase free枪头6200μL EP管72ml棕色EP管8黑色平底酶标板/PCR96孔板(高管)9核酸酶清除剂10无酶水11离心管架本实验操作根据酶标仪增益功能不同,分为3种类型进行详细说明,用户可根据自己酶标仪的具体功能,选择对应的实验操作方法。
1.具备自动优化+手动设置增益值的酶标仪,以下以Tecan Spark为例;2.只能选择自动优化或者高中低增益值的酶标仪,以下以SpectraMax iD5为例;3.对于不具备自动优化的酶标仪,比如Fluoroskan FL,这种酶标仪不推荐使用,此时建议使用具有FAM通道的qPCR仪,以下以Thermofisher7500为例。

pcr实验基本流程
一。
PCR 实验准备工作那可是相当重要。
1.1 首先得把实验材料备齐喽,像引物、模板 DNA、dNTPs、Taq 聚合酶,还有各种缓冲液,缺了啥都不行,这叫“兵马未动,粮草先行”。
1.2 仪器设备也得准备妥当,PCR 仪得性能良好,离心机、移液器都得调试好,不然关键时刻掉链子,那可就麻烦大了。
二。
实验操作得一丝不苟。
2.1 先把反应体系配好,各种成分的量都得严格按照要求来,多了少了都可能影响结果,这就好比做饭,盐多了太咸,盐少了没味。
2.2 加样的时候得小心谨慎,千万别把样品弄混了,一旦出错,那就是“一失足成千古恨”。
2.3 把样品放进 PCR 仪,设置好反应程序,温度、时间都得精准,这就像开车掌握好油门和刹车,才能稳稳当当到达目的地。
三。
实验结束后的工作也不能马虎。
3.1 反应结束后,要对产物进行检测,琼脂糖凝胶电泳就是常用的方法,通过电泳图谱能看出实验结果好不好。
3.2 对实验结果进行分析和判断,看看是不是达到了预期的目标,如果不理想,就得找找原因,是操作失误还是实验设计有问题,“吃一堑,长一智”,下次可不能再犯同样的错误。
PCR 实验就像一场精心策划的战斗,每个环节都要考虑周全,操作精准,才能取得胜利,得到满意的结果。

一步一步教你做PCR整天谈论测序,那测序里面关键的一环是什么呢?当然是PCR了。
呵呵,本次小编就写给只会做分析而不会做实验的的生物信息小伙伴们。
很重要奥,所有的分析最终都要落到验证上奥!一、实验原理PCR是体外人工选择性扩增DNA的一种方法,它类似于生物体内DNA的复制。
在生物体内DNA复制需要模板、引物、DNA聚合酶、DNA解旋酶、dNTPs;而体外PCR反应同样需要类似的成分,有模板、引物、PCR Buffer、Taq酶、dNTPs。
其中引物是人工设计的特定序列,实现对特定位置的扩增;PCR Buffer提供一个反应的缓冲环境;反应过程同生物体内一样,DNA双链打开,引物结合模板,延伸形成新链。
而这些过程在生物体内靠DNA解旋酶解链,而在体外在通过控制反应温度实现的。
如常用94℃变性模板DNA打开双链,在退火温度处引物结合到模板上,最后在72℃完成延伸,并反复重复此过程即可实现对特定片段DNA的大量扩增。
到第三循环开始才产生出和靶DNA区段相同的DNA分子,进一步循环地产生出靶DNA区段的指数加倍。
在扩增后期,由于产物积累,使原来呈指数扩增的反应变成平坦的曲线,产物不再随循环数而明显上升,这称为平台效应。
平台期(Plateau)会使原先由于错配而产生的低浓度非特异性产物继续大量扩增,达到较高水平。
因此,应适当调节循环次数,在平台期前结束反应,减少非特异性产物。
到达平台期(Plateau)所需循环次数取决于样品中模板的拷贝。
二、主要的成分模板1.模板可以是多种形式,主要包括基因组DNA、质粒DNA、携带病毒的基因组DNA、PCR产物,cDNA等,但不能为RNA。
对于不同类型的模板,其主要不同在于预变性的时间以及模板的量。
一般对于大的基因组DNA预变性时间10min足够,质粒DNA2min、携带病毒的基因组DNA预变性2min、PCR产物预变性2min即可。
注意cDNA为单链DNA,但仍可做PCR的模板,只不过在第一轮循环时只有一个引物结合,合成另一条链。


核酸引物探针质量技术要求全文共四篇示例,供读者参考第一篇示例:核酸引物探针是分子生物学实验中常用的一种工具,用于特异性检测和测定目标DNA或RNA序列。
其质量的好坏直接影响实验结果的准确性和可靠性。
制定和遵守一定的技术要求对于保证核酸引物探针的质量至关重要。
一、引物序列设计核酸引物探针的设计是其质量的重要基础。
在设计引物时,需要考虑以下几个方面:1. 引物的长度:引物长度通常在18-30个碱基对之间,需要保证引物足够长以确保特异性结合目标序列,同时又不能过长影响反应效率。
2. 引物的GC含量:引物中GC含量的合理范围通常在40%-60%之间,过高或过低的GC含量可能导致引物的特异性降低。
3. 引物的特异性:引物的特异性是指引物与目标序列的互补性,需要确保引物与非目标序列的互补性尽可能小以避免干扰。
4. 引物的末端结构:引物的末端结构需要考虑到PCR扩增、杂交和捕获等不同实验操作的需求,合理设计引物的末端结构有助于提高实验效率。
二、引物检测和验证在使用核酸引物探针前,需要对引物进行检测和验证,以确保其质量符合要求。
主要检测方法包括:1. 电泳检测:通过琼脂糖凝胶电泳或毛细管电泳等方法,检测引物的纯度和长度。
2. 透射电子显微镜观察:可以通过透射电子显微镜观察引物的形态和结构,判断引物的合成质量。
3. 质谱分析:利用质谱分析技术,可以检测引物的精确质量和序列。
三、引物处理和保存引物的处理和保存也对其质量起到重要影响,以下是一些常用的处理和保存方法:1. 复溶:引物在使用前需要以适当的溶剂复溶,避免在处理过程中引物受损。
2. 冻干:可以利用冻干技术将引物保存在干燥状态,减少引物的降解和污染。
3. -20℃或更低温度保存:引物应该保存在-20℃或更低的温度下,避免被高温或光照导致降解。
四、实验操作规范在进行核酸引物探针实验时,需要严格遵守实验操作规范,包括以下几个方面:1. 避免污染:实验前准备工作台、试剂和仪器时要避免污染,以确保实验结果的准确性。
引物和探针设计 –PCR 和定量PCR基本原理引物设计的重要因素针对特殊应用的其他提示引物的质量和纯度目录1247基本原理引物是短的寡核苷酸,充当DNA复制的起始点。
因为几乎所有DNA聚合酶都不能从头合成,所以它们需要一个3'-羟基作为DNA合成的起始点。
这个3'-羟基由相配的引物提供。
引物在体内由RNA聚合酶(称为引物酶)生成。
这些引物(在此为小RNA)由DNA聚合酶用作延长的起始点。
在延长过程中,RNA 引物降解并由DNA取代。
体外扩增反应,如聚合酶链反应(PCR)或逆转录(RT),需要引物。
通过选择特异的引物序列,DNA 片段的所需区域可得到扩增。
对于大多数PCR反应,决定整个反应成功与否的最重要因素是引物的序列和质量。
在开始引物设计之前,必须弄清以下几点:PCR的目的(例如定量检测、克隆、基因分型)PCR类型(定量PCR、RT-PCR、长片段PCR)样品材料(基因组DNA、RNA、微小RNA)可能的问题(例如假基因、SNP)1引物设计的重要因素2有一些不同的软件工具可用于引物设计和序列分析。
它们能简化相配引物对的搜索,一般考虑以下标准。
最流行的软件为Primer 3,它是大多数基于网络引物设计应用的基础。
典型的引物长度为18-30个碱基。
短的引物(15个核苷酸以下)能非常高效地结合---但是它们的专一性不够。
非常长的引物能提高专一性,但是退火效率低,从而导致PCR 产物量低下。
应避免编码单一序列和重复序列的引物。
引物长度和专一性引物的GC 含量应介于40%和60%之间。
应避免聚-(dC )-或聚(dG )-区域,因为它们会降低退火反应的专一性。
聚-(dA )-和聚(dT )-也应避免,因为这会生成不稳定的引物-模板复合物,从而降低扩增效率。
平衡GC含量,避免GC-和AT-富集区域退火温度是基于引物的解链温度(Tm )计算。
最常用的解链温度计算公式显示如下。
“2+4”法则,亦称华莱士法则,对于极短的寡核苷酸(最多14个碱基)有效,该法则提出每个AT 对能将双链DNA 的解链温度提高2°C ,每个GC 对则能提高4°C 。
核酸类检测试剂生产工艺流程下载提示:该文档是本店铺精心编制而成的,希望大家下载后,能够帮助大家解决实际问题。
文档下载后可定制修改,请根据实际需要进行调整和使用,谢谢!本店铺为大家提供各种类型的实用资料,如教育随笔、日记赏析、句子摘抄、古诗大全、经典美文、话题作文、工作总结、词语解析、文案摘录、其他资料等等,想了解不同资料格式和写法,敬请关注!Download tips: This document is carefully compiled by this editor. I hope that after you download it, it can help you solve practical problems. The document can be customized and modified after downloading, please adjust and use it according to actual needs, thank you! In addition, this shop provides you with various types of practical materials, such as educational essays, diary appreciation, sentence excerpts, ancient poems, classic articles, topic composition, work summary, word parsing, copy excerpts, other materials and so on, want to know different data formats and writing methods, please pay attention!一、概述核酸类检测试剂是用于检测DNA和RNA的生物化学试剂,广泛应用于生物医学领域。
简述新一代核酸测序工作技术流程
新一代核酸测序技术,听起来好高大上啊,但其实它就像咱们
玩的拼图游戏。
首先,得把DNA切成小块,然后在每块上都加个接头,方便后面操作。
这就像是把一张大图剪成小块,每块都标上号,方便拼起来。
接下来,就是把这些小块DNA固定好,然后开始复制。
想象一下,就像是在墙上贴满了小磁铁,然后每个磁铁上都吸上了一块铁片,这样一来,每块铁片上都有了DNA的信息。
然后,就是开始解读这些DNA信息了。
通过一系列的反应,就
像是在玩解密游戏,一步步把DNA的密码解开,变成我们能看懂的
文字。
在这个过程中,还会拍很多照片,记录下每一步的反应。
就像
是在给DNA拍电影,每一帧都是它变化的样子。
最后,把这些照片
连起来,就像是把电影剪辑成完整的故事,我们就得到了完整的
DNA序列。
所以你看,新一代核酸测序技术其实就是个高级版的拼图游戏+
解密游戏+电影制作。
就这样,我们能把生命的密码解读出来,真是太神奇了!。
核酸类检测试剂生产工艺流程下载提示:该文档是本店铺精心编制而成的,希望大家下载后,能够帮助大家解决实际问题。
文档下载后可定制修改,请根据实际需要进行调整和使用,谢谢!本店铺为大家提供各种类型的实用资料,如教育随笔、日记赏析、句子摘抄、古诗大全、经典美文、话题作文、工作总结、词语解析、文案摘录、其他资料等等,想了解不同资料格式和写法,敬请关注!Download tips: This document is carefully compiled by this editor. I hope that after you download it, it can help you solve practical problems. The document can be customized and modified after downloading, please adjust and use it according to actual needs, thank you! In addition, this shop provides you with various types of practical materials, such as educational essays, diary appreciation, sentence excerpts, ancient poems, classic articles, topic composition, work summary, word parsing, copy excerpts, other materials and so on, want to know different data formats and writing methods, please pay attention!核酸类检测试剂生产工艺流程1. 原料准备阶段。
标记探针制备
标记探针制备方法是一种用于生物学和医学研究的重要技术,它使用标记探针(例如核酸、碳水化合物或蛋白质)来克隆、检测和定位特定的DNA序列或蛋白质。
该技术在遗传学、分子生物学、转基因研究、微生物学和药物发现等领域有着广泛的应用。
标记探针制备的过程主要包括几个步骤:
1. 选择目标序列:首先,研究者需要选择他们想要克隆或检测的特定DNA序列或蛋白质序列作为标记探针的目标序列。
这可以通过搜索数据库或通过任何其他方法实现。
2. 合成标记探针:接下来,研究者可以使用任何一种合成技术(例如PCR、合成DNA或全基因组合成)来合成标记探针,以实现对特定序列的克隆和检测。
3. 加入标记物:研究者可以使用任何一种方法将标记物(如核酸、荧光素或放射性同位素)加入到已经合成的标记探针中,以便将其与特定序列区分开来。
4. 纯化探针:最后,研究者可以使用纯化技术将标记探针从其他成分中纯化出来,以便用于实验。
标记探针制备技术的使用有助于研究者更好地深入理解和研究人类基因组的结构和功能,也有助于研究者更
快、更准确地确定他们感兴趣的物种的基因组结构,从而为药物发现和基因治疗等应用提供更多有用的信息。