qPCR引物设计
- 格式:ppt
- 大小:699.00 KB
- 文档页数:22

荧光定量pcr的原理方法
荧光定量PCR(Fluorescent Quantitative PCR,qPCR)是一种用荧光信号量化检测PCR产物的方法,用于定量分析目标DNA或RNA的含量。
荧光定量PCR的基本原理如下:
1.引物设计:设计特异性引物,使其能够特异性地扩增目标DNA或RNA序列。
2.模板DNA或RNA的提取:从样品中提取目标DNA或RNA。
3.cDNA合成:对于RNA样品,需要首先将RNA反转录成cDNA,作为PCR 的模板。
4.Real-time PCR扩增反应:将模板DNA或cDNA与引物和荧光探针一起加入PCR反应体系中,进行实时PCR扩增。
PCR反应体系中还包括核苷酸,聚合酶和缓冲液等。
5.荧光信号检测:随着PCR的进行,荧光探针被解旋成单链,释放出与之配对的荧光染料。
荧光染料产生荧光信号,信号强度与扩增产物的数量成正比。
6.荧光信号检测系统:荧光信号检测系统实时检测PCR反应体系中的荧光信号,并将其转换成数值。
7.标准曲线绘制:通过使用已知浓度的标准品进行一系列稀释,绘制出标准曲线。
标准曲线将荧光信号强度与目标DNA或RNA的初始浓度之间建立了一个标准关系。
8.样品定量:通过对样品的荧光信号强度进行测量,并使用标准曲线进行插值计算,确定样品中目标DNA或RNA的初始浓度。
荧光定量PCR具有高灵敏度、高特异性、宽动态范围、低检测限和快速分析等优点,广泛应用于分子生物学和疾病诊断等领域。

cut run的qpcr引物设计规则引物设计在分子生物学实验中起着至关重要的作用,特别是在定量PCR实验中。
cut run是一种新近发展的染色质免疫共沉淀技术,其原理是通过特异性抗体识别靶标蛋白,并利用酶切作用将其与染色质分离,然后使用qPCR来定量分析目标基因的丰度。
在cut run实验中,引物的设计是确保实验成功的关键。
在进行cut run实验前,需要确定目标基因的序列。
这可以通过数据库查询或测序技术来获取。
在得到目标基因序列后,可以使用一些在线工具或软件来设计引物。
例如,Primer3是一种常用的引物设计软件,它可以根据一系列参数,如引物长度、GC含量、Tm值等,自动生成合适的引物序列。
在cut run实验中,引物设计需要遵循一些规则和原则。
首先,引物的长度通常在18-25个碱基对之间,这样可以提高引物的特异性和扩增效率。
此外,引物的GC含量应在40-60%之间,以确保引物在反应温度下具有适当的稳定性。
同时,引物的Tm值应在50-65℃之间,以确保在PCR反应中引物的特异性和扩增效率。
在设计引物时,还需要考虑引物的特异性。
特异性引物能够准确识别目标基因,而不会与其他非靶标序列发生非特异性扩增。
为了确保引物的特异性,可以使用一些在线工具或软件来进行引物序列的BLAST比对,以确保引物只与目标基因序列匹配。
引物的设计还需要考虑引物之间的配对效率。
在cut run实验中,通常需要使用两对引物,一个用于目标基因的扩增,另一个用作内参基因。
内参基因的选择应该是稳定表达且不受实验条件影响的基因。
例如,常用的内参基因有GAPDH、β-actin等。
两对引物的设计应该遵循一致的规则,并且要确保引物的Tm值相近,以确保它们在同一温度下扩增。
在实验中,引物的质量也是非常重要的。
为了确保引物的质量,可以选择可靠的供应商,并避免使用旧的或不合适的引物。
此外,引物的储存也需要注意,应该存放在干燥、避光和低温的环境中,避免引物的降解和污染。

qpcr引物设计原则和注意事项
qPCR引物设计原则和注意事项
qPCR引物设计是指为了检测特定基因序列而设计的一种引物,是实现qPCR技术的关键技术之一。
引物设计的准确性和准确性直接影响qPCR的精确性、可靠性和重复性。
qPCR引物设计的原则是:1)选择被检测序列的5’端两个核苷酸作为引物的开始;2)采用20-23bp的长度,保证引物的灵敏度和特异性;3)引物的Tm值应位于55-60°C,以确保引物有足够的结合力;4)尽量避免引物中存在重复序列;5)尽量避免引物中存在非特异性结合位点。
在设计qPCR引物时,应注意以下几点:1)选择被检测基因序列的特定位置作为引物的起始位点;2)计算引物的Tm值,以确保引物的结合力;3)避免引物中存在重复序列及其它非特异性结合位点;4)加入引物的标记磷酸,以增加引物的特异性;5)检查引物序列中是否存在致突变的核苷酸;6)检查引物序列中是否存在非特异性结合位点。
总之,qPCR引物设计是一项重要且复杂的技术,设计者必须特别注意以上原则和注意事项,以确保设计出的引物具有足够的特异性和灵敏度,以实现qPCR的准确性和可靠性。
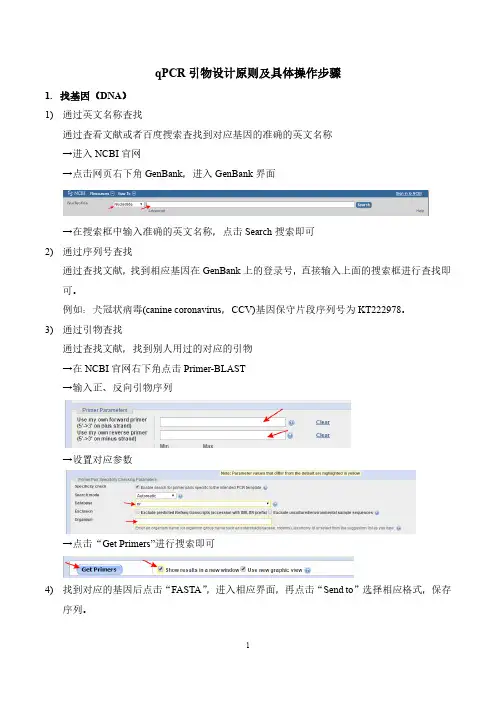
qPCR引物设计原则及具体操作步骤1.找基因(DNA)1)通过英文名称查找通过查看文献或者百度搜索查找到对应基因的准确的英文名称→进入NCBI官网→点击网页右下角GenBank,进入GenBank界面→在搜索框中输入准确的英文名称,点击Search搜索即可2)通过序列号查找通过查找文献,找到相应基因在GenBank上的登录号,直接输入上面的搜索框进行查找即可。
例如:犬冠状病毒(canine coronavirus,CCV)基因保守片段序列号为KT222978。
3)通过引物查找通过查找文献,找到别人用过的对应的引物→在NCBI官网右下角点击Primer-BLAST→输入正、反向引物序列→设置对应参数→点击“Get Primers”进行搜索即可4)找到对应的基因后点击“FASTA”,进入相应界面,再点击“Send to”选择相应格式,保存序列。
2.qPCR引物和TaqMan探针的设计1)引物设计注意事项a)引物长度17bp-25bp为佳。
太短的引物容易导致扩增效率降低;太长的引物会导致出现引物高级结构的几率增加。
两者都会干扰定量结果的准确性b)扩增片段长度为:90-150 bp(最低不能超过70,最高不能超过180)c)引物的Tm值为:最小57℃,最大63℃,最适为60℃,两条引物之间退火温度得差距不超过1℃,推荐使用Primer Premier 5进行Tm值计算;d)引物A、G、C、T整体分布尽量要均匀,避免使用GC或者TA含量高的区域,尤其是3’端,必须避开GC含量不均匀的区域。
e)引物设计时请尽量避开TC或者AG的连续结构。
f)3’端不能超过3个以上碱基互补,自互补碱基数不超过3;3’端最后一个碱基绝对不能搭上g)特异性要有保证,与非特异模板3’端互搭碱基数不超过3,不连续出现4个及以上的GC互搭h)引物3’端最后五个碱基不能包含超过2个以上的G或者Ci)引物的GC含量控制在40%-60%之间为好,最佳为45%-55%之间j)正向或者反向引物应尽量接近探针序列但是不能和探针序列有重合区域k)在Primer-BLAST设计时,在Organism 处选择相应物种l)需跨外显子设计,避免基因组污染2)TaqMan探针设计指南a)探针序列应尽量接近正向或者反向引物,但是不能与之有重合区域;一般相隔1~5个碱基(一般10个以内,最好是1个碱基)。

LncRNA引物设计-⼗年资深qPCR引物设计经验分享及常⽤LncRNA数据库介绍LncRNA引物设计-⼗年资深qPCR引物设计经验分享及常⽤LncRNA数据库介绍LncRNA的qPCR引物如何设计?在设计的时候有没有需要特别注意的地⽅?如何能设计出最好的qPCR引物。
基于这些问题,总结⾃⼰10年qPCR引物设计经验分享给⼤家。
LncRNA的qPCR引物相⽐于编码基因,是要复杂。
导致其复杂的原因并不是引物设计原则上有特别之处,⽽是在于有太多的LncRNA数据库,数据库之间往往没有关联,且不同数据库的维护也不尽相同。
今天的⽂章分两块:LncRNA数据库介绍和LncRNA引物设计注意事项。
LncRNA数据库介绍1、NCBIRefSeqNCBI不多做介绍,通常LncRNA信息我们参考RefSeq数据库中的数据,RefSeq数据库中的数据参考数据,是经过⼈⼯审核,其数据信息可信,注释全⾯。
RefSeq数据库中LncRNA的命名通常是NR_或者XR_开头,后⾯加数字,其外显⼦信息位置,数量信息⾮常完整。
2、Ensembl相同的LncRNA,Ensembl数据库中信息往往要⽐NCBI多很多,特别是转录本数量。
且数据变化⾮常快且变化会很⼤,可能昨天浏览这个数据库中某个LncRNA只有2个转录本,隔天再去看的时候,可能就变成3个甚⾄更多。
NCBI就不同,尽管更新频率也⾮常的快,但是LncRNA的变化通常很⼩,转录本数量基本不变化,序列变化的可能性也⾮常的⼩。
3、UCSCUCSC数据库的LncRNA数据个⼈认为更新相对较慢,但有些LncRNA名称如uc001ylu就需要前往UCSC数据库查询其序列信息。
UCSCGenomeBrowser可以根据基因组的位置、基因ID、转录本等信息进⾏浏览查询。
4、RNAcentralRNAcentral整合了包括Ensembl、GENCODE、Greengenes、HGNC、LNCipedia、lncRNAdb、miRbase、NONCODE、RDP、RefSeq、Rfam、SILVA在内的多个数据库。

NCBI使⽤教程--qPCR引物设计NCBI设计qPCR引物主要⽤到Primer designing tool⼀/打开基因序列页⾯⼆/点击Pick Primers,跳转到到Primer designing tool的页⾯到Primer designing tool的页⾯。
*如果已经有了基因的序列,可以不通过基因查找页⾯,直接进⼊Primer designing tool。
具体的⼊⼝是NCBI——BLAST——Primer-Blast。
三/根据公式对将设计出的引物进⾏参数设计① PCR Template通过查找序列转到Primer designing tool的,那么在PCR Template栏中则会⾃动出现您的基因的编号,⽐如对于human β-actin来讲,其mRNA的页⾯点击Pick Primer之后,PCR Template 栏中会出现其对应编号:NM_001101.3。
如果是直接进⼊Primer designing tool界⾯,则需要将⽬的基因的编号或者序列粘贴进该框中。
在右侧的Range栏中,可以设定正反向引物的结合范围。
② Primer Parameters引物参数栏中,前两⾏是引物的序列栏,⽤于对已有的引物进⾏特异性分析,在设计引物的过程中并不会⽤到,因此留空即可。
在PCR product size⼀⾏中,可以设定PCR引物的扩增产物长度范围,对于qPCR来讲,范围设为50~250时结果⽐较精确,可以先设定⼀个较⼩的范围,如80~150,如果该范围内没有设计出⽐较好的引物,则可以返回扩⼤产物⼤⼩范围。
# of primers to return是指软件设计引物的数量(如果有的话),默认为10对。
引物的Tm值⼀⾏中,可以设定引物的最⼤、最⼩和最佳Tm值,以及正反向引物Tm的最⼤差值。
对于qPCR来讲,默认的即为最佳Tm值60°C,不需做任何修改。
③ Exon/intron selection在抽提RNA的过程中,样本中的基因组DNA污染会显著影响最终的结果,虽然可以使⽤DNase I进⾏处理将RNA样本中的中DNA降解掉,但是很难保证DNA降解完全。


10.26-qPCR引物设计设计原则:1.PCR扩增产物长度 :80-150bp. 引物的产物⼤⼩不要太⼤,⼀般在80-250bp之间都可;80-150bp最为合适(可以延长⾄300 bp)2.引物长度⼀般在15-30碱基之间。
引物长度(primer length)常⽤的是18-27bp,但不应⼤于38bp,因为过长会导致其延伸温度⼤于74℃,不适于Taq DNA 聚合酶进⾏反应。
3.引物GC含量在40%~60%之间,Tm值最好接近72℃。
Tm=58-60度。
GC含量(composition)过⾼或过低都不利于引发反应。
上下游引物的GC含量不能相差太⼤。
另外,上下游引物的Tm值(melting temperature)是寡的解链温度,即在⼀定盐浓度条件下,50%寡核苷酸双链解链的温度。
有效启动温度,⼀般⾼于Tm值5-10℃。
若按公式Tm=4(G+C+2(A+T)估计引物的Tm值,则有效引物的Tm为55-80℃,其Tm值最好接近72℃以使复性条件最佳。
4.引物3'端不能选择A,最好选择T。
引物3'端错配时,不同碱基引发效率存在着很⼤的差异,当末位的碱基为A时,即使在错配的情况下,也能有引发链的合成,⽽当末位链为T时,错配的引发效率⼤⼤降低,G、C错配的引发效率介于A、T之间,所以3'端最好选择T。
5.引物⾃⾝及引物之间不应存在互补序列。
引物⾃⾝不应存在互补序列,否则引物⾃⾝会折叠成发夹结构(Hairpin)使引物本⾝复性。
这种⼆级结构会因空间位阻⽽影响引物与模板的复性结合。
引物⾃⾝不能有连续4个碱基的互补。
两引物之间也不应具有互补性,尤其应避免3'端的互补重叠以防⽌引物⼆聚体(Dimer与Cross dimer)的形成。
引物之间不能有连续4个碱基的互补。
引物⼆聚体及发夹结构如果不可避免的话,应尽量使其△G值不要过⾼(应⼩于4.5kcal/mol)。
否则易导致产⽣引物⼆聚体带,并且降低引物有效浓度⽽使反应不能正常进⾏。

sybr green qpcr引物设计原则实时荧光定量聚合酶链反应(qPCR)是一种高通量检测技术,用于快速准确的检测和定量化DNA。
它的成功离不开准确的引物设计。
SYBR Green qPCR引物设计原则是一种指导性的设计方法,可用于指导研究人员开发有效的qPCR探针引物。
SYBR Green qPCR引物设计原则主要包括:(1)引物结合特异性:引物在qPCR反应中用于特异性结合DNA 模板,以引起聚合反应。
因此,研究人员应密切关注引物体系中两个特异性配对序列(3’端Tm值> 55℃)的准确性,以及引物在不同条件下能够结合DNA模板的特异性。
(2)引物安全性:引物的设计应避免在qPCR反应过程中与目标DNA片段发生不特异性结合,或者它们之间的交叉反应也应该尽可能地被避免。
(3)引物的Tm值:Tm值是指底物在指定温度下(通常在60℃至70℃),一半的引物分子与目标片段结合的温度,这是产生指定特异性酶催化活性的关键因素。
(4)引物体系的GC含量:该体系应控制在30%-70%之间,以消除组成成分对反应条件的影响。
(5)引物长度:通常,qPCR引物呈放射形式排列,其反应条件最为理想。
同时,引物长度应尽可能降低,以降低反应成本,提高反应的效率。
一般情况下,引物的长度为17-24bp,3’端的长度应不小于17bp,5’端的长度可以小于17bp,最好尽量控制在20bp左右。
(6)引物的单体纯度:反应受到低纯度引物影响会变慢,结果不可靠,因此引物的纯度应高于95%。
(7)灵敏性:灵敏性是指qPCR反应可以检测到的最低DNA模板浓度。
灵敏性越高,实验的结果越准确,一般来说,灵敏度达到1ng/ul 或更高水平。
(8)模板效应:模板效应是指反应影响模板浓度的引物反应比。
当t>Ct的情况出现,这是由于引物失效或模板效应,其中模板效应是由引物/模板量比变化引起的。
此外,在引物设计策略中,还应特别注意一些重要参数,如引物杂交温度(Tm)、安全性、灵敏度等,以及产品的单体纯度、杂质团规格等。

荧光实时定量PCR引物设计I.靶的选择和试验设计1.针对目的基因序列选择合适的扩增片断查看以下三个网站是否有合适的已经证实的QRT-PCR的扩增引物,探针以及反应条件.RTPrimerDB (http://medgen.ugent.be/rtprimerdb), 相对物种较多PrimerBank (/primerbank/index.html) 人,鼠Real Time PCR Primer Sets () 人,mouse,rat如果没有合适的或者已经证实的可以提供参考,以下的设计方案仅供参考:A.最广泛使用的商业化的软件Beacon Designer()B.DIY的软件Primer ExpressC.如果Beacon Designer 无法得到您说需要的结果,或者获得到设计方案的备选数目不够的话,可以选择Sigma-Genosys )的服务方案,详细周到D.一个免费的基于网页构架的引物和探针设计程序:/products/probe_design.asp您可以挑选其中的4-6对引物进行试验,选择引物的扩增效率和灵敏度高的.这个软件可以直接与NCBI的网站进行比对,并且用NCBI的ePCR进行虚拟的电子PCR 引物设计简介DNA引物长度:15-25 个碱基GC含量:50%左右如果引物的与AT区域富集结合,可以考虑用LNA替换几个碱基,较少引物的长度以及避免引物次级结构和3’端二聚体的影响.由于引物和模板和探针与靶点之间的分之间的相互竞争,分之内杂交,倒转重复等等会引起引物的引发探针对结合效率达降低,因此我们选择引物二聚体的△G为负值,即:<10 kcal/mol.没有连续的G/C.引物探针的保存一般遵循以下原则:正向和反向引物保存在-20度, 浓度为10mM 或者10×工作浓度.探针应该避光保存,贮存在-70度,最好以冻干粉状态,工作浓度的液体保存一般两周左右。
2.输入靶序列,用BLASTn在/blast进行比对3.检查比对序列的多态性以及可能的错误避免这些区域来进行引物和探针设计4.在靶序列中避免直接待重复区,在重复区进行杂交容易使得引物非获得产物性结合,降低DNA的扩增效率以及减少分析的灵敏度。

PCR 之引物设计--------基因序列查询篇•确定目标基因的形态:DNA or RNA•查找目标基因•比对目标基因同源性和特点•选取目标基因序列•选取目标基因的目标区段第一步:如何查基因:1、mRNA 序列的查询;以查大鼠cort为例;/search 选择11、在NCBI上搜索到目的基因,找到该基因的mRNA,Copy该mRNA序列作为软件查询序列的候选对象。
该mRNA文件的命名:Rattusnorvegicuscortistatin (Cort), mRNA;NM_012835:是唯一的编号;把以下序列存成txt文件:Rattusnorvegicuscortistatin (Cort), mRNA* Comment* Features* SequenceLOCUS NM_012835 438 bp mRNA linear ROD 11-FEB-2008DEFINITION Rattusnorvegicuscortistatin (Cort), mRNA.ACCESSION NM_012835VERSION NM_012835.1 GI:6978682KEYWORDS .SOURCE Rattusnorvegicus (Norway rat)ORGANISM RattusnorvegicusEukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi;Mammalia; Eutheria; Euarchontoglires; Glires; Rodentia;Sciurognathi; Muroidea; Muridae; Murinae; Rattus.REFERENCE 1 (bases 1 to 438)AUTHORS Xidakis,C., Mastrodimou,N., Notas,G., Renieri,E., Kolios,G.,Kouroumalis,E. and Thermos,K.TITLE RT-PCR and immunocytochemistry studies support the presence ofsomatostatin, cortistatin and somatostatin receptor subtypes in ratKupffer cellsJOURNAL Regul.Pept. 143 (1-3), 76-82 (2007)PUBMED 17481746REMARK GeneRIF: RT-PCR and immunocytochemistry studies support the presence of cortistatin in rat Kupffer cells.REFERENCE 2 (bases 1 to 438)AUTHORS Bourgin,P., Fabre,V., Huitron-Resendiz,S., Henriksen,S.J., Prospero-Garcia,O., Criado,J.R. and de Lecea,L.TITLE Cortistatin promotes and negatively correlates with slow-wave sleepJOURNAL Eur. J. Neurosci. 26 (3), 729-738 (2007)PUBMED 17686045REMARK GeneRIF: The capacity of CST-14 to increase SWA, together with preprocortistatin's inverse correlation with time spent in SWS, suggests a potential role in sleep homeostatic processes.REFERENCE 3 (bases 1 to 438)AUTHORS Deghenghi,R., Avallone,R., Torsello,A., Muccioli,G., Ghigo,E. and Locatelli,V.TITLE Growth hormone-inhibiting activity of cortistatin in the ratJOURNAL J. Endocrinol. Invest. 24 (11), RC31-RC33 (2001)PUBMED 11817718REFERENCE 4 (bases 1 to 438)AUTHORS de Lecea,L., Criado,J.R., Prospero-Garcia,O., Gautvik,K.M., Schweitzer,P., Danielson,P.E., Dunlop,C.L., Siggins,G.R.,Henriksen,S.J. and Sutcliffe,J.G.TITLE A cortical neuropeptide with neuronal depressant andsleep-modulating propertiesJOURNAL Nature 381 (6579), 242-245 (1996)PUBMED 8622767COMMENT PROVISIONAL REFSEQ: This record has not yet been subject to final NCBI review. The reference sequence was derived from U51919.1.Summary: inhibits growth hormone secretion; may act as aneuropeptide to mediate signaling via a somatostatin receptorsubtype [RGD].FEATURES Location/Qualifierssource 1..438/organism="Rattusnorvegicus"/mol_type="mRNA"/strain="Sprague-Dawley"/db_xref="taxon:10116"/chromosome="5"/map="5q36"gene 1..438/gene="Cort"/note="cortistatin"/db_xref="GeneID:25305"/db_xref="RATMAP:41189"/db_xref="RGD:2383"CDS 30..368/gene="Cort"/note="Preprocortistatin"/codon_start=1/product="cortistatin"/protein_id="NP_036967.1"/db_xref="GI:6978683"/db_xref="GeneID:25305"/db_xref="RATMAP:41189"/db_xref="RGD:2383"/translation="MGGCSTRGKRPSALSLLLLLLLSGIAASALPLESGPTGQDSVQDATGGRRTGLLTFLAWWHEWASQDSSSTAFEGGTPELSKRQERPPLQQPPHRDKKPC KNFFWKTFSSCK"sig_peptide 30..110/gene="Cort"mat_peptide 324..365/gene="Cort"/product="cortistatin-14"ORIGIN1 aaagcacagacttcaggtctccaaggaggatgggtggctgcagcacaagaggcaagcggc61 cgtcagccctcagtctgctgctgctgctgctgctctcggggatcgcagcctctgccctcc121 ccctggagagcggtcccaccggccaggacagtgtgcaggatgccacaggcgggaggagga181 ccggccttctgactttccttgcctggtggcatgagtgggcttcccaagacagctccagca241 ccgctttcgaagggggtaccccggagctgtctaagcggcaggaaagaccacccctccagc301 agcccccacaccgggataaaaagccctgcaagaacttcttctggaaaaccttctcctcgt361 gcaagtagcccgagcctgaccggagcctgaccggccaccctgtgaatgcagccgtggcct421 gaataaagagtgtcaagt//其中origin后面的序列,是我们用来设计的序列。
使用Oligo设计QPCR引物—以果蝇LaminC序列为例普通PCR的基础上,加上荧光信号,在扩增目的片段的过程中通过荧光信号的富集来得到Ct值,从而确定基因转录表达含量。
[1]引物设计原则[1]考虑引物与DNA片段结合的特异性、强度以及是否会产生引物二聚体等因素,因此有如下原则:◆引物长度大于16bp,18~24个核苷酸;◆引物与靶序列间的Tm值不应该过低。
两条引物之间尽量接近,差值不超过4℃;◆引物不应该有发夹结构,不能有4bp以上回文序列;◆两引物间不应有大于4bp以上的互补序列或同源序列,在3’端不应有任何互补碱基;◆碱基尽可能均匀分布,GC含量在40~60%;◆引物尽量不形成二级结构。
◆但是qPCR与其他PCR的扩增目的稍微有所不同,qPCR需要通过快速扩增实现对转录本的定量。
因此一般要求扩增的片段比较短,一般在200bp以下,考虑是否会引物特异性高低问题出现杂峰等。
扩增长度,建议在100~300bp 间比较好扩增;引物长度在20~25bp 较好,而且上下游引物间不要相差超过5bp;再就是看看Tm 值,60℃左右,相差不要超过1℃。
●引物长度一般为15-27 bp,最适为18-22 bp(注意此处所指的长度是结合到模板的长度,不包括5'端加修饰后的长度),过长会导致延伸温度大于74℃,不适于Taq DNA聚合酶进行反应。
上下游引物长度差不超过4 bp,Tm差不超过2℃。
●引物序列在模板其他位置应当没有相似性较高的序列,尤其是3'端,否则容易导致错配。
引物3'端避免出现3个以上的连续碱基如GGG或CCC。
●引物3'端的末位碱基对Taq酶合成效率有较大影响,末位碱基为A的错配率明显高于其他碱基,应当避免在引物的3'端使用碱基A。
另外,引物二聚体或发夹结构也可能导致PCR反应失败。
5'端序列对PCR 影响不太大,因此常用来引进修饰位点或标记物。
QPCR引物设计要点一、设计对象及分析目的1.分析对象则为设计对象;但具体的实验对象因具体的实验方法而异;2.表达水平分析(定量):RNA(mRNA, lncRNA, rRNA, tRNA, miRNA, piRNA);3.存在分析(定性):DNA (CHIP-QPCR), RNA (RIP-QPCR);4.等位基因拷贝数分析:DNA (LOH);5.甲基化分析:重亚硫酸盐转化后的基因组DNA序列(可以假设CpG位点的C全部发生甲基化,或CpG位点的C全部未发生甲基化);6.设计模板:序列号或具体的序列(见图1)二、设计参数以在线软件为例:/tools/primer-blast/ 1.PCR产物的长度(PCR product size):一般为80-200bp,最好为:90-140 bp(图2);2.引物的退火温度(Primer melting temperatures, TM):59-60℃,最优为60℃(在实际进行QPCR反应时,TM值为60℃)(图2);3.跨外显子(span an exon-exon junction):其中一条引物落在相邻两个外显子的交界上,其作用是避免所提取的总RNA污染了基因DNA所造成的假阳性(图3),如果设计模板为基因组DNA(重亚硫酸盐处理的DNA序列)则不需遵守;4.跨内含子(separated by at least one intron):正反引物分别落在相邻的两个外显子上,其作用是避免所提取的总RNA污染了基因DNA所造成的假阳性(图3),如果设计模板为基因组DNA(重亚硫酸盐处理的DNA序列)则不需遵守;5.BLAST检索引物的特异性,根据设计对象的属性选择合适的数据库种类(图4),输入检测样本的种属(图5);6.引物的要求:长度为18-26bp,最优为23bp, 引物的GC含量为50-60%,引物中连续出现的相同碱基的数目不超过4(图6);7.计算引物TM值的参数:QPCR反应体系中的一价盐离子,二价盐离子,dNTP的浓度和引物的浓度均可影响TM值(图7);8.引物尽量不含已知的SNP位点(图8),否则存在出现碱基错配的风险;。
荧光定量PCR(qPCR)对基因表达的分析一、实验试剂SYBR Green premix(Takara,RR420A)二、试验设备和材料无RNase的离心管、PCR管、枪头。
三、操作步骤(一)引物设计原则(1)设计引物的长度一般为18–28个核苷酸。
(2)扩增产物长度80-150 bp最好,最长是300 bp。
(3)避免重复核苷酸延伸段。
(4)目标为50% GC含量,有助于防止错配稳定化。
(5)选择Tm值匹配的引物(相差在5°C范围内),60℃左右最好。
(6)避免一个Assay中采用的所有引物之间以及各引物内出现序列互补。
(二)总RNA提取(TaKaRa MiniBEST Universal RNA Extraction Kit,9767)准备工作:Buffer RL中加入50×DTT;Buffer RWB中加入无水乙醇;配制70%DEPC乙醇。
1. 动物培养细胞的 RNA 提取(1)细胞的裂解。
①倒出培养液,使用 1×PBS清洗一次。
②向培养细胞中加入适当量(Table 1 中推荐的使用量)的裂解 Buffer RL(使用前请确认已加入 50×DTT Solution),水平放置片刻,使裂解液均匀分布于细胞表面并裂解细胞,然后使用移液枪吹打细胞使其脱落。
(6 孔细胞培养板每孔加入 350μL)③将内含细胞的裂解液转移至离心管中,用移液枪反复吹吸直至裂解液中无明显沉淀。
(2)裂解液室温静置2 min。
注:对于基因组含量较多的材料或者材料起始量较大时,可以直接按步骤 7 进行(否则 DNA 含量过高可能造成 gDNA Eraser Spin Column 堵塞),如基因组含量较低或材料起始量较少时,可以按步骤 3-6 进行。
(3)将gDNA Eraser Spin Column放到2 mL 的Collection Tube(试剂盒提供)上。
(4)将裂解液转移入到gDNA Eraser Spin Column中。
引物设计不求⼈!⼿把⼿教你设计RT-qPCR引物!RT-qPCR 全称 Real-time polymerase chain reaction, 即实时聚合酶链锁反应,是⼀种在DNA扩增反应中,以萤光染⾊剂检测每次聚合酶链锁反应(PCR)循环后产物总量的⽅法。
OK~这⾥对RT-qPCR的原理,应⽤及数据分析就不再赘述了,因为今天我们主要讲的是如何设计出好的PCR引物。
相信科研⼩伙伴们都已经⽤过Pubmed来设计引物了,基本就是 “傻⽠式操作”就解决了!但这⾥还需提醒⼩伙伴们两点:1. 需跨越⾄少⼀个外显⼦连接(⼀般我们⽤来做RT的RNA都是通过Trizol抽提法分离出来的,为了避免基因组的DNA也有可能被扩增出来,就要⾄少跨越⾄少⼀个exon-exon junction 了)。
2. 不要忘记选对“物种”!(这⾥以⼈种⽰例)但是Pubmed设计出来的引物⼀般不尽如⼈意,⽐如说引物之间存在极⼤的互补性,3’端的互补重叠,形成引物⼆聚体等等。
因为⽤Pubmed设计引物本⾝能设置的参数条件就很少,所以它才是最简单的。
所以,今天我要给⼤家介绍两款实⽤的软件,Primer Premier 6.0 和 Oligo 7.0。
今天主要讲讲第⼀款,⾄于第⼆款,改天再聊啦!当然,这两款软件,要想⽤全功能版本,都是需要付费的哦!不过...,我们伟⼤的祖国有强⼤的某宝,这⾥就不多说了哈。
好了,我们进⼊今天的正题……(说了这么多废话,终于要进⼊正题了!!!)⾸先,我们选⼀个基因举例来说,PPARA,⾄于为什么会选这个基因呢?因为啊,PPAR⽬前是肿瘤学研究中的⼀个“明星基因”,它的全称是Peroxisome proliferator-activated receptor, 即过氧化物酶体增殖物激活型受体,⽬前发现有三种亚型,PPAR a,PPARbeta/delta,PPAR r。
好吧…….听着好复杂的样⼦啊!总之,管它到底是什么呢,下⾯来说说它的引物如何设计。
qPCR引物设计总结1、产物扩增长度建议:80-150bp2、引物长度:17bp---25bp3、 3’端尽量避免⾼GC或者AT含量⾼区域4、 3’端最后⼀个碱基最好为G或者C,避免使⽤T5、两个引物Tm最好相差不要超过1°C,Tm值在60°C—65°C为佳(使⽤Primer 5 设计)6、引物GC含量控制在40%---60%之间7、引物特异性检测(NCBI)注:通常设计qPCR引物时会将引物设计在跨两个外显⼦,⽬的减少遗留DNA造成误差,但是有时候这样设计的引物不能满⾜以上条件,所以强烈建议提取RNA时使⽤gDNA酶处理,这样完全可以不考虑跨外显⼦。
举例:⼤⿏的MyD88序列ATGTCTGCGGGAGGCCCCCGCGTGGGATCCGTGTCCGTGGACTCCTACTTGTTCTCTCTA CCGTTGGTCGCGCTCAACGTGGGAGTGAGGCGCCGCCTCTCGCTGTTCTTGAACCCGCGG ACAACCGCGGCGGCCGACTGGACCTCGCTGGCGGAGGAGATGGGTTTCGAGTACTTGGAG ATCCGCGAGTTTGAGACGCGCCCCGATCCCACTCGCAGTTTGTTGGATGCCTGGCAGGGG CGCTCTGGCTCGTCGGTCGGCAGGCTGCTAGAGTTGCTAGCCTTGTTAGACCGTGAGGAT ATACTGTATGAACTGAAGGACCGCATCGAGGAGGACTGCCAGAAATACATACGCAACCAG CAGAAACAGGAGTCTGAGAAGCCCTTGCAGGTGGCCAGAGTGGAGAGCAGTGTCCCACAG ACAAAGGAACTGGGAGGCATCACCACCCTCGATGACCCCCTGGGGCAAACGCCGGAGCTT TTCGACGCCTTCATCTGCTACTGCCCCAGTGATATTGAGTTTGTGCAGGAGATGATCCGG CAACTAGAACAGACAGACTATCGGCTTAAATTGTGTGTGTCCGACCGCGATGTCCTGCCT GGCACCTGCGTGTGGTCCATCGCCAGCGAGCTCATTGAGAAAAGGTGTCGTCGCATGGTG GTGGTTGTTTCTGACGATTACCTGCAAAGTAAGGAATGTGACTTCCAGACCAAGTTTGCT CTCAGCCTGTCTCCAGGTGTCCAACAGAAGCGACTGATCCCTATCAAGTACAAAGCAATG AAGAAGGACTTTCCTAGTATCCTACGGTTCATCACTATCTGCGACTATACCAACCCTTGC ACCAAGTCCTGGTTCTGGACCCGTCTTGCCAAGGCTCTGTCCCTGCCCTGA在prime.5⾥⾯点击search之后,修改参数,主要调节Tm和GC就⾏了之后,选取打分较⾼的引物,如前五个:5‘---3’1F:GTCGCATGGTGGTGGTTGTT 1R: TGATAGGGATCAGTCGCTTCTGT2F:AAAGGTGTCGTCGCATGGTG 2R: GTGGTACGCTGCTGTGGAAA3F:TCGTCGCATGGTGGTGGTT 3R:TGATAGGGATCAGTCGCTTCTGT4F:TCGGCAGGCTGCTAGAGTTG 4R:GTTTCTGCTGGTTGCGTATGTATTT5F: AGGACTGCCAGAAATACATACGC 5R:CCTTTGTCTGTGGGACACTGCTC按照以上7条总结:为了不使3‘为T,所以使⽤2号和5号引物最后检查引物特异性,通过NCBI 进⾏blast⽐对即可,⽬的是引物这个基因特有的。
qpcr引物设计原则和注意事项
qPCR引物设计的原则和注意事项包括:
1. 选择稳定的Tm值:qPCR引物的Tm值应在60°C - 63 °C,它应尽量接近被检测目标序列的Tm值;
2. 尽量降低引物间的胁迫竞争性作用:两个引物之间如果存在太多高度相似的区域,会导致引物间的竞争性作用,影响qPCR结果;
3. 避免引物之间的碱基配对:引物末端的碱基是控制引物稳定性的关键,因此不宜出现自我碱基配对来影响引物的稳定性;
4. 保证引物的GC含量:一般而言,引物GC含量应在30% - 80%之间,这是因为太高的GC含量会导致引物的热稳定性降低;
5. 保证引物的熵和质量:引物熵和质量是决定引物热稳定性的重要因素;
6. 避免引物的折叠卷曲:当引物的长度大于20个碱基时,它可能会向内折叠,从而影响它的热稳定性;
7. 注意引物的质量控制:引物的质量应在良好的水平,以便有效检测。