新一代高通量测序技术SOLiD简介
- 格式:doc
- 大小:33.50 KB
- 文档页数:3


新一代高通量测序技术SOLiD简介目前市场上有四种高通量测序仪,分别是Solexa,454 (GS-FLX),SOLiD和Polonator。
根据测序原理,它们可以被分为两大类:使用合成法测序(Sequencing by Synthesis)的Solexa和454,及使用连接法测序(Sequencing by Ligation)的Polonator和SOLiD。
这些高通量测序仪的共同点是不需要大肠杆菌系统进行DNA模板扩增,且测序所得序列较短:其中的454序列最长,为200~300个碱基,其余三种序列都只有几十个碱基。
测序原理及序列长度的差异决定了各种高通量测序仪具有不同的应用领域。
这就要求我们在熟悉各种高通量测序仪内在技术特点的基础上进行选择。
基因组所引进的SOLiD (Sequencing by Oligonucleotide Ligation and Detection)是ABI(Applied Biosystems)公司生产的高通量测序仪。
目前这台SOLiD运行稳定,SOLiD实验及数据分析小组也可以为大家提供专业的技术服务。
所以接下来的关键是如何把SOLiD测序仪应用到符合其技术特点的科研项目中。
本短文将简单介绍SOLiD测序流程,双碱基编码原理及数据分析原理,以帮助大家了解SOLiD测序仪的技术特点和应用范围。
1.SOLiD关键技术及其原理SOLiD使用连接法测序获得基于“双碱基编码原理”的SOLiD颜色编码序列,随后的数据分析比较原始颜色序列与转换成颜色编码的reference序列,把SOLiD颜色序列定位到reference上,同时校正测序错误,并可结合原始颜色序列的质量信息发现潜在SNP位点。
1.1. SOLiD文库构建使用SOLiD测序时,可根据实际需要,制备片段文库(fragment library)或末端配对文库(mate-paired library)。
简单地说,制备片段文库就是在短DNA片段(60~110 bp)两端加上SOLiD 接头(P1、P2 adapter)。



新一代测序技术(下)-SOLiD (ABI)摘要:2005年454公司首推划时代的新一代测序仪,从而引发了测序市场上454、Illumina、ABI等公司在新一代测序技术上的比拼高潮。
也正是这种你追我赶,让绘制人类全基因组图谱由过去的耗费亿美元和13年时间,骤然缩短到如今SOLiD 3运行一次即获得50GB可定位测序数据。
生物通6月重磅推出新一代测序技术专题,将逐个为你解析Illumina、ABI、Roche(454)的新一代测序仪,并汇集世界各大基因组中心诸多牛人对新一代测序仪的选择标准及使用反馈。
敬请期待!过去20年,美国应用生物系统公司(ABI)在测序方面一直占据着垄断地位。
自公司的共同创始人Leroy Hood在上世纪80年代中期设计了第一台自动荧光测序仪之后,生命科学研究就摆脱了手工测序的繁琐和辛劳,骄傲地迈入自动测序的新时代。
直到2005年,454推出了FLX焦磷酸测序平台,ABI的领先地位开始有些动摇。
之后,ABI迅速收购了一家测序公司——Agencourt Personal Genomics,并在2007年底推出了SOLiD 新一代测序平台。
从SOLiD 到如今的SOLiD 3,短短一年多时间,它已经上演了一出精彩的“一级方程式赛车”。
SOLiD全称为supported oligo ligation detetion,它的独特之处在于以四色荧光标记寡核苷酸的连续连接合成为基础,取代了传统的聚合酶连接反应,可对单拷贝DNA片段进行大规模扩增和高通量并行测序。
就通量而言,SOLiD 3系统是革命性的,目前SOLiD 3单次运行可产生50GB 的序列数据,相当于17倍人类基因组覆盖度。
而其无以伦比的准确性、系统可靠性和可扩展性更让它从其他新一代测序平台中脱颖而出。
为什么SOLiD能轻松实现貌似不可能的任务?让生物通带你从测序原理入手,一探究竟。
SOLiD工作流程SOLiD系统能支持两种测序模板:片段文库(fragment library)或配对末端文库(mate-paired library)。


百泰派克生物科技
高通量测序
高通量测序技术,又称为二代测序技术(Next Generation Sequencing, NGS)是
在一代测序技术上发展而来的新一代测序技术。
高通量测序技术克服了一代测序技术通量低、成本高的缺点,使同时分析几十万到几百万条序列成为现实,在大幅度提高通量的同时,保证了测序结果的准确性,降低了测序成本。
高通量测序技术的问世使得我们能快速、细致、全面、深入的分析一个物种的基因组和转录组序列,揭开这些遗传物质的神秘面纱。
随着高通量测序技术的发展,不同公司相继开发了不同的测序平台,如Roche公司的454测序平台、Illumina公司的Solex测序平台以及ABI公司的Solid平台等。
高通量测序平台在测序通量以及速度上有着显著的优势,使其成为当前基因组以及转录组测序的首选技术。
百泰派克生物科技基于Illumina高通量测序平台提供高效快速的转录组高通量测
序服务,可在单核苷酸水平上检测任何物种的整体转录水平,在分析转录本的结构和表达水平的同时,还可以发现未知的转录本和稀有的转录本,并能准确识别可变剪切位点和编码序列单核苷酸多态性(cSNP),从而提供最全面的转录组信息,欢
迎免费咨询。

高通量测序技术及其在微生物学研究中的应用一、本文概述随着生物技术的飞速发展,高通量测序技术(High-Throughput Sequencing Technology,HTS)已经成为微生物学研究领域的重要工具。
其原理基于下一代测序(Next Generation Sequencing,NGS)技术,通过并行化处理和大规模测序,实现了对生物样本中DNA或RNA序列的高效、快速、低成本测定。
本文旨在全面介绍高通量测序技术的基本原理、技术特点以及在微生物学研究中的广泛应用,包括但不限于基因组测序、转录组测序、宏基因组测序等方面,以期对广大科研工作者和学者在这一领域的深入研究提供有益的参考和启示。
我们将对高通量测序技术的基本原理进行阐述,包括测序平台的选择、样本制备、测序流程以及数据分析等关键环节。
接着,我们将重点介绍高通量测序技术在微生物学研究中的应用,包括基因组测序在微生物种类鉴定、基因组结构分析、进化关系研究等方面的应用;转录组测序在微生物基因表达调控、代谢途径解析、抗药性机制等方面的应用;以及宏基因组测序在环境微生物群落结构分析、生物多样性评估、新功能基因挖掘等方面的应用。
我们还将探讨高通量测序技术在微生物学研究中的优势和挑战,包括测序通量高、成本低、速度快等优势,以及数据分析复杂、生物信息解读困难等挑战。
我们将对高通量测序技术在微生物学研究中的未来发展趋势进行展望,以期为相关领域的研究提供有益的参考和借鉴。
二、高通量测序技术概述高通量测序技术,也被称为下一代测序技术(Next Generation Sequencing,NGS),是近年来生物信息学领域的一次重大技术革命。
该技术能够在短时间内对大量的DNA或RNA分子进行测序,显著提高了测序的通量和效率。
与传统的桑格测序法相比,高通量测序技术具有更高的测序速度、更低的成本和更高的准确性,因此被广泛应用于各种生物学研究中。
高通量测序技术的基本原理是边合成边测序。


一、概述固态纳米孔测序(solid-state nanopore sequencing)是一种新兴的高通量DNA测序技术。
该技术通过利用纳米孔对DNA单链进行电信号检测,实现对DNA序列的测定。
相较于其他测序技术,固态纳米孔测序具有速度快、精准度高、成本低等优势,因此备受关注。
二、材料固态纳米孔测序主要需要以下材料:1. 固态纳米孔:固态纳米孔是指由固体材料制成的纳米尺寸的通道。
目前常用的固态纳米孔有氮化硅(SiN)和氧化铝(Al2O3)等。
这些材料具有良好的生物相容性和化学稳定性,能够有效防止电解液中的离子产生反应而破坏纳米孔结构。
2. DNA分子:DNA分子是固态纳米孔测序的待测样品。
DNA分子可以通过PCR扩增、化学合成等方式获取。
在进入纳米孔之前,需要将DNA分子加工成单链形式,并加上适当的标签以便于检测。
3. 离子电解液:离子电解液是指在纳米孔两侧的电极之间加入的液体。
电解液中的离子可以通过纳米孔形成的通道进行传输,从而产生电信号。
常用的电解液有KCl、NaCl等,其中KCl是最为常用的。
4. 电极:电极用于施加电压和检测电信号。
电极一般由金属制成,如银、铝、铜等。
电极需要与纳米孔上下游相连,以便于电信号的检测和控制。
5. 电子设备:电子设备包括放大器、数据采集卡、计算机等。
这些设备用于放大和记录电信号,并进行数据处理和分析。
三、工作原理固态纳米孔测序的工作原理基于DNA分子通过纳米孔时的电信号变化。
简单来说,当DNA分子通过纳米孔时,会改变纳米孔内的离子浓度分布,导致纳米孔两侧的电势差发生变化。
该变化可以通过电极检测到,并转化为数字信号存储和处理。
根据这个原理,固态纳米孔测序需要在以下几个方面进行优化:1. 纳米孔尺寸和形状:纳米孔的尺寸和形状会直接影响到电信号变化的大小和形态。
一般来说,纳米孔的直径应该与DNA单链的直径相当,并且需要具有适当的深度和宽度,以便于DNA分子的通过和检测。
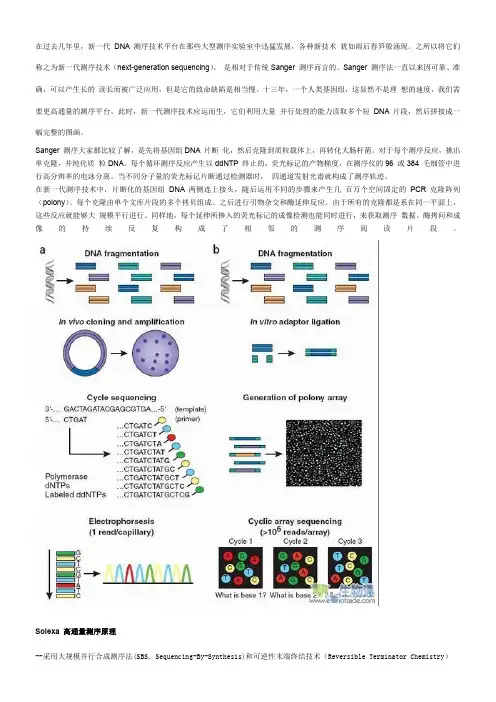
在过去几年里,新一代DNA 测序技术平台在那些大型测序实验室中迅猛发展,各种新技术犹如雨后春笋般涌现。
之所以将它们称之为新一代测序技术(next-generation sequencing),是相对于传统Sanger 测序而言的。
Sanger 测序法一直以来因可靠、准确,可以产生长的读长而被广泛应用,但是它的致命缺陷是相当慢。
十三年,一个人类基因组,这显然不是理想的速度,我们需要更高通量的测序平台。
此时,新一代测序技术应运而生,它们利用大量并行处理的能力读取多个短DNA 片段,然后拼接成一幅完整的图画。
Sanger 测序大家都比较了解,是先将基因组DNA 片断化,然后克隆到质粒载体上,再转化大肠杆菌。
对于每个测序反应,挑出单克隆,并纯化质粒DNA。
每个循环测序反应产生以ddNTP 终止的,荧光标记的产物梯度,在测序仪的96 或384 毛细管中进行高分辨率的电泳分离。
当不同分子量的荧光标记片断通过检测器时,四通道发射光谱就构成了测序轨迹。
在新一代测序技术中,片断化的基因组DNA 两侧连上接头,随后运用不同的步骤来产生几百万个空间固定的PCR 克隆阵列(polony)。
每个克隆由单个文库片段的多个拷贝组成。
之后进行引物杂交和酶延伸反应。
由于所有的克隆都是系在同一平面上,这些反应就能够大规模平行进行。
同样地,每个延伸所掺入的荧光标记的成像检测也能同时进行,来获取测序数据。
酶拷问和成像的持续反复构成了相邻的测序阅读片段。
Solexa 高通量测序原理--采用大规模并行合成测序法(SBS, Sequencing-By-Synthesis)和可逆性末端终结技术(Reversible Terminator Chemistry)--可减少因二级结构造成的一段区域的缺失。
--具有高精确度、高通量、高灵敏度和低成本等突出优势--可以同时完成传统基因组学研究(测序和注释)以及功能基因组学(基因表达及调控,基因功能,蛋白/核酸相互作用)研究----将接头连接到片段上,经PCR 扩增后制成Library 。
solid测序技术原理Solid测序技术是一种高通量的测序技术,它的原理是将DNA样品分离成单个的DNA分子,并通过特殊的化学处理和光学检测方法,将DNA的碱基序列逐个确定下来。
Solid测序技术的原理可以分为四个步骤:DNA片段制备、DNA连接、DNA扩增和测序。
第一步是DNA片段制备。
在这一步中,DNA样品会经过特殊处理,将其分离成短小的DNA片段。
这些DNA片段通常长度在100到200个碱基对之间。
这些片段是后续测序的基础,因为较短的片段更容易被处理和测序。
第二步是DNA连接。
在这一步中,DNA片段会被连接到固定的玻璃片上。
连接的过程会使用特殊的连接酶,将DNA片段与玻璃片上的连接位点相连接。
连接的目的是将DNA片段固定在特定位置,以便后续的处理和测序。
第三步是DNA扩增。
在这一步中,DNA片段会被扩增成大量的复制品。
扩增的过程使用聚合酶链式反应(PCR)技术,通过多次复制和扩增,将DNA片段的数量增加到足够多的水平。
这样可以确保在后续的测序过程中有足够的信号产生,并提高测序的准确性和可靠性。
第四步是测序。
在这一步中,DNA片段的碱基序列会被逐个确定下来。
测序的过程使用特殊的荧光染料标记的核苷酸,通过检测荧光信号的强度和位置,确定DNA片段中每个碱基的序列。
这一步需要高度敏感的光学设备和复杂的数据处理算法,以确保测序的准确性和可靠性。
Solid测序技术的原理优点是高通量、高准确性和高可靠性。
由于其高通量的特点,可以同时测序多个DNA样品,大大提高了测序的效率。
同时,由于其高准确性和高可靠性,可以对DNA片段的碱基序列进行精确的确定,避免了传统测序技术中的错误和偏差。
总结一下,Solid测序技术的原理是将DNA样品分离成单个的DNA分子,并通过特殊的化学处理和光学检测方法,将DNA的碱基序列逐个确定下来。
这种技术具有高通量、高准确性和高可靠性的特点,可以广泛应用于基因组学、遗传学和生物医学研究等领域。
solid测序技术原理
Solid测序技术是一种新型的DNA测序技术,由华盛顿大学科学家Dr.Stephen Quake开发于2007年。
Solid测序技术采用的是一种改进的羟基偶联反应(Hydroxyl Radical Coupled Reaction,或简称HRC)研究方法,它能够精确地识别出DNA的序列,称为序列品质(sequence quality),并且能够有效地检测到基因的变异。
Solid测序的基本原理是将DNA序列的每个核苷酸配对到一个模板核酸上,然后利用HRC反应。
HRC反应是一种使用氧化物进行DNA序列分析的技术,它可以将氧化物连接到基因物质,从而改变其化学结构,从而可以检测到基因序列中变异的部分。
这一反应会产生四种不同的氧化物,每一种氧化物都会对应不同的DNA碱基,以此来确定出DNA序列。
Solid测序的其他优势是它能够迅速地识别出基因的变异,以及它能够检测到有效的大量DNA序列。
Solid测序还有很多好处,它可以测量出保守的碱基,以及检测出一系列基因序列的变异。
因为Solid测序不需要任何大量的消耗材料,所以它也被定义为“节约型”技术。
Solid测序技术比其他DNA测序技术更加准确、快速、可重复性更强、成本更低,所以它在各个领域,如医药基因组学、细胞分子生物学研究中的应用实用性大大提升。
目前,Solid测序技术已经成为医学研究领域中被广泛应用的一种新型DNA测序技术。
- 1 -。
solid测序技术原理引言solid测序技术(Sequencing by Oligonucleotide Ligation and Detection)是一种高通量测序技术,被广泛应用于基因组学、遗传学、癌症研究等领域。
本文将详细介绍solid测序技术的原理和应用。
solid测序技术概述solid测序技术是一种二代测序技术,首次由Applied Biosystems公司于2007年发布。
与其他二代测序技术相比,solid测序技术具有高准确性、高产出和高通量的优势。
solid测序技术流程solid测序技术主要包括DNA片段化、连接适配体、抗体依附、扩增和测序等步骤。
下面将对每个步骤进行详细介绍。
DNA片段化首先,需要将待测序的DNA样本进行片段化处理。
这可以通过机械切割、酶切或超声波破碎等方式实现。
连接适配体接下来,将DNA片段与适配体连接。
适配体是一种短的DNA序列,包含引物序列和特定的标识序列。
适配体的引物序列用于后续扩增和测序,标识序列用于将不同的DNA片段区分开来。
抗体依附连接适配体后,将DNA片段进行抗体依附。
抗体是一种特异性结合DNA的分子,可以用来捕获和固定DNA片段。
扩增抗体依附后,进行PCR扩增。
通过多轮PCR扩增,可以生成大量的DNA复制物。
每一轮PCR都会在适配体的引物上添加特定的标记序列,增加测序的准确性。
测序准备好扩增的DNA后,进行solid测序。
solid测序使用特殊的测序引物,通过多轮测序循环,在每一轮循环中,测序引物与DNA复制物首尾相连,然后通过荧光标记的碱基识别和离婺法确定DNA序列。
solid测序技术优势solid测序技术相比其他测序技术具有以下优势:1.高准确性:solid测序技术的错误率低于其他二代测序技术,可以达到0.1%以下。
2.高产出:solid测序技术可以同时测序数以亿计的DNA片段,产出高达几百Gb的测序数据。
3.高通量:solid测序技术的平台基于微阵列技术,可以并行测序大量的DNA片段,提高测序效率。
solid 测序原理
固体测序(Solid sequencing)是一种基于不同碱基特异荧光信
号的测序技术,其原理是在固体表面上固化 DNA 库,通过链
扩增和测序反应来确定 DNA 序列的碱基。
该技术与 Illumina
测序相似,但不同之处在于固体测序使用的是短 DNA 片段的
端开环化进行测序,而不是像 Illumina 测序那样使用 DNA 片
段的终端加入。
固体测序需要将目标 DNA 片段连接到载体 bead 上,然后将bead 固定在测序芯片上。
随后,每个 bead 内的 DNA 被链扩增,以产生许多属于同一 DNA 片段的 copies,这些 copies 都
被夹缝在一个相当微小的区域内。
在测序反应中,特定的碱基会引起一个特定的荧光信号,在该信号被检测后,DNA 片段
的下一个碱基就被反应掉,并且所有荧光信号都同时记录下来,以生成一条 DNA 片段的读取。
这种测序技术的主要优点是读取长度相对较长,同时也比其他测序技术更适合整个基因组组装。
然而,该技术需要高质量的输入 DNA 和高适应的计算资源,因此在某些情况下使用成本
比较高。
solid 测序原理
固体阵列测序(solid sequencing)是一种基于合成DNA片段
的测序技术,使用这种技术可以将一小部分DNA样品转换成
大量的DNA片段。
这些片段可以被分配到数百万个独立的区域,从而成为一个高通量的、平衡地管理的测序反应。
图像信号处理也是solid测序技术中的一个重要部分,这种技
术会在每个测序循环的末尾记录下荧光信号的强度。
通过波长分解,将荧光信号分解成符合序列基序的4个荧光染料信号。
最终,每个固定的DNA链基因由荧光信号的存在或缺失生成,从而得到DNA测序结果。
该技术的一个优势是具有很高的准确性,可以达到小于1%的
误差率。
另一个优势是可以产生很长的读长(长相同的重复段被测序多次以获得更高的准确性),最长可以达到100bp。
这
使得solid测序在一些应用中有着很好的应用前景,尤其是在
基因定位和重组研究中。
solid测序技术原理
Solid测序技术原理
Solid测序技术是一种高通量测序技术,被广泛应用于基因组学、转录组学和表观基因组学等领域。
它的原理基于串联测序和图像分析,能够高效地获取DNA或RNA序列信息。
在Solid测序技术中,首先需要将DNA或RNA样本进行文库构建,即将DNA或RNA片段连接到测序适配器上。
接下来,文库中的DNA或RNA片段会被固定在微小的固相颗粒上,形成单一分子单端序列。
然后,这些固相颗粒会被固定在晶格中,使每个颗粒都可以被分配一个独特的标识序列。
在测序过程中,首先会进行两轮的引物扩增,通过引物的连接来标记每个固相颗粒上的DNA或RNA片段。
然后,通过加入荧光标记的核苷酸,可以标记每个碱基的序列。
接下来,通过固定在玻璃芯片上的固相颗粒,利用荧光显微镜观察每个颗粒上的荧光信号,并记录下来。
这样就可以得到每个碱基的序列信息。
Solid测序技术的原理在于通过不断循环的荧光标记和观察,可以逐个碱基地确定DNA或RNA片段的序列。
由于每个固相颗粒都带有独特的标识序列,可以保证测序结果的准确性和可靠性。
此外,Solid测序技术还可以通过多重引物扩增和并行测序的方式,大大提高测序的效率和吞吐量。
总的来说,Solid测序技术的原理是基于串联测序和图像分析,通过固相颗粒上的固定和荧光标记,可以高效地获取DNA或RNA片段的序列信息。
这种高通量测序技术在基因组学、转录组学和表观基因组学等领域有着广泛的应用,为研究人员提供了强大的工具和技术支持。
新一代高通量测序技术SOLiD简介目前市场上有四种高通量测序仪,分别是Solexa,454 (GS-FLX),SOLiD和Polonator。
根据测序原理,它们可以被分为两大类:使用合成法测序(Sequencing by Synthesis)的Solexa和454,及使用连接法测序(Sequencing by Ligation)的Polonator和SOLiD。
这些高通量测序仪的共同点是不需要大肠杆菌系统进行DNA模板扩增,且测序所得序列较短:其中的454序列最长,为200~300个碱基,其余三种序列都只有几十个碱基。
测序原理及序列长度的差异决定了各种高通量测序仪具有不同的应用领域。
这就要求我们在熟悉各种高通量测序仪内在技术特点的基础上进行选择。
基因组所引进的SOLiD (Sequencing by Oligonucleotide Ligation and Detection)是ABI(Applied Biosystems)公司生产的高通量测序仪。
目前这台SOLiD运行稳定,SOLiD实验及数据分析小组也可以为大家提供专业的技术服务。
所以接下来的关键是如何把SOLiD测序仪应用到符合其技术特点的科研项目中。
本短文将简单介绍SOLiD测序流程,双碱基编码原理及数据分析原理,以帮助大家了解SOLiD测序仪的技术特点和应用范围。
1.SOLiD关键技术及其原理SOLiD使用连接法测序获得基于“双碱基编码原理”的SOLiD颜色编码序列,随后的数据分析比较原始颜色序列与转换成颜色编码的reference序列,把SOLiD颜色序列定位到reference上,同时校正测序错误,并可结合原始颜色序列的质量信息发现潜在SNP位点。
1.1. SOLiD文库构建使用SOLiD测序时,可根据实际需要,制备片段文库(fragment library)或末端配对文库(mate-paired library)。
简单地说,制备片段文库就是在短DNA片段(60~110 bp)两端加上SOLiD 接头(P1、P2 adapter)。
而制备末端配对文库,先通过DNA环化、Ecop15I酶切等步骤截取长DNA片段(600bp到10kb)两末端各25 bp进行连接,然后在该连接产物两端加上SOLiD接头。
两种文库的最终产物都是两端分别带有P1、P2 adapter的DNA双链,插入片段及测序接头总长为120~180 bp。
1.2:油包水PCR我们知道,文库制备得到大量末端带P1、P2 adapter但内部插入序列不同的DNA双链模板。
和普通PCR一样,油包水PCR也是在水溶液进行反应,该水相含PCR所需试剂,DNA模板及可分别与P1、P2 adapter结合的P1、P2 PCR引物。
但与普通PCR不同的是,P1引物固定在P1磁珠球形表面(SOLiD将这种表面固定着大量P1引物的磁珠称为P1磁珠)。
PCR反应过程中磁珠表面的P1引物可以和变性模板的P1 adapter负链结合,引导模板合成,这样一来,P1引物引导合成的DNA链也就被固定到P1磁珠表面了。
油包水PCR最大的特点是可以形成数目庞大的独立反应空间以进行DNA扩增。
其关键技术是“注水到油”,基本过程是在PCR反应前,将包含PCR所有反应成分的水溶液注入到高速旋转的矿物油表面,水溶液瞬间形成无数个被矿物油包裹的小水滴。
这些小水滴就构成了独立的PCR 反应空间。
理想状态下,每个小水滴只含一个DNA模板和一个P1磁珠,由于水相中的P2引物和磁珠表面的P1引物所介导的PCR反应,这个DNA模板的拷贝数量呈指数级增加,PCR反应结束后,P1磁珠表面就固定有拷贝数目巨大的同来源DNA模板扩增产物。
A BI公司提供的SOLiD 实验手册已经把小水滴体积及水相中DNA模板和磁珠的个数比等重要参数进行了技术优化和流程固定,尽可能提高“优质小水滴”(水滴中只含一个DNA模板一个P1磁珠)的数量,为后续SOLiD 测序提供只含有一种DNA模板扩增产物的高质量P1磁珠。
1.3.含DNA模板P1磁珠的固定SOLiD测序反应在SOLiD玻片表面进行。
含有DNA模板的P1磁珠共价结合在SOLiD玻片表面。
磁珠是SOLiD测序的最小单元。
每个磁珠SOLiD测序后形成一条序列(具体SOLiD测序过程请见图5)。
1.4. SOLiD双碱基编码原理及测序流程SOLiD“双碱基编码原理”实质上是阐明了荧光探针的颜色类型与探针编码区碱基对的对应关系。
SOLiD连接反应的底物是8碱基单链荧光探针混合物。
连接反应中,这些探针按照碱基互补规则与单链DNA模板链配对。
如图1“底物探针”所示,探针5’末端可分别标记“CY5,Texas Red,CY3,6-FAMTM”4种颜色的荧光染料,并且这四种颜色用数字“3,2,1,0”示意;探针3’端1~5位为随机碱基,可以是“A,T,C,G”四种碱基中的任何一种碱基,其中第1、2位构成的碱基对是表征探针染料类型的编码区,“双碱基编码矩阵”规定了该编码区16种碱基对和4种探针颜色的对应关系,而3~5位的“n”表示随机碱基,6~8位的“z”指的是可以和任何碱基配对的特殊碱基,由上可知,SOLiD连接反应底物中共有45 种底物探针。
单向SOLiD测序包括五轮测序反应。
每轮测序反应含有多次连接反应(一般情况下,片段文库是7次,mate-paired文库是5次,所以片段文库共有35个连接反应,而末端配对文库共有25次连接反应)。
每轮测序反应的第一次连接反应由与P1引物区域互补的“连接引物”介导。
这五种连接引物长度相同,但在P1引物区域的位置相差一个碱基(分别用n,n-1,n-2,n-3,n-4表示),都含有5’端磷酸,所以可以介导连接反应的进行。
现以图5所示一个磁珠上发生的SOLiD测序反应为例进行说明。
第一轮测序的第一次连接反应由连接引物“n”介导,由于每个磁珠只含有均质单链DNA模板(也就是每个磁珠表面的单链DNA模板序列都是一样的),所以这次连接反应掺入一种8碱基荧光探针,SOLiD测序仪记录反应模板序列第1、2位碱基序列的探针第1、2位编码区颜色信息,随后的化学处理断裂探针3’端第5、6位碱基间的化学键,并除去6~8位碱基及5’末端荧光基团,暴露探针第5位碱基5’磷酸,为下一次连接反应作准备。
由此我们知道第一次连接反应使合成链多了5个碱基,所以第二次连接反应得到反应模板序列第6、7位碱基序列的颜色信息,而第三次连接反应得到的是第11、12位碱基序列的颜色信息… … 以此类推,第一轮测序反应获取了模板链7个碱基对的颜色信息。
如图5所示,由于第二轮连接引物n-1比第一轮错开一位,所以第二轮得到是以0,1位起始的7个碱基对的颜色信息。
五轮测序反应反应后,按照第0、1位,第1、2位... …的顺序把对应于模板序列的颜色信息连起来,就得到由“0,1,2,3”组成的SOLiD原始颜色序列。
1.5. 数据分析原理SOLiD测序完成后,获得了由颜色编码组成的SOLiD原始序列(图6.a)。
理论上来说,按照“双碱基编码矩阵”(图4),只要知道所测DNA序列中任何一个位置的碱基类型,就可以将SOLiD 原始颜色序列“解码”成碱基序列。
但由于双碱基编码规则中双碱基与颜色信息的兼并特性(一种颜色对应4种碱基对),前面碱基的颜色编码直接影响紧跟其后碱基的解码,所以一个错误颜色编码就会引起“连锁解码错误”,改变错误颜色编码之后的所有碱基(图6.1)。
和所有其它测序仪一样,测序错误在所难免,关键是对测序错误的评价和后续处理。
为避免“连锁解码错误”的发生,SOLiD数据分析软件不直接将SOLiD原始颜色序列解码成碱基序列,而是依靠reference序列进行后续数据分析。
SOLiD序列分析软件首先根据“双碱基编码矩阵”把reference碱基序列转换成颜色编码序列,然后与SOLiD原始颜色序列进行比较,来获得SOLiD 原始颜色序列在reference的位置,及两者的匹配性信息。
Reference转换而成的颜色编码序列和SOLiD原始序列的不完全匹配主要有两种情况:“单颜色不匹配”和“两连续颜色不匹配”(图6)。
由于每个碱基都被独立地检测两次(图5),且SNP位点将改变连续的两个颜色编码(图6.2),所以一般情况下SOLiD将单颜色不匹配处理成测序错误,这样一来,SOLiD分析软件就完成了该测序错误的自动校正;而连续两颜色不匹配也可能是连续的两次测序错误,SOLiD分析软件将综合考虑该位置颜色序列的一致性及质量值来判断该位点是否为SNP。
2.SOLiD测序技术的应用2.1. 基因组测序全基因组重测序。
研究者可以基因组DNA为初始样本构建SOLiD文库(fragment文库及mate-paired文库),以恰当的全基因组序列为reference,可以快速鉴定SNP,indel及基因组结构变化。
特定基因组区域测序。
除应用于传统的ChIP-seq,SOLiD技术平台还可以结合芯片技术,富集特定基因组序列进行深度测序,快速鉴定SNP。
其关键技术流程如下:SOLiD fragment文库经适当循环数PCR扩增得到足量样品DNA(约30ug), 文库扩增产物与Agilent芯片(或其它自订制芯片)杂交,然后对芯片探针紧密结合的洗脱产物进行常规Emulsion PCR及SOLiD测序。
SOLiD结合芯片技术对基因组特定区域的进行深度测序,可发现低频率SNP(如肿瘤样本中特定基因的体细胞突变)。
2.2. RNA-seq高通量测序仪的问世,使得测序成本大大降低,提供了不依赖现有基因模型的大规模基因表达谱研究手段,促进了针对细胞全部转录产物(small RNA 等non-coding RNA,低拷贝protein-coding RNA及其可变剪接体)的深度挖掘及后续功能研究。
目前有两种SOLiD试剂盒促进SOLiD测序仪在转录组上的应用。
SOLiD small RNA 试剂盒以含5’段磷酸及3’段羟基的s mall RNA为初始样本,2天就可完成与SOLiD RNA特异adapter 连接,逆转录,PCR扩增等步骤, 得到SOLiD fragment 文库。
SOLiD whole transcriptome expression 试剂盒针对序列较长的non-coding RNA或mRNA。
该试剂盒使用RNA H将mRNA或去除rRNA 的总RNA片段化并回收酶切产物,其后实验流程和SOLiD s mall RNA完全相同。
这两种试剂盒以RNA为初始样本,并且所用的RNA 特异adapter方向确定,所以最后测序所得序列的方向也就确定了。
而传统方法大多以双链cDNA为初始样本,难以确定测序所得序列来自转录本的正义链还是反义链而干扰后续数据分析。