CRISPR-Cas9细胞-动物KOKI实验基本流程-2
- 格式:pdf
- 大小:357.95 KB
- 文档页数:9

基因编辑技术CRISPRCas9的详细操作步骤基因编辑技术CRISPR-Cas9是一种革命性的工具,能够精确切割和修改DNA 序列。
它已被广泛应用于实验室研究、农业、医学和生物技术领域。
CRISPR-Cas9的操作步骤分为设计目标序列、构建修饰体、细胞转染、筛选、验证等几个阶段。
以下是基因编辑技术CRISPR-Cas9的详细操作步骤:1. 设计目标序列首先,确定要编辑的基因序列。
识别目标区域,通常选择高度保守的DNA 区域,以最大程度减少非特异性修饰。
目标序列的设计需要遵循一些规则,例如避免重复、剪切酶的PAM序列靠近目标区域等。
2. 构建修饰体根据设计的目标序列,构建CRISPR修饰体。
修饰体通常由CRISPR核酸(包括crRNA或sgRNA)和Cas9核酸组成。
crRNA(或sgRNA)负责定位到目标序列,Cas9则负责剪切目标序列。
合成修饰体的DNA或RNA序列后,可以使用PCR扩增或化学合成的方法得到修饰体。
3. 细胞转染将构建好的修饰体转染至目标细胞中。
转染可以选择不同的方法,例如化学法、电穿孔法、超声波法或病毒载体等。
转染后,修饰体会逐渐进入目标细胞,并开始作用于基因组。
4. 筛选在转染后,大部分细胞仍然具有未被编辑的基因组。
为了筛选出完成编辑的细胞,可以使用筛选方法。
最常用的方法是通过引入荧光蛋白标记的修饰体,利用流式细胞术或显微镜检查标记的细胞。
5. 验证对于筛选出来的细胞,需要进行进一步的验证。
最常用的方法是通过测序确认基因组是否发生了预期的编辑。
此外,也可以使用T7E1酶切或限制性内切酶消化等方法,进一步验证编辑效果的准确性。
总结起来,基因编辑技术CRISPR-Cas9的详细操作步骤包括设计目标序列、构建修饰体、细胞转染、筛选和验证等几个阶段。
通过遵循这些步骤,研究人员可以实现对目标基因组的精确编辑,进而揭示基因功能、治疗遗传疾病,并在农业和生物技术领域开拓新的应用前景。

CRISPR-Cas9体系实验流程(2)一、材料试剂准备1)质粒:pSpCa9(BB)(AddgeneplamidID:42230),若实验用gRNA为PCR扩增纯化产物,则需要该质粒做为U6的模版。
pUC19(Invitrogen,cat.no.15364-011)可用于构建gRNA,若用PCR 产物进行转染,则需要该质粒来进行共转染,做为DNAcarrier。
上述三种质粒,根据实验选择gRNA的表达方式(PCR产物/单载体系统/双载体系统)来确定具体使用哪种。
2)超纯水,DNae/RNae-free(LifeTechnologie,cat.no.10977-023) 3)高保真聚合酶,KapaHiFi(KapaBioytem),PfuUltra(Agilen),HerculaeIIfuionpolymerae均可,只需保真效果好,扩增过程不产生突变。
4)TaqDNApolymeraewithtandardTaqbuffer(NEB,cat.no.M0273S)用于一般检测。
5)QIAquickgele某tractionkit(Qiagen,cat.no.28704)6)QIApreppinminiprepkit(Qiagen,cat.no.27106)7)FatDigetBbI(BpiI)(Fermenta/ThermoScientific,cat.no.FD1014),如需要将gRNA构建到pSpCa9(BB)-2A-GFP质粒上,则需要该酶。
8)T7DNAligaewith2某rapidligationbuffer(Enzymatic,cat.no.L602L).或者T4DNAligae,二者无区别。
12)T4polynucleotidekinae(NewEnglandBioLab,cat.no.M0201S)13)PlamidSafeATP-dependentDNae(Epicentre,cat.no.E3101K)14)Adenoine5′-triphophate,10mM(NewEnglandBioLab,cat.no.P0756S)15)SOCmedium(NewEnglandBioLab,cat.no.B9020S)二、实验流程或者人工手动选择,在目的区域5′-NGG(即PAM)上游20bp片段均可做为targetite,一般targetite可选在正义链或者反义链,二者均可。

基于CRISPR Cas9技术基因敲除小鼠(Cas9-KO)的制作方法一、CRISPR/Cas9靶向基因敲除小鼠制作的基本技术原理:通过CRISPR/Cas9基因敲除技术,crRNA通过碱基配对与tracrRNA(trans-activating RNA)结合,形成双链RNA。
这一tracrRNA:crRNA二元复合体指导Cas9蛋白在crRNA引导序列靶标的特定位点剪切双链DNA。
在与crRNA引导序列互补的位点,Cas9蛋白的HNH核酸酶结构域剪切互补链而Cas9 RuvC-like 结构域剪切非互补链,实现敲除目的基因的功能,制备基因敲除小鼠模型。
二、具体步骤如下:一)模型制作策略制作:利用生物信息学手段(NCBI&IMPC&MGI),分别仔细分析目的基因敲除后小鼠的生存能力及繁育能力,并结合邻近基因的影响,最终选择合适的敲除区域进行敲除方案的设计,出具相应的制作策略。
二)载体的设计和构建:使用麻省理工学院的CRISPR Design工具(/),依据中靶Score的高低及脱靶Score的高低设计一对长度为20bp的针对靶标DNA的寡聚核苷酸链序列用于制备sgRNA,并在该靶区域设计引物用于后续阳性小鼠的基因鉴定。
1、制备sgRNA的实验方法步骤:1)线性化pUC57-GDNA-T7载体中提pUC57-GDNA-T7载体,用BsaI线性化过夜。
胶回收保存备用。
2)引物退火及加磷酸将上下游引物(干粉)稀释,再进行引物退火及加磷酸。
3)连接&阳性菌落筛选取步骤二中的加磷酸产物与线性化载体pUC57-GDNA-T7进行连接,该连接反应在干式恒温器中进行。
对连接产物进行转化,涂板,37°C培养箱过夜培养。
再用PCR&测序的方法筛选阳性克隆,再将测序正确的克隆进行甘油菌保种,-80°C保存备用4)制备转录模板以构建好的sgRNA载体为模板进行PCR扩增,将PCR产物切胶回收,回收产物离心后倒掉上清留DNA沉淀,再溶解DNA。

《利用CRISPR-Cas9系统构建DUSP9基因敲除小鼠胚胎干细胞系》篇一一、引言在生物学研究中,基因敲除是一种重要的技术手段,能够精确地操控特定基因的表达,进而研究基因在生物体中的功能。
近年来,随着基因编辑技术的发展,尤其是CRISPR-Cas9系统的广泛应用,基因敲除技术得到了极大的推进。
本文旨在探讨利用CRISPR-Cas9系统构建DUSP9基因敲除小鼠胚胎干细胞系的方法和步骤,为进一步研究DUSP9基因的功能提供技术支持。
二、CRISPR-Cas9系统简介CRISPR-Cas9系统是一种基于细菌免疫系统的基因编辑技术,具有高效、精确的基因编辑能力。
该系统通过设计特定的RNA 引导序列与Cas9蛋白结合,形成复合物,识别并切割靶点DNA 序列,从而实现基因的敲入、敲除或替换等操作。
三、DUSP9基因敲除小鼠胚胎干细胞系的构建1. 靶点设计:首先需要确定DUSP9基因的敲除靶点,设计相应的sgRNA和Cas9蛋白序列。
通过分析DUSP9基因的序列,找到合适的位置作为靶点。
2. 胚胎干细胞系的获取:选取适合的小鼠胚胎干细胞系,确保其处于稳定的生长状态。
3. CRISPR-Cas9系统介导的基因编辑:将设计好的sgRNA和Cas9蛋白通过特定的方式(如质粒转染、病毒载体等)引入小鼠胚胎干细胞中。
在细胞内,sgRNA和Cas9蛋白形成复合物,识别并切割DUSP9基因的靶点。
4. 克隆筛选和鉴定:经过一段时间的培养和筛选,挑选出发生基因突变的小鼠胚胎干细胞克隆。
通过PCR、测序等分子生物学手段,鉴定克隆中DUSP9基因的敲除情况。
5. 细胞系建立:将鉴定成功的DUSP9基因敲除小鼠胚胎干细胞进行传代培养,建立稳定的细胞系。
四、实验结果与讨论通过上述步骤,我们成功构建了DUSP9基因敲除小鼠胚胎干细胞系。
在实验过程中,我们观察到CRISPR-Cas9系统对DUSP9基因的切割效率较高,成功获得了多株基因敲除克隆。

CRISPR/Cas9基因编辑技术具体步骤及方法CRISPR/Cas9 是一种能够对基因组的特定位点进行精确编辑的技术。
其原理是核酸内切酶Cas9蛋白通过导向性RNA(guide RNA, gRNA)识别特定基因组位点并对双链DNA进行切割,细胞随之利用非同源末端连接(Non homologous End Joining,NHEJ)或者同源重组(Homologous Recombination, HR)方式对切割位点进行修复,实现DNA水平基因敲除或精确编辑。
CRISPR基因敲除利用CRISPR / Cas9 进行单基因敲除目前研究最透彻、应用最广泛的II 型-CRISPR/Cas9 系统由两部分组成:1. 单链的guide RNA(single-guide RNA,sgRNA)2. 有核酸内切酶活性的Cas9 蛋白CRISPR/Cas9 系统利用sgRNA 来识别靶基因DNA,并引导Cas9 核酸内切酶剪切DNA(图1)。
当基因组发生双链DNA 断裂后,细胞通过非同源性末端接合(Non-homologous end joining, NHEJ) 将断裂接合,在此过程中,将随机引入N 个碱基的缺失或增加,若N 非3 的倍数,则目的基因发生移码突变,实表1 CRISPR/Cas9 基因敲除与RNAi 比较CRISPR过表达利用CRISPR / Cas9 进行单基因过表达通过修饰CRISPR/Cas9 系统中的一些元件,形成一种蛋白复合物-协同激活介质(SAM),可实现对多数细胞内源基因的特异性激活。
该系统灵活方便,为研究基因功能提供了极为便利的工具。
CRISPR-SAM 系统由三部分组成:1. 失去核酸酶活性的dCas9(deactivated Cas9)-VP64 融合蛋白2. 含2 个MS2 RNA adapter 的sgRNA3. MS2-P65-HSF1 激活辅助蛋白CRISPR-SAM 系统中的MS2-P65-HSF1 激活辅助蛋白就是SAM,全称为SynergisticActivation Mediator( 协同激活调节器),这也就是CRISPR-SAM 的命名由来。

CRISPR/Cas9敲除细胞系构建步骤及方法一、技术简介CRISPR/Cas9是最新出现的一种由RNA指导的Cas9核酸酶对靶向基因进行编辑的技术,也是目前研究最热的基因编辑技术。
由于其具有构建方法简单快捷、基因修饰效率高、成本低廉、实验周期短、适用范围广等诸多优点,目前已成功应用于人类细胞、斑马鱼、大/小鼠等多种动植物的基因组精确修饰。
二、实验流程1. 预实验1.1 Cas9导入细胞方法:尝试各种方法,如脂质体类转染、电转、慢病毒感染、腺病毒感染等,确定高效导入Cas9方法。
1.2 药物浓度预实验:降低后续阳性克隆筛选和检测工作难度。
1.3 单克隆培养情况:确认细胞是否可以单克隆培养。
2. 基因敲除(敲入)2.1 靶点设计:一般在不同转录产物的共同外显子上设计3个靶点,靶点位置尽量在基因CDS的前1/3,ATG之后,最好能破坏重要的domain和所有的转录产物isoform。
第一批合成构建3个,效果不佳或时间紧张的可一次构建6个。
2.2 载体构建和病毒包装:根据预实验结果,选择合适的普通载体或病毒载体(普通Cas9载体、慢病毒Cas9载体和腺病毒Cas9载体)。
2.3 内源活性筛选:转染细胞或感染细胞48h后,使用Puro或Blasticidin筛选48h,提取基因组DNA。
使用T7E1酶验证打靶载体的活性,将有效的突变型PCR产物测序验证。
2.4 Donor载体(基因敲入):根据筛选的gRNA靶点位置,构建Donor普通载体或腺病毒载体,共转染/感染Cas9-gRNA和Donor。
2.5 单克隆筛选:无限稀释到每孔1个细胞的数量,每株细胞铺至少2个96孔板。
细胞数量足够后,验证内源活性并送测。
2.6 获得突变型:如需纯合子,则可能需要重复步骤3-5。

⼿把⼿教你学会CRISPRCas9基因敲除技术(需要挑选成对的靶点⼀般在正义链和反义链上分。
h ttp:///a/214091994_177233(需要挑选成对的靶点⼀般在正义链和反义链上分别挑选靶点配对)C RISPR/Cas 是进⾏基因编辑的强⼤⼯具,可以对基因进⾏定点的精确编辑。
在向导 RNA(guide RNA, gRNA)和 Cas9 蛋⽩的参与下,待编辑的细胞基因组 DNA 将被看作病毒或外源 DNA,被精确剪切。
⼀、寻找⽬的基因的靶标使⽤在线设计⽹站 CRISPR direct,如需直接复制⽹址,可在⽣物学霸后台对话框回复 direct即可。
靶点挑选要点:1. 基因敲除靶点应设计在起始密码⼦附近(包括起始密码⼦)或者起始密码⼦下游的外显⼦范围内。
2. 不同Cas9/gRNA 靶点在基因敲除效率上有较⼤差异,因此同时设计构建2~3 个靶点的基因敲除载体再从中选出敲减效果较佳的靶点。
3. N1-N20NGG 靠近 PAM 的碱基对靶点的特异性很重要,前 7~12 个碱基的错配对 Cas9 切割效率影响较⼩。
设计好的靶点序列应在基因库中进⾏ BLAST 检测。
4. Cas9Nicknase 需要挑选成对的靶点。
⼀般在正义链和反义链上分别挑选相距 20~30bp 的靶点配对。
多对靶点的敲除效率常有较⼤差异。
由于基因敲除实验时间长,在正式对⽬的细胞进⾏敲除前对靶点进⾏验证和挑选⾮常必要。
⼆、插⼊⽚段设计插⼊寡核苷酸序列设计(必须 PAGE 纯化寡核苷酸):正向序列5’ACACCGNNNNNNNNNNNNNNNNNNNGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCGTT3’反向序列3’TGTGGCNNNNNNNNNNNNNNNNNNNCAAAATCTCGATCTTTATCGTTCAATTTTATTCCGATCAGGCAA5’插⼊⽚段的合成1. ⽤⽔将寡核苷酸稀释为 100 µM。

CRISPRCas9基因敲除原理及实验建议
CRISPR/Cas9基因敲除原理及实验建议
CRISPR Cas9已经成为了最受欢迎的基因编辑技术之⼀,在2016年的国⾃然基⾦中也有很多项⽬是关于 CRISPR Cas9的。
⽬前在市场上已经有很多Cas9的基因敲除试剂盒,这些试剂盒的操作流程较为简单,客户可让公司直接帮忙设计gRNA,乃⾄最后的载体验证全包。
公司会根据您的要求收取不同的费⽤,如果只是合成载体,不验证,那么会便宜些。
如果要合成载体同时⼜验证,那么价格⼜会贵⼀些。
下⾯是对Cas9基因敲除试剂盒的⼀个详细说明,我们可以从中了解Cas9基因敲除所需要的敲除原理,基本试剂,基本步骤和研究⽅法,希望对⼤家可以有帮助。
建议⼤家直接依靠公司来进⾏设计,因为⾃⼰做的话前期的摸索过程可能会长⼀些。
⽬前有在线的软件来设计gRNA,如张锋实验室推出的gRNA设计软件等,后⾯就是分⼦克隆⽅⾯的实验。
从实验条件和时间成本考虑,对于⼤部分的临床医⽣⽽⾔,选择试剂盒要⽐选择⾃⼰做载体进⾏验证要好得多。

基因敲除小鼠的实验流程1.设计基因敲除小鼠实验方案在开始实验之前,需要明确研究目的,确定需要敲除的基因,并设计相应的实验方案。
一般可以使用 CRISPR-Cas9 系统来实现基因敲除,在设计基因敲除实验方案时,需要选择合适的 sgRNA 序列,以及设计恰当的引物用于检测突变。
2.获得基因敲除小鼠的胚胎干细胞为了实现基因敲除,需要获得基因敲除小鼠的胚胎干细胞。
一种常用的方法是利用胚胎干细胞对外源DNA的高度易感性,将敲除基因的质粒DNA转染到小鼠胚胎干细胞中。
3.筛选敲除基因的胚胎干细胞株系将转染了敲除基因的胚胎干细胞以悬浮培养的方式进行培养,培养一段时间后,利用一定的筛选条件来筛选出含有敲除基因的胚胎干细胞株系。
筛选条件可包括对抗生素的使用或筛选标记基因的表达。
4.制备敲除基因小鼠的固定胚胎干细胞系通过体外培养,将敲除基因的胚胎干细胞系定植在培养皿上,培养数代以后,将其冻存,以备后续的实验使用。
5.实施敲除基因小鼠的胚胎干细胞基因改造将固定的胚胎干细胞系重新激活,转染 Cas9 和 sgRNA,利用CRISPR-Cas9 系统使这些细胞具有敲除基因的突变。
6.识别敲除基因的胚胎干细胞阳性克隆株对转染了 Cas9 和 sgRNA 的胚胎干细胞进行筛选,通过 PCR、Western blot、Southern blot等技术方法识别出敲除了目标基因的阳性克隆株。
7.将敲除基因的胚胎干细胞注入小鼠的早期胚胎取出已受精的小鼠卵母细胞,利用显微操作将敲除基因的胚胎干细胞注入到小鼠的早期胚胎中。
利用体外受精或者通过体内或体外的胚胎移植方式将基因敲除干细胞注入受体小鼠。
8.制备基因敲除小鼠的嵌合小鼠将已注入敲除基因的胚胎干细胞的受体小鼠进行嵌合以产生基因敲除小鼠。
嵌合可以通过体内或体外的胚胎移植方式进行。
9.筛选识别基因敲除小鼠对产生的嵌合小鼠进行筛选,确认敲除基因是否成功。
可以通过 PCR、Western blot、Southern blot等技术方法对小鼠体细胞或组织进行分析。
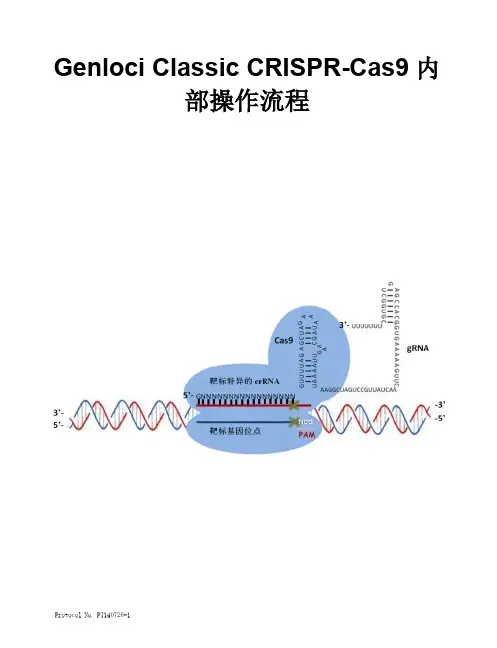
Genloci Classic CRISPR-Cas9内部操作流程Protocol No. PT140726-1Protocol No. PT140726-11,CRISPR-Cas9基因编辑实验流程图如下:pGK1.1U6 CACC G CAAA 4 55’ -5’3’-3’ -- ~20bp2,操作步骤实验前,请您务必做好以下验证实验:A.单细胞生长情况,确保单个细胞可以正常生长形成单克隆,即,低密度的细胞在培养皿中可以形成单克隆。
B.目的基因的表达情况分析,为防止因染色体缺失等情况导致靶基因缺失,首先需要PCR扩增靶基因,并对PCR结果进行测序,确保靶基因的完整存在;其次,您还可以用RT-PCR分析靶基因的活跃度。
1. 设计Oligo DNA序列首先,您需要在靶标DNA区域中设计一对20bp左右的oligo DNA,您可以通过以下在线工具设计:●麻省理工学院的CRISPR Design:/●德国癌症研究中心的E-Crisp:/E-CRISP/designcrispr下面我们选择麻省理工学院的CRISPR Design工具来做设计举例,以Fut8基因为例,一次只能输入大小为23~250bp的基因片段,最好一次只输入一个外显子,避免Guide序列跨内含子的。
点击“Download as genbank”按钮,出现以下界面:“Fut8”Array根据左边的score的高低选取合适的Guide序列,以Guide#1序列为例,2条单链oligo的序列如下(红色字体部分是要与Bbs I酶切后的载体相互补的部分):Fut8-F: cacc G AATGAGCATAATCCAACGCCFut8-R: aaac GGCGTTGGATTATGCTCATTC※注意:oligo DNA设计序列的第一个碱基必须是G,如果你选取的Guide序列的第一个碱基不是G,可自行加一个G上去。
另外,需在位点上下游各设计一条引物,用于后续PCR或测序检测阳性克隆,引物能扩增约300bp 的DNA片段,上游引物距突变位点约100bp,下游引物距突变位点约200bp。

基因编辑技术CRISPRCas9的操作步骤与技巧概述基因编辑技术CRISPR-Cas9是一种革命性的基因工程方法,它使得科学家们能够准确、高效地修改生物体的基因组。
CRISPR-Cas9系统利用CRISPR序列指导Cas9酶进行DNA切割,并借助细胞自然的修复机制来实现精确的基因修饰。
本文将介绍CRISPR-Cas9技术的操作步骤,以及实验中需要注意的技巧和注意事项。
操作步骤:1. 设计合适的gRNA(引导RNA)序列:gRNA是用来指导Cas9酶精确切割特定DNA序列的RNA分子。
设计一个合适的gRNA序列是CRISPR-Cas9技术操作的第一步。
确保目标DNA序列位点的选择和gRNA序列的设计遵循一定的规则和原则,以提高操作的效率和准确性。
2. 合成gRNA和Cas9蛋白:合成合适的gRNA是CRISPR-Cas9技术的关键。
根据设计的gRNA序列,合成相应的gRNA。
同时,合成Cas9蛋白或利用表达载体将Cas9蛋白表达进入细胞中。
3. 转染gRNA和Cas9蛋白:将合成的gRNA和Cas9蛋白转染到目标细胞中。
可以使用合适的转染试剂和方法,如化学转染、电转染或病毒转染等。
确保gRNA 和Cas9蛋白能够成功地进入细胞内。
4. DNA切割和修复:一旦Cas9与gRNA形成复合物进入细胞核,Cas9便能够准确地识别和切割目标DNA序列。
DNA的切割会引发细胞内自然的修复机制,可以选择使用非同源末端连接(NHEJ)或同源重组(HDR)来修复切割的DNA。
NHEJ修复机制常常带来插入缺失或错配,而HDR可以实现特定的DNA改造。
5. 筛选和检测:在进行CRISPR-Cas9基因编辑后,需要对细胞或生物体进行筛选和检测,以鉴定编辑效果和确认目标基因的改变。
可以利用PCR、Westernblot、DNA测序等方法验证基因的编辑和修饰效果,并在细胞或生物体水平进行相关功能性分析。
技巧和注意事项:1. 合适的gRNA选择:选择合适的gRNA序列对于CRISPR-Cas9技术的成功应用至关重要。
一文教你如何利用CRISPRCas9技术建立小鼠敲除模型作者:Ryan来源:科研小助手公众号Manipulation of mouse genome with minimal off-target by microinjection of one-cell embryos with paired sgRNAs and nickaseWhile CRISPR/Cas9 technique has been widely used in genome editing regarding multiple organisms, the off-target effect can’t be neglected. Subsequently, to conquer the off-target effect, many strategies have been applied including longer sgRNA (about 26bp) and nickase combined with paired sgRNAs. Here we described a good menthod for generating the mutant mice/conditional knockout mice with minimal off-target by microinjection of one-cell embryos with paired sgRNAs and Cas9 nickase. Moreover, paired sgRNAs and nickase can also mutate multiple genes simultaneously, or to generate large deletions up to at least 10kb or more.Comparison of Cas9 and nickase (Cas9D10A)Figure 1. Cas9 nickase strategy. Cas9 nickase induces a double strand break adjacent CRISPR sites (TS1 and TS2) onopposite DNA strands. Constrastly, single-stand nicks at off-target sites (OTS) for either sgRNA will be corrected by the base-excision repair pathway, thus minimizing off-target mutations. P, PAM site.Plasmidsused in the protocolT7-Nickase(Cas9-D10A):T7-sgRNA:ReagentsPlasmids: T7-Nickase(Cas9-D10A) and T7-sgRNAmMESSAGE mMACHINE®T7 Ultra kit (Ambion, AM1345)MEGAshortscript TM Kit(Ambion, AM1354)RNeasy Mini Kit (QIAGEN,74104)MEGAclear TM Kit(Ambion, AM1908)RNAsecure TM Reagent(Ambion, AM7005)QIAprep Spin Miniprep Kit(QIAGEN, 27104)MiniElute PCRPurification Kit (QIAGEN, 28004)BsaI (NEB, R0535S)AgeI (NEB, R0552S)DraI (TAKARA, D1037A)T4 DNA Ripid ligation Kit(NEB,M2200S)PMSG (Sansheng, China,50IU/ml in normal saline, Aliquot and store at -80℃)HCG (Sansheng, China,50IU/ml in normal saline, Aliquot and store at -80℃)EmbryoMax® Injection Biffer (Millipore, MR-095-10F)ProteinaseK(Merck,1245680100, 20 mg/ml in water, Aliquot and store at -20℃)Lysis buffer (10 μMTris-H Cl, 0.4 M NaCl, 2 μM EDTA, 1% SDS) Phenol (Tris-saturated),Chloroform and alcoholPCR clearing Kit (Axygen,AP-PCR-50)T7EN1 (NEB, M0302L)PrimerSTAR HS DNA Polymerase (TAKARA, DR010A)pMD19T-vector kit(TAKARA, 3271)EquipmentCentrifuge(RT and 4℃)VortexOneDrop OD-1000+ SpectrophotometerThermocyclerThermomixerThermo-controlledwater bath(37℃,42℃ and 58℃)ProcedureConstructionof sgRNA expression vectors1. Design of paired sgRNA oligos.Select paired sgRNAs in a tail-to-tail orientation and separated by 10-30 bp, which have the sequence 5’-CCN(52-72)GG.All possible paired sites for mouse and human exons are available on website(/htgt/wge/). For each sgRNA, the 5’-GGN(19)GGmotif is preferred, however, 5’-GN(20)GG or 5’-N(21)GG are also satisfactory. BLAT or BLAST the sgRNA target sites in UCSC or ENSEMBL genome browsers to find those with few or no highly related sites in the genome.Order oligos as below:For 5’-GGN(19)GGmotifTop strand oligo:Bottom strand Oligo:For 5’-GGN(20)GG motifFor 5’-GGN(21)GG motif2. Annealing oligos prior to cloning.4.5μl Top Oligo (100 μM)4.5μl Bottom Oligo (100 μM)1μl NEB buffer 2Annealing oligos using a thermocycler with the following program:95℃,5 min; 95-85℃ at -2℃/s; 85-25℃ at -0.1℃/s; hold at 4℃.3. Preparation of T7-sgRNA plasmid.2 μg T7-sgRNA plasmid.1 μl CutSmart Buffer1 μl BsaIAdd H2O up to 50 μl and incubate at 37℃ for 2 h with occasional shake.Purify the digeston product using MinElutePCR Purification Kit.4. Ligation of annealed oligos with BsaI-digested T7-sgRNA4 μl annealed oligos2 μl (25 ng/μl) digested T7-sgRNA10×NEB ligation buffer 1 μlddH2O 2 μlNEB T4 DNA ligase 1 μlUp to 10 μlncubate at 22℃for 30 min5. Transformation and plate on Kan+plate (50 μg/ml).6. Confirm correct Insertion of sgRNA oligosby sequencing using M13-47 primer.7. Mini-prep T7-sgRNA plasmid using QIAprepSpin Miniprep Kit.Transcriptionof sgRNAs in vitro1. Ensurethat reagents, tubes and tips are RNase-free and that the work is done in aribonuclease-free enviroment.2. Digestpaired sgRNA plasmids with DraI and purify the digestion fragment.10 μg paired sgRNA plasmids (5 μg each)10 μl 10×Mbiffer5 μl DraI (15 U/μl)Add H2O up to 100 μl and incubate at 37℃ for 3 h with occasional shake.Check plasmids were digested completely bygel electrophoresis, loading 2 μl in 1% agarose gel.Two bands (1621 and 1152 bp) will beobserved. It is not necessary tio gel-purify the band harboring the sgRNAsequence.Add 4 μl RNAsecure and incubate at 60 ℃ for 10 min in a thermomixer.Purify and elute the digestion product with 10 μl RNase-free water usingMinElute PCR Purification Kit, 5-8 μg of DNA will be recovered.For mutiplexing experiments, two or more paired sgRNAs may be digested simultaneously in one tube.Alternatively, the transcription template containing the T7 promoter sgRNAsequence may be prepared by PCR amplification from a bacterial colony using thefollowing primers and PCR program:sgRNA-For:5’-TCTCGCGCGTTTCGGTGATGACGGsgRNA-Rev:5’-AAAAAAAGCACCGACTCGGTGCCACTTTTTCProgram:94℃,5min; ((98 ℃ ,10s; 72-62℃, -1℃/cycle, 15s; 72 ℃, 30s) 10 cycles, (98℃, 10s;62 ℃, 15s; 72 ℃, 30s) 25 cycles); 72 ℃, 5 min; hold at 4℃.Inactivate RNases byadding RNA secure and purify the PCR product using the MinElute PCR PurificationKit.3. Invitro transcription of sgRNAs using MEGAshortscriptTMKit.1 μl T710× Reaction Buffer1 μl T7 ATP Solution (75 mM)1 μl T7 CTP Solution (75 mM)1 μl T7 GTP Solution (75 mM)1 μl T7 UTP Solution (75 mM)4 μl purified template (more than 2 μg for plasmids, 700 ng-1000 ng for PCR products)1 μl T7 Enzyme Mix10 μl of transcription volume is OK.Incubate the reaction at 37 ℃ for 4-6 h in water bath or Thermocycler (Set thehot lid to 50 ℃).Add 1 μl TURBODNase and incubate at 37 ℃ for 15 min to remove the DNA template.4. Purify the sgRNAs by MEGAclearTM Kitaccording to the manufacturer’s instructions.RNA elutionoption 2 in the manual is preferred.Precipitatewith 5 M Ammonium Acetate and ethanol.Resuspendthe pellet using the 30 μl RNase free water.20-50 μgRNA will be obtained depending on the quality of DNA template.5.Assess sgRNA yield using the One Drop OD-1000+Spectrophotometer (or equivalent) and sgRNA quality by gel electrophoresis. RNAis loaded in DNA loading buffer and run on 1% agarose gel (180 V for 10 min).6.Aliquot and store at -80 ℃. The sgRNAs are stablefor one year without freeze-thaw cycles.Transcription of Nickase (Cas9-D10A) in vitro1. Ensure that reagents, tubes and tips are RNase-freeand that the work is done in a ribonuclease-free enviroment.2. Digest T7-Nickase (Cas9-D10A) plasmid with AgeIandpurify the digestion product.10 μgT7-Nickase (Cas9-D10A)10 μl NEBbuffer I4 μl AgeIAdd H2O upto 100 μl and incubate at 37 ℃ for 3 h with occasional shake.Add 4 μlRNAsecure and incubate at 60 ℃ for 10 min in a thermomixer.Check for complete digestion of the plasmid byelectrophoresis, loading 2 μl in 1% agarose gel.Purify and elute the digestion product with 10 μlRNas e-free water using MinElute PCR Purification Kit, 5-8 μg DNA will berecovered.3.In vitro transcribe Cas9-D10A using mMESSAGE mMACHINE® T7 Ultra Kit according to the manufacturer’s instruction.4. Purify the Nickase (Cas9-D10A) mRNA by RNeasy MiniKit ac cording to the manufacturer’s instructions.5. Assess sgRNA yield using the One Drop OD-1000+Spectrophotometer (or equivalent) and sgRNA quality by gel electrophoresis. RNAis loaded in DNA loading buffer and run on a 1% agarose gel (180V for 10 min).A yield of 30-60 μg mRNA is expected.Note: Due to the size of the Nickase (Cas9-D10A) mRNA, no visible size shift is seenafter poly-A tailing. The mRNA quality is good if a smear is not observed.6. Aliquot and store at -80 ℃. Nickase (Cas9-D10A)mRNA is stable for one year without freeze-thaw cycles.Collection of zygotes1. Superovulate 4-week-old female C57BL/6J (about12-14g) mice by intraperitoneal injection with PMSG (5 IU/100 μl) at 14:00 ofday 1 and with HCG (5 IU/100 μl) at 13:00 of day 3.2. Cross superovulated females with males (C57BL/6J orCBA).3. Identify plugged females at 9:00 of day4. Collectone-cell embryos as decribed in Reyon, D. et al, 2012.Preparation of microinjection mixture1. Thaw aliquot of the Cas9-D10A mRNA and sgRNAs onice. Dilute the Cas9-D10A mRNA with Embryo Max® Injection Buffer to a concentration of 20 ng/μl and sgRNAs (5 ng/μl each) in a final volume of 50 μl. Pipette the mixture upand down several times2. Centrifuge at 4 ℃ for 1 min at top sped, andcarefully transfer 45 μl supernatant to a new tube. Always keep the tube onice.Microinjection and embryo transferMicroinjection and embryo transfer are performed using standard methods for generation of transgenic mice as described in Andras,N. et al., 2003, Cold Spring Harb Protoc. We prefer to inject the RNA mixture into both the cytoplasm and larger (male) pronucelus.Genotyping founders1. Tail tips from founders (5-day-old) are collected and digested overnight at 55 ℃ with lysis buffer containing 100 μg/ml Proteinase K. Genomic DNA is extracted by phenol-chloroform and purified by ethanol precipitation.2. Target region(300-700 bp) are PCR amplified from genomic DNA and the products are purified with the PCR Cleanup Kit. Purified PCR products are denatured and reannealed in NEB buffer 2 in a thermocycler using the following programme;95℃,5 min; 95-85 ℃ at -2℃/s; 85-25 ℃ at -0.1℃/s; hold at 4℃.3. Hybridized PCR products are digested with 0.5 μlT7EN1 at 37℃ for 30 min and separated by 2% agarose gel. Mutant founders will yield lower molecular weight cleavage bands.4. Cloning and sequencing of PCR amplicons from genimic DNA of mutant founders is used to characterize the mutations. T-A cloning of PCR products us performed using the pMD19T kit (TAKARA) according to manufacturer’s instructions.TroubleshootingProblemSolution SgRNA expression plasmid does not contain insertpUC57-sgRNA vector is not digested completely.Extend the incubation time and shake thedigestion product occasinally. Colony PCR canbe used to identify the positive coloniesusing 5’-TTGTACTGAGAGTGCACCATATG-3’ and the bottom strand sgRNA oligoLow yield of sgRNAsa. Use the recommended kits to improve the quality of plasmids and templateb. Increase the amount of template or use the PCR product as template. Electrophoresis of sgRNAs shows more than one band a. sgRNAs can form dimers. Always keep sgRNAson ice. A low amount of dimer will not affectthe function of sgRNA.b. DNA template is incompletely digested.Circular template can produce longertranscripts. Extend the incubation time and shake the digestion product occasionally. c. DNA template contamination. Add more TURBO DNase and extend incubation time.Cas9-D10A mRNA produces a smear on an agrose gel a. Use RNAsecure to inactivate RNase contaminationb. Use the recommended kits to improve thequality of the DNA template.(QIAGEN Mini-prepand PCR clean-up kits are recommended)Time Taken4 days for the construction of sgRNA expression vectors.1 day for the in vitro transcription and preparation of sgRNAs. 1 day for the in vitro transcription and preparation of Cas9-D10A mRNA.4 days for the superovulationb of females, collection of 1-cell embryos and microinjections1 week for the genotyping of founder animals.。
crisprcas9实验流程下载温馨提示:该文档是我店铺精心编制而成,希望大家下载以后,能够帮助大家解决实际的问题。
文档下载后可定制随意修改,请根据实际需要进行相应的调整和使用,谢谢!并且,本店铺为大家提供各种各样类型的实用资料,如教育随笔、日记赏析、句子摘抄、古诗大全、经典美文、话题作文、工作总结、词语解析、文案摘录、其他资料等等,如想了解不同资料格式和写法,敬请关注!Download tips: This document is carefully compiled by theeditor. I hope that after you download them,they can help yousolve practical problems. The document can be customized andmodified after downloading,please adjust and use it according toactual needs, thank you!In addition, our shop provides you with various types ofpractical materials,such as educational essays, diaryappreciation,sentence excerpts,ancient poems,classic articles,topic composition,work summary,word parsing,copy excerpts,other materials and so on,want to know different data formats andwriting methods,please pay attention!CRISPR-Cas9 实验流程如下:1. 设计 sgRNA:根据目标基因的序列,设计特异性的 sgRNA(single guide RNA)。
crisprcas9基因敲除技术流程下载温馨提示:该文档是我店铺精心编制而成,希望大家下载以后,能够帮助大家解决实际的问题。
文档下载后可定制随意修改,请根据实际需要进行相应的调整和使用,谢谢!并且,本店铺为大家提供各种各样类型的实用资料,如教育随笔、日记赏析、句子摘抄、古诗大全、经典美文、话题作文、工作总结、词语解析、文案摘录、其他资料等等,如想了解不同资料格式和写法,敬请关注!Download tips: This document is carefully compiled by theeditor.I hope that after you download them,they can help yousolve practical problems. The document can be customized andmodified after downloading,please adjust and use it according toactual needs, thank you!In addition, our shop provides you with various types ofpractical materials,such as educational essays, diaryappreciation,sentence excerpts,ancient poems,classic articles,topic composition,work summary,word parsing,copy excerpts,other materials and so on,want to know different data formats andwriting methods,please pay attention!CRISPR/Cas9基因敲除技术是一种革命性的基因编辑工具,广泛应用于基础研究、疾病模型构建和潜在的基因治疗领域。
CRISPRCas9基因编辑操作步骤及详细说明实验材料与方法一、细胞培养人宫颈癌细胞 HeLa,常规培养使用含 10% FBS 的 DMEM 培养基 ( 含 1.5 mg/L-Glutamine,100 U/mL Penicillin,100 μg/mL Streptomycin) 中,37ºC 5% CO2 饱和湿度培养箱中培养。
二、基因信息及双 gRNA 设计基因信息及分析1.hsa-mir-152 基因信息:pubmed2.hsa-mir-152 基因位于蛋白编码基因 COPZ2 内含子内,敲除hsa-mir-152 基因不会影响该蛋白编码3.hsa-mir-152 precursor 序列(87 bp):TGTCCCCCCCGGCCCAGGTTCTGTGATACACTCCGACTCGGGCTCTGGAGCAGTCAGTGCATGACAGAACTTGGGCCCGGAAGGACC双 gRNA 设计使用在线 gRNA 设计软件在 hsa-mir-152 precursor 基因组序列两侧设计双 gRNA注:dgRNA 即为双 gRNA.三、慢病毒侵染实验材料及试剂DMEM 培养基 + 10% FBSD-Hank’s SolutionTrypsin-EDTA Solution96 孔板24 孔板Lentivirus- 病毒液(GenePharma)步骤靶细胞侵染实验1.靶细胞铺板:24-well,加入2.5×105 cells/well(根据细胞种类调整),0.5 mL 完全培养基,37℃,5% CO2 过夜;2.稀释病毒:稀释液(靶细胞维持液培养基)400 μL + 终浓度 5 μg/mL Polybrene,将慢病毒原液按 1:9 加入到稀释液中;3.移去 Step1 中细胞培养液,加入 Step2 稀释后的病毒液,同时建立对照(blank、negative),37℃,5% CO2 过夜;4.12~24 小时移去细胞侵染后的病毒液,加入 0.5 mL 完全培养基,37℃,5% CO2 过夜;5.根据细胞状态和类型,如果必要分出 1/3~1/5,加入0.5 mL 完全培养基,继续培养 24~48 小时,荧光倒置显微镜下观察结果。
CRISPR-CAS9基因敲除原理
CRISPR/Cas9(Clustered Regularly Interspaced Short Palindromic Repeats)是最新出现的一种由RNA指导Cas核酸酶对靶向基因进行特定DNA修饰的技术。
CRISPR 是细菌和古细菌为应对病毒和质粒不断攻击而演化来的获得性免疫防御机制。
在这一系统中,crRNA (CRISPR-derived RNA)通过碱基配对与tracrRNA(trans-activating RNA)结合形成双链RNA,此tracrRNA/crRNA二元复合体指导Cas9蛋白在crRNA引导序列靶定位点剪切双链DNA达到对基因组DNA进行修饰的目的。
Cas9结合gRNA,gRNA 的长度约为80个核苷酸,包含两个区域:gRNA 5' 端前20个核苷酸对应于靶标DNA,能结合在靶DNA 上的约60个核苷酸(gRNA 长度取决于表达gRNA 的质粒)形成一个发夹结构,这个结构能帮助gRNA 与Cas9结合,并由此指导与DNA 的结合。
通过gRNA上的靶点序列,在目标基因组上找到靶点序列,并揭开双螺旋,Cas9将剪切DNA双链,造成DNA双链断裂。
Cas9使用简单,可满足多个靶点同时操作。
Insertion /deletion
HDR
gRNA
Cas9
Donor vector
基因敲除小鼠流程:。
一、CRISPR-Cas9 细胞基因敲除敲入实验基本操作流程1)设计sgRNA :1.1、确定待敲除基因的靶位点根据提供的物种、基因名称或者基因ID在NCBI或ENSEMBLE中进行查找。
找到该基因CDS 区,分析相应的基因组结构,明确CDS的外显子部分。
按照基因本身的性质,选择候选的待敲除位点,确定待敲除位点。
对于蛋白编码基因,如果该蛋白具重要结构功能域,可考虑将基因敲除位点设计在编码该结构域的外显子;如果不能确定基因产物性质,可选择将待敲除位点放在起始密码子A TG后的外显子上。
如果是microRNA,可以将待敲除位点设计在编码成熟microRNA的外显子或在编码成熟microRNA的外显子的5’和3’侧翼序列。
1.2、设计识别靶位点的一对DNA Oligos确定待敲除位点后,选择23-至250bp的外显子序列输入到在线免费设计sgRNA的软件Input 框中(/),然后进行设计运算,软件会自动输出sgRNA序列(网站设计一般很慢或数据输出不完整,可使用我的内部软件,2天内输出全部结果,无物种限制)。
一般地,基因特异的sgRNA模板序列为位于PAM序列(Protospacer AdjacentMotif)前间区序列邻近基序,这是一种见于crRNA分子的短核苷酸基序,可以被Cas9蛋白特异性识别并切割)的20个nt。
而PAM序列的特征为NGG(其中N为任意核苷酸)。
因此,sgRNA模板序列选择非常方便,即使没有软件,研究者也可手工进行选择。
不过,在线软件可以给出该序列在基因组中存在相似序列的情况,即可能的脱靶位点。
因此,利用在线软件可以选择脱靶机会小的序列作为sgRNA模板序列。
根据选择的sgRNA模板序列,合成一对序列互补的DNA Oligos (同时设计检测目的基因的引物一起合成)。
1、/2、/mpg/crispr_design/3、/~slin/cas9.html4、/E-CRISP/5、/6、/crispr/,Drosophila7、/index.jsp8、/casot/index.php9、/ZiFiT/ChoiceMenu.aspx10、/根据酶切方式,选择合适接头,例如,PX458等质粒sgRNA靶点oligo如下(Bbs1酶切)5‘-CACCGNNNNNNNNNNNNNNNNNNNN-3’3‘- CNNNNNNNNNNNNNNNNNNNN-CAAA-5‘(1)对于sgRNA的长度,一般应为20 nt左右;(2)对于sgRNA序列的碱基组成,可选3'末端含GG的sgRNA,同时sgRNA种子序列尽量避免以4个以上的T结尾,GC%含量最佳为40%~60%;(3)sgRNA的种子序列与脱靶位点的匹配数尽可能低(4)如果构建U6或T7启动子驱动sgRNA的表达载体,需考虑sgRNA的5' 碱基为G或GG,以提高其转录效率;(5)对于sgRNA靶向基因的结合位置,如需造成基因移码突变,需尽量靠近基因编码区的A TG下游,最好位于第一或第二外显子;(6)检查sgRNA靶向结合位点基因组序列是否存在SNPs;(7)如采用Cas9单切口酶,设计paired-gRNA需考虑成对sgRNA的间距;(8)全基因脱靶效应分析,需考虑脱靶位点最大允许几个错配碱基数,建议最少5个碱基。
重点考察种子序列和非种子序列碱基错配数,以及脱靶位点是否位于基因编码区等,另外还可考察是否存在碱基插入或缺失的脱靶位点。
特异性的sgRNA最好存在较少的脱靶位点,不存在种子序列完全匹配的脱靶位点,不存在含有1和2个碱基错配的脱靶位点。
实验前,首先应选择合适的软件进行sgRNA设计和全基因组脱靶效应评估,筛选2~3个活性可能高且脱靶效率最低的sgRNA。
再通过实验验证,依据实验结果选择最优的sgRNA。
2)构建sgRNA表达载体:将两条引物按照如下体系,退火后连接进sgRNA表达载体中,转化DH5a、Stbl3细菌,挑取单克隆测序鉴定。
Annealing and cloning procedure:1. Anneal each pair of oligos:1 µl oligo 1 (100 μM)1 µl oligo2 (100 μM)8 µl H2O10 µl totalAnneal in a thermocycler at 95℃ 5 min and then leave on the bench at RT to cool down for 1hr. Dilute the annealed oligo 1:250 (250 - fold).2. Set up digestion reaction: (pX330/pX335载体和内切酶Bbs I为演示案例)X µl pX330/pX335 or other backbone vector (at least 2 µg)2 µl 10X NEBuffer 2.11 µl Bbs I (NEB) Use more if cutting more DNAH2O to final V olume of 20 µlIncubate the digestion reaction at 37o C for at least 4hr. Run ~200ng on agarose gel to ensure COMPLETE digestion. When digest is complete, heat inactivate (65°C for 20 min) or column purify linearized plasmid.3. Set up ligation reaction:X µl pX330/pX335 BbsI digested vector (100ng)2 µl annealed oligo duplex from step 1 (1:250 dilution)2 µl 10x DNA ligase buffer (make sure fresh, else A TP or DTT may be shot)1 µl T4 ligaseY µl H2O to 20 µl final volume- Incubate the ligation reaction according to manufacturer recommendations.4. Transformation with 1 - 2 ul of the final product into competent cells.5. Pick colony and sequence verify with U6 sequencing primer (U6seqF: ACTA TCA TA TGCTTACCGTAAC)3)质粒制备:用去内毒素试剂盒抽提质粒,测定浓度,琼脂糖电泳检测质粒质量。
sgRNA表达质粒Cas9表达质粒(或二合一质粒)4)培养细胞和转染a)准备培养皿,培养细胞,细胞汇合度70%左右时进行转染。
b)按照Lipofectamine® 2000(或Dfector TM原代细胞转染试剂)转染说明书转染细胞,如果是分开表达的质粒(质粒质量比1:1)c)转染48h或72h后提取细胞基因组DNA,提前设计好检测on-target或预测的off-target引物,PCR扩增目的片段。
(引物在切口两端不对称设计,100:200或者200:300bp)5)T7E1酶切法检测突变体T7 核酸内切酶I 是一种具有底物结构选择性的酶。
该酶以不同的活性作用于不同的DNA底物。
切割特定底物时,必须控制酶量和反应时间。
反应温度超过42℃时,会增加非特异性核酸酶活性。
避免反应温度超过55℃,会导致酶活性降低。
a) PCR扩增出带有突变位点(CRISPR/Cas9的target site)DNA片段,长度约500bp,突变位点最好不位于PCR片段中央,这样将切割出两条大小不同的带。
b)突变体DNA与野生型DNA的PCR产物按如下体系进行混合,进行加热变性、退火复性处理。
(如果测混合克隆,直接将PCR产物变性,退火复性处理)表1. T7E1酶切体系和反应条件c)上述反应体系分别加入0.5ul T7E1酶,37℃反应30 min后,跑2%的琼脂糖凝胶电泳检测分析酶切结果。
图2-1. T7E1酶切法检测图图2-2. T7E1酶切法检测图6)测序评估:将PCR产物连接进T载体中,转化进DH5a细菌,随机挑取15-20个单菌落进行测序,评估目标片段被切割后碱基变化和切割效率。
图3. 单克隆测序7)PAGE胶检测突变体:制备非变性PAGE胶,检测PCR产物,检测on-target 或预测的off-target sites位点(可选方法)。
图4. PAGE胶检测突变体模式图(REF. SCIENTIFIC REPORTS,2014)8)稀释法筛单克隆先从A1孔(细胞原液约1000个细胞/孔,数量根据细胞状态调整)至H1孔方向对半稀释,然后第一列再横向对半稀释,显微镜观察含单细胞的孔。
9)设计ssODN寡核苷酸(KI)Designing an ssODN template for HR-mediated repair5’ — (~60 - nt 5’ homology) NNN NGG (~60 - nt 3' homology) —3’如果用donor质粒,同源臂理论上越长越好,比如KI 1K左右的基因,同源臂设计在1K bp左右。
二、利用CRISPR系统建立基因敲除动物品系实验过程:2.1 确定待敲除基因的靶位点2.2 设计识别靶位点的识别的一对DNA Oligo(引物)2.3 构建可表达sgRNA的Cas9质粒2.4 体外转录sgRNA 和Cas9 RNA2.5 将sgRNA和Cas9 RNA直接注射入受精卵检测sgRNA活性2.6 将有活性的sgRNA和Cas9 RNA直接注射入受精卵建立Founder2.7 将Founder自交得到F12.8 F1自交得到F22.1 确定待敲除基因的靶位点同前2.2 设计识别靶位点的一对DNA Oligo(引物)同前2.3 构建可表达sgRNA的Cas9质粒同前,选择带有T7启动子的质粒2.4 体外转录sgRNA 和Cas9 RNA使用特定引物,以上述质粒为模板,以高保值酶分别对Cas9和sgRNA进行PCR扩增,循环数设为30个,20 ul反应体系。