原位杂交技术(in situ hybridization)
- 格式:doc
- 大小:24.50 KB
- 文档页数:5

原位杂交名词解释
原位杂交(in situ hybridization)是一种分子生物学技术,用
于检测和定位特定的核酸序列在细胞或组织中的分布情况。
该技术利用一段含有探针的核酸序列与待检测样品中的相应核酸序列进行互补杂交,通过标记的探针的位置和数量来确定目标序列的位置。
原位杂交的操作一般分为以下几个步骤:
1.固定样本:首先需要将待检测的细胞或组织样本固定在载玻
片或载体上,使其不被破坏。
2.脱氧核酸的解性处理:将样本中的DNA或RNA进行解性处理,使其成为单链的状态,以利于与探针的杂交。
3.制备探针:设计和合成一段与目标核酸片段互补的核酸探针,并进行标记。
标记可以使用荧光标记剂、酶标记剂等。
4.杂交:将制备好的探针加到固定的样品上,在合适的温度和
离子平衡条件下,使探针与待检测的样品中的目标序列发生互补杂交。
5.洗涤:通过一系列洗涤步骤,去除未与目标序列相互补的探针。
6.检测和成像:利用显微镜或其他成像设备观察和记录探针的
标记位置和数量,以确定目标序列在样品中的分布情况。
原位杂交技术可以用于很多领域,如遗传学、细胞生物学和病理学等。
它可以用来研究基因表达、染色体异常、病原体感染等问题,帮助科学家更好地了解细胞和组织的结构和功能。
此外,原位杂交还可以用来诊断疾病,如肿瘤、遗传病等,通过监测特定基因的表达情况,帮助医生做出正确的诊断和治疗方案。
总的来说,原位杂交是一种研究和定位目标核酸序列在细胞或组织中分布情况的技术,具有高灵敏度和高特异性的优点,可以广泛应用于生命科学的各个领域。


miRNA的常用检测方法研究1.原位杂交(in situ hybridization)原位杂交技术是一种广泛应用于miRNA检测的方法。
该技术基于miRNA和其互补的探针(probe)间的互补配对,通过观察上调或下调的信号强度来判断miRNA的存在及相对表达量。
此方法可能不如其他技术在定量方面准确。
2.RT-qPCR实时荧光定量PCR(RT-qPCR)被广泛应用于miRNA检测。
该方法通过引物探针的设计,特异性的扩增目标miRNA,利用荧光信号实现高灵敏度、高特异性和低误差的miRNA检测。
此方法可以获得很高的检测精度和速度,但是相对于其他技术成本较高。
3.Northern blottingNorthern blot是一种利用RNA凝胶电泳、转移和探针杂交检测RNA分子量、丰度及空间分布的方法。
该方法适用于测定miRNA的大小,但是灵敏度较低,需要较大的miRNA数量才能获得可靠的结果。
因此其在miRNA检测中的使用较少。
4.MicroarraymiRNA microarray是一种同时检测大量miRNA在样品中存在与否和表达量的方法。
它通过载入大量的探针来检测miRNA,具有高通量和快速的检测速度。
但是由于探针的种类、长度和相互竞争存在的问题,其实验结果的可重复性和准确性容易受到影响。
5.Next-generation sequencing随着测序技术的发展,高通量测序技术也被应用于miRNA的检测分析。
该方法通过高速测序技术,直接测定miRNA的序列和表达量,具有高度准确性和可重复性。
与其他方法相比,其天然的多样性和广泛性也是其独特优势之一。
但是其数据量大、需较长时间的数据处理和计算,并且实验成本相对较高。
总之,miRNA检测技术各有优劣,不同方法的选择应视具体的实验需求而定。
现阶段综合应用多种技术进行miRNA检测,在不同的研究环节中互补和交叉验证,是对miRNA检测更好的选择。

原位核酸分子杂交技术原位核酸分子杂交技术简称原位杂交(in situ hybridization ISH)是应用已知碱基顺序并带有标记物的核酸探针与组织、细胞中待检测的核酸按碱基配对的原则进行特异性结合而形成杂交体,然后再应用与标记物相应的检测系统,通过组织化学或免疫组织化学方法在被检测的核酸原位形成带颜色的杂交信号,在显微镜或电子显微镜下进行细胞内定位。
这一技术为研究单一细胞中DNA和编码各种蛋白质、多肽的相应mRNA的定位提供了手段,使从分子水平研究细胞内基因表达及有关基因调控提供了有效的工具。
可视为组织化学或免疫细胞化学中革命性的突破。
原位杂交技术已应用于基础研究如基因组图(Gene mapping),转基因检测,基因表达定位(localization of gene expression),核DNA和RNA的mRNA的排列和运输(arrangement and transport of mNA),复制(replication)和细胞的分类(Sorting of cells)。
临床研究应用在细胞遗传学(Cytogenetics),产前诊断(Prenatal diagnosis),肿瘤和传染性疾病的诊断,生物学剂量测定(biological dosimetry)和病毒学的病原学诊断等。
随着核酸探针的制备,标记方法和基本操作方法的不断改进,新的技术不断涌现,相信在不久的将来,原位杂交技术将会更广泛的被应用于各个学科,并不断为生命科学提供新的资料,开拓新的领域。
第一节原位核酸分子杂交技术的发展原位杂交1969年美国耶鲁大学Gall和Pardue首先创立的,他们用爪蟾核糖体基因探针与其卵母细胞杂交,确定该基因定位于卵母细胞的核仁中。
与此同时,Buongiorno Nardelli 和Amaldi,John及其同事等相继利用同位素标记核酸探针进行了细胞或组织的基因定位。
Orth(1970)应用3H标记的兔乳头状瘤病毒cRNA探针与兔乳头状瘤组织的冰冻切片进行杂交,首次用原位杂交技术检出了病毒DNA在细胞中的定位。

分子生物学研究中的原位杂交技术1. 引言原位杂交技术(In Situ Hybridization,ISH)是一种分子生物学研究中常用的重要技术方法,它在研究基因功能、表达、定位和疾病等方面具有广泛的应用。
通过掌握ISH技术的基础知识和基本操作,可以为分子生物学的深入研究提供强有力的工具。
2. 背景知识ISH技术是通过将DNA或RNA探针与待检测物品(如细胞、组织、染色体等)发生靶向杂交反应,从而探究DNA和RNA序列在待检测物品内的分布、表达及功能等。
利用双链DNA分子中序列互补的特性,ISH技术可以检测同源性的DNA或RNA序列,并确定它们在待检测物品内的位置。
ISH技术有多种类型,其中包括原位DNA杂交(In Situ DNA Hybridization,ISDH)、细胞核流式原位杂交(Fluorescence InSitu Hybridization of Interphase Nuclei,FISH)、原位RNA杂交(In Situ RNA Hybridization,ISR)等。
FISH是目前应用最广泛的一种ISH技术,它能够在细胞核级别上进行检测,解决了ISDH只能检测染色体水平的限制。
3. 原位DNA杂交 (ISDH)ISDH技术是通过从待检测物品中提取DNA标记探针,利用其与待检测物品DNA发生互补杂交来实现。
它能够检测到DNA分子的位置和数量,并确定待检测物品中特定DNA序列的分布。
ISDH的操作步骤主要包括:(1)待检测物品的准备和固定;(2)DNA探针的制备和标记;(3)探针与待检测物品DNA的杂交;(4)洗涤和显色。
4. 细胞核流式原位杂交 (FISH)FISH技术是通过使用荧光探针,将荧光标记的DNA探针与待检测物品的染色体DNA或RNA发生互补杂交反应,直接在细胞核水平上检测DNA序列的位置和数量。
FISH技术不仅能够在正常染色体结构和分布的情况下对基因进行检测,还能够检测基因突变、重排等变异情况。

rna原位杂交技术RNA原位杂交技术(RNA in situ hybridization)是一种用于研究细胞和组织中特定RNA分子表达的重要实验方法。
它通过特异性杂交的原理,能够对细胞和组织中特定的mRNA或非编码RNA进行定位和可视化,从而揭示基因表达的时空分布情况,为研究基因功能和疾病发生机制提供了有力的工具。
RNA原位杂交技术的基本原理是利用互补的核酸序列进行杂交。
在实验中,首先需要制备一段具有特异性的标记RNA探针,该探针的序列与目标RNA的互补部分相匹配。
标记通常使用放射性同位素或荧光染料进行,以便在细胞或组织中可视化。
在进行RNA原位杂交实验时,首先需要固定样本,以保持细胞和组织的形态结构。
然后,通过脱水和透明化等处理,将样本与RNA 探针进行杂交。
杂交后,通过洗涤去除未结合的探针,然后进行探针的检测。
对于放射性标记的探针,可以使用放射自显影的方法进行可视化;对于荧光标记的探针,可以使用荧光显微镜观察。
RNA原位杂交技术在生命科学研究中具有广泛的应用。
它可以用于研究胚胎发育过程中基因表达的时空分布,揭示基因在不同发育阶段和不同组织中的表达模式。
此外,RNA原位杂交技术还可以用于研究肿瘤的发生和发展机制,通过检测癌细胞中特定基因的表达情况,揭示肿瘤细胞的分化状态和侵袭能力。
近年来,随着高通量测序技术的发展,RNA测序已经成为研究基因表达的主流方法。
然而,与RNA测序相比,RNA原位杂交技术具有直接观察细胞和组织中RNA表达的优势。
它可以提供细胞和组织层面的信息,揭示基因在空间上的分布和相互作用。
因此,RNA 原位杂交技术与RNA测序技术相辅相成,共同推动了基因表达研究的深入发展。
尽管RNA原位杂交技术在研究中具有重要意义,但也存在一些局限性。
首先,由于RNA原位杂交实验需要杂交探针与目标RNA的互补配对,因此对探针的设计和合成具有一定的挑战性。
其次,在实验过程中,样本的固定和处理等步骤可能会对RNA的结构和表达产生一定的影响,因此需要进行严格的实验控制。

1.原位杂交(in situ hybridization, ISH):原位杂交技术是将细胞或组织切片固定于载玻片上,使细胞中DNA或RNA在保持原来位置条件下,与标记的核酸探针进行原位杂交反应,通过放射自显影检测和显微镜观察,可以对所用材料中被杂交的核酸分子进行定位、定量分析或观察基因表达(mRNA)的水平。
通常以检测基因表达即组织细胞中mRNA含量的方法称为RNA原位杂交。
2.脂质体(liposome):(1)某些细胞质中的天然脂质小体。
(2)由连续的双层或多层复合脂质组成的人工小球囊。
借助超声处理使复合脂质在水溶液中膨胀,即可形成脂质体。
它可以作为生物膜的实验模型,或在临床上用于捕获外源性物质(如药物、酶或其他制剂)后将它们更有效地运送到靶细胞,经同细胞融合而释放。
3.脂筏(lipid rafts):一种非均一性富集固醇和鞘脂的高动态小型域。
约10~200 nm大小,能使细胞过程隔室化。
有时小型筏会借助蛋白质-蛋白质以及蛋白质-脂质的相互作用稳定地形成较大平台。
4.易化扩散(facilitated diffusion):一些非脂溶性(或亲水性)的物质,如葡萄糖、氨基酸、核苷酸以及细胞代谢物等,不能以简单扩散的方式通过细胞膜,但它们在载体蛋白的介导下,不消耗细胞的代谢能量,顺物质浓度梯度或电化学梯度进行转运,这种方式称为易化扩散或帮助扩散。
5.协同运输(co transport):一种分子的穿膜运输依赖于另一种分子同时或先后穿膜的运输方式。
后者从高浓度到低浓度的运输可为前者逆浓度梯度的运输提供能量。
分为对向运输和共运输两类。
6.巴氏小体(Barr body):在哺乳动物体细胞核中,除一条X染色体外,其余的X染色体常浓缩成染色较深的染色质体,此即为巴氏小体,是性染色体异固缩的结果。
又称X小体,通常位于间期核膜边缘。
在人类,男性细胞核中很少或根本没有巴氏小体,而女性则有1个。
7.细胞融合(cell fusion):两个或几个体细胞融合成为一个细胞的过程。

原位杂交(In situ hybridization)一、目的掌握核酸探针原位杂交操作技术,并利用该技术对单细胞的靶目标进行定位,用于细胞生物学基础研究。
二、原理原位杂交技术(in situ hybridization)是分子生物学和组织化学成功结合的产物,是特定标记的已知序列核酸作为探针与细胞或组织切片中核酸进行杂交并对其实行检测的方法。
其基本原理是含互补序列的标记DNA或RNA片段,即探针,在适宜的条件下与细胞内特定的DNA或RNA形成稳定的杂交体。
原位杂交能在成分复杂的组织中进行单一细胞的研究而不受同一组织中其它成分的影响,因此对于那些细胞数量少且散在于其他组织中的细胞内DNA或RNA研究更为方便;由于原位杂交不需要从组织中提取核酸,对于组织中含量极低的靶序列有极高的敏感性,并可完整地保持组织与细胞的形态,更能准确地反映出组织细胞的相互关系及功能状态。
三、仪器设备烘箱,切片机,展片机,染色缸,湿盒,原位PCR仪,显微镜、镊子、量筒、烧杯、吸水纸、枪与枪头、载玻片、盖玻片、冰盒等。
四、材料和试剂1.材料:带有病原体的水生动物,如感染WSSV病毒的对虾、感染虹彩病毒的水生动物等。
2.试剂:Davidson’s AFA固定液:330 ml 95%乙醇220 ml 福尔马林(37~39%甲醛水溶液)115 ml 冰醋酸335 ml H2O混匀后封口,室温放置;DIG标记与检测试剂盒(Roche公司);TNE:50 mmol/L Tris-HCl 6.57 g Tris Base10 mmol/L NaCl 0.58 g NaCl1 mmol/L EDTA 0.37 g EDTAddH2O 900 ml (定容至1L)用HCl调pH至7.4,高压灭菌,4℃保存;0.4% 甲醛:37~39%% 甲醛 5.4 mlddH2O 495 ml;蛋白酶K溶液(Pr.K,10 mg/ml):10 mg Pr.K 溶于1 ml TNE中(用时现配);20×SSC:3 mol/L NaCl 175.32 g NaCl0.3 mol/L柠檬酸钠88.23 g 柠檬酸钠ddH2O 900 ml (定容至1L)调pH至7.0,高压灭菌,4℃保存;2×SSC:20×SSC 100 mlddH2O 900 ml0.45 µm 滤膜过滤,4℃保存;20×Denhardt’s溶液:0.4%牛清血蛋白0.4 g牛清血蛋白0.4%聚蔗糖0.4 g聚蔗糖0.4%聚乙烯吡咯烷酮0.4 g聚乙烯吡咯烷酮ddH2O 100 ml0.45 µm 滤膜过滤,4℃保存;25%硫酸葡聚糖:25 g硫酸葡聚糖溶于80 ml ddH2O(定容至100 ml),-20℃保存;杂交液:4×SSC50% 甲酰胺1×Denhardt’s5% 硫酸葡聚糖0.5 mg/ml鲑精DNA4℃保存;BufferI:100 mmol/L Tris-HCl 12.11 g Tris Base150 mmol/L NaCl 8.77 g NaCl用HCl调pH至7.5,定容至1L,高压灭菌,4℃保存,;BufferII:0.5% Blocking agent 0.5 g Blocking agentBufferI 100 ml低温加热溶解,4℃保存一星期;BufferIII:100 mmol/L Tris-HCl 12.11 g Tris Base100 mmol/L NaCl 5.84 g NaCl50 mmol/L MgCl2 10.16 g MgCl2•6H2OddH2O 990 ml(定容至1L)用HCl调pH至9.5,0.45 µm 滤膜过滤,4℃保存;BufferIV:10 mmol/L Tris-HCl, pH8.0, 1 mmol/LEDTA;显色液(NBT/BCIP使用液):20 µl NBT/BCIP + 1ml BufferIII。


原位杂交技术(insituhybridization,ISH)我们常说,科学家也是艺术家,在明确真理探索科学的道路上,往往会创造出很多极具美感的艺术作品,比如下面的这些实验结果图,美吧~---Olson, B. D. and Downes, G. B.---in situ Hybridization of wild type Drosophila embryos所以今天小笔为大家介绍的就是能做出这种美美图的新技能,原位杂交实验~基本原理简单点说就是碱基互补配对原则。
官方来讲的话,就是我们将外源核酸(也就是常说的分子探针)与组织、细胞上待检测的DNA或RNA进行配对,形成核酸杂交分子,再经过一定手段将这个杂交分子的位置显示出来。
实验技术的更新是很快的,在明确了该技术的可行性后,科学家进一步改进并进行分类,主要有以下几类:荧光原位杂交技术:DNA分子原位杂交技术;在原技术手段上增加了荧光元素,技术手段较为先进,易掌握,运用广泛,一会儿小编会着重介绍哦~毕竟,能够在我们自己的实验中运用上,才是我们的最终目标嘛。
多彩色荧光原位杂交技术:显而易见,是荧光原位杂交的升级版,采用多个荧光来检测,目前的发展及运用也是较多的。
原位PCR:将原位杂交结合PCR技术发展起来的实验方法。
说实话,了解的比较少,我就不班门弄斧了,呵呵。
基因组原位杂交技术:该技术在我们的领域可能运用的毕竟少,主要是根据物种DNA同源性差异实现,在农作物等的杂交育种上运用较广,我就不详细介绍啦~明确了以上的分类及原理后,接下来小编主要讲讲在我们的领域中运用较广的荧光原位杂交方法。
结合示意图可以更好的理解哦~实验材料(重要的,基础材料不赘述)1. 4%多聚甲醛(1x PBS配置):固定剂2. 蛋白激酶K:使得靶核酸暴露,增加探针和靶核酸结合。
3. 杂交缓冲溶液实验步骤取材:动物组织取材(小鼠采用心脏灌流法)后在4%的多聚甲醛中固定4小时左右, 1x PBS洗5次,每次30分钟,用25%蔗糖脱水过夜(均在4°进行)。

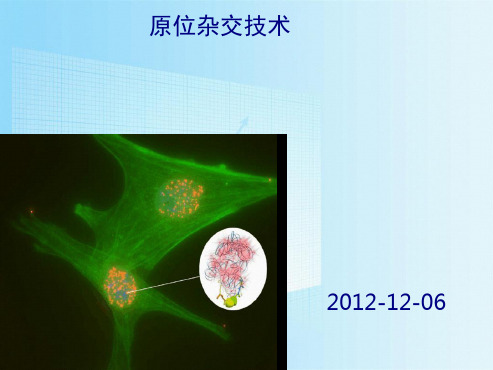

原位杂交技术一、概述(一)概念:原位杂交( in situ hybridization, ISH )是应用生物化学中核酸分子杂交( nucleic acid molecular hybridization )的原理,在组织切片、细胞涂片或印片上原位检测某种DNA或RNA序列的一项技术。
是将分子杂交与组织学相结合的一项技术,也称之为杂交组织化学(hybridization histochemistry)、细胞杂交(cytologicalhybridization)或原位杂交组织化学(in situ hybridizationhistochemistry)。
(二)历史和现状可以检测组织细胞本身的或外源的DNA或RNA序列。
二、原位杂交的基本原理核酸分子杂交的基本原理是利用核酸分子(DNA , RNA)的碱基对形成氢键的互补性(A=T , A=U , C=G),用一标记的核酸探针去检测与之碱基互补的靶核酸,这与免疫学中抗原-抗体的反应相似。
优点:①分子杂交的特异性强、灵敏度高,同时又有组织细胞化学染色的可见性。
②既可用新鲜组织,又可用石蜡包埋组织作回顾性研究。
③所需样本量少,可用活组织细针穿刺和细胞涂片。
④应用范围广泛,可对特定的基因(如癌基因、病毒基因)DNA、mRNA的表达进行定位、定性和定量、组织细胞分布和杂交电镜的亚细胞定位研究。
三、探针(probe)是含有互补顺序的外源性被标记的DNA或RNA片段,是在原位杂交中用于检测细胞内特定DNA或RNA顺序定位的特殊试剂,探针的碱基序列是已知的,只能与特定的核酸分子结合上。
用于原位杂交的探针有不同种类,可用于不同靶核酸的探测。
(一)探针种类、来源和应用1. 双链cDNA探针2. 单链cDNA探针3. 寡核苷酸探针4. cRNA探针(二)探针标记的种类1.放射性标记2.非放射性标记最常用的为生物素(光敏生物素、补骨脂素生物素)和地高辛(digxigenin , DIG),此外还有荧光素、酶类,如辣根过氧化酶(HRP)和碱性磷酸酶(ALP)等。


原位核酸分子杂交技术原位核酸分子杂交技术简称原位杂交(in situ hybridization ISH)是应用已知碱基顺序并带有标记物的核酸探针与组织、细胞中待检测的核酸按碱基配对的原则进行特异性结合而形成杂交体,然后再应用与标记物相应的检测系统,通过组织化学或免疫组织化学方法在被检测的核酸原位形成带颜色的杂交信号,在显微镜或电子显微镜下进行细胞内定位。
这一技术为研究单一细胞中DNA和编码各种蛋白质、多肽的相应mRNA的定位提供了手段,使从分子水平研究细胞内基因表达及有关基因调控提供了有效的工具。
可视为组织化学或免疫细胞化学中革命性的突破。
原位杂交技术已应用于基础研究如基因组图(Gene mapping),转基因检测,基因表达定位(localization of gene expression),核DNA和RNA的mRNA的排列和运输(arrangement and transport of mNA),复制(replication)和细胞的分类(Sorting of cells)。
临床研究应用在细胞遗传学(Cytogenetics),产前诊断(Prenatal diagnosis),肿瘤和传染性疾病的诊断,生物学剂量测定(biological dosimetry)和病毒学的病原学诊断等。
随着核酸探针的制备,标记方法和基本操作方法的不断改进,新的技术不断涌现,相信在不久的将来,原位杂交技术将会更广泛的被应用于各个学科,并不断为生命科学提供新的资料,开拓新的领域。
第一节原位核酸分子杂交技术的发展原位杂交1969年美国耶鲁大学Gall和Pardue首先创立的,他们用爪蟾核糖体基因探针与其卵母细胞杂交,确定该基因定位于卵母细胞的核仁中。
与此同时,Buongiorno Nardelli 和Amaldi,John及其同事等相继利用同位素标记核酸探针进行了细胞或组织的基因定位。
Orth(1970)应用3H标记的兔乳头状瘤病毒cRNA探针与兔乳头状瘤组织的冰冻切片进行杂交,首次用原位杂交技术检出了病毒DNA在细胞中的定位。


原位杂交的基本原理
原位杂交技术的基本原理是利用核酸分子单链之间有互补的碱基序列,将有放射性或非放射性的外源核酸(即探针)与组织、细胞或染色体上待测DNA或RNA互补配对,结合成专一的核酸杂交分子,经一定的检测手段将待测核酸在组织、细胞或染色体上的位置显示出来。
为显示特定的核酸序列必须具备3个重要条件:组织、细胞或染色体的固定、具有能与特定片段互补的核苷酸序列(即探针)、有与探针结合的标记物(曾呈奎等2000)。
RNA原位核酸杂交又称RNA原位杂交组织化学或RNA原位杂交。
该技术是指运用cRNA或寡核苷酸等探针检测细胞和组织内RNA表达的一种原位杂交技术。
其基本原理是:在细胞或组织结构保持不变的条件下,用标记的已知的RNA核苷酸片段,按核酸杂交中碱基配对原则,与待测细胞或组织中相应的基因片段相结合(杂交),所形成的杂交体 (Hybrids)经显色反应后在光学显微镜或电子显微镜下观察其细胞内相应的mRNA、rRNA和tRNA分子。
RNA原位杂交技术经不断改进,其应用的领域已远超出DNA原位杂交技术。
尤其在基因分析和诊断方面能作定性、定位和定量分析,已成为最有效的分子病理学技术,同时在分析低丰度和罕见的mRNA表达方面已展示了分子生物学的一重要方向。
原位杂交技术(in situ hybridization)是以标记的核酸分子为探针,在组织细胞原位检测特异核酸分子的技术。
这一技术不需要从组织细胞中提取核酸,对组织中含量极低的靶序列有很高的灵敏度,并可保持组织与细胞的结构完整,反映特异核酸分子的定位。
特别是配合使用能定位特异蛋白分子的免疫细胞化学技术,就能对生理或病理条件下从DNA 到mRNA到蛋白质这样一个基因表达过程进行定性和定位的分析,是基因表达研究强有力的手段。
(一)原位杂交技术的原理
原位杂交技术的原理是:使含有特异序列、经过标记的核酸单链即探针,在适宜条件下与组织细胞中的互补核酸单链即靶核酸发生杂交,再以放射自显影或免疫细胞化学方法对标记探针进行探测,从而在细胞原位显示特异的DNA或RNA分子。
1、核酸分子杂交
DNA双链分子在一定条件下可以发生变性和复性的过程。
分子杂交技术利用的就是两条互补的DNA单链能够复性的性质。
分子杂交是碱基互补的两条异质核酸单链在一定条件下缔合形成双链分子的过程。
这一过程相当于DNA的复性,只是核酸单链的来源不同。
原位杂交技术中,以经过标记的已知核酸分子为探针,以细胞内与探针序列互补的特异核酸分子为靶分子,造成一定条件使探针与靶核酸分子在原位发生杂交,然后再对其探测。
2、探针标记
探针是经过标记的核酸分子,与待测DNA或RNA序列互补。
探针主要有cDNA、RNA和寡核苷酸。
探针标记物有放射性的和非放射性的两类。
常用的放射性标记物主要有35S、32P、33P 和3H。
非放射性标记物主要有荧光素、生物素、地高辛和溴脱氧尿嘧啶等。
通常标记物被
-UTP、生物素-UTP、地高辛-dUTP 等。
然后通过各种酶促反应,如随机引物法或PCR法,使标记分子参入探针。
3、杂交
不同来源的序列互补的单链核酸分子在一定条件下借氢键相连而形成双链杂交分子的过程为杂交。
杂交可发生于RNA与DNA、DNA与DNA、RNA与RNA之间。
形成的双链杂交分子为杂交体(hybrids)。
4、杂交体的探测
对杂交体的探测根据探针标记物的不同而采用各种方法,目的是使标记的杂交体在光镜或电镜下可见。
⑴放射自显影如果探针是同位素标记的,显示杂交反应的方法就是光镜或电镜水平的放射自显影技术。
方法是在切片上涂覆核子乳胶膜后置于暗盒中于4℃自显影一段时间,然后经显影和定影后观察银离子在细胞和细胞器分布情况。
⑵免疫细胞化学对于非同位素标记的探针,可根据免疫细胞化学原理用直接法或间接法将探针标记物显示出来。
也就是采用那些在光镜或电镜下具有可见性的免疫细胞化学标记
联结的抗地高辛抗体,再加入酶的底物来显示杂交体。
(二)实验方法
原位杂交技术的实验方法主要包括:标记探针的准备,组织样品制备,杂交和杂交体探测。
1.标记探针的准备对cDNA、RNA和寡核苷酸三种探针的选择,要考虑
所要检测的靶核酸分子的性质,如cDNA 和RNA 适宜于检测mRNA分子,寡核苷酸对mRNA的杂交效率和特异性都不如RNA探针。
2.组织样品制备光镜原位杂交标本的固定多使用4%多聚甲醛,常规石蜡包埋和切片,但在操作过程中应提防RNA酶污染。
电镜原位杂交标本的固定也采用4%多聚甲醛。
与免疫细胞化学技术一样,电镜水平的原位杂交可以在未经包埋的组织切片上进行,称包埋前技
术;也可以在超薄切片上进行,称包埋后技术。
冷冻切片能较好的保存靶核酸因而较易获得杂交信号,但对细胞结构的保存较差。
3.杂交杂交是在一定温度和离子强度条件下让标记探针与组织中靶分子结
合的过程。
杂交的效率和特异性是优化杂交条件的出发点,也是需要兼顾的两个方面。
影响因素主要是探针的结构、杂交温度、杂交液中的甲酰胺浓度和离子强度、杂交后漂洗等。
要通过实验对条件进行优化。
4.杂交体探测根据探针标记物的不同采用放射自显影方法或免疫细胞化学
方法显示杂交结果。
5.基本实验过程
探针标记→纯化→(变性)
↓→涂覆乳胶→放射自显影
原位杂交→杂交体探测:→加酶联抗体→加酶底物显色
↑→金银染色
样品制备→切片→通透处理
2 荧光原位杂交技术
荧光原位杂交(Fluorescence in situ hybridization,FISH)技术是在已有的放射性原位杂交技术的基础上发展起来的一种非放射性DNA分子原位杂交技术。
它利用荧光标记的核酸片段为探针,与染色体上或DNA显微切片上的特异fl-N:~行杂交,通过荧光检测系统(荧光显微镜)检测信号DNA序列在染色体或DNA显微切片上的目的DNA序列,进而确定其杂交位点。
FISH技术检测时间短,检测灵敏度高,无污染,已广泛应用于染色体的鉴定、基因定位和异常染色体检测等领域。
FISH是原位杂交技术大家族中的一员,因其所用探针被荧光物质标记(间接或直接)而得名,该方法在80年代末被发明,现已从实验室逐步进入临床诊断领域。
基本原理是荧光标记的核酸探针在变性后与已变性的靶核酸在退火温度下复性;通过荧光显微镜观察荧光信号可在不改变被分析对象(即维持其原位)的前提下对靶核酸进行分析。
DNA荧光标记探针是其中最常用的一类核酸探针。
利用此探针可对组织、细胞或染色体中的DNA进行染色体及基因水平的分析。
荧光标记控针不对环境构成污染,灵敏度能得到保障,可进行多色观察分析,因而可同时使用多个探针,缩短因单个探针分开使用导致的周期过程和技术障碍。