隐马尔可夫模型的多序列比对研究
- 格式:pdf
- 大小:303.49 KB
- 文档页数:4

基因序列分析是生物信息学领域的重要研究内容之一。
利用马尔可夫模型进行基因序列分析可以帮助研究者理解基因的结构和功能,从而为疾病的治疗和预防提供重要的信息。
本文将介绍利用马尔可夫模型进行基因序列分析的基本原理和方法,希望读者能够通过本文了解基因序列分析的基本知识,并能够在实际研究中应用马尔可夫模型进行基因序列分析。
1. 马尔可夫模型简介马尔可夫模型是一种描述随机过程的数学模型,它具有“马尔可夫性质”,即未来的状态仅仅取决于当前的状态,与过去的状态无关。
在基因序列分析中,可以利用马尔可夫模型描述DNA序列中碱基的分布规律,从而推断基因的结构和功能。
2. 马尔可夫模型在基因序列分析中的应用在基因序列分析中,马尔可夫模型通常被用来预测DNA序列中的隐含Markov 模型和隐含马尔可夫模型,以及用在基因识别中。
通过对已知基因序列的训练,可以建立马尔可夫模型,然后利用该模型对未知的基因序列进行预测和分析。
3. 利用马尔可夫模型进行DNA序列的建模在利用马尔可夫模型进行基因序列分析时,首先需要对DNA序列进行建模。
通常情况下,可以将DNA序列中的碱基分为四类:A、C、G和T。
然后,可以利用马尔可夫模型描述碱基之间的转移概率。
以二阶马尔可夫模型为例,可以建立一个4*4的矩阵,表示从一个碱基转移到另一个碱基的概率。
4. 马尔可夫模型参数的估计在建立马尔可夫模型之后,需要对模型的参数进行估计。
参数估计的方法通常包括极大似然估计和贝叶斯估计。
通过对已知的训练数据进行统计分析,可以估计马尔可夫模型中的转移概率和初始状态概率。
5. 利用马尔可夫模型进行基因识别基因识别是基因序列分析的重要任务之一。
利用马尔可夫模型可以对DNA序列进行分析,从而识别其中的基因区域。
通过对DNA序列进行标记,可以利用马尔可夫模型进行概率推断,从而识别基因区域和非基因区域。
6. 马尔可夫模型在基因序列比对中的应用除了基因识别外,马尔可夫模型还可以应用于基因序列比对。

生物信息学中的计算方法和工具生物信息学是生命科学中的一个重要领域,它研究如何从大量的生物数据中提取信息,以更好地理解生物学现象和生物学系统的运作规律。
在生物学的研究中,生物信息学可用于DNA、RNA、蛋白质等生物分子的序列分析,以及基因功能研究、治疗和预防疾病、新药开发等。
本文将重点介绍生物信息学领域中的计算方法和工具。
基本概念生物信息学中的计算方法和工具主要涉及以下方面:1. 序列比对:指将两段或多段序列进行对比,以确定它们的相似程度和差异点。
比对方法包括全局比对、局部比对和多序列比对等。
2. 基因预测:指对一个序列或一组序列进行分析,以确定其中是否存在基因序列和其位置、结构和功能等。
基因预测通常使用的方法包括基于序列或基于比对的方法。
3. 基因注释:指为已知或新发现的基因序列提供更多相关信息的过程。
根据序列相似性和功能分析,可以对其进行已知基因注释、预测基因注释、轨迹注释等。
4. 进化分析:研究生物种系的进化关系、起源和分化过程,主要方法包括序列比对、物种树和系统发育树分析等。
5. 蛋白质结构预测:指根据氨基酸序列对蛋白质结构进行模拟和预测的方法。
此外,还可以通过生物标记分析、三维结构分析、功能域分析等多种方法进行细化分析。
生物信息学计算方法和工具1. BLASTBLAST是生物信息学领域最常用的序列比对工具之一。
它可以通过比对数据库中所有已知序列,快速找出输入序列与之相似的序列,并提供序列相似度和信心度评估等信息。
2. HMMERHMMER是一种基于隐马尔可夫模型(HMM)的序列比对工具,主要用于蛋白质序列的域注释和拓扑域分析。
HMMER与BLAST相比,在序列的相对差异较大情况下,更具优势。
3. NCBI EntrezNCBI Entrez是一个基于网络的生物学检索系统,它允许通过NCBI中不同数据库与序列进行搜索。
4. ClustalWClustalW是一种多序列比对工具,它可以对两个或多个序列进行全局或局部比对,并产生序列的进化关系树。

三种分析蛋白结构域的方法蛋白质是生命体内重要的功能分子,它们通过其特有的三维结构来实现其功能。
蛋白结构域是指蛋白质结构中具有独立功能和收缩性的区域。
分析蛋白结构域的方法对于理解蛋白的功能和机制有重要意义。
以下是三种常用的分析蛋白结构域的方法。
第一种方法是比对分析。
比对分析是通过比对已知结构域的蛋白质序列和结构与待研究蛋白质序列和结构进行对比,以此来鉴定待研究蛋白质中的结构域。
比对分析常用的工具有BLAST和HMMER等。
BLAST(基本局部序列比对工具)通过比对两个蛋白序列的共同片段来确定相似性,可以帮助确定蛋白质的结构域。
HMMER(隐含马尔可夫模型比对工具)则建立了一个隐含马尔可夫模型,将待研究的蛋白质序列与已知结构域的蛋白质序列进行比对,以此来确定结构域。
第二种方法是结构预测。
结构预测是通过计算机程序对蛋白质序列进行建模,以预测其三维结构。
常见的结构预测方法有基于比对的序列相似性建模、基于物理力学的方法和基于机器学习的方法等。
基于比对的序列相似性建模方法通过比对已知结构域的蛋白质序列与待研究蛋白质序列来构建模型,以此来预测待研究蛋白质的结构域。
基于物理力学的方法则基于分子力学和物理化学原理,通过计算机模拟来推测蛋白质的结构。
基于机器学习的方法则使用已知结构域的蛋白质数据来训练算法,以此来预测待研究蛋白质的结构域。
第三种方法是功能簇分析。
功能簇分析是通过聚类算法来将蛋白质分为不同的簇,以确定其中的结构域。
常见的聚类算法有层次聚类、基于密度的聚类和K均值聚类等。
层次聚类是将样本逐步合并成不同的簇,直到达到预定的停止条件。
基于密度的聚类则是根据样本的密度将其分为不同的簇。
K均值聚类是将样本分为K个不同的簇,使得簇内的样本之间的差异最小化。
通过功能簇分析可以鉴定出具有相似功能的蛋白质结构域。
综上所述,比对分析、结构预测和功能簇分析是常用的分析蛋白结构域的方法。
这些方法能够帮助鉴定蛋白质中的结构域,进而理解其功能和机制。

生物信息学中多序列比对算法的研究与改进生物信息学是研究生物学领域中的大规模生物数据的收集、存储、处理和分析的一门学科。
其中,多序列比对是生物信息学中一个重要的任务,可以帮助我们理解生物序列的相似性和差异性,从而揭示生物进化、功能和结构的信息。
本文将介绍多序列比对算法的基本原理、常用的方法和一些改进策略。
多序列比对的基本原理是将多个生物序列在一定的比对模型下进行比对,找到它们之间的共有特征和差异。
而这种比对过程是通过构建一个比对矩阵来完成的,该矩阵记录了每对序列之间的相似性得分。
常用的比对模型包括全局比对、局部比对和连续比对。
全局比对是将所有序列从头至尾进行比对,适合于序列相似性较高且较短的情况。
常用的算法有Needleman-Wunsch算法和Smith-Waterman算法。
Needleman-Wunsch算法使用了动态规划的思想,通过计算不同序列位置之间的得分矩阵,找到最优的比对方案。
Smith-Waterman算法是对Needleman-Wunsch算法的改进,它引入了负得分以处理局部比对的情况。
局部比对是将序列的某个片段与其他序列进行比对。
这种比对方法适用于序列相似性低或存在插入/缺失的情况。
常用的算法有BLAST、FASTA和PSI-BLAST。
BLAST算法使用了快速查找的技术,先找到一些高度相似的序列片段,再进行进一步的比对。
FASTA算法也是通过生成比对矩阵来找到相似片段,但它比BLAST更加灵敏。
PSI-BLAST算法将多次比对与序列数据库的搜索相结合,用于找到蛋白质序列中的保守和演化区域。
连续比对是将序列中的一个或多个连续子序列与其他序列进行比对。
这种比对方法可用于寻找序列中的结构域和功能区域。
常用的算法有HMMER和COBALT。
HMMER算法使用了隐马尔可夫模型和HMM-profile来比对序列中的结构域,具有较好的准确性和灵敏性。
COBALT算法是一种基于Conserved Domain Database (CDD) 的比对方法,通过利用数据库中的结构域信息来找到序列中的结构域。

多序列比对算法多序列比对算法是一种可用来比较多个相应序列之间的结构和功能差异的序列分析工具。
它可以被用来比较两个或多个序列,以发现它们之间的结构和功能差异。
多序列比对算法的核心思想是比较序列之间的相似性,搜索最适合的生物序列分析方法。
它是一种两个或多个生物分子序列之间的相似性分析和比较方法。
多序列比对算法具有广泛的应用,主要用于生物信息学中许多重要的问题,如深入了解功能性蛋白质、研究RNA结构和功能、设计药物靶点等。
它还可以用于挖掘基因的结构和功能,提升蛋白质的结构和功能,研究核酸、蛋白质和多种细胞的进化古迹,以及研究蛋白质的生物信息学。
多序列比对算法可以利用其卓越的识别能力找出两个或多个相互关联的序列中的相似片段。
许多多序列比对算法都基于概率模型,能够更准确地找出两个或多个序列之间有用的序列特征。
例如,多序列比对算法可以用于研究序列模式和相似性预测。
多序列比对算法可以应用于基因组定位,获得与序列相关的基因、转录因子和调控元件的功能和结构信息。
这种序列比对算法也可以用来预测基因在基因组中的位置,提供关于生物活性的结构信息,甚至可以用来挖掘复杂的信号转导网络中的基础关系。
此外,多序列比对算法可用于发现复杂对称结构,这对于研究生物体结构是非常有用的。
它也可以用于预测氨基酸序列或基因组中的结构和功能,它们是一种可以被用来预测基因表达状态和发挥作用的分子模型。
因此,多序列比对算法在生物序列分析领域占据了重要的地位。
多序列比对算法有很多种,如Smith-Waterman算法、Needleman-Wunsch算法、BLAST算法、Sequence Alignment算法、Clustal算法、HMM算法和全局模式填充法等,它们使用不同的功能来完成多序列比对任务。
Smith-Waterman算法是一种计算最长公共子字串的动态规划算法,可以找到最佳比对结果。
而Needleman-Wunsch算法是一种全局比对算法,该算法可以找到两个序列之间最佳比对结果,甚至可以是局部比对的变体。




生物信息学中的基因序列比对方法生物信息学是一门研究生命科学中生物数据的存储、检索、分析和解释的学科。
在生物信息学中,基因序列比对是一项重要的分析技术,它可以用于比较不同生物物种之间的基因组序列,以及找到相同或类似的序列模式。
基因序列比对不仅可以帮助研究者理解基因的功能和演化,还能发现与一些重要生物学问题相关的基因变异。
在生物信息学中,常用的基因序列比对方法包括全局比对、局部比对和迭代比对。
全局比对是一种用于比较两个较长序列的方法。
其中最著名的算法就是史密斯-沃特曼算法(Smith-Waterman algorithm)。
这个算法通过引入一个“得分矩阵”来评估两个序列的相似性。
得分矩阵中不同的配对得分反映了不同碱基(A、T、C、G)之间的相似程度。
该算法通过计算每个位置处的最高得分来确定两个序列的最佳比对位置。
全局比对方法适用于两个序列相似性较高且长度相近的情况。
局部比对是一种主要用于比较较短序列或在指定区域内比较的方法。
其中最著名的算法是基于隐马尔可夫模型(Hidden Markov Model, HMM)的Smith-Waterman算法的改进——Gotoh算法。
与全局比对不同的是,局部比对方法将序列的一部分(而不是整个序列)与其他序列进行比较。
这种方法可以在相似性不高、但存在区域相似的序列中找到最佳的比对。
迭代比对是一种通过多次迭代比对来提高比对准确性的方法。
迭代比对通常由两个步骤组成:第一步是使用一种快速算法,如BLAST(Basic Local Alignment Search Tool),利用预先构建的数据库搜索相似的序列。
在第二步中,将这些相似序列与查询序列进行进一步的比对,以获得更准确的结果。
迭代比对方法可以帮助研究者发现比较遥远、相似性较低的基因序列。
此外,还有一些其他的基因序列比对方法,如滑动窗口比对、多重比对和北斗星比对。
滑动窗口比对是一种通过将一个固定大小的窗口滑过一个较大的序列来寻找局部相似性的方法。

生物信息学复习题名词解释1. Homology (同源):来源于共同祖先的序列相似的序列及同源序列。
序列相似序列并不一定是同源序列。
(直系同源):指由于物种形成的特殊事件来自一个共同祖先的不同物种中的同源序列,它们具有相似的功能。
(旁系(并系)同源):指同一个物种中具有共同祖先,通过基因复制产生的一组基因,这些基因在功能上的可能发生了改变。
基因复制事件是促进新基因进化的重要推动力。
(异同源):通过横向转移,来源于共生或病毒侵染而产生的相似的序列,为异同源。
Score:The sum of the number of identical matches and conservative (high scoring) substitutions in a sequence alignment divided by the total number of aligned sequence characters. Gap总是不计入总数中。
6.点矩阵(dot matrix):构建一个二维矩阵,其X轴是一条序列,Y轴是另一个序列,然后在2个序列相同碱基的对应位置(x,y)加点,如果两条序列完全相同则会形成一条主对角线,如果两条序列相似则会出现一条或者几条直线;如果完全没有相似性则不能连成直线。
7. E值:得分大于等于某个分值S的不同的比对的数目在随机的数据库搜索中发生的可能性。
衡量序列之间相似性是否显着的期望值。
E值大小说明了可以找到与查询序列(query)相匹配的随机或无关序列的概率,E值越小意味着序列的相似性偶然发生的机会越小,也即相似性越能反映真实的生物学意义,E值越接近零,越不可能找到其他匹配序列。
值:得分为所要求的分值比对或更好的比对随机发生的概率。
它是将观测得到的比对得分S,与同样长度和组成的随机序列作为查询序列进行数据库搜索进行比较得到的HSP(高分片段对)得分的期望分布联系起来计算的。
通常使用低于来定义统计的显着性。
生物信息学中的序列比对方法研究序列比对是生物信息学中常用的一项基础技术,用于确定两个或多个生物序列之间的相似性和差异。
这对于理解生物系统的演化关系、预测蛋白质结构和功能以及研究基因组变异等具有重要的意义。
本文将介绍生物信息学中常用的序列比对方法和它们的优缺点。
1. 单序列比对方法单序列比对方法主要用于比较一个序列与数据库中的其他序列的相似性,例如BLAST(Basic Local Alignment Search Tool)和FASTA(Fast All)。
BLAST是一种快速而准确的比对算法,它通过在查询序列与目标序列中找到相似的片段并计算分数来确定序列之间的相似性。
BLAST将查询序列与目标序列比对的过程分为两步:首先,寻找高分数的核苷酸(或氨基酸)片段;其次,对这些片段进行扩展以确定整个比对序列。
BLAST是一种非常快速的比对方法,适用于大规模的数据库搜索。
FASTA是另一种常用的序列比对方法,它通过计算两个序列之间的局部相似性来确定它们的相似性。
FASTA使用快速而高效的算法,可以找到目标序列中完全或部分匹配的片段,并通过得分来评估相似性。
FASTA比对方法适用于较小规模的数据库搜索,并具有较高的灵敏度。
2. 多序列比对方法多序列比对方法主要用于比较多个序列之间的相似性,例如ClustalW、MUSCLE和MAFFT。
ClustalW是一种经典的多序列比对算法,它通过将多个序列进行两两比对并计算相似性得分来构建一个序列矩阵。
然后,ClustalW将该矩阵用于构建进化树,并生成一种优化的多序列比对结果。
ClustalW适用于较小规模、低复杂度的序列。
MUSCLE(Multiple Sequence Comparison by Log-Expectation)是一种基于概率模型的多序列比对方法。
它通过最大化序列之间的整体相似性得分来构建多序列比对结果。
MUSCLE相对于ClustalW而言,能够获得更准确的多序列比对结果。
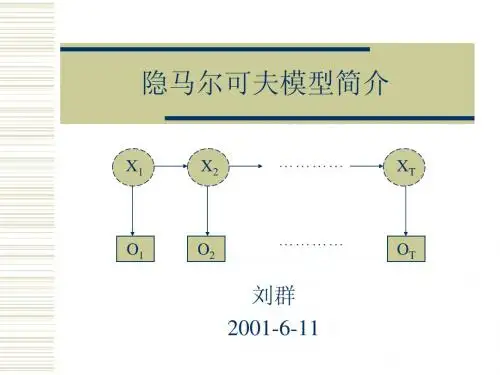
自然语言处理实验报告课程:自然语言处理系别:软件工程专业:年级:学号:姓名:指导教师:实验一隐马尔可夫模型与序列标注实验一、实验目的1掌握隐马尔可夫模型原理和序列标注2使用隐马尔可夫模型预测序列标注二、实验原理1.隐马尔可夫模型隐马尔可夫模型是马尔可夫链的一种,它的状态不能直接观察到,但能通过观测向量序列观察到,每个观测向量都是通过某些概率密度分布表现为各种状态,每一个观测向量是由一个具有相应概率密度分布的状态序列产生。
所以,隐马尔可夫模型是一个双重随机过程----具有一定状态数的隐马尔可夫链和显示随机函数集。
2. 使用隐马尔可夫模型做预测需要的处理步骤收集数据:可以使用任何方法。
比如股票预测问题,我们可以收集股票的历史数据。
数据预处理:收集完的数据,我们要进行预处理,将这些所有收集的信息按照一定规则整理出来,从原始数据中提取有用的列,并做异常值处理操作。
样本生成:根据收集的数据生成样本。
训练模型:根据训练集,估计模型参数。
序列预测并分析结果:使用模型对测试集数据进行序列标注,计算准确率,进行误差分析,可以进行可视化。
三、实验数据收集1.训练数据由于训练数据需要进行大量标注工作,所以训练数据选择了现有的已标注的人民日报1998语料库。
所有文章都已分词完毕,如:1998,瞩目中华。
新的机遇和挑战,催人进取;新的目标和征途,催人奋发。
英雄的中国人民在以江泽民同志为核心的党中央坚强领导和党的十五大精神指引下,更高地举起邓小平理论的伟大旗帜,团结一致,扎实工作,奋勇前进,一定能够创造出更加辉煌的业绩!2.测试数据测试数据使用搜狗实验室的新闻数据集,由于该数据集也是没有标注的数据集,所以手动标注了少量用于测试。
四、实验环境1.Python3.7和JDK1.8五、实验步骤1.数据收集及数据预处理训练数据使用人民日报1998语料库,所以不需要进行太多预处理,主要是测试数据集,我们使用搜狗实验室的新闻数据集,以下是收集和处理过程。
比对序列的算法
序列比对是生物信息学中的一项重要任务,它可以帮助我们理解生物序列之间的相似性和差异性,从而推断它们的进化关系、功能和结构等信息。
序列比对的算法有很多种,下面我将介绍一些常见的序列比对算法。
一、全局比对算法
全局比对算法是将两个序列的整个长度进行比对,它的目标是找到两个序列之间的最佳匹配。
其中最常用的算法是Needleman-Wunsch算法,该算法使用动态规划的方法进行比对,具有精确性和准确性,但计算复杂度较高。
二、局部比对算法
局部比对算法是将两个序列中的一部分进行比对,它的目标是找到两个序列中最相似的片段。
其中最常用的算法是Smith-Waterman算法,该算法也使用动态规划的方法进行比对,具有较高的准确性和灵敏性,但计算复杂度也较高。
三、基于快速哈希的比对算法
基于快速哈希的比对算法是将序列转换成哈希值,然后比对哈希值,具有较高的速度和较低的计算复杂度。
其中最常用的算法是BLAST算法,该算法使用局部
比对的方法,先将查询序列切成短片段,然后比对数据库中的序列,最后将所有匹配的片段进行组合,得到最终的比对结果。
四、基于马尔可夫模型的比对算法
基于马尔可夫模型的比对算法是将序列转换成马尔可夫模型,然后比对模型,具有较高的准确性和灵敏性。
其中最常用的算法是HMMER算法,该算法使用隐马尔可夫模型进行比对,具有较高的精确性和速度。
以上是常见的几种序列比对算法,每种算法都有其优缺点和适用范围,选择合适的算法需要根据具体的应用场景和需求进行评估和选择。
生物信息学中的蛋白质结构预测方法研究引言蛋白质结构是决定其功能的关键因素之一。
在生物信息学领域,蛋白质结构预测是一个重要的研究课题。
随着计算机技术和生物学研究的发展,越来越多的预测方法被提出和应用。
本文将探讨几种常用的蛋白质结构预测方法,并分析其优缺点。
一、序列比对方法序列比对法是最早也是最常用的蛋白质结构预测方法之一。
该方法基于蛋白质序列之间的相似性,通过比较与已知结构蛋白质的序列,来预测新的蛋白质结构。
其中,最常用的是基于比对结果生成模型的隐马尔可夫模型(HMM)方法。
序列比对方法的优点在于计算速度快、适用范围广。
然而,由于蛋白质的序列与结构之间并不是一一对应关系,所以该方法存在一定的局限性。
此外,序列比对方法对于含有跨膜区域的蛋白质预测的效果较差。
二、基于模板的方法基于模板的方法是通过利用已知结构的蛋白质模板,来预测新蛋白质的结构。
该方法假设结构相似的蛋白质在相同或相似序列之间存在较高的保守性。
基于此假设,可以将已知结构中的残基与待预测蛋白质进行比对,从而推断出新蛋白质的结构。
基于模板的方法与序列比对方法相比,可以在一定程度上提高预测的准确性。
然而,该方法的前提是已知结构的蛋白质与待预测蛋白质存在相似的序列,因此对于序列没有现有模板的蛋白质,该方法并不适用。
三、基于物理化学原理的方法基于物理化学原理的方法是通过计算蛋白质的力学和热学性质,来预测其结构。
这些方法通常基于分子力学模拟、能量计算和统计力场等原理,可以考虑蛋白质中的非共价相互作用、氢键以及电性等因素。
基于物理化学原理的方法在预测蛋白质结构中具有很高的准确性。
然而,由于其计算复杂度较高,所需的计算资源也会相应增加。
此外,该方法对初始结构的选择和参数的设置要求较高,因此需要有一定的专业知识。
四、机器学习方法机器学习方法是近年来在蛋白质结构预测中得到广泛应用的方法。
该方法通过训练数据集,构建预测模型,并利用模型预测新的蛋白质结构。
常见的机器学习方法包括神经网络、支持向量机等。