同源性分析
- 格式:pptx
- 大小:818.91 KB
- 文档页数:5


关于分子生物学中的同源性分析作者:李尚伟杜娟来源:《教育教学论坛》2014年第51期摘要:同源性是指在进化过程中源于同一祖先的分支之间的关系,包括直系同源和旁系同源。
直系同源基因描述在不同物种中来自于共同祖先的基因,而旁系同源基因描述在同一物种内由于基因复制而分离的同源基因。
同源性与相似性是两个不同的概念,前者是质的概念,后者突出量的描述,但它们之间又有一定关系,可以通过相似性来推测序列是否具有同源性。
介绍了用BLAST进行序列同源性分析的方法。
关键词:同源性;直系同源;旁系同源;相似性;BLAST中图分类号:G642.0 文献标志码:A 文章编号:1674-9324(2014)51-0184-02在分子生物学的教学及研究中,经常对核苷酸或氨基酸序列进行比对以确定基因之间或蛋白质之间的同源关系,进而根据同源性来推测物种间的亲缘关系。
基因或蛋白质之间的同源关系包括直系同源和旁系同源,序列间的同源性可用相似性或一致性来进行量化,用相似性(一致性)来判断序列是否同源。
一、同源性的概念在生物学中,同源性(homology)是指在进化过程中源于同一祖先的分支之间的关系。
我们可以在生物学的不同层次(如形态性状、分子性状等)上进行同源性分析,形态性状由于进行上或个体发育上的共同来源而呈现出本质上的相似性,但其功能不一定相同,那么它们就是同源的,如马的前肢与鸟的翅就是同源器官。
在分子水平上同源性主要是指基因的核苷酸序列或蛋白质的氨基酸序列之间的相似程度。
同源基因或蛋白质(homolog)指遗传上从某一共同祖先经趋异进化而形成的具有不同序列的基因或蛋白质。
同源性是一个相对的概念,在一定水平和范围内对其研究才有意义[1]。
二、直系同源与旁系同源同源关系包括两种类型:直系同源(ortholog)和旁系同源(paralog)。
这里我们主要以同源基因为例来进行讨论,同源蛋白质是同样的情况。
同源基因是遗传上来自某一共同祖先DNA序列的基因,包括直系同源基因和旁系同源基因。

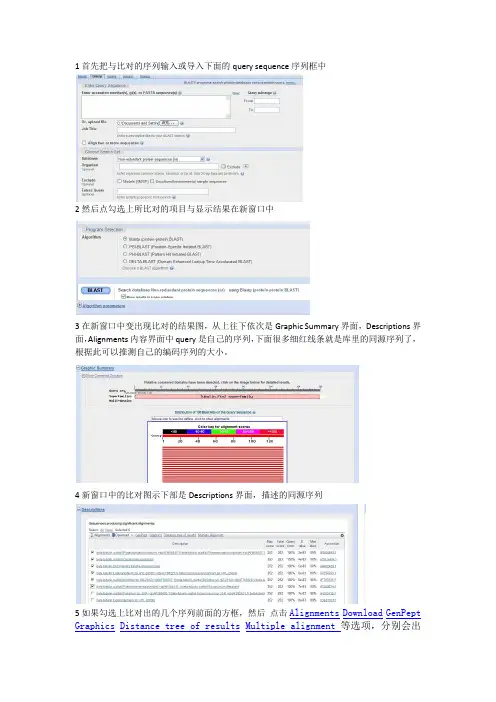
1首先把与比对的序列输入或导入下面的query sequence序列框中
2然后点勾选上所比对的项目与显示结果在新窗口中
3在新窗口中变出现比对的结果图,从上往下依次是Graphic Summary界面,Descriptions界面,Alignments内容界面中query是自己的序列,下面很多细红线条就是库里的同源序列了,根据此可以推测自己的编码序列的大小。
4新窗口中的比对图示下部是Descriptions界面,描述的同源序列
5如果勾选上比对出的几个序列前面的方框,然后点击Alignments Download GenPept Graphics Distance tree of results Multiple alignment等选项,分别会出
现两个序列比对结果,下载所勾选的序列,对应的蛋白序列,基因图示,进化树图像和所有勾选的序列的同源性比对结果图。


耐碳青霉烯大肠埃希菌分子特征和同源性分析摘要:耐碳青霉烯大肠埃希菌(Carbapenem-resistant Escherichia coli,CRE)是一种严峻的抗生素耐药菌,已成为医疗机构感染控制的重要难题。
探究CRE分子特征和同源性对于深度了解该菌的分子机制和进一步探究其治疗方案具有重要意义。
本文通过对已公开的CRE菌株数量较多、分布区域较广、伴随临床感染病例较多的3个亚种(E. coli O157:H7、E. coli K12、E. coli B)进行基因组序列分析,探究其分子特征和同源性。
结果显示,这三个亚种中,多数菌株表现出CRE的耐药特征和β-内酰胺类酶产生能力,其中最主要的酶类为KPC、NDM、VIM和IMP。
此外,在这些亚种中也发现了一些共同的抗菌基因和质粒。
基于这些结果,我们认为,CRE的分子特征和同源性表明,该菌株可能源自多个分支,且不同地理区域间存在着分布不均和菌株多样性的现象。
因此,在控制CRE感染和研发新药时,需要依据其分子特征和同源性进行更加有针对性的探究。
关键词:耐碳青霉烯大肠埃希菌;分子特征;同源性分析;β-内酰胺酶;质除了已经报道过的KPC、NDM、VIM和IMP酶外,CRE还可以通过多种机制表现出耐药性,例如通过改变外膜蛋白、使用外泌体和细胞间信号传递等方式。
同时,CRE也可能带有多种抗菌基因和质粒,这些基因和质粒也可能参与CRE的耐药机制。
同源性分析显示CRE在遗传结构上存在较高的多样性,可能源自不同的遗传分支并在不同的地理区域产生分布不均。
因此,在防治CRE方面,需要思量其分子特征和同源性的差异,并制定相应的针对性策略。
例如,在临床上可以通过对不同类型的CRE分子特征的分子诊断和分子分型,以及依据不同抗菌基因和质粒的分析来针对CRE耐药机制进行精准治疗。
此外,研发新型抗生素或利用已有的抗生素联合应用也可能是控制CRE的有效手段之一。
总之,深度探究CRE的分子特征和同源性,对于预防和治疗CRE感染有着重要的意义此外,在控制CRE感染方面,预防是最为重要的措施。

在分子生物学的教学及研究中,经常对核苷酸或氨基酸序列进行比对以确定基因之间或蛋白质之间的同源关系,进而根据同源性来推测物种间的亲缘关系。
基因或蛋白质之间的同源关系包括直系同源和旁系同源,序列间的同源性可用相似性或一致性来进行量化,用相似性(一致性)来判断序列是否同源。
一、同源性的概念在生物学中,同源性(homology)是指在进化过程中源于同一祖先的分支之间的关系。
我们可以在生物学的不同层次(如形态性状、分子性状等)上进行同源性分析,形态性状由于进行上或个体发育上的共同来源而呈现出本质上的相似性,但其功能不一定相同,那么它们就是同源的,如马的前肢与鸟的翅就是同源器官。
在分子水平上同源性主要是指基因的核苷酸序列或蛋白质的氨基酸序列之间的相似程度。
同源基因或蛋白质(homolog)指遗传上从某一共同祖先经趋异进化而形成的具有不同序列的基因或蛋白质。
同源性是一个相对的概念,在一定水平和范围内对其研究才有意义[1]。
二、直系同源与旁系同源同源关系包括两种类型:直系同源(ortholog)和旁系同源(paralog)。
这里我们主要以同源基因为例来进行讨论,同源蛋白质是同样的情况。
同源基因是遗传上来自某一共同祖先DNA序列的基因,包括直系同源基因和旁系同源基因。
直系同源基因,又称直向或垂直同源基因,指的是这样一些基因,它们起源于这些基因所在物种的最近共同祖先的一个祖先基因。
这些基因通常具有相同的功能,但并不是绝对的,当我们比较直系同源基因时,可能会发现有的基因失去了原来的功能或者进化出了新的功能[2-5]。
因此,直系同源基因描述在不同物种中来自于共同祖先的基因。
旁系同源基因,又称横向或并行同源基因,指在一个特定的基因组中由于基因复制产生的同源基因。
当我们比较旁系同源基因时,发现它们可能彼此具有了新的功能,也可能成为假基因了[2-4]。
旁系同源基因描述在同一物种内由于基因复制而分离的同源基因。
如图1所示,祖先球蛋白基因(globin gene)经过复制后分离产生了α球蛋白和β球蛋白基因,这两类基因就是旁系同源基因。


耐甲氧西林表皮葡萄球菌及耐药性检测和同源性分析目的:了解我院MRSE的检出率及耐药的特点,并对MRSE菌株的同源性进行研究。
为临床合理用药和耐药菌的防治提供参考依据。
方法:采用头孢西丁纸片扩散法及mecA基因PCR法检测出MRSE菌株,并对两种检测方法加以比较。
结果:128株临床分离表皮葡萄球菌中,用头孢西丁纸片法及PCR法对MRSE 检出率分别为79.69%(102/128)和85.16%(109/128),两种方法对MRSE 检出率差异无统计学意义。
以PCR法检出mecA基因作为判断MRSE的金标准,头孢西丁纸片法灵敏度为88.07%、特异度为68.42%、符合率为88.16%。
MRSE对多种抗菌药物耐药显著;除对B-内酰胺类明显耐药外,对红霉素和克林霉素的耐药率均高达98.17%,结果显示部分MRSE菌株间谱型相同。
结论:头孢西丁纸片法是筛选和确认MRSE菌株的一种可靠、简便的方法。
MRSE为多重耐药菌株,耐药形势严峻,部分菌株间存在克隆传播现象,临床必须加强细菌耐药性监测。
标签:耐甲氧西林表皮葡萄球菌;耐药性;抗菌药物1资料与方法1.1标本来源128株表皮葡萄球菌均来自我院2005年1月~2007年12月间住院患者的各类送检标本,主要来源于痰(44.53%)、分泌物(15.62%)、血液(10.93%)、创伤口(7.81%)等。
1.2方法1.2.1头孢西丁纸片法检测MRSE参照2005年CLSI的标准进行。
秤量19g M-H琼脂溶解于500ml双蒸水中,高压灭菌后,M-H琼脂冷却至50-60℃左右,倾注25ml制备药敏平皿。
将培养好的细菌液体,稀释到0.5个麦氏浊度,用棉棒蘸取原菌液均匀涂布在药敏平皿上,然后贴上30ug头孢西丁纸片,在35℃下培养24小时观察结果。
头孢西丁纸片法的判定标准为:抑菌环直径=24 mm判定为MRSE。
1.2.2mecA基因的检测根据美国临床及实验室标准协会(CLSI)标准,通过PCR方法检测出耐甲氧西林的表皮葡萄球菌(MRSE)。


利用系统进化分析软件对序列进行同源性分析1.0 目的1.1 为了保持国际上各个耐药性实验室的高检验水准,进行分子进化分析是有很重要作用的。
它不仅可以保证流行病学目的顺利实现,而且有利于发现检测阶段可能产生的潜在的交叉污染。
1.2 利用此软件进行分析所得的信息对于进行艾滋病毒在人群中传播的流行病学研究具有十分重要的意义。
1.3 通过对实验室之前分析的数据与当前数据进行比较,可以发现在此前实验过程中由于标本处理不当所导致的潜在的实验室污染。
1.4 对于确保得到高质量的实验结果并及时发现可能出现在实验室里的问题具有重要意义。
2.0 仪器设备2.1 计算机一台。
2.2 Windows 95 以上的操作系统。
2.3 BioEdit 以及MEGA 4 分析软件。
2.3.1 均为免费软件,可以从互联网上下载,在计算机上进行安装。
3.0 操作过程3.1 局限及要求3.1.1 经过序列编辑软件拼接处理后的txt 文件或fasta 文件均可,例如ChromasPro软件。
3.1.2 可以在MEGA 上分析的分子序列或距离矩阵数据。
3.1.3 Mega 4 软件只能将长度相等的序列转换为MEGA 输出文件,因此,任何多序列文件必须通过BioEdit 软件进行对齐修剪,然后才能进入通过Mega 软件转换成*.meg 格式进行分析。
3.1.4 该数据集的大小受限于计算机上可用的物理(RAM )和虚拟内存。
3.1.5 分析的序列必须包括两个或两个以上长度相同的序列,所有序列分析之前必须用MEGA 软件对齐。
3.1.6 核苷酸和氨基酸序列应该用英文字母连续书写,不区分大小写。
一些特殊的特殊符号,例如表示对齐缺口,碱基缺失等的符号也可以包含在序列中。
3.1.7 空格和制表符经常用于数据文件中,因此会被MEGA 忽略。
ASCII 字符,如(.)(- )(?),一般都作为特殊符号来表示序列中的不同碱基,分别表示第一个序列,对齐缺口及碱基缺失。
表2 不同时期各处理MCN ‰双因素方差分析结果T able 2 Analysis of each treatment of different time on the MCN ‰double fac 2tor varlance变异来源S ource of aberrance DF SS MS F A 因素A factor 20.290.150.045B 因素B factor 71188.00169.7150.66033A ×B14 1.520.190.057误差Eroor48160.56 3.35总变异Chief aberrance711350.37注:33差异极显著,P <0.01。
样分析可视为3次重复实验,所得数据可做统一处理。
B 因素内部极显著(P <0.01),说明各个处理组水平诱导蚕豆根尖细胞产生的MC N ‰是不相同的。
为了具体测定3种水样诱导蚕豆根尖细胞产生的MC N ‰之间的差异,对每一处理所得的MC N ‰平均值列表进行多重比较分析。
结果见表3。
表3 各处理间MCN ‰多重Duncan 检验T able 3 Multiple Duncan exam ination of various water sam plesⅠⅡa ⅤⅡb ⅢⅣⅡc Ⅱd 13.743312.683312.513311.263310.30338.79338.6233Ⅱc 5.1233 4.0633 3.8933 2.6433 1.6830.17Ⅳ 4.9533 3.8933 3.7233 2.4733 1.51Ⅲ 3.4433 2.3833 2.21330.96Ⅱb 2.4833 1.42 1.25Ⅴ 1.230.17Ⅱa1.06注:3差异显著P <0.05;33差异极显著P <0.01。
多重比较结果显示,Ⅱa 、Ⅱb 、Ⅱc 、Ⅱd 间存在极显著差异,即环磷酰胺对蚕豆根尖细胞的致突变性具有明显的剂量—效应关系,当环磷酰胺在0.5μg ΠL 以上浓度时,蚕豆根尖细胞的MC N ‰与CK 比较相差明显,所以,环磷酰胺可以作为该检测法的阳性指示物和衡量各检测点废水致突变性强弱及其变化。
生物信息学中的同源性搜索与分析生物信息学是一门复杂而又重要的学科,涉及到许多领域,其中之一就是同源性搜索与分析。
同源性是指两个或多个生物序列(DNA、RNA、蛋白质序列等)之间具有相同的祖先,其中表现为相同或相似的序列段。
同源性搜索与分析是指利用计算机技术从海量的生物序列数据库中寻找具有相同或相似序列的生物分子,对其进一步分析和研究,从而揭示生物分子之间的结构、功能和进化关系。
本文将从同源性的概念、同源性搜索与分析的方法、应用和前景等方面阐述生物信息学的重要作用和发展趋势。
一、同源性的概念同源性是生物学中的一个基本概念,主要用于描述不同生物分子之间的相似性。
同源性可以是两个或多个蛋白质序列的部分或全部相同,也可以是两个或多个DNA或RNA序列的部分或全部相同。
一般来说,同源性的相似性越高,意味着两个生物分子之间的功能和结构越相似。
同源性的发现可以为生物分子的功能研究提供重要线索,而同源性分析则可以用于构建生物分子之间的进化树,揭示它们之间的起源和演化路径。
二、同源性搜索与分析的方法同源性搜索与分析是生物信息学中的一种常见研究方法,它需要运用计算机技术从数据库中寻找具有相同或相似序列的生物分子,并对其进行比对和分析。
历经多年的发展,同源性搜索与分析的方法和工具已经非常成熟,常用的算法包括Smith-Waterman算法、Needleman-Wunsch算法、BLAST算法、HMM算法等。
其中,BLAST算法是最经典也是应用最广泛的同源性搜索和比对算法之一。
BLAST算法采用序列比对的思想,通过预先构建一台索引数据库,在查询序列和数据库中比对相似序列时减少比对的计算量。
BLAST算法将查询序列和索引库中的序列比对,并计算分值和E值,从而判断它们之间的相似性。
BLAST算法的速度非常快,可对多个数据库进行比对和查询,可以进行基于蛋白质序列、核酸序列等多种序列比对,而且还可以采用多序列比对的方式对多个序列进行对比和分析。
004 16s rRNA 序列同源性分析与细菌系统分类鉴定中国预防医学科学院营养与食品卫生研究所 (北京 100050)焦振泉 刘秀梅综述 孟昭赫审校 摘要 本文介绍了16s r RN A 序列测定及同源性分析的方法,并阐述了其在细菌系统分类鉴定中的重要作用。
关键词 16s rR NA 序列同源性分析 细菌 分类鉴定 近10多年来,随着分子生物学理论和技术的迅速发展,特别是作为生物技术里程碑的聚合酶链反应(PCR )技术的出现及核酸测序技术的不断完善,产生了许多新的分类方法,如:质粒图谱、限制性片段长度多态性分析、脉冲场凝胶电泳、PCR 指纹图、rDNA 指纹图、16s r RNA 序列分析等。
它们主要是对细菌染色体进行直接的DNA 分析或对染色体外的DNA 片段进行分析,从遗传进化的角度去认识细菌,从分子水平进行分类与鉴定,使细菌的分类越来越科学和精确,特别是16s rRNA 序列分析方法的出现使细菌进化可以通过试验研究来证实。
这是细菌分类史上的一次革命,必将使人们对生物进化及其与其它生物学科关系的认识更加深入。
1 16s rRNA 的结构与性质16s r RNA 为原核生物核糖体中一种核糖体RNA 。
目前,在细菌的系统分类学研究中最有用的和最常用的分子钟是r RNA ,其种类少,含量大(约占细菌RNA 含量的80%),分子大小适中,存在于所有的生物中,特别是其进化具有良好的时钟性质,在结构与功能上具有高度的保守性,素有“细菌化石”之称。
rRNA 在大多数原核生物中都具有多个拷贝[1],5s 、16s 和23s rRNA 的拷贝数相同[2],16s rRNA 由于大小适中,约1.5kb 左右,既能体现不同菌属之间的差异,又能利用测序技术来较容易地得到其序列,故被细菌学家及分类学家所接受[3]。
所以,“细菌系统学研究特设委员会”建议依据系统发育关系分类。
通过对其序列的分析,可以判定不同菌属、菌种间遗传关系的远近。
同源性分析标准操作规程一、同源性分析的应用包括感染暴发的判断,感染病原菌的确定及感染源的寻找。
二、基本方法1.细菌的表型特征分型技术(如血清型、耐药表型等)。
2.基因分型技术(如.PFGE、Rep—PCR、AFLP等)。
三、操作步骤(一)表型分型(血清型、耐药表型)1.标本孵育:从患者感染部位采集标本,并接种于相应培养基(培养瓶)中孵育。
2.分离菌种:分离病原菌(细菌或真菌),并鉴定细菌(真菌)种类。
3.血清分型:如可能则对同种细菌进行血清分型,如军团菌等。
4.药敏试验:根据细菌(真菌)种类选择药敏卡(纸片),进行药敏试验。
5.结果分析:分析血清型和药敏谱,如结果相同或药敏相差不大则提示有同源性。
(二)基因分型(PFGE)1.菌栓制备:将细菌悬液与2%低熔点琼脂糖凝胶混合,制备菌栓。
2.细菌消化:分别用含溶菌酶和(或)蛋白酶K的裂解液对菌栓进行消化。
3.洗涤菌栓:可用无菌水反复清洗或PMSF中和多余的蛋白酶K。
4.酶切:按照各种不同限制性内切酶的说明进行酶切。
5.制胶:用PF(讵电泳凝胶模具制备:PF(逼级琼脂糖凝胶。
6.电泳:根据不同细菌酶切片段的大小选择适当的脉冲参数,进行电泳。
7.凝胶成像:胶块染色后在凝胶成像仪下成像。
8.结果分析:根据Tenover等制定的标准对凝胶图像进行分析,判断菌株之间的同源性。
四、注意事项1.不同的分型方法,在分型能力、重复性、分辨能力、操作和成本等方面都不尽相同,可根据情况进行选择。
2.表型分型方法的分辨能力普遍比基因分型方法低,但简便、快速,适用于对感染暴发的初筛。
3.基因分型方法需要特殊实验器材,操作较繁琐且耗时,但为感染暴发同源性分析的“金标准”。
检验科要创作条件,开展该项业务。
临床微生物检测的基因同源性分析医院感染的流行病学研究是临床微生物学工作者的重要课题,在医院感染监测的研究中,一个很重要的问题就是如何确定感染途径和传播途径,以采取有效预防和控制措施,从而防止医院感染或者暴发流行,因此对微生物鉴定和菌株同源性分析提出了更高的要求。
目前,传统的细菌鉴定和同源性分析技术已不能很好地满足医院感染诊断和流行病学调查的需要,从型、亚型、株,甚至分子水平上去认识细菌变得愈来愈重要了。
近年来分子生物学的理论和技术在细菌感染诊断中的渗透和广泛应用,使得细菌鉴定、耐药基因的检测、分子流行病学调查变得更加准确、简洁和快速。
细菌DNA 同源性分析技术如脉冲场凝胶电泳、聚合酶链反应、DNA 探针杂交以及序列分析等方法是目前在分子水平上分析细菌的主要手段,它在鉴定细菌感染的爆发、确定院内交叉感染、感染病原菌之间的基因同源性等方面有着重要的作用,本文将对常用基因同源性分析方法的机理和过程做简要综述。
一、质粒分型(Plasmid profile assay):1、原理:质粒是可移动的染色体外元件,可以自发丢失或者被宿主稳定地获得,因此流行病学上相关的分离菌株有可能表现出不同的质粒图谱。
把质粒提取出来,进行常规的琼脂糖电泳分析,就可以知道分离菌株携带质粒的大小和数目。
2、实验过程:a.质粒抽提;b.0.8%琼脂糖电泳;c.EB染色、凝胶成像。
3、实验方法的评价:优势:a.步骤比较简单,对实验仪器要求不高。
b.在评价那些从局限的时间和地点(如一个医院中的急性爆发)分离出来的菌株非常有效。
缺点:a.实验结果的重复性不好。
b.分辨力不高。
二、染色体DNA的限制性内切核酸酶分析(Restriction Endonuclease Assay REA)1、原理:限制性内切核酸酶(REA)在特异的核酸识别序列切割DNA,DNA被消化后,所得到的限制性片段的数目和大小是由酶的识别位点和DNA的组成共同决定的。
在传统的限制性内切核酸酶分析中,人们用含有相对多的限制性位点的内切核酸酶消化细菌的DNA,这样就会得到成百上千条长度在0.5-50Kb范围内的DNA片段。