人全外显子组序列捕获及第二代测序
- 格式:docx
- 大小:235.89 KB
- 文档页数:4

全外显子组测序的具体方法及步骤
全外显子组测序(Whole Exome Sequencing,WES)是指利用序列捕获或者靶向技术将全基因组外显子区域DNA 富集后再进行高通量测序的基因组分析方法。
与全基因组重测序相比,全外显子组测序只需针对外显子区域的基因序列测序,覆盖度更深、数据准确性更高,更加简便、经济、高效。
技术优势
高性价,强分析,快速交付
外显子组测序主要用于识别和研究与疾病相关的编码区的基因组变异。
结合大量的公共数据库提供的外显子数据和正常人群数据库, 有利于更好地排除无害突变及解释变异信息之间的关联和致病机理。
技术路线
技术参数
样本要求。

第三代测序技术(单分子实时DNA测序)与第二代测序技术(高通量测序技术)简介第三代测序技术(单分子实时DNA测序)与第二代测序技术(高通量测序技术)简介第三代测序技术简介如果有人告诉你用显微镜实时观测单分子DNA聚合酶复制DNA,并用它来测序,你一定会认为他异想天开,没有一点生物的sense。
我最初就是这样认为的,然而它不仅可以实现,而且已经实现了~这个就是被称为第三代的测序技术,Pacific Biosciences公司推出的“Single Molecule Real Time(SMRT) DNA Sequencing”(单分子实时DNA测序)。
我有幸在NIH听到了这个技术发明人Stephen Turner博士的讲座,根据自己粗浅的理解记录整理一下。
要实现单分子实时测序,有三个关键的技术。
第一个是荧光标记的脱氧核苷酸。
显微镜现在再厉害,也不可能真的实时看到“单分子”。
但是它可以实时记录荧光的强度变化。
当荧光标记的脱氧核苷酸被掺入DNA链的时候,它的荧光就同时能在DNA链上探测到。
当它与DNA链形成化学键的时候,它的荧光基团就被DNA聚合酶切除,荧光消失。
这种荧光标记的脱氧核苷酸不会影响DNA聚合酶的活性,并且在荧光被切除之后,合成的DNA链和天然的DNA链完全一样。
第二个是纳米微孔。
因为在显微镜实时记录DNA链上的荧光的时候,DNA链周围的众多的荧光标记的脱氧核苷酸形成了非常强大的荧光背景。
这种强大的荧光背景使单分子的荧光探测成为不可能。
Pacific Biosciences公司发明了一种直径只有几十纳米的纳米孔[zero-mode waveguides (ZMWs)],单分子的DNA聚合酶被固定在这个孔内。
在这么小的孔内,DNA链周围的荧光标记的脱氧核苷酸有限,而且由于A,T,C,G这四种荧光标记的脱氧核苷酸非常快速地从外面进入到孔内又出去,它们形成了非常稳定的背景荧光信号。
而当某一种荧光标记的脱氧核苷酸被掺入到DNA链时,这种特定颜色的荧光会持续一小段时间,直到新的化学键形成,荧光基团被DNA聚合酶切除为止(见图)。

2)DNA的质量监测通常有两个方法:首先OD260/OD280比值应该在1.8左右(1.7-1.9),否则意味着DNA样品中存在大量的蛋白质或RNA污染。
其次,琼脂糖电泳分析时应主要以超螺旋条带为主。
最多不超过三条带(分别为超螺旋DNA,线性化DNA和环状DNA)。
否则意味质粒DNA的质量不高,应该重新制备。
2.限制性内切酶的活性1)限制性内切酶一般需要低温保存,而且反复的升降温过程对酶活性的损害很明显。
因而为了确保在有效期内的限制性内切酶不会失活,限制性内切酶的日常保存和使用应当很小。
2)建议购买具有保温功能的冻存盒保存限制性内切酶(-20度),而且取用限制性内切酶时,也应该使用具有保温功能的冻存盒,尽量防止酶的温度反复出现大的波动。
3.限制性内切酶的用量1)限制性内切酶的单位定义通常为:在合适的温度下,完全消化1ugDNA底物所需的酶量定义为一个单位。
2)在这个单位定义中,有几个不确定因素:首先是底物,不同的酶单位定义是选择的底物可能不同(常用的几个底物DNA包括:Lambda DNA ,AD2 DNA 和一些质粒DNA);第二个不确定因素是限制性内切酶在底物DNA上的酶切位点的个数。
由于单位定义中要求完全消化,因而底物上某个酶的酶切位点的个数的多少,就直接影响了该酶的单位定义。
3)因而,在进行酶切时,用1ul酶(一般10IU/ul)消化1ugDNA的通常做法是很不科学的,这也导致在实际工作中,大家要进行多次预实验才能确定最合适酶切条件。
4)以前,我推荐了一个在线的双酶切设计软件,double digestion designer, 可以精确地计算酶切时的限制性内切酶的用量。
使用中,能够注意到,用来进行双酶切的两个酶的用量有时竟然相差近20倍(EcoRI + NheI),而且发现,小片段PCR产物(100-500bp)进行酶切时,需要的酶量比质粒DNA酶切时用量多10倍以上。
5)该软件目前可以免费使用,用户名和密码都是test。

全外显子测序的技术要点全外显子测序是一种高通量测序技术,可以同时测定一个生物体的所有外显子区域的DNA序列。
全外显子测序技术的出现,极大地促进了人类基因组学的发展和疾病研究的进展。
下面将介绍全外显子测序技术的要点。
1. 序列捕获:全外显子测序的第一步是捕获外显子区域的DNA序列。
这一步骤使用特定的引物或探针,使外显子区域的DNA片段与其结合,然后通过化学方法将其他DNA片段去除。
这样可以高效地富集外显子区域的DNA序列。
2. 高通量测序:全外显子测序使用高通量测序技术,如Illumina测序平台。
这种测序技术可以同时测定大量的DNA序列,从而提高测序的速度和效率。
高通量测序技术的出现,使得全外显子测序成为可能。
3. 数据分析:全外显子测序产生的数据量庞大,需要进行复杂的数据分析。
数据分析的主要步骤包括测序质量评估、序列比对、变异检测等。
这些分析可以帮助研究人员鉴定外显子中的遗传变异,从而进一步研究其与疾病的关联。
4. 疾病研究应用:全外显子测序技术在疾病研究中具有广泛的应用价值。
通过对疾病样本和正常样本进行比较,可以发现与疾病相关的遗传变异。
这些变异可能是疾病的致病原因,也可以作为疾病的标志物进行诊断和预后的判断。
5. 个体化医疗:全外显子测序技术为个体化医疗提供了基础。
通过分析患者的外显子序列,可以了解其个体差异和易感基因型。
这对于制定个性化的治疗方案和预防策略非常重要。
6. 遗传咨询:全外显子测序技术的应用还使得遗传咨询工作更加准确和全面。
通过对患者及家族成员的全外显子测序,可以准确地识别携带疾病相关基因突变的个体,为其提供个性化的遗传咨询和风险评估。
7. 数据存储和共享:全外显子测序产生的数据量巨大,对于数据的存储和共享提出了挑战。
科研机构和数据库需要建立完善的数据管理系统,确保数据的安全性和可访问性,以促进全外显子测序数据的广泛应用。
8. 未来发展:随着测序技术的不断发展,全外显子测序技术也将不断改进。


这里为您介绍二代测序的相关流程和应用。
随着人类基因组工程的完成,对于低花费的测序技术的需求促进了高通量二代测序技术的发展。
这些新的测序平台允许进行高通量测序,具有广泛的应用:全基因组从头测序或者重测序目标序列重测序转录组分析微生物组研究基因调控研究NGS 序列二代测序仪器有很多种组合,在通量、片段长度、准确度、每一轮测序成本、每百万碱基对测序成本、初始成本、规格和技术方面存在存在差异。
从规格和初始成本的角度而言,二代测序仪器可轻松地分类为更窄的范围,也就是所谓的“台式测序仪”和高通量仪器。
台式测序仪使得任何实验室都可以像使用real-time PCR一样,自己进行测序。
这些仪器可以和一些靶标序列富集技术相结合,用在一些临床的应用中,其中:选定的靶标基因用于深度分析,以检测稀有的突变,或者检测多样样本中(比如癌症样本)中的突变。
目前,这些仪器的通量在10 Mb到 Gb之间,但是随着硬件,软件和试剂的持续改善,通量也在稳步增加。
高通量测序仪非常适合于大量的,基因组范围的研究,每次测序能测定600 Gb的序列。
一些这样的高通量和高精度的平台,能测定的片段长度相对较短,这对于高重复性的序列和未知基因组的从头测序就可能成为问题。
与此相反,也有一些仪器能测序的片段较长(达到2500 bp),但是其精度和测序能力(90 Mb)要低很多。
还有一些测序能力位于两者之间的仪器(~800 bp,700 Mb)。
因此,应用决定了哪一种仪器是最合适的。
有一种新的方法被称作“纳米孔测序”。
这种技术中,根据一个DNA链通过一个合成的或者蛋白纳米孔道所引起的电流的改变,可以确定通过这个孔道的碱基。
这理论上可以仅用一步就测序一个完整的染色体,而不需要生成新的DNA链。
DNA测序二代DNA测序的工作流程如下:DNA样本制备文库构建和验证文库分子大规模平行克隆扩增测序二代测序DNA样本的质量控制首先,评价基因组DNA的质量是非常必要的(完整性和纯度)。

外显子捕获结题报告2010-11-22内容1 项目信息 (1)2 工作流程介绍 (2)2.1 Agilent液相捕获平台 (2)2.2 NimbleGen 液相捕获平台 (3)2.3 生物信息分析流程 (4)3 分析报告 (5)结果 (5)3.1 标准生物信息分析 (5)3.1.1 数据产出统计 (5)3.1.2 目标区域单碱基深度分布图 (6)3.1.3外显子捕获测序的均一性 (7)3.1.4一致序列组装和SNP检测 (7)3.1.5 SNP注释 (8)3.1.6插入/缺失(indels)检测 (9)3.1.7插入/缺失(indels)注释 (9)3.2个性化分析 (9)3.2.1氨基酸替换预测 (9)3.2.2群体SNP检测和等位基因频率估计 (12)3.2.3孟德尔遗传病分析 (13)3.2.4 NGS-GW AS 分析 (14)3.2.5正向选择信号的检测 (14)4 数据分析方法说明 (15)4.1信息分析软件及常用参数介绍 (15)4.2参考数据库 (16)4.3数据文件格式 (17)1 项目信息2 工作流程介绍采用Aglient SureSelect外显子靶向序列富集系统和NimbleGen SeqCap EZ人全外显子捕获系统。
这两个系统都采用液相系统进行高特异性和高覆盖率的外显子区域捕获。
2.1 Agilent液相捕获平台图2.1 Aglient外显子捕获和测序流程基本流程:首先将基因组DNA随机打断成150-200bp左右的片段,随后在片段两端分别连接上接头制备杂交文库。
文库经纯化后经过LM-PCR的线性扩增与SureSelect Biotinylated RNA Library (BAITS)进行杂交富集,再经过LM-PCR 的线性扩增,文库检测合格后即可上机测序(Hiseq2000测序仪)。
对每个捕获文库进行高通量测序并保证测序深度达到要求,原始图像文件经过Illuminabasecalling Software 1.7进行碱基读取,获得读长为90bp双末端序列(reads)。

【NGS原创系列⼆】DNA建库那些事⼉众所周知,⽬前市场上主要以⼆代测序为主,⼆代测序过程中第⼀步是⽂库构建,本期为⼤家介绍⼀下常见⽂库构建的种类。
针对于illumina测序平台,DNA类的⽂库主要分为:DNA⼩⽚段⽂库、DNA⼤⽚段⽂库、Exon⽂库、PCR-Free⽂库和单细胞⽂库等。
DNA⼩⽚段建库DNA⼩⽚段⽂库是指⽚段⼤⼩在1kb以下的普通DNA⽂库(200bp、350bp、500bp等),DNA⼩⽚段⽂库可⽤来进⾏⼈重测序,动植物、微⽣物的de novo和重测序,16s rRNA测序,宏基因组测序等项⽬类型的⽂库构建。
DNA⼩⽚段⽂库的构建主要包含以下2种⽅法:01传统⽂库构建流程传统建库流程主要分为以下⼏个步骤:(1)将待测序的DNA分⼦⽤超声波打碎成200-500bp长的序列⽚段(2)将打碎后的基因组进⾏末端补平(3)在补平后的⽚段3’端加A和5’端进⾏磷酸化(4)对修复后的⽚段进⾏接头连接(5)对接头连接后DNA⽚段进⾏PCR扩增(6)对扩增后的⽂库进⾏磁珠纯化DNA⼩⽚段⽂库建库原理图如下:02转座法建库流程转座法建库相对于传统建库,其实验⽅法简单快速,5分钟内即可同时完成DNA⽚段化和接头连接,⽽且所需要的DNA量少。
⽬前市⾯上能提供具有⾃主知识产权专利的Tn5转座法建库试剂盒的公司就只有Illumina公司和TransGen公司。
由于转座酶与所输⼊的基因组量有⼀定的⽐例关系,因此我司开发了2款转座酶法建库试剂盒:(1)针对50ng起始量DNA(2)针对1ng起始量DNA转座法建库原理图如下:DNA⼤⽚段建库DNA⼤⽚段⽂库,⼜称之为末端配对(mate-paired)⽂库,⽚段长度⼤于1kb,主要⽤于动植物,微⽣物的de novo测序。
DNA⼤⽚段⽂库的构建主要分为以下⼏个步骤:(1)将待测序的基因组打断(2)将打碎的基因组进⾏末端修复和进⾏Biotin标记(3)将标记好的基因组环化(4)将环化后的基因组打碎(5)磁珠捕获⽚段化的基因组(6)对捕获后的基因组进⾏末端修复加A和加接头(7)对接头连接后的基因组进⾏PCR扩增(8)将PCR扩增后的⽂库进⾏分选回收DNA⼤⽚段⽂库建库流程图如下:Exon⽂库⼈类外显⼦组总共约30 Mb,占⼈类基因组约1%,外显⼦具有⾼度的保守型,且⼤部分疾病的致病位点位于外显⼦区。

国际儿科学杂志2021年1月第48卷第1期Int J Pediatr,Jan 2021,Vol. 48 ,No. 1•综述.• 9 •全外显子组测序技术在新生儿遗传病诊断中的应用魏豪(综述)颜景斌(审校)上海市儿童医院上海医学遗传研究所上海交通大学附属儿童医院200040通信作者:颜景斌,Email:189****8323@【摘要】新生儿期的遗传病往往具有临床症状不典型、病情较为严重等特点,从而造成诊断和治疗的困难。
随着测序技术的不断发展,第二代测序技术以其高通量、低成本、快速化检测等优势逐渐应用于临床领域。
全外显子组测序(whole exome sequencing,WES)技术作为第二代测序技术的一种,仅对基因组外显子区域进行序列捕获、富集并测序,再利用生物信息学手段对W E S产生的大量数据进行分析和筛选,从而找到遗传病的变异基因或位点。
W ES技术因其具有检查结果提示全面和报告周期短的优势,逐渐成为新生儿遗传病分子诊断的重要手段。
【关键词】全外显子组测序技术;新生儿;遗传病诊断基金项目:国家重点研发计划“精准医学研究”重点专项(2016YFC0905102)DOI: 10. 3760/cma. j. issn. 1673-4408. 2021.01.003Application of whole exome sequencing technology in the diagnosis of neonatal genetic diseasesWei Hao,Yan JingbinShanghai Children's Hospital,Shanghai Institute of Medical Genetics,Shanghai Jiao Tong University,Shanghai 200040, ChinaCorresponding author:Yan Jingbin, Email:189****************【Abstract】Genetic diseases that have shown clinical symptoms in neonatal period are often character-ized with atypical symptoms and serious condition, which contributes to difficulties in diagnosis and treatment With the progress of sequencing technology, the next-generation sequencing technology is gradually applied to the clinical field with its advantages of high throughput, low cost and rapid detection. As one of next-generation sequencing technologies, the whole exome sequencing technology (WES) captures, enriches andsequences the genomic exon regions, and then the large amount of WES data is analyzed by bioinformaticsmethods and screened to find variant site in gene that causes genetic disease. WES technology has gradually become an important means of diagnosis in neonatal genetic diseases because of its advantages of comprehensiveresults and short reporting period.【Key words 】Whole exome sequencing technology; Neonates ;Genetic disease diagnosisFund program:uPrecision Medicine Research>,Key Special Project of National Key Research and Development Plan (2016YFC0905102)DOI :10.3760/cma. j. issn. 1673-4408.2021.01.003罹患遗传病的新生儿往往具有多系统的临床表 型,与普通新生儿相比具有更高的发病率和病死率,大约有1/3的新生儿遗传病患儿在5岁之前死亡[1]。

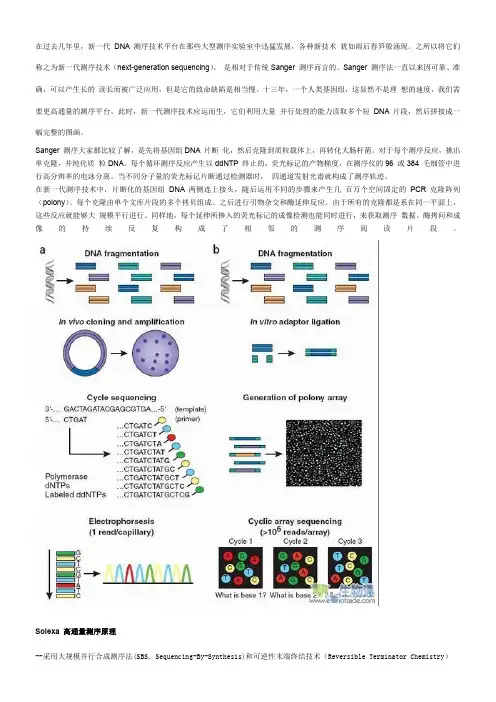
在过去几年里,新一代DNA 测序技术平台在那些大型测序实验室中迅猛发展,各种新技术犹如雨后春笋般涌现。
之所以将它们称之为新一代测序技术(next-generation sequencing),是相对于传统Sanger 测序而言的。
Sanger 测序法一直以来因可靠、准确,可以产生长的读长而被广泛应用,但是它的致命缺陷是相当慢。
十三年,一个人类基因组,这显然不是理想的速度,我们需要更高通量的测序平台。
此时,新一代测序技术应运而生,它们利用大量并行处理的能力读取多个短DNA 片段,然后拼接成一幅完整的图画。
Sanger 测序大家都比较了解,是先将基因组DNA 片断化,然后克隆到质粒载体上,再转化大肠杆菌。
对于每个测序反应,挑出单克隆,并纯化质粒DNA。
每个循环测序反应产生以ddNTP 终止的,荧光标记的产物梯度,在测序仪的96 或384 毛细管中进行高分辨率的电泳分离。
当不同分子量的荧光标记片断通过检测器时,四通道发射光谱就构成了测序轨迹。
在新一代测序技术中,片断化的基因组DNA 两侧连上接头,随后运用不同的步骤来产生几百万个空间固定的PCR 克隆阵列(polony)。
每个克隆由单个文库片段的多个拷贝组成。
之后进行引物杂交和酶延伸反应。
由于所有的克隆都是系在同一平面上,这些反应就能够大规模平行进行。
同样地,每个延伸所掺入的荧光标记的成像检测也能同时进行,来获取测序数据。
酶拷问和成像的持续反复构成了相邻的测序阅读片段。
Solexa 高通量测序原理--采用大规模并行合成测序法(SBS, Sequencing-By-Synthesis)和可逆性末端终结技术(Reversible Terminator Chemistry)--可减少因二级结构造成的一段区域的缺失。
--具有高精确度、高通量、高灵敏度和低成本等突出优势--可以同时完成传统基因组学研究(测序和注释)以及功能基因组学(基因表达及调控,基因功能,蛋白/核酸相互作用)研究----将接头连接到片段上,经PCR 扩增后制成Library 。
《中国产前诊断杂志(电子版)》 2020年第12卷第2期·综述· 全外显子组测序在产前诊断中的应用及其发展方向林晓莹 魏佳雪 (广东省第二人民医院产前诊断中心,广东广州 510317)【摘要】 血清学筛查、超声检查和染色体核型分析是传统产前诊断的主要手段,而近年来,产前分子诊断成为产前诊断的重要内容,荧光原位杂交技术(fluorescenceinsituhybridization,FISH)、荧光定量PCR技术、多重连接依赖探针扩增技术(multiplexligation dependentprobeamplification,MLPA)和染色体微阵列分析(chromosomemicroarrayanalysis,CMA)等已广泛应用于产前诊断的临床实践中。
而对于那些超声检查异常,核型分析和染色体微阵列分析结果为阴性的产前诊断病例中,全外显子组测序(wholeexomesequencing,WES)往往能提供更多的遗传信息,从而提高其检出率,有效提高产前诊断的准确性和特异性。
同时,WES在产前诊断的临床实践中也面临着如方法局限性、遗传咨询、社会伦理学等方面的挑战。
因此,本文主要针对WES目前在产前诊断的应用情况进行讨论,并对其今后发展方向进行展望。
【关键词】 全外显子组测序;产前诊断;分子诊断【中图分类号】 R714.55 【文献标识码】 A犇犗犐:10.13470/j.cnki.cjpd.2020.02.012 通信作者:魏佳雪,E mail:weijiaxue@hotmail.com 我国是出生缺陷高发国家,每年新增出生缺陷数约90万例,其中出生时临床明显可见的出生缺陷约有25万例[1]。
同时,胎儿异常能在2%~5%的妊娠中被发现,且围产期死亡例数的20%[2 5]。
因此,产前诊断是防治出生缺陷的重要内容。
出生缺陷病因包括环境因素和遗传因素,根据《全球出生缺陷报告》,全球每年出生遗传相关出生缺陷婴儿近800万,占出生总人数6%[6]。
高通量测序技术及原理介绍高通量测序技术(High-throughput sequencing)又称“下一代”测序技术(“Next-generation”sequencing technology),以能一次并行对几十万到几百万条DNA分子进行序列测定和一般读长较短等为标志。
高通量测序技术应用测序技术推进科学研究的发展。
随着第二代测序技术的迅猛发展,科学界也开始越来越多地应用第二代测序技术来解决生物学问题。
比如在基因组水平上对还没有参考序列的物种进行从头测序(de novo sequencing),获得该物种的参考序列,为后续研究和分子育种奠定基础;对有参考序列的物种,进行全基因组重测序(resequencing),在全基因组水平上扫描并检测突变位点,发现个体差异的分子基础。
在转录组水平上进行全转录组测序(whole transcriptome resequencing),从而开展可变剪接、编码序列单核苷酸多态性(cSNP)等研究;或者进行小分子RNA测序(small RNA sequencing),通过分离特定大小的RNA分子进行测序,从而发现新的microRNA分子。
在转录组水平上,与染色质免疫共沉淀(ChIP)和甲基化DNA免疫共沉淀(MeDIP)技术相结合,从而检测出与特定转录因子结合的DNA区域和基因组上的甲基化位点。
这边需要特别指出的是第二代测序结合微阵列技术而衍生出来的应用--目标序列捕获测序技术(Targeted Resequencing)。
这项技术首先利用微阵列技术合成大量寡核苷酸探针,这些寡核苷酸探针能够与基因组上的特定区域互补结合,从而富集到特定区段,然后用第二代测序技术对这些区段进行测序。
目前提供序列捕获的厂家有Agilent和Nimblegen ,应用最多的是人全外显子组捕获测序。
科学家们目前认为外显子组测序比全基因组重测序更有优势,不仅仅是费用较低,更是因为外显子组测序的数据分析计算量较小,与生物学表型结合更为直接。
292新医学2024年4月第55卷第4期DOI: 10.3969/j.issn.0253-9802.2024.04.010研究论著3例CHDFIDD患儿的分子遗传学分析及文献复习赵静 宣小燕 李红英 陈政芳 范晓萱 赵晓科【摘要】目的 分析先天性心脏缺陷、面部畸形和智力发育障碍[CHDFIDD,细胞周期蛋白依赖性激酶13(CDK13)相关疾病)]患儿的临床表型及基因突变情况,探讨其遗传学病因。
方法 采用芯片捕获高通量测序技术对3例CHDFIDD患儿及其父母的基因组DNA进行全外显子组测序,对疑似致病突变进行Sanger测序验证和生物信息分析。
以“CDK13 基因”“CDK13相关疾病”为检索词,检索中国知网、万方数据库建库至2024年2月的文献;以“CDK13”“CDK13-related disorder ”“CHDFIDD”为检索词,检索PubMed数据库建库至2024年2月的文献,对相关文献进行复习。
结果 全外显子组测序结果均提示3例患儿存在CDK13基因杂合突变,分别为c.2572C>T (p.Leu858Phe)、c.2579G>A(p.Arg860Gln)和c.2602C>T (p.Arg868Trp),Sanger测序也证实了3种突变,结合临床表型,3例患儿均被确诊为CHDFIDD。
3例患儿在各自家系中表现为新发突变;但不排除患儿双亲之一为该突变的生殖系嵌合体。
根据美国医学遗传学与基因组学学会的指南,3个突变位点均可能致病。
文献复习检索到14篇相关文献共108例CHDFIDD病例,其中c.2572C>T突变未见文献报道。
结论 CDK13基因突变可能是该3例患儿的遗传学病因。
本研究丰富了CDK13基因突变谱,为CHDFIDD相关疾病的诊疗提供了参考。
【关键词】先天性心脏缺陷、面部畸形和智力发育障碍;CDK13基因;CDK13相关疾病;全面性发育迟缓;智力障碍Molecular genetic analysis of three children with CHDFIDD and literature review Zhao Jing, Xuan Xiaoyan, Li Hongying,Chen Zhengfang, Fan Xiaoxuan, Zhao Xiaoke. Children’s Hospital of Nanjing Medical University, Nanjing Children’s Hospital,Nanjing 210008, ChinaCorresponding author: Zhao Xiaoke, E-mail:******************.com【Abstract】Objective To analyze the clinical and genetic characteristics of three children with congenital heart defects,dysmorphic facial features and intellectual developmental disorders(CHDFIDD). Methods Three children presenting with CHDFIDD were enrolled. Genomic DNA was extracted from peripheral venous blood of the children and their parents. Whole-exome sequencing(WES) was performed using chip-capture high-throughput sequencing technology. Suspected causative mutations were veri fi ed by Sanger sequencing and bioinformatic analysis. Using “CDK13 gene” and “CDK13-related diseases” as search terms,literatures of CNKI and Wanfang database were retrieved until February 2024. Using “CDK13”,“CDK13-related disorder” and “CHDFIDD” as search terms, literatures from the establishment of PubMed database untio February 2024 was retrieved, and the relevant literature was reviewed. Results WES revealed heterozygous variants of the CDK13 gene in three children, including c.2572 C>T (p.Leu858Phe), c.2579 G>A (p.Arg860Gln), and c.2602C>T (p.Arg868Trp), which were veri fi ed as de novo variants by Sanger bined with the clinical phenotype, all 3 children were diagnosed with CHDFIDD. However, the possibility that one of the a ff ected children’s parents was a germline chimera for the mutation could not be excluded. According to the ACMG guidelines,all three mutation sites were classi fi ed as likely pathogenic. A total of 14 studies consisting of 108 cases were retrieved. Among them,c.2572C>T has not been reported. Conclusions The de novo variants of the CDK13 gene may be the genetic cause of developmental delay/intellectual disability in these three children. The fi ndings in the present study expand the spectrum of CDK13 gene mutations,providing reference for the diagnosis of CHDFIDD.【Key words】Congenital heart defects, dysmorphic facial features and intellectual developmental disorders; CDK13 gene;CDK13-related disorder;Global developmental delay;Intellectual disability基金项目:江苏省妇幼保健协会科研基金(FYX202013)作者单位:210008 南京,南京医科大学附属儿童医院南京儿童医院通信作者:赵晓科,E-mail:******************.com2024年4月第55卷第4期293新医学先天性心脏缺陷、面部畸形和智力发育障碍(CHDFIDD)又被称为细胞周期蛋白依赖性激酶13(CDK13)相关疾病,是由CDK13 (OMIM:603309)基因突变所致的常染色体显性遗传性疾病,其临床特征主要包括发育迟缓或智力障碍、可识别的颅面特征、小头畸形、先天性心脏异常和大脑结构异常等[1]。
什么是高通量测序?高通量测序技术(High-throughput sequencing,HTS)是对传统Sanger测序(称为一代测序技术)革命性的改变, 一次对几十万到几百万条核酸分子进行序列测定, 因此在有些文献中称其为下一代测序技术(next generation sequencing,NGS )足见其划时代的改变, 同时高通量测序使得对一个物种的转录组和基因组进行细致全貌的分析成为可能, 所以又被称为深度测序(Deep sequencing)。
什么是Sanger法测序(一代测序)Sanger法测序利用一种DNA聚合酶来延伸结合在待定序列模板上的引物。
直到掺入一种链终止核苷酸为止。
每一次序列测定由一套四个单独的反应构成,每个反应含有所有四种脱氧核苷酸三磷酸(dNTP),并混入限量的一种不同的双脱氧核苷三磷酸(ddNTP)。
由于ddNTP 缺乏延伸所需要的3-OH基团,使延长的寡聚核苷酸选择性地在G、A、T或C处终止。
终止点由反应中相应的双脱氧而定。
每一种dNTPs和ddNTPs的相对浓度可以调整,使反应得到一组长几百至几千碱基的链终止产物。
它们具有共同的起始点,但终止在不同的的核苷酸上,可通过高分辨率变性凝胶电泳分离大小不同的片段,凝胶处理后可用X-光胶片放射自显影或非同位素标记进行检测。
什么是基因组重测序(Genome Re-sequencing)全基因组重测序是对基因组序列已知的个体进行基因组测序,并在个体或群体水平上进行差异性分析的方法。
随着基因组测序成本的不断降低,人类疾病的致病突变研究由外显子区域扩大到全基因组范围。
通过构建不同长度的插入片段文库和短序列、双末端测序相结合的策略进行高通量测序,实现在全基因组水平上检测疾病关联的常见、低频、甚至是罕见的突变位点,以及结构变异等,具有重大的科研和产业价值。
什么是de novo测序de novo测序也称为从头测序:其不需要任何现有的序列资料就可以对某个物种进行测序,利用生物信息学分析手段对序列进行拼接,组装,从而获得该物种的基因组图谱。
第二代测序技术及其法医学运用基因组DNA 碱基序列差异是不同个体间最本质的遗传差异,对碱基序列的测定,人们一直在努力探索。
到目前为止测序技术经历了三代发展。
第一代测序技术以Sanger双脱氧核苷酸链终止法和Maxim降解法为代表,但由于Sanger 双脱氧核苷酸链终止法比Maxim 化学降解法需要更少的有毒化学药品,放射性同位素标记[1],因此它成为了DNA 测序常用的技术,而且其测序准确性高,被认为是确定基因序列的金标准。
第一代测序技术,因其操作简单,读长长,而被广泛运用。
但第一代测序无法实现一次大样本测序,而且耗时长,成本高。
以高通量、低成本为主要特点的第二代测序技术应运而生。
二代测序技术(又称下一代测序技术,next gen eratio n seque ncin g ,NGS主要包括Roche公司的454测序技术、lllumine公司的Solexa测序技术和ABI公司的SOLID测序技术。
二代测序技术比起一代测序技术在测序通量、成本、时间等方面都有突破。
二代测序技术最主要的特点就是通量高,一次能对多个样本同时测序,但是二代测序技术因其高通量的同时会带来相对短的读长,这是二代测序技术发展面临的主要问题。
第三代测序技术(又称为next-next-generation sequencing是以单分子测序为主要特征,而不经过前期文库的构建、PCR 扩增目的基因片段,使测序更为精确。
目前对DNA 单分子直接测序的第三代测序技术还在研究。
第二代测序技术操作简单、通量高、成本低,仍是目前应用较为广泛的技术。
本文就二代测序技术及其法医学运用做简要介绍。
1 第二代测序技术Roche公司的454测序技术,利用焦磷酸测序的原理,通过DNA延伸时产生的焦磷酸盐来确定碱基顺序。
即如果加入的未标记的dNTP 与模板链互补,在DNA聚合酶的作用下产生等摩尔的焦磷酸盐(ppi),焦磷酸盐在硫酸化酶、荧光素酶的作用下转变成荧光,根据加入的dNTP 的类型和荧光信号的强度,可以确定模板DNA 核苷酸的序列。
人全外显子组序列捕获及第二代测序
概述
外显子组是指全部外显子区域的集合,该区域包含合成蛋白质所需要的重要信息,涵盖了与个体表型相关的大部分功能性变异。
外显子组序列捕获及第二代测序是一种新型的基因组分析技术:外显子序列捕获芯片(或溶液)可在同一张芯片上以高特异性和高覆盖率捕获研究者感兴趣的目标外显子区域,后续利用Solexa/SOLiD/Roche 454测序直接解析数据。
与全基因组重测序相比,外显子组测序只需针对外显子区域的DNA 即可,覆盖度更深、数据准确性更高,更加简便、经济、高效。
可用于寻找复杂疾病(如:癌症、糖尿病、肥胖症等)的致病基因和易感基因等的研究。
同时,基于大量的公共数据库提供的外显子数据,我们能够结合现有资源更好地解释我们的研究结果。
目前,SBC提供的外显子组序列捕获芯片是NimbleGen Sequence Capture 2.1M Human Exome Array及Agilent SureSelect Target Enrichment System(Human Exome)。
技术路线
以Nimblegen外显子捕获结合Solexa测序为例加以说明:基因组DNA首先被随机打断成500bp左右的片段,随后在DNA片段两端分别连接上接头。
经过PCR库检合格后的DNA 片段与NimbleGen 2.1M Human Exome Array芯片进行杂交。
去除未与芯片结合的背景DNA 后,将经过富集的外显子区域的DNA片段洗脱下来。
这些DNA片段又随机连接成长DNA片段
后,再次被随机打断并在其两端加上测序接头,经过LM-PCR的线性扩增,在经qPCR质量检测合格后即可上机测序。
外显子组测序的实验流程示意图()
生物信息学分析流程图
研究内容
1.外显子组捕获与测序
将基因组DNA随机打断成片段,通过与人全外显子捕获芯片杂交富集外显子区域,通过第二代测序技术对捕获的序列进行测序。
2.基本数据分析
数据产出统计:对测序结果进行图像识别(Base calling),去除污染及接头序列;统计结果包括:测定的序列(Reads)长度、Reads数量、数据产量。
3. 高级数据分析
高级数据分析内容包括:
(1)Clean reads序列与参考基因组序列比对;
(2)目标外显子区域测序深度分析;
(3)目标外显子区域一致序列组装;
(4)目标外显子区域SNP检测及在CCDS数据库中的注释;
(5)可变剪切、基因融合、外显子融合分析。
技术特点
相关产品技术参数
SureSelect Human All Exon 50Mb Kit(New)
捕获量:50Mb
捕获对象:
1、GENCODE project中发现的外显子(约12M)
2、NCBI Consensus CDS database(CCDS, March 2009)中的外显子
3、Sanger V13 database中的miRNA
4、多于300条的human non-coding RNAs(例如snoRNAs和scaRNAs)
* All Exon 50Mb试剂盒是Agilent与Wellcome Trust Sanger Institute、 Gencode consortium合作开发的人全外显子捕获试剂盒。
在前款产品(SureSelect Human All Exon Kit)的基础上,加入了WT Sanger实验室发现的人外显子(12Mb),合计为50Mb。
这款产品是目前市面上覆盖最全面的人全外显子捕获试剂盒。
SureSelect Human All Exon Kit
捕获量:38Mb
捕获对象:
1、NCBI Consensus CDS database(CCDS)中的外显子(约为人类基因组的1.22%)
2、Sanger V13 database中的miRNA
3、多于300条的human non-coding RNAs(例如snoRNAs和scaRNAs)
SureSelect Human All Exon Plus Kit
捕获量:44Mb
捕获对象:
在SureSelect Human All Exon Kit的基础上加入6M可以自主选择的捕获区域(需要用e-Array设计)
All Exon Kit与All Exon 50Mb Kit的覆盖范围对比。