RT引物序列
- 格式:doc
- 大小:28.50 KB
- 文档页数:2

pcr知识点总结归纳PCR(Polymerase Chain Reaction),即聚合酶链反应,是一种用于抑制、合成、扩增DNA的技术。
PCR技术广泛应用于科学研究、临床诊断、法医鉴定和生物工程等领域。
PCR技术的出现不仅提高了DNA的扩增速度,而且在很大程度上解决了DNA分析的难题和可行性问题。
PCR技术的关键在于DNA的扩增,通过特定的引物(primer)和热稳定的DNA聚合酶在不同温度下进行多次循环反应,使目标DNA片段扩增成百上千倍。
PCR技术的应用可以在较短时间内获得充足的DNA,为后续的实验提供了可行性基础。
PCR技术是分子生物学研究的重要工具,掌握PCR技术的原理和操作方法对于分子生物学研究者来说至关重要。
下面将对PCR技术的知识点进行总结和归纳,包括PCR的基本原理、PCR反应体系、PCR引物设计、PCR技术的优缺点以及PCR技术在生物学研究中的应用等方面。
一、PCR的基本原理PCR技术是通过DNA的酶解、DNA的引物延伸、DNA的片段合成来实现DNA扩增。
PCR主要由以下三个步骤组成:变性、退火和延伸。
1. 变性步骤:将DNA的双链解链成两条单链,即使DNA解链。
2. 退火步骤:将引物与DNA模板结合成双链,即使DNA复性。
3. 延伸步骤:在退火变性条件下将引物作为起始核酸,然后DNA聚合酶将其扩增成DNA。
通过以上三个步骤的循环反应即可实现DNA的多次扩增。
二、PCR反应体系PCR反应体系主要包括DNA模板、引物、DNA聚合酶、四种dNTPs、缓冲液和辅助剂等。
1. DNA模板:PCR反应的起始材料,可以是基因组DNA、cDNA或其他DNA模板。
2. 引物:在退火步骤中将与DNA模板特异性结合,为DNA的扩增提供起始核酸。
3. DNA聚合酶:用于合成DNA,PCR反应中通常采用热稳定的DNA聚合酶如Taq DNA聚合酶。
4. dNTPs:即脱氧核苷酸三磷酸盐,即dATP、dCTP、dGTP和dTTP,是DNA聚合的四种脱氧核苷酸单体。

RT-PCR常用引物序列RT-PCR引物序列基因来源引物序列产物大小(kb)β-actin 人有意义链CCTCG CCTTT GCCGA TCC 反义链GGA TC TTCAT GAGGT AGTCA GTC 0.62 kbβ-actin* 大鼠有意义链TACAA CCTCC TTGCA GCTCC 反义链GGA TC TTCA T GAGGT AGTCA GTC 0.62kbβ-actin 小鼠有意义链GTCGT ACCAC AGGCA TTGTG A TGG反义链GCAAT GCCTG GGTAC ATGGT GG 0.49 kbGAPDH 人有意义链GGTGA AGGTC GGAGT CAACG反义链CAAAG TTGTC ATGGA TGHACC 0.50kbGAPDH 大鼠有意义链GATGC TGGTG CTGAG TATGR CG反义链GTGGT GCAGG ATGCA TTGCT CTGA 0.20 kbDynein 小鼠有意义链GCGGG CGCTG GAGGA GAA反义链GGA TC TTCA T GAGGT AGTCA GTC 12.3 kbPolymerase ε 人有意义链CGCCA AATTT CTCCC CTGAAA反义链CCGTA GTGCT GGGCA ATGTT C 6.8 kbPolymerase ε 人有意义链AAGGC TGGCG GATTA CTGCC反义链GA TGC TGCTG GTGAT GTACT C 3.5 kbTuberous Sclerosis 人有意义链GGAGT TTATC ATCAC CGCGG AAATA CTGAG AG反义链TATTT CACTG ACAGG CAATA CCGTC CAAGG 5.3 kb18S rRNA 大豆有意义链CTTTC GATGG TAGGA TAGTG GCCT反义链CAATG A TCCT TCCGC AGGTT CACCT AC 1.5 kb*引物不会扩增假基因PCR引物序列基因来源引物序列产物大小(kb)HIV gag region 病毒SK 38ATTAAT CACTA TCCAG TAGGA GAAATSK 39TTTGG TCCTG TCTTA TGTCC AGAAT GC 0.11kbβ-globin 人(29923)GGTGT TCCCT TGATG TAGCA CA(34016)CCAGG ATTTT TGATG GGACA CG 4.1kbβ-globin 人(31194)GCTGC TCTGT GCATC CGAGT GG(34016)CCAGG ATTTT TGATG GGACA CG 2.8kb序列来源:nvitrogen 公司大家用什么稀释引物?引物应该用TE稀释。

RT-PCR的步骤引言逆转录聚合酶链式反应(RT-PCR)是一种常用的分子生物学技术,用于从RNA样本中扩增特定的DNA片段。
该技术可以检测和定量特定基因的表达水平,并在疾病诊断、药物研究等领域发挥重要作用。
本文将介绍RT-PCR的步骤及其基本原理。
步骤1. 提取RNA在进行RT-PCR之前,首先需要从细胞或组织中提取RNA。
常用的RNA提取方法包括酚-氯仿法、硅胶酸盐柱法等。
通过这些方法,可以将总RNA或特定种类的RNA从样本中纯化出来,为后续的实验步骤提供RNA样本。
2. 逆转录(RT)逆转录是将RNA转录为相应的cDNA(互补DNA)的过程。
逆转录反应中使用逆转录酶,该酶具有转录RNA为cDNA的能力。
在逆转录反应中,逆转录酶合成一个与RNA模板互补的cDNA链。
常见的逆转录酶包括M-MLV逆转录酶和SuperScript逆转录酶等。
3. PCR扩增PCR扩增是通过引物(寡核苷酸序列)将需要扩增的目标DNA片段进行反复循环扩增的过程。
在PCR反应中,每轮反应包括三个步骤:解链、引物结合和延伸。
通过不断循环这三个步骤,DNA片段将被指数级扩增。
4. 凝胶电泳PCR扩增后,我们需要将扩增产物进行凝胶电泳,以确认是否扩增成功,并确定目标DNA片段的大小。
在凝胶电泳中,将PCR产物加载到琼脂糖凝胶孔中,并施加电场,1使DNA片段在凝胶中迁移。
通过与已知大小的DNA分子量标准进行比较,可以确定PCR扩增产物的大小。
结论RT-PCR技术已经成为分子生物学领域中不可或缺的工具。
通过逆转录和PCR扩增,可以快速、准确地扩增RNA样本中感兴趣的DNA片段,为疾病诊断和基因表达研究提供了重要的技术支持。
了解RT-PCR的步骤和基本原理,有助于更好地应用该技术进行实验和数据解读。
2。

RTPCR的基本原理一、什么是RTPCRRTPCR(Reverse Transcription Polymerase Chain Reaction)是一种基因检测技术,常用于检测RNA的数量和序列特征。
通过该技术,可以对RNA进行逆转录合成DNA,然后利用聚合酶链反应(PCR)放大DNA的特定片段。
RTPCR技术在医学、生物学和疾病诊断领域具有广泛的应用。
二、RTPCR的基本步骤RTPCR技术主要包括逆转录、PCR放大和检测三个步骤。
1. 逆转录逆转录是将RNA转录为DNA的过程。
在逆转录过程中,需要使用逆转录酶、RNA 模板和引物(primers)进行反应。
逆转录酶能够将RNA模板中的核苷酸序列反转录成互补的DNA链。
引物是一小段DNA或RNA序列,在逆转录过程中与RNA模板发生互补配对,作为起始合成新DNA链的起点。
2. PCR放大PCR放大是将逆转录得到的DNA片段进行指数级扩增的过程。
在PCR反应体系中,需要加入逆转录产物、DNA聚合酶、引物和dNTPs(脱氧核苷酸三磷酸盐)。
PCR 反应通过循环加热和冷却的步骤,在不同温度下引发DNA的分离、复性和扩增。
反复的PCR循环能够使目标DNA片段的数量成倍增加。
3. 检测PCR放大后的DNA片段可以进行进一步的检测和分析。
常用的检测方法包括凝胶电泳、荧光探针或荧光染料结合等技术。
通过这些方法,可以检测到目标DNA片段的数量和特异性。
三、RTPCR的应用RTPCR技术在医学和生物学领域有广泛的应用。
1. 疾病诊断RTPCR可以检测病原体的基因序列,用于疾病的早期诊断和追踪。
例如,在新型冠状病毒肺炎(COVID-19)疫情中,RTPCR被广泛应用于检测病毒的核酸,以帮助诊断和追踪病例。
2. 基因表达分析RTPCR可以检测和分析基因的表达水平。
通过检测特定基因的转录水平,可以了解基因在不同组织、不同发育阶段或不同环境条件下的表达差异。
这对于研究基因功能和调控机制非常重要。

RT—PCR引物设计原则与方法近刚开始做PCR,参考了很多与引物设计相关得帖子,结合自己使用primer5与oligo得体会,想谈谈自己设计引物得方法与步骤,恳请各位前辈指正、ﻫﻫRT—PCR引物设计原则与方法ﻫﻫ在NCB I上搜索到该基因,找到该基因得mRNA,在CDS选项中,找到编码区所在位置,在下面得origin 中,Copy该编码序列作为软件查询序列得候选对象。
ﻫ打开Primer Premier5,点击sequence, 出现输入序列窗口,Copy目得序列在输入框内(选择As),此窗口内,序列也可以直接翻译成蛋白。
点击Primer,进入引物窗口。
ﻫﻫ此窗口可以链接到“引物搜索”、“引物编辑”以及“搜索结果"选项,点击Search按钮,进入引物搜索框,选择“PCR pri mers”,“Pairs”,设定搜索区域与引物长度与产物长度。
在Search Parameters里面,可以设定相应参数。
一般若无特殊需要,参数选择默认即可,但产物长度可以适当变化,因为100~200bp得产物电泳跑得较散,所以可以选择300~500bp。
ﻫ点击OK,软件即开始自动搜索引物,搜索完成后,会自动跳出结果窗口,搜索结果默认按照评分(Rating)排序,点击其中任一个搜索结果,可以在“引物窗口"中,显示出该引物得综合情况,包括上游引物与下游引物得序列与位置,引物得各种信息等。
ﻫ对于引物得序列,可以简单查瞧一下,避免出现下列情况:3’端不要以A结尾,最好就是G或者C,T也可以;3'不要出现连续得3个碱基相连得情况,比如GGG或 CCC,否则容易引起错配。
此窗口中需要着重查瞧得包括:Tm应该在55~70度之间,GC%应该在45%~55%间,上游引物与下游引物得Tm 值最好不要相差太多,大概在2度以下较好。
该窗口得最下面列出了两条引物得二级结构信息,包括,发卡,二聚体,引物间交叉二聚体与错误引发位置。

问题一:如何在pubmed上找到目标基因的DNA序列以及其内含子和外显子情况。
步骤:打开NCBI主页面,选GENE,输入基因名称-》显示出很多不同种但名字相同的基因(fig.1)-》找到你要的种属,打开-》显示基因信息,找到”Genomic regions, transcripts, and products”,点击基因名称,links到geneback(fig.2)-》显示了该基因DNA的信息(fig.3),可以看到这是基因组DNA,从mRNA选项中可以看到JION后面的是外显子的位置,该基因一个共有3个内含子,4个外显子,以及外显子的起至位置,为了以后的操作,可以把这个DNA序列和外显子的起至位置记下来。
问题二:如何在pubmed上找到目标基因的mRNA序列。
步骤:打开NCBI主页面,选GENE,输入基因名称-》显示出很多不同种但名字相同的基因-》找到你要的种属,打开-》显示基因信息,找到“mRNA and protein”前面那个NM***********,号码(fig.4),把它记下来(后面的blast 要用)并点开-》显示这个基因的mRNA信息(Fig.5),最后的ORIGIN是CDNA序列,把它记下来,一会PRIMER的时候要用。
问题三:如何设计跨内含子的引物(利用primer-blast)1)打开primer-blast网站:()2)将mRNA编号(NM***********)输入序列框中,real time PCR 一般产物长度为80~150bp也可以超一点,但是会影响扩增效率,这样就不能用ddCT法计算了,酌情考虑。
温度在55~63和GC含量在40%~60%,两条链不要差太远。
3)会得到若干条引物(fig。
6),红条中的黄圈标记的小白色缺口是内含子所在的位置,跨过小缺口就是跨了内含子。
根据下面列出的引物具体的温度GC含量之类的再进行选择。
4)将初步选择的引物用PRIMER 5验证引物二聚体,发卡结构等,方法见下个问题“问题四:用PRIMER 5查找已知引物的产物位置”。

RTPCR的原理实验步骤及应用概述逆转录聚合酶链式反应(Reverse Transcription Polymerase Chain Reaction,简称RT-PCR),是一种常用的分子生物学实验技术,用于检测和定量分析RNA分子在细胞中的表达水平。
本文将介绍RT-PCR的原理、实验步骤及应用。
原理RTPCR利用逆转录酶(Reverse Transcriptase,简称RT)将RNA模板逆转录为cDNA(complementary DNA),然后通过聚合酶链式反应(Polymerase Chain Reaction,简称PCR)对cDNA进行扩增,最终得到大量的目标DNA片段。
RT-PCR的基本原理如下:1.逆转录:反转录酶RT利用RNA模板合成与之互补的cDNA。
在逆转录过程中,需要引入一条逆转录引物(反向互补RNA链)作为反转录的起始点。
2.PCR扩增:将逆转录合成的cDNA作为模板,引入一对特异性引物,通过PCR反应进行DNA扩增。
PCR反应分为三个步骤:变性、退火和扩增。
3.变性:将反应温度升高至95°C,使DNA双链变性为两条单链。
4.退火:将反应温度降低至特异性引物的退火温度,使引物与模板序列互相结合。
5.扩增:将反应温度升高至适合DNA聚合酶的活性温度,使DNA聚合酶逆反应合成DNA。
通过多轮的PCR反应,可以扩增出大量的目标DNA片段,其数量呈指数级增长。
实验步骤1. 样品处理首先需要从待检测的样品中提取总RNA,常用的提取方法有酚氯仿法和柱式纯化法。
提取得到的总RNA需要通过比色法或者用分光光度计进行测量,确保样品的质量和浓度。
2. 逆转录反应逆转录反应是将RNA模板转录为cDNA的过程。
需要准备逆转录试剂盒,主要包括逆转录酶、随机引物、dNTPs(脱氧核苷酸三磷酸盐)和逆转录缓冲液。
按照试剂盒说明书的配方,将总RNA与逆转录试剂混合,进行逆转录反应。
反应结束后,需要进行热灭活,以停止反应。

RT-PCR技术简介和RT-PCR引物的选择RT-PCR简介RT-PCR是将RNA的反转录(RT)和cDNA的聚合酶链式扩增(PCR)相结合的技术。
首先经反转录酶的作用从RNA合成 cDNA,再以cDNA为模板,扩增合成目的片段。
RT-P CR技术灵敏而且用途广泛,可用于检测细胞中基因表达水平,细胞中RNA病毒的含量和直接克隆特定基因的cDNA序列。
作为模板的RNA可以是总RNA、mRNA或体外转录的RNA产物。
无论使用何种RNA,关键是确保RNA中无RNA 酶和基因组DNA的污染。
使用天为时代公司的总RNA提取系统(如目录号 DP405和DP406),所获得的RNA的纯度高,基因组DNA污染少,用于RT-PCR系统可得到满意结果。
用于反转录的引物可视实验的具体情况选择随机引物、Oligo dT 及基因特异性引物中的一种。
对于短的不具有发卡结构的真核细胞mRNA,三种都可。
RT-PCR引物的选择随机引物适用于长的或具有发卡结构的RNA。
适用于rRNA、 mRNA、tRNA 等所有RNA 的反转录反应。
主要用于单一模板的RT-PCR反应。
Oligo dT 适用于具有PolyA尾巴的RNA。
(原核生物的 RNA、真核生物的 Oligo dT rRNA和tRNA不具有PolyA尾巴。
)由于Oligo dT要结合到PolyA 尾巴上,所以对RNA样品的质量要求较高,即使有少量降解也会使全长cDNA合成量大大减少。
基因特异性引物与模板序列互补的引物,适用于目的序列已知的情况。
天为时性引物代公司的SuperScript One-Step System特别适合于与基因特异性引物连用。
RT-PCR影响因素RT-PCR反应受多个因素影响,如硫酸镁的浓度, 引物退火的温度,扩增的循环数等。
◇建议选择0.5-3.0 mM (相差0.5 mM)的硫酸镁作初步实验。
◇对于具有较高Tm的引物,增加退火和延伸时的温度对反应有利。
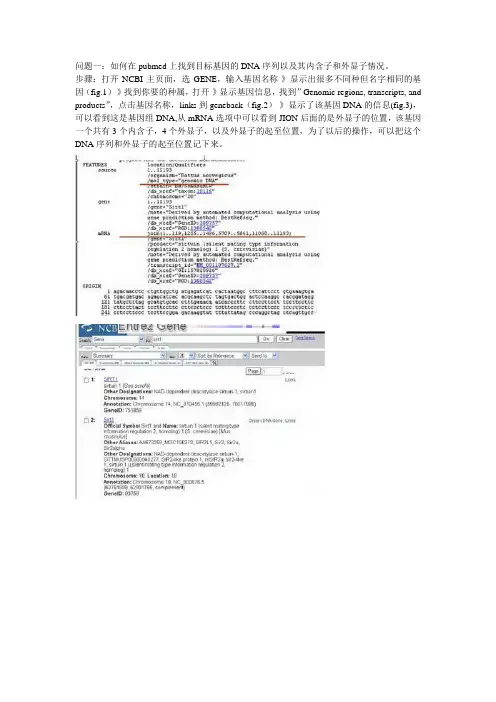
问题一:如何在pubmed上找到目标基因的DNA序列以及其内含子和外显子情况。
步骤:打开NCBI主页面,选GENE,输入基因名称-》显示出很多不同种但名字相同的基因(fig.1)-》找到你要的种属,打开-》显示基因信息,找到”Genomic regions, transcripts, and products”,点击基因名称,links到geneback(fig.2)-》显示了该基因DNA的信息(fig.3),可以看到这是基因组DNA,从mRNA选项中可以看到JION后面的是外显子的位置,该基因一个共有3个内含子,4个外显子,以及外显子的起至位置,为了以后的操作,可以把这个DNA序列和外显子的起至位置记下来。
问题二:如何在pubmed上找到目标基因的mRNA序列。
步骤:打开NCBI主页面,选GENE,输入基因名称-》显示出很多不同种但名字相同的基因-》找到你要的种属,打开-》显示基因信息,找到“mRNA and protein”前面那个NM***********,号码(fig.4),把它记下来(后面的blast要用)并点开-》显示这个基因的mRNA信息(Fig.5),最后的ORIGIN是CDNA序列,把它记下来,一会PRIMER的时候要用。
问题三:如何设计跨内含子的引物(利用primer-blast)1)打开primer-blast网站:(/tools/primer-blast/)2)将mRNA编号(NM***********)输入序列框中,real time PCR 一般产物长度为80~150bp也可以超一点,但是会影响扩增效率,这样就不能用ddCT法计算了,酌情考虑。
温度在55~63和GC含量在40%~60%,两条链不要差太远。
3)会得到若干条引物(fig。
6),红条中的黄圈标记的小白色缺口是内含子所在的位置,跨过小缺口就是跨了内含子。
根据下面列出的引物具体的温度GC含量之类的再进行选择。
4)将初步选择的引物用PRIMER 5验证引物二聚体,发卡结构等,方法见下个问题“问题四:用PRIMER 5查找已知引物的产物位置”。

rt pcr的常用方法rt-PCR( real-time PCR )是一种用于检测和定量新型冠状病毒(SARS-CoV-2)核酸的技术。
下面是 rt-PCR 的常用方法及其拓展:1. 设计引物:rt-PCR 的第一步是设计引物。
引物是探针分子,用于结合目标核酸,并用于在 rt-PCR 中扩增目标核酸。
引物的质量和准确性对 rt-PCR 的结果至关重要。
通常,引物由两个互补的短链组成,分别位于引物的两端。
引物的长度和结构可以影响 rt-PCR 的反应时间和结果。
2. 提取核酸:从患者或样本中提取足够的核酸是 rt-PCR 的第一步。
通常,需要使用核酸提取试剂盒,将患者或样本中的核酸提取出来。
核酸提取的准确性和效率对 rt-PCR 的结果也有很大的影响。
3. 混合核酸:将提取的核酸与引物一起混合,并使用 rt-PCR 试剂盒进行扩增。
扩增过程中,引物将与目标核酸结合,并生成一个反应物。
这个反应物可以在rt-PCR 中测量,并计算出病毒的数量。
4. 控制温度和湿度:rt-PCR 的反应需要一定的温度和湿度条件。
通常,需要使用专门的 rt-PCR 设备,并将设备放置在适当的温度和湿度环境中。
这有助于提高 rt-PCR 的准确性和效率。
5. 数据分析:一旦扩增了目标核酸,就需要对数据进行分析。
通常,使用rt-PCR 数据分析软件对数据进行处理和可视化。
这可以帮助确定扩增是否成功,并确定病毒的数量。
rt-PCR 是一种快速、准确和灵敏的方法来检测和定量新型冠状病毒。
通过设计合适的引物,提取足够的核酸,并将核酸与引物一起混合,并控制温度和湿度,可以提高 rt-PCR 的准确性和效率。

rtpcr的常用方法一、RT-PCR的基本原理1. 逆转录反应(Reverse Transcription, RT):RT-PCR的第一步是将RNA转录成互补的DNA,这一步叫做逆转录反应。
逆转录反应通过引入RNA逆转录酶(Reverse Transcriptase)和随机引物(Random Primers),将RNA模板转录成cDNA(反转录DNA)。
2. 聚合酶链反应(Polymerase Chain Reaction, PCR):逆转录完成后,所得的cDNA经过稀释和变性等预处理后,使用DNA聚合酶(DNA Polymerase)和两个特异性引物,通过多轮的循环反应来扩增目标DNA区域。
二、RT-PCR的基本步骤1.RNA提取和纯化:从样品中提取并纯化RNA,以保证所得的RNA具有较高的纯度和完整性。
2.反转录反应:将RNA转录成cDNA,这一步可通过两种方式进行:使用随机引物、dNTPs、逆转录酶等直接反转录,或通过特异性引物(即RT引物)进行引物延伸。
3. PCR扩增反应:将逆转录所得的cDNA作为模板,使用特异性引物和DNA聚合酶进行DNA扩增。
PCR的循环条件通常是:变性(denaturation)、退火(annealing)和延伸(extension)。
4.电泳分析:将PCR产物经过聚丙烯酰胺凝胶电泳,通过对比分子量标准品,判断扩增产物的大小。
三、RT-PCR的优点和局限性1.优点(1)可以从一个RNA样本中扩增目标序列,从而可以检测低丰度的mRNA。
(2)推导出目标基因的相对表达量和定量结果。
(3)能够对基因表达的动态变化进行实时监测。
(4)扩增模板的选择性强,通过控制引物的设计,能够选择性地扩增特定的目标。
2.局限性(1)需要对RNA进行提取和纯化,这一步骤可能会引入一定程度的变异,影响结果。
(2)逆转录过程中可能发生评性引物引起的“板块效应”,导致不同通量的逆转录效率不同。
miRNA RT-PCR引物设计原理对于miRNA RT-PCR实验来说,最核心的实验原理和步骤就是miRNA引物设计思路。
之前大家熟知的miRNA RT-PCR引物主要有两种类型:1、Oligod(T)特异的RT引物(QIAGEN产品为主)由特异序列+(T)20左右+兼并碱基V或VN组成。
(所有miRNA可以公用一个Oligod(T)的RT引物,但是RNA在反转录前需要进行末端Poly(A)加尾)2、茎环状结构的RT引物(ABI产品为主)由可以自身呈环茎状的特异序列+6到8个miRNA3’端反向互补碱基组成。
(一条miRNA序列特异对应一个茎环状结构的RT引物)。
这里主要给大家介绍美国signosis公司的一种新方法。
基于自有专利技术,美国Signosis开发了一种高灵敏、高分辨的实时PCR 方法,用于检测miRNA的表达,其应用寡核苷酸连接和基于实时PCR的SYBR green。
该方法可用于总RNA或细胞裂解物中的miRNA表达的定量分析,无需进行cDNA转换。
该方法中,靶miRNA分子与二对oligo连接,形成RNA/DNA复合物。
当序列完全匹配时,这二对oligo与miRNA连接,二对oligo的节点通过DNA酶连接。
miRNA之间单个寡核苷酸的差异就会阻止杂交或连接。
寡核苷酸对连接上后,连接分子可进行实时PCR。
以下是signosis新方法的原理图:Signosis的新方法有效地解决了许多同源miRNA中只有单个碱基的不同而无法分辨的问题。
并且省去了反转录成cDNA的步骤,同时配有相应的细胞裂解液产品使实验更简便、更可靠。
想了解更多关于该方法的信息可登陆signosis官方网站:/中国总代网站:/。
引物设计不求⼈!⼿把⼿教你设计RT-qPCR引物!RT-qPCR 全称 Real-time polymerase chain reaction, 即实时聚合酶链锁反应,是⼀种在DNA扩增反应中,以萤光染⾊剂检测每次聚合酶链锁反应(PCR)循环后产物总量的⽅法。
OK~这⾥对RT-qPCR的原理,应⽤及数据分析就不再赘述了,因为今天我们主要讲的是如何设计出好的PCR引物。
相信科研⼩伙伴们都已经⽤过Pubmed来设计引物了,基本就是 “傻⽠式操作”就解决了!但这⾥还需提醒⼩伙伴们两点:1. 需跨越⾄少⼀个外显⼦连接(⼀般我们⽤来做RT的RNA都是通过Trizol抽提法分离出来的,为了避免基因组的DNA也有可能被扩增出来,就要⾄少跨越⾄少⼀个exon-exon junction 了)。
2. 不要忘记选对“物种”!(这⾥以⼈种⽰例)但是Pubmed设计出来的引物⼀般不尽如⼈意,⽐如说引物之间存在极⼤的互补性,3’端的互补重叠,形成引物⼆聚体等等。
因为⽤Pubmed设计引物本⾝能设置的参数条件就很少,所以它才是最简单的。
所以,今天我要给⼤家介绍两款实⽤的软件,Primer Premier 6.0 和 Oligo 7.0。
今天主要讲讲第⼀款,⾄于第⼆款,改天再聊啦!当然,这两款软件,要想⽤全功能版本,都是需要付费的哦!不过...,我们伟⼤的祖国有强⼤的某宝,这⾥就不多说了哈。
好了,我们进⼊今天的正题……(说了这么多废话,终于要进⼊正题了!!!)⾸先,我们选⼀个基因举例来说,PPARA,⾄于为什么会选这个基因呢?因为啊,PPAR⽬前是肿瘤学研究中的⼀个“明星基因”,它的全称是Peroxisome proliferator-activated receptor, 即过氧化物酶体增殖物激活型受体,⽬前发现有三种亚型,PPAR a,PPARbeta/delta,PPAR r。
好吧…….听着好复杂的样⼦啊!总之,管它到底是什么呢,下⾯来说说它的引物如何设计。
rt pcr原理
RT-PCR(Reverse Transcription Polymerase Chain Reaction)是
一种常用的分子生物学技术,用于检测RNA分子的存在和浓度。
其原理基于聚合酶链式反应(PCR)和逆转录(RT)两
个步骤的结合。
首先,逆转录步骤将RNA转录成相应的DNA分子。
这一步
骤使用逆转录酶将特定的RNA模板转录成互补的DNA链,
生成称为cDNA的复制的DNA。
逆转录酶会在上述反应中合
成新的DNA链,并在合成的DNA链上加上一些特殊的引物
序列。
接下来,PCR步骤用于扩增目标DNA片段。
PCR反应通过转
录酶链式反应引发DNA的扩增。
反应液中的DNA片段会被PCR引物引导,并在加入热稳定的聚合酶的情况下,经历一
系列的循环性变性、退火和延伸,从而扩增起始 DNA区域。
这些循环热压的步骤会导致指数级别的DNA增加,从而产生
大量特定DNA片段。
扩增反应进行完毕后,可以采用多种方法来检测扩增产物。
最常见的方法是使用凝胶电泳,将扩增产物根据大小进行分离并观察。
此外,还可以使用DNA染料或探针等,在实验室条件
下观察和量化。
总的来说,RT-PCR技术通过结合逆转录和PCR两个步骤,
可以将RNA分子转录成DNA,并对目标DNA片段进行扩增。
这一技术在基因表达分析、病毒检测和遗传研究等领域得到了广泛应用。
β-actin mouse primer 序列-回复在分子生物学和基因表达研究中,基因的测定和定量是非常重要的。
为了确定一个基因的表达水平,研究人员通常使用RT-PCR(逆转录聚合酶链反应)技术来扩增目标基因的片段,并对扩增的产物进行测定和定量分析。
在RT-PCR中,引物是扩增过程中的关键因素之一。
β丝氨酸(β-actin)是一种常用的参考基因,用于在实验中测量和标准化其他基因的表达水平。
首先,我们需要知道β-actin 基因的序列和相应的引物序列。
β-actin 是一种高度保守的测序,其序列在不同物种之间的变化很小。
在小鼠中,β-actin 基因的序列如下:5'-ATGGATGATGATATCGCCGCGCTG-3'(前向引物)5'-ATGAAACGCTCGGTCAGGATCTTCA-3'(反向引物)这些引物序列可以直接用于RT-PCR 扩增β-actin 基因片段。
下面将详细介绍一步一步的实验过程。
第一步是RNA的提取。
假设我们想要从小鼠组织中提取总RNA。
我们可以使用商业化的RNA提取试剂盒,按照说明书的指导进行操作。
一般情况下,将组织样本置于离心管中,加入适量的试剂并破碎组织,然后提取总RNA。
提取的总RNA可以通过紫外光比色法或琼脂糖凝胶电泳进行质量和纯度的检测。
第二步是反转录过程,将总RNA转录成cDNA。
反转录实验要求使用逆转录酶和适用的引物。
在这里,我们将使用以下反转录试剂:- M-MLV逆转录酶- 随机引物- dNTPs- 反转录缓冲液- RNase抑制剂- DEPC水首先,在冰上配置逆转录的反应混合液。
将以下试剂加入1.5mL离心管中:- 2μg总RNA- 1μL随机引物- 1μL dNTPs- 1μL M-MLV逆转录酶- 1μL RNase抑制剂- 2μL反转录缓冲液- 13μL DEPC水混合并离心管轻轻旋转。
然后,将反应混合液加热至65C离解RNA二级结构,然后冷却至42C。
PCR内参:选还是不选?,PCR内参,RT生物帮> 专题> 实验技术> 分子> PCR内参:选还是不选?导读:内参即是内部参照,它们在各组织和细胞中的表达相对恒定,在检测基因的表达水平变化时常用它来做参照物。
常用的PCR内参有GAPDH 、β- actin 、18sRNA、28sRNA 、B2M、ACTB、SDHA、HPRT1、ARBP内参基因等。
本专题就PCR实验中遇见的有关于内参的一些问题作出总结,以供读者参考。
PCR内参基因介绍GAPDH简介内参基因β-Actin简介实时荧光定量PCR 中内参基因的选择内参基因的概念和作用PCR内参基因介绍基因表达转录分析中内参基因的选择目前基因表达的转录分析多采用单一看家基因作为内参来校正目标基因的表达量.实验中以人肝癌BEL-7402细胞为研究对象,应用[详细]海带配子体18S rRNA 基因作为内参基因的应用通过基因克隆和序列测定,获得了海带( Laminaria japonica) 配子体的18S rRNA 基因序列,其长度为1 716bp ,GenBank 登录号为EU293553。
以蓝[详细]白纹伊蚊β2肌动蛋白基因片段作为基因表达内参目的获取白纹伊蚊β2肌动蛋白基因序列并探讨其作为基因表达内参照的作用。
方法根据昆虫β2肌动蛋白核苷酸序列[详细]常用的内参基因及其使用范围包括GAPDH 、β- actin(BETA-actin) 、18sRNA、28sRNA 、B2M、ACTB、SDHA、HPRT1、ARBP内参基因等。
[详细]常用的β-actin 引物序列常用的β-actin 引物序列[详细]RT-PCR常用引物序列引物应该用TE稀释。
oligo在酸性条件下是不稳定的,容易降解,如果用水溶解引物,无法保证pH值,如果合成引物纯化不好,造[详细]PCR内参常见问题汇总内参能跑出来但为何我的cDNA总是不稳定?我要做荧光定量试验,师姐做的定性,我能共用RT-PCR电泳照片有问题qPCR 目的基因与内参是不是要在同样体系里扩增斑马鱼的内参基因组怎么选择啊绝对定量需要用到内参基因吗选择内参基因的原则PCR内参常见问题汇总RT-PCR内参基因选择:除了β-actin还能用什么?实时定量PCR试验中,内参对于实验具有重要意义。
RT-PCR引物设计原则和方法在NCBI上搜索到该基因,找到该基因的m RNA,在CDS选项中,找到编码区所在位置,在下面的origin中,Copy该编码序列作为软件查询序列的候选对象。
打开Primer Premier5,点击File-New-DNA sequence, 出现输入序列窗口,Copy 目的序列在输入框内(选择As),此窗口内,序列也可以直接翻译成蛋白。
点击Primer,进入引物窗口。
此窗口可以链接到“引物搜索”、“引物编辑”以及“搜索结果”选项,点击Search按钮,进入引物搜索框,选择“PCR primers”,“Pairs”,设定搜索区域和引物长度和产物长度。
在Search Parameters里面,可以设定相应参数。
一般若无特殊需要,参数选择默认即可,但产物长度可以适当变化,因为100~200bp的产物电泳跑得较散,所以可以选择300~500bp。
点击OK,软件即开始自动搜索引物,搜索完成后,会自动跳出结果窗口,搜索结果默认按照评分(Rating)排序,点击其中任一个搜索结果,可以在“引物窗口”中,显示出该引物的综合情况,包括上游引物和下游引物的序列和位置,引物的各种信息等。
对于引物的序列,可以简单查看一下,避免出现下列情况:3’端不要以A结尾,最好是G或者C,T也可以;3’不要出现连续的3个碱基相连的情况,比如GGG或CCC,否则容易引起错配。
此窗口中需要着重查看的包括:Tm应该在55~70度之间,GC%应该在45%~55%间,上游引物和下游引物的Tm值最好不要相差太多,大概在2度以下较好。
该窗口的最下面列出了两条引物的二级结构信息,包括,发卡,二聚体,引物间交叉二聚体和错误引发位置。
若按钮显示为红色,表示存在该二级结构,点击该红色按钮,即可看到相应二级结构位置图示。
最理想的引物,应该都不存在这些二级结构,即这几个按钮都显示为“None”为好。
但有时很难找到各个条件都满足的引物,所以要求可以适当放宽,比如引物存在错配的话,可以就具体情况考察该错配的效率如何,是否会明显影响产物。