甲基化检测方法(亚硫酸氢盐修饰后测序法)
- 格式:doc
- 大小:71.00 KB
- 文档页数:7

DNA亚硫酸氢盐修饰和纯化操作步骤修饰设计:使用CpGenome TM kit使胞嘧啶转化为尿嘧啶的步骤如下。
中等温度碱性pH下使DNA变性成为单链形式暴露出碱基。
试剂一,一种包含亚硫酸氢根的钠盐,可使未甲基化的胞嘧啶磺化和水解脱氨,产生一种尿嘧啶磺酸盐中间产物。
然后DNA在另一种盐﹙试剂二﹚存在的条件下与一种微粒载体﹙试剂三﹚结合,并通过重复离心和在70%的乙醇中重悬浮脱盐。
向尿嘧啶的转化是通过在90%的乙醇中反复碱性脱磺酸基作用和脱盐完成的。
DNA最终在TE缓冲液中通过加热从载体上洗脱下来。
第一步:试剂准备(1)3 M NaOH原料(用前现配)把1g干NaOH片剂溶解在8.3mL水中。
使用此类腐蚀性碱,注意小心谨慎和实验操作。
(2)20 mM NaOH/90% EtOH(用前现配)配制1mL该溶液需:900μl 100%的乙醇,93.4μl水,6.6μl 3M的氢氧化钠。
(3)溶解试剂Ⅰ(用前现配)打开前将试剂瓶加温至室温。
对每份待修饰的样本,称取0.227g DNA修饰试剂Ⅰ加入0.571mL水中。
充分涡旋振荡混合。
使用该试剂时要小心谨慎,因为它对呼吸系统和皮肤有刺激性。
用大约20μl 3M NaOH调整pH至5.0,用pH试纸检测pH值。
试剂Ⅰ避光保存以免分解。
为了最佳效果,试剂应在配置后立即使用。
(4)溶解试剂Ⅱ打开前将试剂瓶加温至室温。
将1μl β-巯基乙醇加入20mL去离子水中。
每份待修饰的DNA样本需将750μl该溶液加入到1.35g DNA修饰Ⅱ。
充分混合确保完全溶解。
过量的试剂可用箔纸包裹的容器、2℃-8℃、避光保存长达6周。
第二步:DNA修饰程序1、在带有螺旋形瓶盖的1.5-2.0mL的微量离心管中:将7.0μl 3M NaOH加入到含有1.0 μg DNA的100μl水中(10ng/μl),混匀。
注意:如果样本含有的DNA量不到1.0μg,就向样本DNA中加入2 μl DNA修饰试剂Ⅳ并加水至总体积100μl。

DNA甲基化检测实验一、重亚硫酸盐的测序法实验流程(BSP)(Bisulfite Genomic Sequence)原理:结合重亚硫酸盐的测序法是一种灵敏的能直接检测分析基因组DNA甲基化模式的方法。
重亚硫酸盐处理后,用针对改变后的DNA序列设计特异性引物并进行聚合酶链式反应(PCR)。
PCR产物中原先非甲基化的胞嘧啶位点被胸腺嘧啶所替代,而甲基化的胞嘧啶位点保持不变。
PCR产物克隆后进行测序。
通过这个方法能得到特定位点在各个基因组DNA 分子中的甲基化状态。
该方法特点是:•特异性高,它能够提供特异性很高的分析结果,这是所有其他研究甲基化的分析方法所不能比拟的;•灵敏度高,可以用于分析少于100个细胞的检测样品。
用微量的基因组DNA进行分析就能得到各个DNA分子精确的甲基化位点分布图。
重亚硫酸盐测序法技术实验流程A.DNA制备用DNA抽提试剂盒(Promega, cat. no. A1125)抽提组织,细胞培养物,石蜡包埋组织切片样品中的基因组DNA。
B.重亚硫酸盐处理C.DNA纯化用Wizard DNA clean-up kit (Promega, cat. no. A7280)纯化重亚硫酸盐处理后的DNA样品。
D.PCR扩增E.PCR产物琼脂糖电泳后回收纯化F.PCR产物连接到pMD19-T (Takara) 载体中克隆及测序。
G.用分析软件对各样本测序结果进行甲基化程度分析二、甲基化特异性的PCR实验流程(methylation-specific PCR, MSP)原理:甲基化特异性的PCR是一种灵敏度高且操作相对简单的甲基化研究方法。
重亚硫酸盐处理DNA后,基因组DNA发生的由甲基化状态决定的序列改变。
随后进行引物特异性的PCR。
该方法引物设计是关键。
MSP中设计两对引物,即一对结合处理后的甲基化DNA链(引物对M),另一对结合处理后的非甲基化DNA链(引物对U)。
检测MSP扩增产物,如果用引物M能扩增出片段,则说明检测位点存在甲基化;若用引物U扩增出片段,则说明被检测的位点不存在甲基化(图3)。

甲基化检测方法(亚硫酸氢盐修饰后测序法)第一部分基因组DNA的提取。
这一步没有悬念,完全可以购买供细胞或组织使用的DNA提取试剂盒,如果实验室条件成熟,自己配试剂提取完全可以。
DNA比较稳定,只要在操作中不要使用暴力,提出的基因组DNA应该是完整的。
此步重点在于DNA的纯度,即减少或避免RNA、蛋白的污染很重要。
因此在提取过程中需使用蛋白酶K及RNA酶以去除两者。
使用两者的细节:1:蛋白酶K可以使用灭菌双蒸水配制成20mg/ml;2:RNA酶必须要配制成不含DNA酶的RNA酶,即在购买市售RNA酶后进行再处理,配制成10mg/ml。
否则可能的后果是不仅没有RNA,连DNA也被消化了。
两者均于-20度保存。
验证提取DNA的纯度的方法有二:1:紫外分光光度计计算OD比值;2:1%-1.5%的琼脂糖凝胶电泳。
我倾向于第二种方法,这种方法完全可以明确所提基因组DNA的纯度,并根据Marker的上样量估计其浓度,以用于下一步的修饰。
第二部分亚硫酸氢钠修饰基因组DNA如不特别指出,所用双蒸水(DDW)均经高压蒸汽灭菌。
1:将约2ugDNA于1.5mlEP管中使用DDW稀释至50ul;2:加5.5ul新鲜配制的3M NaOH;3:42℃水浴30min;水浴期间配制:4:10mM对苯二酚(氢醌),加30ul至上述水浴后混合液中;(溶液变成淡黄色)5:3.6M亚硫酸氢钠(Sigma,S9000),配制方法:1.88g亚硫酸氢钠使用DDW稀释,并以3M NaOH滴定溶液至PH 5.0,最终体积为5ml。
这么大浓度的亚硫酸氢钠很难溶,但加入NaOH后会慢慢溶解,需要有耐心。
PH一定要准确为5.0。
加520ul至上述水浴后溶液中。
6:EP管外裹以铝箔纸,避光,轻柔颠倒混匀溶液。
7:加200 ul 石蜡油,防止水分蒸发,限制氧化。
8:50℃避光水浴16h。
一般此步在4pm开始做,熟练的话不到5pm即可完成,水浴16h正好至次日8am 以后收,时间上很合适。

甲基化检测方法(亚硫酸氢盐修饰测序法)基本原理在于:样本经亚硫氢酸盐处理后,甲基化的胞嘧啶(C)保持不变,但非甲基化的胞嘧啶被转化成脲嘧啶,因此在利用该处理产物作为模板的PCR产物中,甲基化的胞嘧啶还是胞嘧啶,但非甲基化胞嘧啶变成了脲嘧啶(胸腺嘧啶),此时检测到的胞嘧啶(C)即是样品中本身的甲基化位点.第一部分基因组DNA的提取。
DNA提取试剂盒,如果实验室条件成熟,自己配试剂提取完全可以。
DNA比较稳定,只要在操作中不要使用暴力,提出的基因组DNA应该是完整的。
此步重点在于DNA的纯度,即减少或避免RNA、蛋白的污染很重要。
因此在提取过程中需使用蛋白酶K及RNA酶以去除两者。
使用两者的细节:1:蛋白酶K可以使用灭菌双蒸水配制成20mg/ml;2:RNA酶必须要配制成不含DNA酶的RNA酶,即在购买市售RNA酶后进行再处理,配制成10mg/ml。
否则可能的后果是不仅没有RNA,连DNA也被消化了。
两者均于-20度保存。
验证提取DNA的纯度的方法有二:1:紫外分光光度计计算OD比值;2:1%-1.5%的琼脂糖凝胶电泳。
我倾向于第二种方法,这种方法完全可以明确所提基因组DNA的纯度,并根据Marker的上样量估计其浓度,以用于下一步的修饰。
第二部分亚硫酸氢钠修饰基因组DNA如不特别指出,所用双蒸水(DDW)均经高压蒸汽灭菌。
1:将约2ugDNA于1.5mlEP管中使用DDW稀释至50ul;2:加5.5ul新鲜配制的3M NaOH;3:42℃水浴30min;水浴期间配制:4:10mM对苯二酚(氢醌),加30ul至上述水浴后混合液中;(溶液变成淡黄色)5:3.6M亚硫酸氢钠(Sigma,S9000),配制方法:1.88g亚硫酸氢钠使用DDW稀释,并以3M NaOH滴定溶液至PH 5.0,最终体积为5ml。
这么大浓度的亚硫酸氢钠很难溶,但加入NaOH后会慢慢溶解,需要有耐心。
PH一定要准确为5.0。
加520ul至上述水浴后溶液中。

甲基化检测方法(亚硫酸氢盐修饰后测序法)第一部分基因组DNA的提取。
这一步没有悬念,完全可以购买供细胞或组织使用的DNA提取试剂盒,如果实验室条件成熟,自己配试剂提取完全可以。
DNA比较稳定,只要在操作中不要使用暴力,提出的基因组DNA应该是完整的。
此步重点在于DNA的纯度,即减少或避免RNA、蛋白的污染很重要。
因此在提取过程中需使用蛋白酶K及RNA酶以去除两者。
使用两者的细节:1:蛋白酶K可以使用灭菌双蒸水配制成20mg/ml;2:RNA酶必须要配制成不含DNA酶的RNA酶,即在购买市售RNA酶后进行再处理,配制成10mg/ml。
否则可能的后果是不仅没有RNA,连DNA也被消化了。
两者均于-20度保存。
验证提取DNA的纯度的方法有二:1:紫外分光光度计计算OD比值;2:1%-1.5%的琼脂糖凝胶电泳。
我倾向于第二种方法,这种方法完全可以明确所提基因组DNA的纯度,并根据Marker的上样量估计其浓度,以用于下一步的修饰。
第二部分亚硫酸氢钠修饰基因组DNA如不特别指出,所用双蒸水(DDW)均经高压蒸汽灭菌。
1:将约2ugDNA于1.5mlEP管中使用DDW稀释至50ul;2:加5.5ul新鲜配制的3M NaOH;3:42℃水浴30min;水浴期间配制:4:10mM对苯二酚(氢醌),加30ul至上述水浴后混合液中;(溶液变成淡黄色)5:3.6M亚硫酸氢钠(Sigma,S9000),配制方法:1.88g亚硫酸氢钠使用DDW稀释,并以3M NaOH滴定溶液至PH 5.0,最终体积为5ml。
这么大浓度的亚硫酸氢钠很难溶,但加入NaOH后会慢慢溶解,需要有耐心。
PH一定要准确为5.0。
加520ul至上述水浴后溶液中。
6:EP管外裹以铝箔纸,避光,轻柔颠倒混匀溶液。
7:加200 ul 石蜡油,防止水分蒸发,限制氧化。
8:50℃避光水浴16h。
一般此步在4pm开始做,熟练的话不到5pm即可完成,水浴16h正好至次日8am 以后收,时间上很合适。

全基因组甲基化研究技术介绍
1.测序方法:
(1) 甲基化敏感的限制性内切酶测序(MSRE-Seq):利用甲基化敏感的限制性内切酶切割DNA,然后通过高通量测序对剪切后的DNA片段进行测序分析,确定DNA甲基化位点。
(2) 亲和捕获甲基化DNA测序(MethylC-Seq):通过亲和捕获和富集甲基化的DNA片段,然后进行测序分析,确定DNA甲基化位点。
(3) 甲基化依赖的化学修饰酶测序(MBD-Seq):利用甲基化依赖的化学修饰酶(如MBD2)来富集甲基化的DNA片段,然后进行测序分析,确定DNA甲基化位点。
(4) 甲基化的二代测序(BS-Seq):将DNA进行亚硫酸盐处理,将非甲基化的胞嘧啶转化为尿嘧啶,而甲基化的胞嘧啶不受影响,然后进行高通量测序分析,确定DNA甲基化位点。
2.芯片方法:
(1)MeDIP芯片:利用MeDIP(甲基化的DNA免疫沉淀)技术将甲基化的DNA片段与抗体结合,然后将DNA片段通过芯片进行检测和分析。
(2)MBD芯片:利用MBD(甲基化结合域)结合甲基化的DNA片段,然后通过芯片进行检测和分析。
(3)hMeDIP芯片:利用hMeDIP(5-羟甲基胞嘧啶的甲基化免疫沉淀)技术将5-羟甲基胞嘧啶甲基化的DNA片段与抗体结合,然后通过芯片进行检测和分析。
该方法可以区分DNA甲基化和5-羟甲基胞嘧啶甲基化。
总的来说,全基因组甲基化研究技术通过测序或芯片的方法分析DNA 甲基化的模式和作用机制,帮助我们深入了解基因组的表观遗传学调控,为研究生物发育、疾病发生等提供重要的信息。
这些技术的发展将进一步推动基因组学、表观基因组学和遗传学研究的进展。

甲基化验证方法
嘿,朋友们!今天咱来聊聊甲基化验证方法。
你说这甲基化啊,就像是基因上的小装饰,可别小瞧了它,它能对咱们的身体产生大影响呢!
那怎么来验证这个小装饰有没有安好呢?首先咱得知道有几种常见的办法。
比如说亚硫酸氢盐测序法,这就好比是基因世界里的侦探,能精准地找出甲基化的位置。
它就像是一个细心的侦探,一点点地排查线索,不放过任何一个甲基化的蛛丝马迹。
还有甲基化特异性 PCR 呀,这就像是一个有特异功能的小助手,专门识别甲基化的区域,一找一个准。
你想想,它能快速地分辨出哪些是甲基化了的,哪些还没有,是不是很厉害?
另外,还有一些其他的方法呢,比如芯片法。
这就好像是一张大地图,能一下子把基因上的甲基化情况都给呈现出来,让我们一目了然。
咱说这些方法,各有各的优点和适用场景。
就像咱生活中不同的工具,有的适合修修补补,有的适合大工程。
你要是想知道某个特定基因的甲基化情况,那可能亚硫酸氢盐测序法就比较合适;要是想大规模地筛查,那芯片法可能就更能派上用场啦。
你说这甲基化验证方法是不是很神奇?就像给基因穿上了不同的衣服,展现出不同的样子。
咱得好好研究研究,才能更好地了解咱们身体里的这些小秘密呀。
那我们在选择方法的时候可得仔细了,要根据自己的需求和条件来。
不然选错了方法,那不就像拿着锤子去拧螺丝,白费力气嘛!而且在操作过程中也要小心谨慎,就像对待珍贵的宝贝一样,可不能马虎大意。
总之,甲基化验证方法是我们探索基因世界的重要工具,我们得好好利用它们,解开基因的神秘面纱。
让我们一起加油,去发现更多关于基因的奇妙之处吧!这就是我对甲基化验证方法的一些看法,你们觉得呢?。

MGMT甲基化检测实验步骤一.亚硫酸氢钠处理DNA(将未甲基化的胞嘧啶脱氨基变为尿嘧啶)使用EZ DNA(北京中西远大科技有限公司)甲基化试剂盒处理样本。
二.DNA样本的处理1.取130微升的CT Conversion Reagent和20微升的DNA样本,加入1.5毫升的离心管中。
混合。
2.准备好水浴锅,将离心管放入98℃水浴锅中10分钟,64℃水浴锅中2.5个小时。
(不能立即开始实验的,将样本放入4℃冰箱中,保存。
)3.将离心管中的溶液吸出,加入到Zymo-Spin TM的萃取柱中。
4.取600微升的M-Binding Buffer加入到萃取柱中,摇晃混匀。
5.转速>10000转/分钟的离心机中,离心30秒。
倒掉液体。
6.取100微升的M-Wash Buffer加入萃取柱,离心30秒,倒掉液体。
7.取200微升的M-Desulphonation Buffer加入萃取柱,等待15~20分钟,使其充分浸润。
然后离心30秒。
8.取200微升的M--Wash Buffer加入离心管中,离心30秒。
重复此步骤一次,随后将收集柱丢弃,萃取柱放入1.5毫升离心管中。
9.取10微升的M-Elution Bufer加入到萃取柱中,离心30秒,萃取柱丢弃,离心管中得到处理好10微升DNA样本。
三.巢式PCR(体系,程序)第一轮PCR:模板(处理后的DNA),引物:MGMT--JF/R ,dd水,PCR MIX Golden Star。
体系为:20微升=模板DNA 4微升+引物2微升+dd水4微升+Golden Star 10微升(一管)反应条件:95℃ 10min95℃ 30s →52℃ 30s → 40个循环72℃ 30s →72℃ 10min4℃ forever第二轮PCR:模板(第一轮PCR的产物),引物:m-MGMT-F/R及u-MGMT-F/R,dd 水,PCR MIX Golden Star。
体系为:20微升=模板DNA 1微升+引物(m-MGMT-F/R)2微升+dd水7微升+Golden Star 10微升(一管)20微升=模板DNA 1微升+引物(u-MGMT-F/R) 2微升+dd水7微升+Golden Star 10微升(一管)反应条件:95℃ 10min95℃ 30s →64℃ 30s → 40个循环72℃ 30s →72℃ 10min4℃ forever四.PAGE电泳检测配置PAGE胶:12%的胶: 100ml 12ml30%丙烯酰胺: 40ml 4.8ml 10xTBE 10ml 1.2ml 10%过硫酸铵 0.7ml 84μl TEMED 35μl 4.2μl H2O 49.3ml 5.9ml。

甲基化检测方法(亚硫酸氢盐修饰后测序法)-2此步细节:1:在使用注射器时,一定要用力均匀且轻,如使用暴力,会将小柱内的薄膜挤破,失去作用。
2:乙酸铵、糖原不需新鲜配制,糖原配好后放在-20度保存,乙酸铵室温即可,因为这样浓度的乙酸铵非常难溶,一旦放在4度,取出用时也会有很多溶质析出。
3:异丙醇、70%乙醇都不需要新鲜配制,但如果用量大,现场配也很方便。
此步关键是在树脂与DNA的结合上,这就再次强调第二部分调亚硫酸钠PH值得重要性。
因为树脂与DNA结合需要有一个适当的PH,如前一步没做好,此步树脂不能与DNA很好结合,将会带来灾难性后果,即DNA随着液体被挤出了,洗脱时实际已没有任何DNA了。
第四部分修饰后DNA用于PCR这一步也没有悬念。
我主要谈一下这里面的几个比较棘手的问题:1:引物问题:我感觉自己设计引物有相当的难度,我曾设计过几对引物,并且试验了一下,但以失败告终。
如果时间充裕、作的又是比较新的基因文献不多,自己设计引物没有问题。
如果不是这样,还是参考文献更好些。
首先查阅SCI分值高的文献,然后是著名实验室的文献,如果国内有做的,更好了,可以直接联系咨询。
查到序列后,一定要和Genbank中的序列进行比对,防止有印刷错误造成的个别碱基的差别。
然后再到google上搜一下,看用的人多否,体系条件是否一样。
用的人多、体系条件一样,表明可重复性比较强。
我也是按此行事,算比较顺利。
2:Taq酶问题:有文献用高保真的金牌 Taq(Platinum),但我感觉只要体系正确、变性退火等条件合适,一般的热启动酶是可以的。
我开始使用的是Takara的LA Taq,很好用,配有10x含mg++的LA缓冲液。
有时候用没了,暂时以Takara 的普通Taq酶也可以。
如何选择,可以根据自己的情况。
初作者还是用好一点的酶。
3:PCR的条件:变性一般都选择95度,3min。
其余我感觉还是根据文献,退火可以根据你的引物的退火温度在小范围内尝试。

单细胞DNA甲基化测序数据处理流程与分析方法1. 内容简述单细胞DNA甲基化测序是一种高分辨率的基因表达和表观遗传学研究方法,它允许研究者检测单个细胞的DNA甲基化状态。
这种技术为理解细胞异质性、基因调控机制以及疾病发展中的表观遗传变化提供了有力工具。
样本制备:首先,从生物体中提取单细胞,然后利用亚硫酸盐转化技术将DNA中的甲基化修饰转换为羟基化修饰,以供后续测序。
文库构建:转化后的DNA被随机打断成小片段,并加上特定的接头序列,以便进行PCR扩增和测序。
测序:构建好的文库被加载到测序芯片上,通过高通量测序技术进行测序。
数据分析:获得的原始数据需要经过一系列清洗、比对、标准化等处理步骤,以获得高质量的甲基化数据集。
甲基化状态分析:识别每个细胞中的甲基化位点,并比较不同细胞之间的甲基化差异。
差异甲基化分析:识别在不同实验条件下(如疾病状态、环境压力等)甲基化模式的差异。
生物信息学分析:使用统计软件和算法对数据进行深度挖掘,发现与特定生物学过程或疾病相关的甲基化模式。
通过对这些数据的综合分析,研究者可以揭示细胞功能的动态变化、基因表达的调控机制以及表观遗传学在疾病发生中的作用。
1.1 单细胞DNA甲基化测序技术简介简称SCDBS)是一种高通量、高分辨率的分析方法,用于研究单个细胞中基因组水平的DNA甲基化状态。
该技术通过测序和分析单细胞中的甲基化位点序列,揭示了基因表达差异、发育过程、疾病发生机制等方面的信息。
随着高通量测序技术的快速发展,SCDBS已经成为生物学研究的重要工具之一。
SCDBS的主要流程包括:样品准备、文库构建、测序、数据处理和分析等步骤。
需要将单细胞样本进行处理,如去除血浆等杂质,保证测序结果的准确性。
通过构建文库来存储待测的DNA片段,通常采用Illumina测序平台进行高通量测序。
对测序数据进行质量控制和过滤,以去除低质量序列和伪迹。
利用生物信息学工具对数据进行处理和分析,包括聚类分析、差异基因表达分析、甲基化模式比较等。
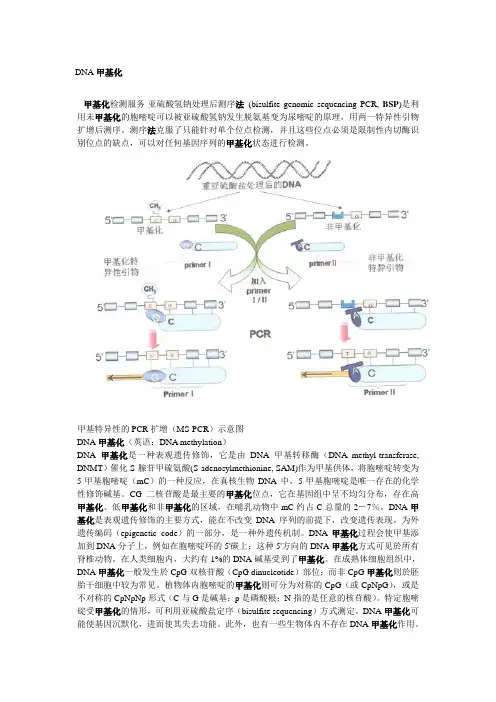
DNA甲基化甲基化检测服务-亚硫酸氢钠处理后测序法(bisulfite genomic sequencing PCR, BSP)是利用未甲基化的胞嘧啶可以被亚硫酸氢钠发生脱氨基变为尿嘧啶的原理,用两一特异性引物扩增后测序。
测序法克服了只能针对单个位点检测,并且这些位点必须是限制性内切酶识别位点的缺点,可以对任何基因序列的甲基化状态进行检测。
甲基特异性的PCR扩增(MS-PCR)示意图DNA甲基化(英语:DNA methylation)DNA甲基化是一种表观遗传修饰,它是由DNA甲基转移酶(DNA methyl-transferase, DNMT)催化S-腺苷甲硫氨酸(S-adenosylmethionine, SAM)作为甲基供体,将胞嘧啶转变为5-甲基胞嘧啶(mC)的一种反应,在真核生物DNA中,5-甲基胞嘧啶是唯一存在的化学性修饰碱基。
CG二核苷酸是最主要的甲基化位点,它在基因组中呈不均匀分布,存在高甲基化、低甲基化和非甲基化的区域,在哺乳动物中mC约占C总量的2-7%。
DNA甲基化是表观遗传修饰的主要方式,能在不改变DNA序列的前提下,改变遗传表现。
为外遗传编码(epigenetic code)的一部分,是一种外遗传机制。
DNA甲基化过程会使甲基添加到DNA分子上,例如在胞嘧啶环的5'碳上:这种5'方向的DNA甲基化方式可见於所有脊椎动物。
在人类细胞内,大约有1%的DNA碱基受到了甲基化。
在成熟体细胞组织中,DNA甲基化一般发生於CpG双核苷酸(CpG dinucleotide)部位;而非CpG甲基化则於胚胎干细胞中较为常见。
植物体内胞嘧啶的甲基化则可分为对称的CpG(或CpNpG),或是不对称的CpNpNp形式(C与G是碱基;p是磷酸根;N指的是任意的核苷酸)。
特定胞嘧碇受甲基化的情形,可利用亚硫酸盐定序(bisulfite sequencing)方式测定。
DNA甲基化可能使基因沉默化,进而使其失去功能。
DNA甲基化检测实验一、重亚硫酸盐的测序法实验流程(BSP)(Bisulfite Genomic Sequence)原理:结合重亚硫酸盐的测序法是一种灵敏的能直接检测分析基因组DNA甲基化模式的方法。
重亚硫酸盐处理后,用针对改变后的DNA序列设计特异性引物并进行聚合酶链式反应(PCR)。
PCR产物中原先非甲基化的胞嘧啶位点被胸腺嘧啶所替代,而甲基化的胞嘧啶位点保持不变。
PCR产物克隆后进行测序。
通过这个方法能得到特定位点在各个基因组DNA 分子中的甲基化状态。
该方法特点是:•特异性高,它能够提供特异性很高的分析结果,这是所有其他研究甲基化的分析方法所不能比拟的;•灵敏度高,可以用于分析少于100个细胞的检测样品。
用微量的基因组DNA进行分析就能得到各个DNA分子精确的甲基化位点分布图。
重亚硫酸盐测序法技术实验流程A.DNA制备用DNA抽提试剂盒(Promega, cat. no. A1125)抽提组织,细胞培养物,石蜡包埋组织切片样品中的基因组DNA。
B.重亚硫酸盐处理C.DNA纯化用Wizard DNA clean-up kit (Promega, cat. no. A7280)纯化重亚硫酸盐处理后的DNA样品。
D.PCR扩增E.PCR产物琼脂糖电泳后回收纯化F.PCR产物连接到pMD19-T (Takara) 载体中克隆及测序。
G.用分析软件对各样本测序结果进行甲基化程度分析二、甲基化特异性的PCR实验流程(methylation-specific PCR, MSP)原理:甲基化特异性的PCR是一种灵敏度高且操作相对简单的甲基化研究方法。
重亚硫酸盐处理DNA后,基因组DNA发生的由甲基化状态决定的序列改变。
随后进行引物特异性的PCR。
该方法引物设计是关键。
MSP中设计两对引物,即一对结合处理后的甲基化DNA链(引物对M),另一对结合处理后的非甲基化DNA链(引物对U)。
检测MSP扩增产物,如果用引物M能扩增出片段,则说明检测位点存在甲基化;若用引物U扩增出片段,则说明被检测的位点不存在甲基化(图3)。
BSP检测技术亚硫酸氢盐处理后测序(bisulfite sequencing PCR,BSP)法是检测基因甲基化的经典方法,其原理为:用亚硫酸氢钠修饰处理基因组DNA,所有未发生甲基化的胞嘧啶(C)被转化为尿嘧啶(U)而甲基化的胞嘧啶则不变。
基因组DNA经亚硫酸盐处理后,设计BSP引物扩增目的片段,此时尿嘧啶(U)全部转化为胸腺嘧啶(T),最后对PCR产物进行测序就可以判断CpG位点是否发生甲基化。
BSP实验流程:引物设计及注意事项:引物设计软件:Methyl Primer Express v1.0引物设计注意事项:用于BSP实验的DNA是经过亚硫酸盐处理,将未甲基化的C碱基转变为U碱基,而甲基化的C碱基不变,从而将甲基化的差异转变成碱基上的差异,所以在后续进行PCR扩增引物设计的时候需要注意:1、引物设计不能含有CpG位点,以避免造成发生甲基化和未发生甲基化DNA的差异。
2 、引物扩增的片段能包含尽量多的CpG位点,BSP扩增片段长度300-500bp,包含的CpG位点越多则引物的打分越高。
BSP服务完成时提供:Bisulfite处理的基因组DNA;实验报告(包括BSP产物的直接测序报告)。
结果展示:BSP测序图BSP点状图空点表示为发生甲基化的CpG位点,实点表示发生甲基化的CpG位点BSP柱状图案例分析:利用二代测序和BSP(bisulfite sequencing PCR)研究MLH1 启动子区域甲基化状况在肿瘤内部的异质性同一肿瘤中可能包含不同体细胞变异的细胞,过去对肿瘤的这种异质性研究主要集中在遗传学领域,作者则将目光锁定在表观遗传学方面。
研究人员先利用二代测序手段,对来自33个样本(包括子宫内膜癌,相应的外周血以及正常的子宫内膜组织)的1000个分子的MLH启动子区域的甲基化谱(spectrum of MLH1 promoter methylation)进行研究,后续再利用BSP手段进行验证,发现了不同的子宫内膜癌细胞MLH1启动子区域出人意料的甲基化异质性形式,这种高分辨率的研究手段能够研究肿瘤甲基化的克隆性(clonality of methylation)从而对肿瘤发生过程中对启动子区域异常甲基化的状况进行研究。
基因甲基化检测⽅法有哪些探究了宫颈细胞变化与基因甲基化的关系过后,我们要做的就是如何检测出基因甲基化的存在现象。
随着科学技术的发展,检验技术也在部断提升。
⼤致可以分为两类:特异位点的甲基化检测和全基因组的甲基化分析,后者也称为甲基化图谱分析(methylation profiling)。
下⾯敬善基因来为⼤家介绍⼀些常⽤的⽅法。
甲基化特异性PCR(MS-PCR)这种⽅法经济实⽤,⽆需特殊仪器,因此是⽬前应⽤最为⼴泛的⽅法。
在亚硫酸氢盐处理后,即可开展MS-PCR。
在传统的MSP⽅法中,通常设计两对引物,⼀对MSP引物扩增经亚硫酸氢盐处理后的DNA模板,⽽另⼀对扩增未甲基化⽚段。
若第⼀对引物能扩增出⽚段,则说明该检测位点存在甲基化,若第⼆对引物能扩增出⽚段,则说明该检测位点不存在甲基化。
这种⽅法灵敏度⾼,可⽤于⽯蜡包埋样本,且不受内切酶的限制。
不过也存在⼀定的缺陷,你要预先知道待测⽚段的DNA序列,并设计出好的引物,这⾄关重要。
另外,若存在亚硫酸氢盐处理不完全的情况,那可能导致假阳性。
亚硫酸氢盐处理+测序这种⽅法⼀度被认为是DNA甲基化分析的⾦标准。
它的过程如下:经过亚硫酸氢盐处理后,⽤PCR扩增⽬的⽚段,并对PCR产物进⾏测序,将序列与未经处理的序列进⾏⽐较,判断CpG位点是否发⽣甲基化。
这种⽅法可靠,且精确度⾼,能明确⽬的⽚段中每⼀个CpG位点的甲基化状态,但需要⼤量的克隆测序,过程较为繁琐、昂贵。
联合亚硫酸氢钠的限制性内切酶分析法(COBRA)DNA样本经亚硫酸氢盐处理后,利⽤PCR扩增。
扩增产物纯化后⽤限制性内切酶(BstUI)消化。
若其识别序列中的C发⽣完全甲基化(5mCG5mCG),则PCR扩增后保留为CGCG,BstU I能够识别并进⾏切割;若待测序列中,C未发⽣甲基化,则PCR后转变为TGTG,BstUI识别位点丢失,不能进⾏切割。
这样酶切产物再经电泳分离、探针杂交、扫描定量后即可得出原样本中甲基化的⽐例。
甲基化检测方法甲基化是一种通过在DNA分子中结合甲基基团来添加化学修饰的过程。
这种化学修饰可以影响基因表达模式,从而对细胞功能和发育过程产生深远影响。
因此,甲基化检测方法在研究生物学、遗传学以及疾病发生机制等方面具有重要意义。
亚硫酸氢盐修饰后测序法是一种被广泛应用于甲基化检测的方法之一,本文将对其原理、步骤和应用进行详细介绍。
亚硫酸氢盐修饰后测序法的原理基于DNA中的甲基化位点与亚硫酸氢盐发生化学反应的特性。
亚硫酸氢盐可以与未甲基化的胞嘧啶碱基形成二硫键结合,而与甲基化的胞嘧啶碱基则不能发生此反应。
利用这个原理,可以将未甲基化的胞嘧啶通过亚硫酸氢盐修饰转化为胞嘧啶-4-亚硫酸酯(CMPS),而甲基化的胞嘧啶则保持不变。
然后,通过测序技术对DNA进行测序,可以得知胞嘧啶碱基是否甲基化。
该方法的具体步骤如下:1.DNA提取:从待测样品中提取DNA,可以使用常规的DNA提取方法来纯化DNA。
2.亚硫酸氢盐修饰:将DNA与亚硫酸氢盐(如亚硫酸氢钠)反应,使未甲基化的胞嘧啶碱基发生亚硫酸化修饰。
通过调整反应条件,可以使甲基化的胞嘧啶碱基保持原样。
3.水解:在亚硫酸氢盐修饰后,将DNA进行水解反应,以将DNA降解成较小的核苷酸片段。
4. 测序:将水解后的DNA片段进行测序,可以使用传统的Sanger测序技术或者高通量测序技术(如二代测序)来完成。
通过测序结果的比对分析,可以确定甲基化位点。
总结起来,亚硫酸氢盐修饰后测序法是一种常用的甲基化检测方法。
通过将DNA中的未甲基化胞嘧啶进行化学修饰,然后通过测序技术分析甲基化位点,可以揭示基因组中的甲基化特征以及其在生物学和疾病中的作用。
在未来,随着测序技术的不断发展和改进,亚硫酸氢盐修饰后测序法还将为我们提供更多深入的甲基化信息。
甲基化测量方法
基甲基化检测主要几种:
甲基化特bai异性PCR(Methylation-specific PCRMSP)
用亚硫酸氢盐处理基组DNA所未发甲基化胞嘧啶转化尿嘧啶甲基化胞嘧啶变;随设计针甲基化非甲基化序列引物进行PCR通电泳检测MSP扩增产物用针处理甲基化DNA链引物能扩增片段则说明该位点存甲基化;反说明检测位点存甲基化
2、亚硫酸氢盐测序(Bisulfite sequencing PCRBSP)
用亚硫酸氢盐处理基组DNA则未发甲基化胞嘧啶转化尿嘧啶甲基化胞嘧啶变随设计BSP引物进行PCR扩增程尿嘧啶全部转化胸腺嘧啶PCR产物进行测序判断CpG位点否发甲基化称BSP-直接测序PCR产物克隆至载体进行测序提高测序功率种称BSP-克隆测序
3、甲基化敏扩增态性
(Methylation-Sensitive Amplified Polymorphism,MSAP)
MSAP利用DNA甲基化敏两种同裂酶Hpa IIMsp I基组DNA进行酶切连接由于两种酶能够识别相同限制性酶切位点即CCGG位点DNA序列CCGG位点现同程度甲基化状态别两种酶识别产物式体现
4、焦磷酸测序(Pyrosequencing)
通准确定量单连续CpG 位点甲基化频率焦磷酸测序能检测并定量甲基化水平细微改变序列延伸程根据CT掺入量定量确定单位点C-T比例同位点甲基化变异能准确检测由于焦磷酸测序提供真实序列数据甲基化状态序列形式呈现。
DNA 甲基化重亚硫酸氢盐修饰法(DNA METHYLATION BISULFITE MODIFICATION)实验操作原理及方法一、实验目的:通过本实验,可以检测特定DNA序列的甲基化状态。
二、实验原理:DNA 甲基化是指由S-腺苷甲硫氨酸(SAM)提供甲基基团,在DNA 甲基转移酶(DNA methyltransferases,DNMTs)的作用下,将CpG 二核苷酸的胞嘧啶(C)甲基化为5-甲基化胞嘧啶(5-m C)的一种化学反应。
DNA 甲基化是调节基因转录表达的一种重要的表观遗传的修饰方式。
DNA 甲基化主要在转录水平抑制基因的表达。
DNA 甲基化引起基因转录抑制的机制可能主要有以下3 种:(1)DNA甲基化直接干扰特异性转录因子与各基因启动子中识别位置的结合。
(2)序列特异性的甲基化DNA 结合蛋白与启动子区甲基化CpG 岛结合,募集一些蛋白,形成转录抑制复合物,阻止转录因子与启动子区靶序列的结合,从而影响基因的转录。
(3)DNA 甲基化通过改变染色质结构,抑制基因表达。
重亚硫酸氢盐修饰法检测DNA甲基化的基本原理是基于DNA变性后用重亚硫酸氢盐处理,可将未甲基化胞嘧啶修饰成尿嘧啶。
此反应的步骤是:1、在C-6位点磺化胞嘧啶残基;2、在C-4处水解去氨基来产生尿嘧啶磺酸盐;3、在碱性条件下去硫酸化。
在这个过程中,5-甲基胞嘧啶由于甲基化基团干扰了重亚硫酸氢盐进入到C-6位点而保持着未反应的状态。
在重亚硫酸氢盐处理后,使用针对每个修饰后DNA链的引物进行PCR反应。
在这个PCR产物中,每5-甲基胞嘧啶显示为胞嘧啶,而由未甲基化胞嘧啶转变成的尿嘧啶则在扩增过程中被胸腺嘧啶所取代。
BSP(bisulfate sequencing PCR) :重亚硫酸盐使DNA中未发生甲基化的胞嘧啶脱氨基转变成尿嘧啶,而甲基化的胞嘧啶保持不变,进行PCR扩增。
最后,对PCR产物进行测序,并且与未经处理的序列比较,判断是否CpG位点发生甲基化。
甲基化检测方法(亚硫酸氢盐修饰后测序法)第一部分基因组DNA的提取。
这一步没有悬念,完全可以购买供细胞或组织使用的DNA提取试剂盒,如果实验室条件成熟,自己配试剂提取完全可以。
DNA比较稳定,只要在操作中不要使用暴力,提出的基因组DNA应该是完整的。
此步重点在于DNA的纯度,即减少或避免RNA、蛋白的污染很重要。
因此在提取过程中需使用蛋白酶K及RNA酶以去除两者。
使用两者的细节:1:蛋白酶K可以使用灭菌双蒸水配制成20mg/ml;2:RNA酶必须要配制成不含DNA酶的RNA酶,即在购买市售RNA酶后进行再处理,配制成10mg/ml。
否则可能的后果是不仅没有RNA,连DNA也被消化了。
两者均于-20度保存。
验证提取DNA的纯度的方法有二:1:紫外分光光度计计算OD比值;2:1%-1.5%的琼脂糖凝胶电泳。
我倾向于第二种方法,这种方法完全可以明确所提基因组DNA的纯度,并根据Marker的上样量估计其浓度,以用于下一步的修饰。
第二部分亚硫酸氢钠修饰基因组DNA如不特别指出,所用双蒸水(DDW)均经高压蒸汽灭菌。
1:将约2ugDNA于1.5mlEP管中使用DDW稀释至50ul;2:加5.5ul新鲜配制的3M NaOH;3:42℃水浴30min;水浴期间配制:4:10mM对苯二酚(氢醌),加30ul至上述水浴后混合液中;(溶液变成淡黄色)5:3.6M亚硫酸氢钠(Sigma,S9000),配制方法:1.88g亚硫酸氢钠使用DDW稀释,并以3M NaOH滴定溶液至PH 5.0,最终体积为5ml。
这么大浓度的亚硫酸氢钠很难溶,但加入NaOH后会慢慢溶解,需要有耐心。
PH一定要准确为5.0。
加520ul至上述水浴后溶液中。
6:EP管外裹以铝箔纸,避光,轻柔颠倒混匀溶液。
7:加200 ul 石蜡油,防止水分蒸发,限制氧化。
8:50℃避光水浴16h。
一般此步在4pm开始做,熟练的话不到5pm即可完成,水浴16h正好至次日8am 以后收,时间上很合适。
这一步细节:1:基因组DNA的量不需十分精确,宁多勿少,因为在以后纯化回收步骤中会有丢失,且此方法修饰最多可至4ug。
2:所有试剂均须新鲜配制,所以配液的技术要过关,既要快,又要精确。
3:亚硫酸氢钠溶液呈强酸性,一定用碱将PH调制5.0,否则PH不合适会影响后续纯化吸收。
4:水浴最好达16小时,虽可以短至8小时,但后者修饰会有不完全。
第三部分修饰后DNA纯化回收EP管如无特别说明均为高压蒸汽灭菌的。
1. 将移液器枪头伸入石蜡油层下,先轻轻加压使其中一小段石蜡油排出,然后吸取混合液至一洁净1.5mlEP管中。
2:以下使用Promega Wizard Cleanup DNA纯化回收系统(Promega,A7280)1)70℃水浴预热DDW;配制80%异丙醇;2)加1ml Promega’s Wizard DNA Clean-up resin,轻柔颠倒混匀,使DNA充分与树脂结合;3)由于该试剂盒中仅配备针筒没有针栓,如果有真空负压吸引器,使用起来很方便;如果没有,需要自备3ml-5ml注射器。
将注射器针筒与试剂盒提供的回收小柱紧密连接后,将上述混合物用移液器移至针筒内,用2ml以上的EP管放置小柱下接收废液。
加针栓,轻轻加压,将液体挤出,此时可见小柱内有白色的树脂沉积。
4)将注射器与小柱分离后拔出针栓,再将针筒与小柱连接,向针筒内加入2ml 80%的异丙醇,插入针栓,轻轻加压,将异丙醇挤出。
此为洗涤步骤。
5)将注射器与小柱分离,将小柱置于洁净1.5ml洁净EP管上,离心12000rpm,2min,以甩去残余异丙醇成分,使树脂干燥。
此时,修饰后DNA处于与树脂结合状态。
6)将小柱取下置于另一洁净1.5mlEP管上,移液器加50ul预热好的DDW,室温放置5min。
7)离心12000rpm,20s,此为洗脱步骤,此时EP管内液体即为洗脱的修饰后DNA溶液,终体积为50ul。
3:加5.5ul 新鲜配制的3M NaOH,室温放置15min。
4:加33ul 10M乙酸铵,以中和NaOH,使溶液PH于7.0左右。
5:加4ul 10mg/ml糖原,此作为沉淀指示剂,因为其与乙醇混合后可产生沉淀,便于以后离心后辨别回收物的位置,以防在吸取残余乙醇时将回收物吸走。
其实,加入这些糖原究竟能起多大作用,不好说。
不过有国产糖原卖,包装不大,也很便宜,买来一用,算严格遵守文献的步骤吧。
6:加270ul 冰无水乙醇,置于-20度,过夜沉淀。
有人为沉淀最短可至2小时,但我认为时间长些可能会更好。
并且做到此步骤时,一般会到中午,如果样本多的话要到下午,不妨放置过夜,日程可以轻松些,顺便做些其他试验。
如果想当天做完,没有问题,但我认为最好多沉淀些时候,至少6小时吧(这是经验,我做过最少6小时,也是可以的,再短就不敢发表意见了)7:4度,12000rpm离心,30min,倒去上清液,收集沉淀。
不必吸净。
8:加500ul 70%乙醇,不要将沉淀吹打起来,只要把乙醇加上即可。
轻柔倾斜EP管,旋转一圈,再次离心,4度12000rpm,5min。
离心后倒掉上清,再加同量乙醇,同样再做一遍。
此为洗涤步骤,共2次。
9:倒掉上清,并常温简短离心后,将附壁乙醇离至EP管底,移液器小心将残余液体吸净,室温干燥5min,或沉淀由不透明变为半透明或透明时,加入20ul-30ulDDW,溶解沉淀。
至此,已完成了修饰后DNA的纯化回收,所得为修饰后DNA溶液,可用于此后的进一步实验。
10:-20℃保存DNA溶液。
此步细节:1:在使用注射器时,一定要用力均匀且轻,如使用暴力,会将小柱内的薄膜挤破,失去作用。
2:乙酸铵、糖原不需新鲜配制,糖原配好后放在-20度保存,乙酸铵室温即可,因为这样浓度的乙酸铵非常难溶,一旦放在4度,取出用时也会有很多溶质析出。
3:异丙醇、70%乙醇都不需要新鲜配制,但如果用量大,现场配也很方便。
此步关键是在树脂与DNA的结合上,这就再次强调第二部分调亚硫酸钠PH值得重要性。
因为树脂与DNA结合需要有一个适当的PH,如前一步没做好,此步树脂不能与DNA很好结合,将会带来灾难性后果,即DNA随着液体被挤出了,洗脱时实际已没有任何DNA了。
第四部分修饰后DNA用于PCR这一步也没有悬念。
我主要谈一下这里面的几个比较棘手的问题:1:引物问题:我感觉自己设计引物有相当的难度,我曾设计过几对引物,并且试验了一下,但以失败告终。
如果时间充裕、作的又是比较新的基因文献不多,自己设计引物没有问题。
如果不是这样,还是参考文献更好些。
首先查阅SCI 分值高的文献,然后是著名实验室的文献,如果国内有做的,更好了,可以直接联系咨询。
查到序列后,一定要和Genbank中的序列进行比对,防止有印刷错误造成的个别碱基的差别。
然后再到google上搜一下,看用的人多否,体系条件是否一样。
用的人多、体系条件一样,表明可重复性比较强。
我也是按此行事,算比较顺利。
2:Taq酶问题:有文献用高保真的金牌Taq(Platinum),但我感觉只要体系正确、变性退火等条件合适,一般的热启动酶是可以的。
我开始使用的是Takara的LA Taq,很好用,配有10x含mg++的LA缓冲液。
有时候用没了,暂时以Takara 的普通Taq酶也可以。
如何选择,可以根据自己的情况。
初作者还是用好一点的酶。
3:PCR的条件:变性一般都选择95度,3min。
其余我感觉还是根据文献,退火可以根据你的引物的退火温度在小范围内尝试。
一般和文献报道差别不大。
只是扩增片断特异性的问题。
建议根据文献。
4:做PCR的EP管最好选择进口的,壁薄且厚度均匀,这可以保证温度的迅速变化可以及时传递给管内的反应液,使体系真正在所设定的温度下运行。
5:PCR仪:如果在某一个仪器上作出来了,最好一直用此仪器继续。
不同的仪器“脾气”也不一样,但EP管必要和仪器内的插孔紧密结合方好,留有空隙,我认为会影响温度的传递。
第五部分PCR产物的凝胶回收这一步比较简单,可以购买一个凝胶PCR产物回收试剂盒,国产的就很好、价格也合理,比如TIANGEN的产品(用过)。
把切下来的胶按说明书操作即可。
几个细节:1:PCR产物进行琼脂糖凝胶电泳,要使用新配的电泳液。
凝胶浓度1%-2%均可。
2:凝胶DNA回收时在300nm紫外灯下观察条带位置,切取目的片断所在位置的凝胶,尽量小,保证特异性。
3:紫外照射时间不能过长,否则对DNA有损伤。
4:回收后的DNA如不马上用就储存于-20度,在数月内是很稳定的。
第六部分PCR产物与T载体的连接和转化、蓝白斑筛选1:连接T载体(本实验使用的是Promega的试剂盒)15ul体系T-easy 1ulLigase 1ul2xbuffer 7.5ulDNA 5.5ul4度,过夜。
2:连接产物的转化1)-70度冰箱内取出感受态细菌,融化后置于冰上;2)连接产物15ul 全部加入,至于冰上30分钟;3)42度,90秒钟;4)冰上2分钟;5)800ul LB培养基;6)280rpm,37度,摇床45分钟(将管放水平了摇,保证菌液摇匀);7)8000rpm,1分钟;在超净台内去上清,留100-150ul;8)涂板:37度孵箱过夜;(板为含有氨苄青霉素的固体LB培养基)先涂:X-gar 35ulIPTG 25ul后涂:混悬液过夜后可见板上长出很多蓝色或白色斑点,取白色斑点,尤其是蓝色斑点周围的白色斑点,此处自联率较低。
3:取白斑,划种于新板上新板:先涂:X-gar 35ulIPTG 25ul然后于板底划出分区,进行标记。
根据需要,一般一板作50个克隆没有问题。
针头挑白斑划2道于板上相应区域内。
37度,孵箱过夜。
4:联系测序公司送测序。
一般一个克隆在35-45元。
这一部分的细节:1:涂板要均匀,保证Xgar和IPTG均匀分布在板面上;2:不要让蓝白斑长得太满,否则选取克隆时容易一下挑2个。
甲基特异性的PCR扩增(MS-PCR)示意图。
DNA经重亚硫酸盐处理后,以处理后的产物作为模板,加入甲基化特异性的引物(primerⅠ)或非甲基化的引物(primerⅡ),进行特异性的扩增(如图所示),只有结合完全的甲基化或非甲基化特异性引物的片段才能扩增出产物。
这种方法的优点是:1.避免了使用限制性内切酶及其后续相关问题;2.敏感性高;可用于石蜡包埋样本[12];缺点是:1.要预先知道待测片段DNA的序列;2.引物设计至关重要;3.若待测DNA中5-甲基胞嘧啶分布极不均衡,则检测时较为复杂;4.这种方法只能作定性研究,即只能明确是否存在甲基化;若要求定量,则需用其他的方法进行进一步检测;5.存在重亚硫酸盐处理不完全导致的假阳性。