蛋白质二级结构预测软件
- 格式:ppt
- 大小:136.50 KB
- 文档页数:38


sopma用于蛋白质二级结构的预测教程文章标题:深入理解SOPMA及其在蛋白质二级结构预测中的应用在蛋白质研究领域中,二级结构的预测一直是一个具有挑战性但又至关重要的任务。
SOPMA作为一种常用的工具,能够帮助研究人员对蛋白质的二级结构进行有效的预测。
本篇文章将从浅入深地介绍SOPMA的原理和操作步骤,帮助读者深入理解该工具,并掌握它在蛋白质研究中的应用。
一、SOPMA的原理及操作步骤1.1 SOPMA的概念SOPMA(Self-Optimized Prediction Method with Alignment)是一种基于序列的蛋白质二级结构预测工具,它利用序列的物理化学性质和启发式算法来进行预测。
与其他预测方法相比,SOPMA在准确性和稳定性上有一定的优势。
1.2 SOPMA的操作步骤我们需要准备蛋白质序列的数据,并对其进行格式化处理。
将处理后的序列输入SOPMA软件中,并设置相应的参数。
在运行预测过程后,我们可以获得基于序列的二级结构预测结果。
二、SOPMA在蛋白质二级结构预测中的应用2.1 SOPMA预测的可靠性经过研究人员的验证和比对,SOPMA预测的结果通常具有较高的准确性和稳定性。
这使得SOPMA成为许多蛋白质研究工作中不可或缺的工具。
2.2 SOPMA的优势和局限性在实际应用过程中,SOPMA能够对不同类型的蛋白质进行较为准确的二级结构预测,但在一些复杂的情况下也存在一定的局限性。
在使用SOPMA时需要结合其他方法进行综合分析。
三、个人观点与理解在我看来,SOPMA作为一种蛋白质二级结构预测工具,具有较高的实用价值和可靠性。
通过深入学习和使用SOPMA,我们能够更好地理解蛋白质的结构与功能,为相关领域的研究提供有力支持。
总结与回顾通过本文的介绍,我们对SOPMA的原理和操作步骤有了全面的了解,并且深入探讨了其在蛋白质二级结构预测中的应用。
通过对SOPMA的研究与使用,我们能够更好地理解和挖掘蛋白质的结构与功能,并为相关领域的研究工作提供有力的支持。

ncbi蛋白质序列的二级结构
NCBI(National Center for Biotechnology Information)是一个国际知名的生物医学信息数据库,提供了大量的生物学、生物医学和基因组学等相关数据。
在NCBI数据库中,可以通过查询蛋白质的序列标识(如蛋白质的NCBI Accession号码)来获取该蛋白质的相关信息,包括二级结构信息。
获取蛋白质的二级结构信息可以通过以下步骤进行:
1. 在NCBI的主页(https:///)上的搜索栏中输入蛋白质的序列标识,点击搜索按钮进行搜索。
2. 在搜索结果页面中,找到与蛋白质相关的条目,点击进入对应的记录页面。
3. 在记录页面中,可以找到蛋白质的基本信息、序列信息等。
如果该蛋白质的二级结构信息可用,通常会在“Structure”或“3D structure”等部分提供相关链接。
4. 点击相关链接,可以进入蛋白质的二级结构数据库(如PDB,Protein Data Bank)或相关工具网站,以查看该蛋白质的二级结构信息。
需要注意的是,不是所有蛋白质的二级结构信息都可以在NCBI数
据库中直接获取,有些蛋白质可能没有经过结晶和测定结构的报道,或者相关信息尚未被整理和存储在数据库中。
此外,蛋白质的二级结构信息也可以通过其他生物信息学工具和数据库进行预测和推断。

pep-fold3用法
PEP-Fold3是一款在线蛋白质二级结构预测服务器,它可以根据给定的氨基酸序列预测蛋白质的二级结构。
以下是使用PEP-Fold3进行蛋白质二级结构预测的一般步骤:
1.打开浏览器,访问PEP-Fold3的官方网站。
2.在网站首页上,找到并点击“Start”按钮,开始一个新的预测任务。
3.在打开的页面中,输入待预测的蛋白质氨基酸序列。
可以手动输入序列,也可以上传FASTA格式的序列文件。
4.选择预测参数。
在PEP-Fold3中,可以选择不同的预测参数和算法进行蛋白质二级结构预测。
选择适当的参数可以提高预测的准确性和可靠性。
5.提交预测任务。
完成输入和参数选择后,点击“Submit”按钮提交预测任务。
6.等待预测结果。
PEP-Fold3会自动进行蛋白质二级结构预测,并在完成后将结果发送到用户的邮箱中。
7.分析预测结果。
用户可以下载预测结果文件,并使用文本编辑器或生物信息学软件分析预测结果。
需要注意的是,PEP-Fold3是一种基于机器学习的蛋白质二级结构预测方法,其预测结果的准确性受到多种因素的影响,如序列长度、序列相似性、序列复杂度等。
因此,在进
行蛋白质二级结构预测时,需要综合考虑多种因素,并采用多种方法进行预测和验证。



nps server蛋白质二级结构NPS服务器是一种用于预测蛋白质二级结构的工具。
蛋白质的二级结构包括α-螺旋、β-折叠和无规卷曲等形态。
准确预测蛋白质的二级结构对于了解其功能和性质非常重要,因此NPS服务器在生物学研究中扮演着重要的角色。
蛋白质是生物体内构成细胞的基本组成部分,也是许多生物功能的重要执行者。
蛋白质的二级结构是指多肽链的局部结构排列方式,对于蛋白质的稳定性、功能和相互作用起着关键的作用。
因此,准确预测蛋白质的二级结构对于研究蛋白质的结构和功能具有重要意义。
NPS服务器是一种基于机器学习算法的预测工具,它使用大量的已知蛋白质的二级结构信息作为训练集,通过分析蛋白质的氨基酸序列来预测其二级结构。
NPS服务器的预测准确性已经得到了广泛的验证和应用。
NPS服务器的工作原理是利用训练集中的已知蛋白质的氨基酸序列和二级结构信息,构建一个预测模型。
该模型可以根据新的蛋白质序列,通过比对已知的序列和结构,预测该蛋白质的二级结构。
NPS服务器使用的机器学习算法可以对序列中的氨基酸进行分类,将其归类为α-螺旋、β-折叠或无规卷曲。
NPS服务器预测蛋白质的二级结构的准确性受多个因素的影响,其中包括序列的长度、序列的一致性、序列的相似性以及模型的质量等。
较短的序列和高度相似的序列往往预测准确性较高,而较长的序列和低相似性的序列则可能导致预测结果的不准确。
NPS服务器的应用范围非常广泛。
它可以用于预测新的蛋白质序列的二级结构,从而进一步研究蛋白质的结构和功能。
此外,NPS服务器还可以用于分析蛋白质的结构动态性,预测蛋白质的折叠路径和折叠速率等。
它在药物设计、生物信息学和生物工程等领域都有重要的应用。
然而,需要注意的是,NPS服务器的预测结果并非绝对准确,可能存在一定的误差。
因此,在使用NPS服务器的预测结果时,应该结合其他实验方法和技术进行验证和分析,以获得更加可靠的结果。
总之,NPS服务器是一种用于预测蛋白质二级结构的工具,通过机器学习算法分析蛋白质的氨基酸序列,可以准确预测蛋白质的二级结构。
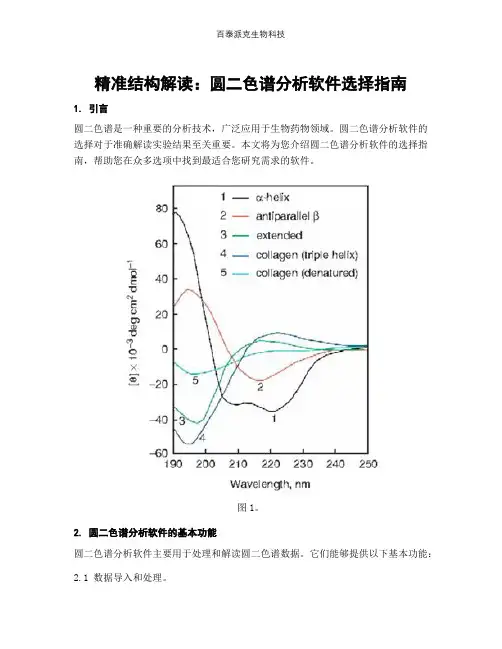
精准结构解读:圆二色谱分析软件选择指南1. 引言圆二色谱是一种重要的分析技术,广泛应用于生物药物领域。
圆二色谱分析软件的选择对于准确解读实验结果至关重要。
本文将为您介绍圆二色谱分析软件的选择指南,帮助您在众多选项中找到最适合您研究需求的软件。
图1。
2. 圆二色谱分析软件的基本功能圆二色谱分析软件主要用于处理和解读圆二色谱数据。
它们能够提供以下基本功能:2.1 数据导入和处理。
优秀的圆二色谱分析软件应具备数据导入和处理的功能。
它们能够读取不同仪器生成的数据文件,并进行预处理,如去除噪音、平滑曲线等。
2.2 结构解析和拟合。
圆二色谱分析软件能够将实验数据与已知结构进行比对,并拟合出最佳的结构模型。
这对于研究人员来说非常重要,因为它能够帮助他们确定分子的构象和构型。
2.3 数据可视化和分析。
优秀的软件还应提供数据可视化和分析的功能。
它们能够生成高质量的图表和图像,帮助研究人员更好地理解实验结果,并进行进一步的数据分析。
3. 圆二色谱分析软件的选择因素在选择圆二色谱分析软件时,有几个关键因素需要考虑:3.1 功能和性能。
首先,您需要考虑软件的功能和性能。
一个好的软件应具备强大的数据处理和结构解析能力,并且能够提供高质量的数据可视化和分析功能。
此外,软件的稳定性和易用性也是需要考虑的因素。
3.2 数据格式兼容性。
不同的圆二色谱仪器可能会生成不同的数据格式。
因此,选择软件时需要确保它能够兼容您实验室使用的仪器生成的数据格式。
这样可以避免数据转换和格式兼容性的问题。
3.3 数据库和参考库。
一些圆二色谱分析软件提供了丰富的数据库和参考库,包括蛋白质、核酸和多肽等。
这些数据库和参考库可以帮助研究人员更好地解读实验结果,并提供结构模型的比对和拟合。
3.4 技术支持和更新。
选择软件时,您还需要考虑技术支持和软件更新的情况。
一个好的软件应该有及时的技术支持和定期的软件更新,以确保软件的稳定性和功能的持续改进。
4. 圆二色谱分析软件的常见选择以下是一些常见的圆二色谱分析软件供您参考:4.1 CDPro。

FTIR计算蛋白质二级结构含量1.简介傅里叶变换红外光谱(F ou ri er Tr an sf o rm In fr ar ed Sp ect r os co py,简称FT IR)是一种常用的分析技术,可以用于研究物质的结构和组成。
在生物化学和蛋白质研究中,利用F TI R可以计算蛋白质的二级结构含量,从而了解其空间构型和功能。
本文将介绍如何使用FT IR进行蛋白质二级结构含量的计算。
2.实验原理2.1傅里叶变换红外光谱F T IR原理是基于物质吸收特定波长的红外光,在物质分子中引起分子振动和拉伸,从而产生特定的光谱图。
通过对所吸收光的频率进行傅里叶变换,可以得到样品的红外光谱。
2.2蛋白质二级结构蛋白质的二级结构是指蛋白质中局部区域的空间构型,包括α-螺旋、β-折叠、无规卷曲和转角等。
不同的二级结构对应不同的峰位和峰形在红外光谱图中的表现。
3.实验步骤3.1样品制备1.选择要研究的蛋白质样品,可以是纯化蛋白质或复杂生物体系中的蛋白质。
2.将蛋白质样品溶解在适当的缓冲溶液中,注意避免样品中存在杂质。
3.2F T I R测量1.准备好F TI R仪器,并对仪器进行校正和调节。
2.将样品溶液放置在专用的FT IR样品盒或对应的样品支架上。
3.将样品放入F TI R仪器中,调整好光谱测量参数,如波数范围和采样间隔。
4.开始记录样品的红外光谱。
3.3数据处理1.导出红外光谱数据,保存为常见数据格式,如Ex ce l或tx t。
2.利用专业的数据处理软件,进行数据绘图和分析。
3.根据红外光谱的峰位和峰形,识别出蛋白质二级结构的特征峰。
4.根据特征峰的面积或峰高,计算蛋白质二级结构的含量。
4.结果和讨论使用FT IR测量蛋白质样品的红外光谱,并根据特征峰的计算方法,可以得到蛋白质二级结构的含量。
该方法具有简便、快速、非破坏性等优点,可以广泛应用于蛋白质研究领域。
然而,需要注意的是,F TI R计算的二级结构含量是一种近似估算,并且对于某些蛋白质样品可能存在一定的误差。

ncbi蛋白质序列的二级结构
NCBI(National Center for Biotechnology Information)是一个提供生物医学和基因组学信息的数据库,它包含了大量的蛋白质序列数据。
蛋白质的二级结构是指蛋白质分子中由氨基酸残基之间的氢键和其他非共价键形成的空间结构。
NCBI数据库中的蛋白质序列可以通过一些工具和算法来预测其二级结构。
一种常用的预测蛋白质二级结构的方法是利用基于序列的预测算法,例如PSIPRED和JPred。
这些算法使用蛋白质序列的氨基酸组成来预测其可能的二级结构,包括α-螺旋、β-折叠和无规卷曲等。
这些预测结果可以在NCBI数据库中的相关蛋白质条目中找到。
另一种方法是利用实验技术,如X射线晶体学和核磁共振等,来直接解析蛋白质的二级结构。
这些实验技术可以提供更准确和直接的二级结构信息,但需要耗费大量时间和资源。
总的来说,NCBI数据库中的蛋白质序列可以通过预测算法和实验技术来研究其二级结构。
这些信息对于理解蛋白质的功能和结构具有重要意义,有助于生物医学和基因组学领域的研究和应用。

DNA序列分析软件介绍Antheprot:蛋白质序列分析软件包ANTHEPROT 4.5是位于法国的蛋白质生物与化学研究院(Institute of Biology and Chemistry of Proteins)用十多年时间开发出的蛋白质研究软件包。
软件包包括了蛋白质研究领域所包括的大多数内容,功能非常强大。
应用此软件包,使用个人电脑,便能进行各种蛋白序列分析与特性预测。
更重要的是该软件能够提供蛋白序列的一些二级结构信息,使用户有可能模拟出未知蛋白的高级结构。
Applied Biosystems Primer Express:这是ABI公司销售附送的软件,可用于设计引物和探针,尤其适用于荧光PCR探针的设计,可以精确计算寡核苷酸与荧光基团鳌合后的Tm值。
可以预测引物与引物之间与模板之间等的二级结构。
Artemis R5:A DNA sequence viewer and annotation tools,一个DNA序列查看器与注释工具,可以以图形形式查看序列的各种分析结果与特性,程序读取EMBL与GENBANK格式的序列与纯DNA序列。
以Java写成,需要安装JRE1.2。
BioEdit是一个序列编辑器与分析工具软件,功能非常强大,使用十分容易。
功能包括:序列编辑、外挂分析程序、RNA分析、寻找特征序列、支持超过20000个序列的多序列文件、基本序列处理功能、质粒图绘制等等。
BLAST (Basic Local Alignment Search Tool)是一套在蛋白质数据库或DNA数据库中进行相似性比较的分析工具。
BLAST程序能迅速与公开数据库进行相似性序列比较。
BLAST结果中的得分是对一种对相似性的统计说明。
BLAST对一条或多条序列(可以是任何形式的序列)在一个或多个核酸或蛋白序列库中进行比对。
BLAST还能发现具有缺口的能比对上的序列。
BLAST是基于Altschul等人在J.Mol.Biol上发表的方法(J.Mol.Biol.215:403-410(1990)),在序列数据库中对查询序列进行同源性比对工作。
了解点冷知识,蛋白质二级结构预测上一次小师弟给大家介绍了从蛋白质一级结构预测相关信息的网站——也即ExPASy ProParam的使用,今天我再给大家介绍一下蛋白质二级结构的预测。
在介绍具体的网站和软件使用之前,我想先介绍一下蛋白质二级结构预测的基础知识。
蛋白质二级结构有如下几种模式:α螺旋(当然,也存在其他形式的螺旋),β折叠,转角(turn),以及无规则卷曲(random coil)。
其中,无规则卷曲并不是一类真正的二级结构,只是作为一种分类,用来表示此类结构没有规则的二级结构。
需要说明的是,无规则卷曲也不是完全随机(random)折叠,其氨基酸分布以及折叠还是有一定规律的,只不过他们的结构非常灵活易变(flexible)。
甚至有研究表明,某些无规则卷曲是有明确而稳定的结构的。
关于二级结构的分类,更详细的可以参考DSSP(Dictionary of Protein Secondary Structure)分类系统,一共将蛋白质二级结构分为8类,其分类依据是二级结构中的氢键结合模式。
Loop,作为另一个在蛋白质二级结构层面的概念,并不是单指上述某一个类型的二级结构,而是一类多样化的二级结构,可以包括转角、无规则卷曲(long loops也被称作无规则卷曲)以及其它连接二级结构的氨基酸链。
Loops多数位于蛋白质分子表面,含有较多亲水氨基酸,具有灵活的构象,可以作为蛋白质的结合位点(比如抗体的抗原结合位点就由6个loop组成)以及酶的催化位点。
二级结构图片在蛋白质二级结构和三节结构之间,还存在两个概念,超二级结构和结构域。
超二级结构在很多教科书中也称作mofit(structural motif: 结构模体/结构基序,或者folding motif:折叠花式)。
超二级结构是指相邻二级结构在三维折叠中相互靠近所形成的组合,分为简单超二级结构和复杂超二级结构。
简单超二级结构一般只包含3个及3个以下的二级结构,我们熟悉的αα,ββ,以及βαβ结构模体就属于简单超二级结构,锌指结构(αββ)也是一种简单的超二级结构。
常用分子生物学软件(一)引言概述:分子生物学软件在当今生物学研究中发挥着重要的作用。
它们以其功能强大和易用性而受到科研人员的青睐。
本文将介绍常用的分子生物学软件,并对它们的主要功能和特点进行详细说明。
正文:一、序列分析软件1. 序列比对软件- BLAST: 用于快速比对蛋白质或核酸序列与已知数据库中的相似序列。
- ClustalW: 对多个序列进行比对,并生成多序列比对结果。
2. DNA/RNA序列分析软件- Primer3: 用于设计引物序列。
- M-fold: 对RNA序列进行二级结构预测。
3. 蛋白质序列分析软件- GRAVY: 计算蛋白质氨基酸序列的相对水溶性。
- ProtParam: 提供氨基酸序列的各种生化性质分析。
4. 基因表达软件- ExPASy Translate: 用于将DNA序列翻译成蛋白质序列。
- Primer-BLAST: 用于设计引物并进行特异性检验。
5. 组学数据分析软件- Galaxy: 提供了一个高度集成的平台,用于处理和分析基因组学数据。
- Cytoscape: 用于可视化和分析分子和基因网络。
二、结构生物学软件1. 分子建模软件- Swiss-PdbViewer: 用于分子可视化和蛋白质模型构建。
- Autodock: 用于模拟蛋白质与小分子之间的相互作用。
2. 蛋白质结构预测软件- Rosetta: 提供了一种高效精确的蛋白质结构预测方法。
- I-TASSER: 通过蛋白质比对和拓扑结构模板识别,预测蛋白质三维结构。
3. 蛋白质结构比对软件- Dali: 用于比对两个或多个蛋白质结构,分析它们之间的结构和功能相似性。
- TM-align: 使用局部结构比对算法,对两个蛋白质的结构进行全局比对。
4. 蛋白质模拟软件- GROMACS: 用于分子动力学模拟和能量最小化。
- NAMD: 适用于分子动力学和分子模拟的高性能软件。
5. 蛋白质结构可视化软件- PyMOL: 用于可视化和分析蛋白质结构。
如何使用SOPMA进行蛋白质二级结构预测在蛋白质科学领域,预测蛋白质的二级结构对于理解蛋白质的功能和性质至关重要。
SOPMA(Self-Optimized Prediction Method with Alignment)是一种常用的工具,用于预测蛋白质的二级结构。
本教程将全面介绍如何使用SOPMA进行蛋白质二级结构预测,并帮助您更深入地理解这一过程。
1. SOPMA的基本原理SOPMA是一种基于序列的二级结构预测方法,它利用了蛋白质序列的保守性和物理化学性质来进行预测。
其原理基于自相似性,通过对蛋白质序列进行比对和分析,识别出其中的重复序列和保守性结构,从而预测出蛋白质的二级结构。
2. 使用SOPMA进行蛋白质二级结构预测的步骤您需要准备待预测的蛋白质序列。
在SOPMA的上线评台或使用其提供的软件工具中,输入蛋白质序列并选择相关参数,如窗口大小和权重。
接下来,点击预测按钮,SOPMA将根据其算法和模型对蛋白质的二级结构进行预测,并给出相应的结果。
3. 理解SOPMA预测结果SOPMA的预测结果通常包括了蛋白质的α-螺旋、β-折叠和无规则卷曲等二级结构元素的具体位置和可能性。
通过分析这些结果,您可以了解蛋白质的结构特征,进而推测其功能和相互作用。
总结与回顾通过本教程,我们全面介绍了如何使用SOPMA进行蛋白质二级结构预测,并帮助您更深入地理解了这一过程。
预测蛋白质二级结构是蛋白质科学研究中的重要一环,它对于理解蛋白质的结构与功能具有重要意义。
在实际应用中,您可以根据SOPMA的预测结果进一步设计实验和验证,从而推动蛋白质科学的发展。
个人观点和理解作为一名蛋白质科学研究者,我深知蛋白质二级结构预测的重要性。
SOPMA作为一种常用的预测工具,其准确性和稳定性得到了广泛认可。
我个人认为,随着技术的不断进步和算法的优化,基于序列的蛋白质二级结构预测将会更加精确和全面,为我们解读蛋白质的奥秘提供更强有力的支持。
alphafold2使用方法
AlphaFold2的使用方法如下:
1. 准备输入数据:将蛋白质序列(FASTA格式)上传至AlphaFold2服务器。
2. 选择模型:AlphaFold2提供了单体模型和多聚体模型,根据研究需求选择合适的模型。
3. 提交任务:在AlphaFold2服务器上提交任务,输入任务名称和选择的数据类型,点击“Submit”按钮。
4. 等待预测结果:AlphaFold2会自动开始预测,等待一段时间后,可以查看预测结果。
5. 分析预测结果:在预测结果页面,可以查看蛋白质的三维结构、结构细节和相关统计信息。
可以使用可视化工具(如PyMOL)来分析和展示预测结果。
6. 其他应用:除了预测蛋白质结构,AlphaFold2还可以用于结构生物学、药物设计等领域。
例如,通过预测蛋白质结构,可以更好地理解蛋白质的功能和相互作用机制,从而为药物设计和疾病治疗提供重要线索。
请注意,AlphaFold2的使用需要一定的生物信息学和计算生物学背景,建
议在使用前进行相关知识和技能的学习和准备。
同时,由于计算资源的限制,AlphaFold2可能需要较长时间才能完成预测任务,因此请耐心等待。