primer premier5引物设计步骤
- 格式:doc
- 大小:21.50 KB
- 文档页数:1

Primer5.0 目的基因序列引物设计
GA TTtCTTGGCTTtATA TA TCTTGT GGAaAGGaCGAAACACCGTGCTCGCTTCGGCAGCAC ATATACTAGTCGACGGGTCTAGACAA TGA TGCTGGGTAA TGACACCAAGCTGGGACTG GTACAGAAAGTCAGAGAACACTTACAGAACGGCA TCTAGACAATGA TGCTGGGTAATA CACTTACAGAACGGCA TCTAGA TGCCGTTCTGTAAGTGTTTGTTGAATGAATGAGTGTT GAACAAACTGCTAAGGTATCTTT ACAAGGTAG
从中扩增出目的基因片段(绿色的部分):
首先:将绿色部分复制到primer5.0引物设计软件中,ctrl+v用鼠标右键不管用
.选择As is后,惦记OK出现:
然后惦记,出现
图中右上角标记,其中惦记S合成的是上游引物,A是下游引物,点击S后出现,然后点击出现
这些就是上游引物,选中后ctrl+c复制到引物合成单中,发给公司,下游引物:回到
点击A出现
用前后调节框,如下,将序列向右移动移到最后,得到下图
注意显示的是3—5,ctrl+c—ctrl+v后悔发现序列变成5---3了,这是软件的好处,因为公司引物合成就是从5—3的
其实上下游引物只是相对的,将得到的引物序列填表后发给公司就好了。

利用软件PrimerPremier5.0进行PCR引物设计的研究利用软件PrimerPremier5.0进行PCR引物设计的研究引言:PCR (Polymerase Chain Reaction) 是一种广泛应用于分子生物学和基因工程领域的技术,通过引物(primers)的选择和设计,能够扩增特定的DNA片段。
PCR引物设计的质量和合理性对PCR扩增的成功与否起着至关重要的作用。
PrimerPremier5.0 是一款被广泛使用的引物设计软件,具有许多功能,可以辅助研究人员选择高质量的引物。
本文旨在研究和探讨利用PrimerPremier5.0软件进行PCR引物设计的方法和优势。
方法:1. 引物设计目标:在PCR实验中,为了获得准确和高效的扩增结果,引物设计时需要考虑以下几个因素:- 引物长度:通常情况下,引物的长度应在18到30个碱基对之间。
- GC含量:GC含量应尽量控制在40%到60%之间,过高或过低都会影响引物的特异性和扩增效率。
- 引物间的互补性:引物之间应避免互补性或三联体结构的形成,以避免非特异性扩增。
- 引物的熔解温度(Tm):引物的Tm值应接近,以确保特异性扩增。
2. PrimerPremier5.0软件的使用:- 安装并启动PrimerPremier5.0软件。
- 导入所需扩增区域的目标序列。
- 在软件中设置引物的参数,如长度、GC含量和Tm值等。
- 执行引物设计程序,软件会根据设置的参数,自动在目标序列中搜索合适的引物。
结果与讨论:通过PrimerPremier5.0软件的使用,可以高效地选择到符合设计要求的引物。
软件会根据设定的参数,自动搜索并筛选出多个候选引物。
1. 引物长度:为了确保引物能够与模板DNA进行特异性结合,通常将引物长度设定在18到30个碱基对之间。
经过软件筛选后,可得到一组长度适当的引物。
2. GC含量:合适的GC含量可以提高引物的稳定性和特异性。
软件会自动根据设定的GC含量范围,筛选出具有适当GC含量的引物。
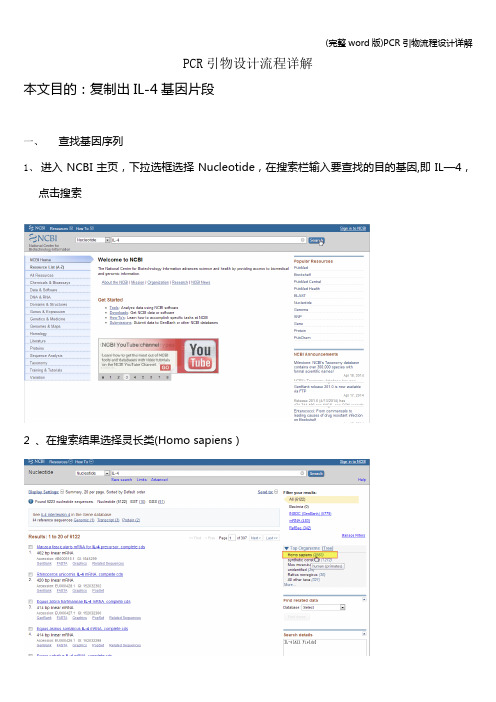
PCR引物设计流程详解本文目的:复制出IL-4基因片段一、查找基因序列1、进入NCBI主页,下拉选框选择Nucleotide,在搜索栏输入要查找的目的基因,即IL—4,点击搜索2 、在搜索结果选择灵长类(Homo sapiens)2、在灵长类IL-4基因中选择需要的mRNA序列3、查看基因的相关信息外显子区域CDs区域4、点击FASTA格式,并将序列保存到文档二、使用primer premier 5。
0设计引物1、建立新文件,将所得的序列复制进输入框内2、点击搜索按钮,搜索引物3、设置引物设计参数(因为在之前查找基因序列的时候获知,外显子区域分别为:1—200、201-248、249-425、426-618,又知在引物设计时引物位置最好跨越一个内含子,PCR产物长度通常为100—150bp,故设定上游引物位置为201—248,下游引物位置为249—425,产物长度为100-150bp)4、确认条件后,显示搜索结果4、双击选中得分最高的引物查看引物情况(上图为上游引物情况,下图为下游引物情况)5、将设计的上下游引物复制出来,保存到文档中三、使用oligo 6.0对设计的引物进行评价1、建立新文件,将从cnki上获得的cDNA复制进输入框,并点击accept接收2、接收后显示出该序列的相关信息3、点击edit按钮录入用primer设计的上游引物,每一次输入新数据后都需要点击accept按钮接收4、同理,录入下游引物5、分析上下游引物二聚体形成情况6、分析上下游引物发卡形成情况7、分析上下游引物GC%8、检测上下游引物与PCR模板其它位置错配情况9、分析PCR整体情况四、引物特异性检验(primer blast)1、进入NCBI主页,并选择blast2、选择primer blast3、在输入框内输入模板序列和上下游引物,并设定对比数据库,点击get primer进行对比4、查看blast结果Blast 结果显示,尽管IL—4与其它基因有相似区,但是引物的3’端没有完全互补。

Primer5.0 目的基因序列引物设计
GA TTtCTTGGCTTtATA TA TCTTGT GGAaAGGaCGAAACACCGTGCTCGCTTCGGCAGCAC ATATACTAGTCGACGGGTCTAGACAA TGA TGCTGGGTAA TGACACCAAGCTGGGACTG GTACAGAAAGTCAGAGAACACTTACAGAACGGCA TCTAGACAATGA TGCTGGGTAATA CACTTACAGAACGGCA TCTAGA TGCCGTTCTGTAAGTGTTTGTTGAATGAATGAGTGTT GAACAAACTGCTAAGGTATCTTT ACAAGGTAG
从中扩增出目的基因片段(绿色的部分):
首先:将绿色部分复制到primer5.0引物设计软件中,ctrl+v用鼠标右键不管用
.选择As is后,惦记OK出现:
然后惦记,出现
图中右上角标记,其中惦记S合成的是上游引物,A是下游引物,点击S后出现,然后点击出现
这些就是上游引物,选中后ctrl+c复制到引物合成单中,发给公司,下游引物:回到
点击A出现
用前后调节框,如下,将序列向右移动移到最后,得到下图
注意显示的是3—5,ctrl+c—ctrl+v后悔发现序列变成5---3了,这是软件的好处,因为公司引物合成就是从5—3的
其实上下游引物只是相对的,将得到的引物序列填表后发给公司就好了。

primer5和Oligo结合设计引物步骤及心得一、引物设计step by step1、在NCBI上搜索到目的基因,找到该基因的mRNA,在CDS选项中,找到编码区所在位置,在下面的origin中,Copy该编码序列作为软件查询序列的候选对象。
2、用Primer Premier5搜索引物①打开Primer Premier5,点击File-New-DNA sequence, 出现输入序列窗口,Copy目的序列在输入框内(选择As),此窗口内,序列也可以直接翻译成蛋白。
点击Primer,进入引物窗口。
②此窗口可以链接到“引物搜索”、“引物编辑”以及“搜索结果”选项,点击Search按钮,进入引物搜索框,选择“PCR primers”,“Pairs”,设定搜索区域和引物长度和产物长度。
在Search Parameters里面,可以设定相应参数。
一般若无特殊需要,参数选择默认即可,但产物长度可以适当变化,因为100~200bp的产物电泳跑得较散,所以可以选择300~500bp.③点击OK,软件即开始自动搜索引物,搜索完成后,会自动跳出结果窗口,搜索结果默认按照评分(Rating)排序,点击其中任一个搜索结果,可以在“引物窗口”中,显示出该引物的综合情况,包括上游引物和下游引物的序列和位置,引物的各种信息等。
④对于引物的序列,可以简单查看一下,避免出现下列情况:3’不要出现连续的3个碱基相连的情况,比如GGG或CCC,否则容易引起错配。
此窗口中需要着重查看的包括:Tm 应该在55~70度之间,GC%应该在45%~55%间,上游引物和下游引物的Tm值最好不要相差太多,大概在2度以下较好。
该窗口的最下面列出了两条引物的二级结构信息,包括,发卡,二聚体,引物间交叉二聚体和错误引发位置。
若按钮显示为红色,表示存在该二级结构,点击该红色按钮,即可看到相应二级结构位置图示。
最理想的引物,应该都不存在这些二级结构,即这几个按钮都显示为“None”为好。

1.安装软件Primer premier5.0。
程序下载:Primer Premier 5.0 /Soft/2005/114.htm 2.程序中双击打开3.点FILE---NEW—DNA SEQUENCE如图所示。
4.输入目的基因片段,可以复制后用ctrl+V键拷贝到栏内,后应加数个N以备后续设计时加酶切位点及保护碱基,如图所示。
5.选中enzyme图标,将所选质粒上的多克隆酶切位点加入左栏6.选中OK键,分析目的基因中所含的酶切位点,选插入位点时就应排除这些酶7.选中primer图标,点S图标,edit primer,开始设计正义链。
8.软件默认引物为25个碱基9.可将鼠标点在设计框的3端从右向左删除7-9个碱基,保留16-18个配对即可10.在引物的5端加入选好的酶切位点并在其左侧加3个保护碱基,入该图加入HIND III酶切位点及TTA保护碱基,完成后点analyze,认为可以后点OK。
11.选中左上角A图标,用鼠标拉动滑块将待选引物放至目的基因末端。
如图,选中EDIT PRIMERS图标,开始设计反义链。
12.从3端删除7-9个碱基同正义链。
13.将酶切位点加在5端,应将产品目录所示的酶切位点序列从右至左加入(注意不要加反)如图加入BamH I酶切位点及CGC3个保护碱基。
完成后点analyze,认为可以后点OK。
14.最后分析结果如图,反义链的FALSE PRIMING可以不考虑,RATING表示引物评分也可以不考虑,主要看Tm值正义链和反义链相差不应超过3度。
GC含量不应超过60%15.该软件有个缺点,不能保存分析结果,只能选择打印16.如果设计RT-PCR检测的引物就如下所示。
同上输入目的基因片段,选SEARCH 图标,选择参数,一般选PCR primers---both—100至250个碱基,引物长短20+/-2,search parametere中的参数可以不选,为默认设置。
点OK。
最近刚开始做PCR,参考了很多与引物设计相关的帖子,结合自己使用primer5和oligo的体会,想谈谈自己设计引物的方法和步骤,恳请各位前辈指正。
RT-PCR引物设计原则和方法在NCBI上搜索到该基因,找到该基因的mRNA,在CDS选项中,找到编码区所在位置,在下面的origin中,Copy该编码序列作为软件查询序列的候选对象。
打开Primer Premier5,点击File-New-DNA sequence, 出现输入序列窗口,Copy 目的序列在输入框内(选择As),此窗口内,序列也可以直接翻译成蛋白。
点击Primer,进入引物窗口。
此窗口可以链接到“引物搜索”、“引物编辑”以及“搜索结果”选项,点击Search按钮,进入引物搜索框,选择“PCR primers”,“Pairs”,设定搜索区域和引物长度和产物长度。
在Search Parameters里面,可以设定相应参数。
一般若无特殊需要,参数选择默认即可,但产物长度可以适当变化,因为100~200bp的产物电泳跑得较散,所以可以选择300~500bp。
点击OK,软件即开始自动搜索引物,搜索完成后,会自动跳出结果窗口,搜索结果默认按照评分(Rating)排序,点击其中任一个搜索结果,可以在“引物窗口”中,显示出该引物的综合情况,包括上游引物和下游引物的序列和位置,引物的各种信息等。
对于引物的序列,可以简单查看一下,避免出现下列情况:3’端不要以A结尾,最好是G或者C,T也可以;3’不要出现连续的3个碱基相连的情况,比如GGG 或CCC,否则容易引起错配。
此窗口中需要着重查看的包括:Tm应该在55~70度之间,GC%应该在45%~55%间,上游引物和下游引物的Tm值最好不要相差太多,大概在2度以下较好。
该窗口的最下面列出了两条引物的二级结构信息,包括,发卡,二聚体,引物间交叉二聚体和错误引发位置。
若按钮显示为红色,表示存在该二级结构,点击该红色按钮,即可看到相应二级结构位置图示。
2. 安装好以后就会在程序中双击打开。
开始设计克隆目的基因的引物。
打开后的界面如图。
点FILE---NEW—DNA SEQUENCE如图所示。
输入目的基因片段,可以复制后用ctrl+V键拷贝到栏内,后应加数个N以备后续设计时加酶切位点及保护碱基,如图所示。
选中enzyme图标,将所选质粒上的多克隆酶切位点加入左栏选中OK键,分析目的基因中所含的酶切位点,选插入位点时就应排除这些酶选中primer图标,点S图标,edit primer,开始设计正义链。
软件默认引物为二-五个碱基可将鼠标点在设计框的3端从右向左删除7-9个碱基,保留16-18个配对即可在引物的5端加入选好的酶切位点并在其左侧加3个保护碱基,入该图加入HIND III酶切位点及TTA保护碱基,完成后点analyze,认为可以后点OK。
选中左上角A图标,用鼠标拉动滑块将待选引物放至目的基因末端。
如图,选中EDIT PRIMERS图标,开始设计反义链。
从3端删除7-9个碱基同正义链。
将酶切位点加在5端,应将产品目录所示的酶切位点序列从右至左加入(注意不要加反)如图加入BamH I酶切位点及CGC3个保护碱基。
完成后点analyze,认为可以后点OK。
最后分析结果如图,反义链的FALSE PRIMING可以不考虑,RATING表示引物评分也可以不考虑,主要看Tm值正义链和反义链相差不应超过3度。
GC含量不应超过60%该软件有个缺点,不能保存分析结果,只能选择打印如果设计RT-PCR检测的引物就如下所示。
同上输入目的基因片段,选SEARCH 图标,选择参数,一般选PCR primers---both—100至250个碱基,引物长短20+/-2,search parametere中的参数可以不选,为默认设置。
点OK。
显示满足设计参数的候选结果,点OK.点SENES选择正义链,RATING表示引物评分,最好选评分高的。
点ANTI-SENSE,相同方式选择反义链。
primer premier5引物设计步骤
引物是在PCR反应中用于扩增目标DNA片段的短链寡核苷酸序列。
Primer Premier 5是一款常用的引物设计软件,以下是引物设计的一般步骤:
1. 目标序列准备:将目标DNA序列输入到Primer Premier 5软件中。
确保序列准确无误,并确定要扩增的目标区域。
2. 引物设计参数设置:根据实验需求和PCR反应条件,设置引物设计的相关参数。
包括引物长度、引物的GC含量、引物间的碱基对数目等。
3. 引物设计策略选择:根据需求选择合适的引物设计策略,如末端限制酶切位点、避免引物间的二聚体形成等。
4. 引物设计和评估:软件将自动生成多个候选引物序列,并根据一定的评估标准对其进行评估,包括引物的互补性、引物之间的二聚体形成、GC含量等。
5. 引物的选择和优化:根据评估结果选择一个或多个最合适的引物序列。
根据需要,可以对引物进行进一步优化,如修改引物长度、GC含量、碱基组成等。
6. 引物合成和实验验证:根据设计的引物序列进行引物合成,并进行PCR 实验验证引物的扩增效果和特异性。
引物设计是PCR实验中至关重要的步骤,引物的质量和合适性直接影响PCR 反应的效果。
Primer Premier 5软件提供了便捷的引物设计工具和功能,可以帮助研究人员高效地设计和评估引物序列。