PCR引物经过流程设计详解
- 格式:doc
- 大小:1.87 MB
- 文档页数:20

2、引物长度一般在15-30碱基之间。
引物长度(primer length)常用的是18-27bp,但不应大于38bp,因为过长会导致其延伸温度大于74℃,不适于Taq DNA 聚合酶进行反应。
3、引物GC含量在40%~60%之间,Tm值最好接近72℃。
GC含量(composition)过高或过低都不利于引发反应。
上下游引物的GC含量不能相差太大。
另外,上下游引物的Tm值(melting temperature)是寡核苷酸的解链温度,即在一定盐浓度条件下,50%寡核苷酸双链解链的温度。
有效启动温度,一般高于Tm 值5-10℃。
若按公式Tm=4(G+C+2(A+T)估计引物的Tm值,则有效引物的Tm为55-80℃,其Tm值最好接近72℃以使复性条件最佳。
4、引物3'端要避开密码子的第3位。
如扩增编码区域,引物3'端不要终止于密码子的第3位,因密码子的第3位易发生简并,会影响扩增的特异性与效率。
5、引物3'端不能选择A,最好选择T。
引物3'端错配时,不同碱基引发效率存在着很大的差异,当末位的碱基为A时,即使在错配的情况下,也能有引发链的合成,而当末位链为T时,错配的引发效率大大降低,G、C错配的引发效率介于A、T之间,所以3'端最好选择T。
6、碱基要随机分布。
引物序列在模板内应当没有相似性较高,尤其是3’端相似性较高的序列,否则容易导致错误引发(False priming)。
降低引物与模板相似性的一种方法是,引物中四种碱基的引物自身不应存在互补序列,否则引物自身会折叠成发夹结构(Hairpin)使引物本身复性。
这种二级结构会因空间位阻而影响引物与模板的复性结合。
引物自身不能有连续4个碱基的互补。
两引物之间也不应具有互补性,尤其应避免3'端的互补重叠以防止引物二聚体(Dimer 与Cross dimer)的形成。
引物之间不能有连续4个碱基的互补。
引物二聚体及发夹结构如果不可避免的话,应尽量使其△G值不要过高(应小于4.5kcal /mol)。

PCR引物设计详细步骤引言PCR(聚合酶链式反应)是一种在分子生物学中常用的技术,用于放大DNA片段。
在PCR过程中,引物的选择非常重要,因为引物的设计质量直接影响到PCR反应的效率和准确性。
本文将详细介绍PCR引物设计的步骤。
步骤1. 确定目标序列首先,需要确定所要放大的目标序列。
这可以是任何你感兴趣的DNA片段,如某个基因的编码区域,特定的DNA序列等。
2. 提取目标序列从已有的DNA样本中提取目标序列。
可以通过DNA提取试剂盒等方法进行提取,确保获得纯净的DNA。
3. 序列比对使用BLAST等工具将目标序列与已知的序列数据库进行比对,以确认目标序列的唯一性和可能存在的变异。
4. 引物设计原则根据目标序列,设计符合以下原则的引物:•引物长度通常在18-25个碱基对之间。
•碱基组成均匀,避免引物中存在大量的G或C碱基,以及连续多个重复的碱基。
•引物之间的互补性尽量避免,以防止二聚体的形成。
•避免引物末端存在碱基的互补序列,以防止非特异性扩增。
5. 引物设计工具使用引物设计工具,如Primer3、NCBI Primer-BLAST等,在目标序列中选择合适的引物。
这些工具可以根据给定的参数,自动设计合适的引物。
6. 引物评估对设计的引物进行评估,包括检查引物的反向互补性、引物的Tm值(熔解温度)、引物的二聚体和自身结构等。
确保引物的质量达到实验要求。
7. 引物合成将设计好的引物发送给合成公司进行合成。
确保引物的纯度和浓度符合要求。
8. PCR反应使用合成的引物进行PCR反应,按照标准的PCR反应体系和条件进行。
根据实验需求调整PCR反应的温度、时间等参数。
9. PCR产物验证通过凝胶电泳等方法验证PCR反应产物的大小和纯度。
确保PCR反应成功,并且没有非特异扩增的产物。
结论PCR引物设计是PCR反应成功的关键。
通过遵循引物设计的原则,结合引物设计工具的辅助,可以设计出合适的引物,弥补PCR技术在DNA放大中的巨大优势,为实验研究提供有效的工具。

实验五 PCR引物设计及评价【实验目的】1、掌握引物设计的基本要求,并熟悉使用Primer premier5.0软件进行引物搜索。
2、掌握使用软件oligo6.0对设计的引物进行评价分析。
【实验原理】一、引物设计原则聚合梅链式反应(polymerase chain reaction)即PCR技术,是一种在体外快速扩增特定基因或DNA 序列的方法,故又称基因的体外扩增法。
PCR技术已成为分子生物学研究中使用最多,最广泛的手段之一,而引物设计是PCR技术中至关重要的一环,使用不合适的PCR引物容易导致实验失败:表现为扩增出目的带之外的多条带(如形成引物二聚体带),不出带或出带很弱,等等。
现在PCR 引物设计大都通过计算机软件进行,可以直接提交模板序列到特定网页,得到设计好的引物,也可以在本地计算机上运行引物设计专业软件。
引物设计原则如下:1、引物应在序列的保守区域设计并具有特异性。
引物序列应位于基因组DNA的高度保守区,且与非扩增区无同源序列。
这样可以减少引物与基因组的非特异结合,提高反应的特异性;2、引物的长度一般为15-30 bp。
常用的是18-27 bp,但不应大于38,因为过长会导致其延伸温度大于74℃,不适于Taq DNA聚合酶进行反应;3、引物不应形成二级结构。
引物二聚体及发夹结构的能值过高(超过4.5kcal/mol)易导致产生引物二聚体带,并且降低引物有效浓度而使PCR反应不能正常进行;4、引物序列的GC含量一般为40-60%。
过高或过低都不利于引发反应。
上下游引物的GC含量不能相差太大;5、引物所对应模板位置序列的Tm值在72℃左右可使复性条件最佳。
Tm值的计算有多种方法,如按公式Tm=4(G+C)+2(A+T);6、引物5'端序列对PCR影响不太大,因此常用来引进修饰位点或标记物。
可根据下一步实验中要插入PCR产物的载体的相应序列而确定。
7、引物3’端不可修饰。
引物3'端的末位碱基对Taq酶的DNA合成效率有较大的影响。


引物设计的详细步骤详细步骤如下:步骤一:了解引物设计的基本原理引物设计是指为特定的DNA序列设计一对合适的引物,以便在PCR反应中扩增目标DNA序列。
引物是PCR反应的关键组成部分,引物的选择和设计对于PCR扩增的成功率和特异性非常重要。
因此,了解引物设计的基本原理对于有效设计合适的引物至关重要。
步骤二:确定PCR反应的目标序列在设计引物之前,我们需要确定PCR反应的目标序列,即我们需要扩增的DNA区域。
这个目标序列可以是已知的基因序列,也可以是未知的区域。
确定目标序列后,我们可以继续设计引物。
步骤三:确定引物的一些基本参数在设计引物之前,我们需要确定一些基本的参数,以便帮助我们选择合适的引物。
这些参数包括引物的长度、GC含量、Tm值以及避免二聚体形成等。
引物长度:通常来说,引物的长度应在18-25个核苷酸之间。
过长的引物可能导致不特异的扩增产物的形成,而过短的引物则可能导致低扩增效率。
GC含量:引物的GC含量对于引物的稳定性和特异性有影响。
在正常情况下,引物的GC含量应在40%-60%之间。
Tm值:引物的Tm值是指引物在PCR反应中的解离温度。
Tm值过低可能导致非特异的扩增产物的形成,而Tm值过高则可能导致低扩增效率。
避免二聚体形成:在设计引物时,我们还需要考虑引物之间的互补性以及避免引物形成二聚体。
引物之间的互补性可能导致引物形成二聚体,从而降低PCR反应的效率和特异性。
步骤四:选择合适的引物设计工具目前有很多在线引物设计工具可供选择,例如NCBI Primer-BLAST、OligoAnalyzer等。
这些工具可以根据输入的目标序列帮助我们快速选择合适的引物。
此外,还可以使用一些商业引物设计软件,如Primer Premier等。
步骤五:进行引物特异性分析设计好引物后,我们需要进行引物特异性分析,确保引物只扩增目标序列而不扩增其他非特异性产物。
这可以通过BLAST或其他相似性工具来完成。
特异性分析的目的是排除可能存在的非特异性扩增产物,以确保PCR反应的准确性和特异性。

pcr扩增的原理和步骤
PCR(聚合酶链反应)是一种用于体外扩增DNA的技术,它能够通过特定的引物和DNA聚合酶实现目标DNA序列的大量复制,使得微量的DNA可以在短时间内得到足够多的复制产物。
PCR的原理可以概括为三个主要步骤:变性、引物结合和延伸。
第一步是变性,即将所需扩增的DNA样本加热至94-98°C,使其双链DNA变为两条单链DNA。
此步骤会破坏氢键,使DNA分子解聚,并暴露出目标序列。
第二步是引物结合,通过在变性的DNA溶液中加入两个特异性的引物。
这两个引物的设计是相互互补的,并且定向分别与目标DNA序列的起始和终止位点结合。
引物与目标序列结合后,温度降低至50-65°C,使引物与带有目标序列的DNA分子发生特异性的酵素结合。
第三步是延伸,即在引物结合的条件下,通过在37-75°C之间的适宜温度下加入DNA聚合酶和适宜的核苷酸三磷酸,使DNA聚合酶从引物的3'末端起始位置开始合成新的DNA链。
在延伸过程中,DNA聚合酶沿着DNA模板链朝着反向方向进行复制,并合成与模板链互补的新链。
以上三个步骤构成了PCR扩增的基本过程,通过不断循环这三个步骤,可以使得目标DNA序列不断复制,并得到大量的
扩增产物。
每个PCR周期将使目标序列的数量成倍增加,因此,经过若干个PCR周期后,可以得到足够多的扩增产物用于后续的分析和应用。

pcr三个步骤(pcr技术三大步骤)本文介绍了PCR的详细操作流程,强调了实验中的注意事项,列举了常用缓冲液的配方。
帮助我们在理解实验的基础上更顺利的操作。
聚合酶链式反应(Polymerase Chain Reaction,PCR)是体外合成双链DNA的一种方法,其原理类似于天然DNA的复制过程,其特异性主要依赖于与其目的片段两端互补的特异引物和高特异性的酶(热启动酶)。
典型的PCR反应有三个步骤:变性,退火,延伸,经过多次循环反应,获得目的片段。
1.DNA模板2.特异性引物3.dNTP Mix4.PCR buffer5.热启动酶1.配置反应体系将各成分依次加入到0.5ml的离心管中扩增使用热启动酶,可在常温下操作,非热启动型的酶要在低温配置反应体系。
2.按照试剂盒说明书设置反应时间和温度,扩增结束后电泳鉴定3.琼脂糖核酸电泳·将电泳所用器具蒸馏水洗净,架好梳子·根据要分离的DNA片段大小制备合适浓度的琼脂糖凝胶,准确称取一定的琼脂糖加入到锥形瓶中,加入30ml左右的电泳缓冲液(TAE或TBE)·在微波炉中加热融化后冷却至40℃左右,充分混匀后倒入电泳槽中·室温下凝固40分钟左右,小心拔出梳子,将凝胶放置电泳槽中准备点样·在电泳槽中加入电泳缓冲液,漫过凝胶表面即可,点样孔内不要有气泡·样品点样前准备好,(在八连管中)加入5ul样品,1ul的6xLoding buffer和1ul的染料混合,用抢将混合后的样品缓缓注入到点样孔中,注意不要串孔·按照正负极(红正黑负),接通电源,电压40-60V,时间30-40min,可根据溴酚蓝的位置判断是否终止电泳·电泳结束,关闭电源,凝胶成像观察,并对比marker确定片段大小不同的琼脂糖浓度分离不同大小的DNA片段:引物引物是决定PCR反应成败的关键,要保证扩增的准确、高效,引物的设计要遵循如下原则:1.引物的设计在cDNA的保守区域,在NCBI上搜索不同物种的同一基因,通过序列分析的软件,得到不同基因相同的序列就是该基因的保守区2.引物的长度:15-28bp,长度大于38bp,会使退火温度升高,不利于普通Taq酶的扩增3.引物GC含量在40%-60%,Tm值最好接近72℃4.因密码子的简并性,引物的3’端最好避开密码子的第三位(最好为T)5.碱基分布错落有致,3’端不超过3个连续G或C6.引物自身不要形成发夹结构,引物设计完成,要BLAST验证模板PCR扩增对模板的要求不高,单链、双链DNA均可作为扩增片段,虽然PCR能够扩增微量的DNA,为了保证扩增的特异性,对不同来源的模板,起始浓度有一定要求,如下:模板可以是纯化后的样品也可以是粗制品,不同来源的模板采取不同的处理方法,实验模板的纯化越简单越好,但模板中如果含有任何的Taq酶抑制剂、蛋白酶、核酸酶等,会干扰反应。
PCR引物设计流程详解本文目的:复制出IL-4基因片段一、查找基因序列1、进入NCBI主页,下拉选框选择Nucleotide,在搜索栏输入要查找的目的基因,即IL—4,点击搜索2 、在搜索结果选择灵长类(Homo sapiens)2、在灵长类IL-4基因中选择需要的mRNA序列3、查看基因的相关信息外显子区域CDs区域4、点击FASTA格式,并将序列保存到文档二、使用primer premier 5。
0设计引物1、建立新文件,将所得的序列复制进输入框内2、点击搜索按钮,搜索引物3、设置引物设计参数(因为在之前查找基因序列的时候获知,外显子区域分别为:1—200、201-248、249-425、426-618,又知在引物设计时引物位置最好跨越一个内含子,PCR产物长度通常为100—150bp,故设定上游引物位置为201—248,下游引物位置为249—425,产物长度为100-150bp)4、确认条件后,显示搜索结果4、双击选中得分最高的引物查看引物情况(上图为上游引物情况,下图为下游引物情况)5、将设计的上下游引物复制出来,保存到文档中三、使用oligo 6.0对设计的引物进行评价1、建立新文件,将从cnki上获得的cDNA复制进输入框,并点击accept接收2、接收后显示出该序列的相关信息3、点击edit按钮录入用primer设计的上游引物,每一次输入新数据后都需要点击accept按钮接收4、同理,录入下游引物5、分析上下游引物二聚体形成情况6、分析上下游引物发卡形成情况7、分析上下游引物GC%8、检测上下游引物与PCR模板其它位置错配情况9、分析PCR整体情况四、引物特异性检验(primer blast)1、进入NCBI主页,并选择blast2、选择primer blast3、在输入框内输入模板序列和上下游引物,并设定对比数据库,点击get primer进行对比4、查看blast结果Blast 结果显示,尽管IL—4与其它基因有相似区,但是引物的3’端没有完全互补。


PCR引物设计的黄金法例1.引物最幸亏模板 cDNA的守旧区内设计。
DNA序列的守旧区是经过物种间相像序列的比较确立的。
在NCBI上搜寻不一样物种的同一基因,经过序列剖析软件(比方DNAman)比对(Alignment ),各基因同样的序列就是该基因的守旧区。
2.引物长度一般在 15~30 碱基之间。
引物长度( primer length )常用的是 18-27 bp,但不该大于 38,由于过长会致使其延长温度大于 74℃,不适于 Taq DNA 聚合酶进行反响。
3.引物 GC含量在 40%~60%之间, Tm值最好靠近 72℃。
GC含量( composition )过高或过低都不利于引起反响。
上下游引物的 GC含量不可以相差太大。
此外,上下游引物的 Tm值( melting temperature )是寡核苷酸的解链温度,即在必定盐浓度条件下, 50%寡核苷酸双链解链的温度。
有效启动温度,一般高于Tm值 5~10℃。
若按公式 Tm= 4(G+C)+2(A+T)预计引物的Tm值,则有效引物的Tm为 55~80℃,其 Tm值最好靠近 72℃以使复性条件最正确。
4.引物 3′端要避开密码子的第 3 位。
如扩增编码地区,引物3′端不要停止于密码子的第 3 位,因密码子的第 3 位易发生简并,会影响扩增的特异性与效率。
5.引物 3′端不可以选择 A,最好选择 T。
引物 3′端错配时,不一样碱基引起效率存在着很大的差别,当末位的碱基为 A 时,即便在错配的状况下,也能有引起链的合成,而当末位链为 T 时,错配的引起效率大大降低,G、C错配的引起效率介于A、T 之间,所以 3′端最好选择 T。
6.碱基要随机散布。
引物序列在模板内应当没有相像性较高,特别是3’端相像性较高的序列,不然简单致使错误引起(False priming )。
降低引物与模板相像性的一种方法是,引物中四种碱基的散布最好是随机的,不要有聚嘌呤或聚嘧啶的存在。

定量pcr引物设计的详细步骤宝子,来给你唠唠定量PCR引物设计的步骤哈。
一、确定目的基因序列。
你得先知道你要研究的目的基因是啥呀。
这就好比你要找一个小伙伴,你得先知道他长啥样。
你可以去一些基因数据库,像NCBI这种超厉害的地方,找到你要的那个基因的序列。
这个序列就像是这个小伙伴的身份证号码,是独一无二的哦。
二、引物设计软件选择。
有好多好用的引物设计软件呢。
比如说Primer Premier,这个就像一个贴心的小助手。
你把目的基因序列输进去,它就能开始帮你设计引物啦。
还有Beacon Designer也很不错哦。
这些软件就像是魔法棒,能在基因的海洋里给你捞出合适的引物来。
三、引物设计参数设置。
这一步很关键哦。
一般来说呢,引物的长度大概在18 - 25个碱基左右就好啦。
太短了就像小短腿,不太稳;太长了又像大长脚,容易出问题。
还有引物的GC含量,最好在40% - 60%之间。
这就像是一个黄金比例,能让引物和模板结合得更牢。
另外,引物自身不能有太多互补的地方,不然它自己就抱成一团,不跟模板好好玩啦。
四、特异性检查。
设计好引物之后,可不能就这么不管了。
得检查一下它的特异性呢。
这就像是给引物做个忠诚度测试。
你可以用BLAST这个工具,把你设计的引物序列放进去,看看它是不是只和你的目的基因结合,要是和其他乱七八糟的基因也有结合,那可不行,就像找错小伙伴啦。
五、引物合成。
如果前面的步骤都没问题啦,那就要把引物合成出来。
这时候就可以找专门的公司啦,就像把设计图交给工匠,让他们做出真正的成品。
合成好的引物拿回来,就可以开始你的定量PCR之旅啦。
宝子,按照这些步骤来,设计定量PCR引物就不是啥难事啦。
加油哦! 。
2010级动科2班杨教童2220103282101511.引物设计的原理和步骤PCR引物设计的目的是为了找到一对合适的寡核苷酸片段,其中在扩增的DNA片断5'端的引物对应于有意链DNA序列,3'端的引物对应于无意链DNA序列,使其能有效地扩增模板DNA序列。
因此,引物的优劣直接关系到PCR的特异性与成功与否。
要设计引物首先要找到DNA序列的保守区。
同时应预测将要扩增的片段单链是否形成二级结构。
如这个区域单链能形成二级结构,就要避开它。
如这一段不能形成二级结构,那就可以在这一区域设计引物。
现在可以在这一保守区域里设计一对引物。
一般引物长度为15~30碱基,扩增片段长度为100~600碱基对。
1.引物设计的原则(1)碱基组成:GC含量应在40%-60%(45%-55% )之间,4中碱基在引物中分配均匀。
没有多聚嘌呤或多聚嘧啶序列,如AAAAA等,没有二核苷酸重复序列,如GCGCGC等;(2)引物长度:引物中与模板互补的区应为18-25个核苷酸长度,上下引物长度差别不能大于3bp,如上游引物为19bp,下游引物为24bp等。
(3)重复或自身互补序列:形成发夹结构,会阻止引物和模板之间的复性。
(4)上下引物的互补性:一个引物的3'末端序列不允许结合到另一个引物的任何位点上,因为PCR中引物浓度较高,会形成引物二聚体。
当一个PCR有多对引物时,注意检查任何一个3'末端都不能和其他任何引物互补。
(5)解链温度(Tm 值):计算出来的两个引物的Tm 值相差不能大于5 ℃,扩增产物的Tm 值与引物的Tm 值相差不能大于10 ℃;引物的Tm 值一般为50-70℃。
这些特性保证了扩增产物在每一个PCR 循环可有效变性。
(6)3’末端3’末端的性质非常关键。
如果可能的话,每个引物的3’末端碱基为G或C;最好不要A,或AA等多聚A;(7)5’端序列添加限制性酶切位点:2.用Primer Premier 5.0 软件设计两对引物(1)在网页上找到要制作引物的一段序列,最好在1000左右,比如:ORIGIN1 atgaatccaa atcaaaagat aataacaatt ggttctgttt ctctcatcat tgccacaata61 tgtttcctta tgcaaattgc tatcctagta actactgtaa cattacattt caagcagcat121 gactacaact cccccgcaaa caaccaagca atgctgtgta aaccaacaat aatagaaaga181 aacacaacag agattgtgta tttgaccaac accaccatag agaaagaaat atgccccaaa241 ctagtagaat atagaaactg gtcaaagccg caatgtaaca ttacagggtt tgcacctttt 301 tccaaggaca attcaattcg gctttctgct ggtggggaca tctgggtgac aagagaacct361 tatgtgtcat gcgatcctga caagtgttat caatttgccc ttgggcaggg aacaacatta421 aacaacggac attcaaataa cactgtacat gataggaccc cttatcgaac cctattgatg481 aatgaattgg gtgttccatt tcatttagga accaggcaag tgtgcatggc atggtccagc 541 tcaagttgtc acgatggaaa agcatggctg catgtttgta taactgggga tgatagcaat 601 gcaacagcta gcttcattta caatgggagg cttgtagata gtattggttc atggtccaaa 661 aatatactca gaacccagga gtcggaatgc gtctgtatca atggaacctg tacagtagta721 atgactgatg ggagcgcttc aggaaaagct gatactaaaa tactattcgt tgaggagggg781 aagatcgttc atgttagcac attgtcagga agtgctcagc atgttgagga gtgctcctgt 841 tatccacgat ttcctggtgt cagatgtgtc tgcagagaca actggaaagg ctccaatagg901 cccatcgtag atataaatgt aaagaattat agcattgttt ccagttatgt atgctcagga961 cttgttggag acacacccag aaaaggcgac agcgtcagca gtagttattg cctagatcct1021 aacaatgaga aaggtggtca tggggtgaaa ggctgggcct ttgatgatgg aaatgacgtg1081 tggatgggaa ggacaatcaa cgagacgtta cgcttaggtt atgaaacctt caaagtcatt1141 gaaggctggt ccaaagctaa ctccaaatta cagacaaata gacaagtcat agttgaaaag1201 ggcgacaggt ccggttattc tggtattttc tccgttgaag gcaaaagctg catcaatcgg 1261 tgcttttatg tggagttgat aaggggaagg aaagaggaaa ctaaagtctg gtggacctca1321 aacagtattg ttgtgttttg tggcacctca ggtacatatg gaacaggctc atggcctgat 1381 ggagcggata tcaatctcat gcctatataa(2)将序列粘贴到制作区域(3)点击Primer后点击Search开始搜索引物,如:上述设计得如下一对引物5’CCTAAGCGTAACGTCTCGT3’TGGGAG GCTTGTAGATAGT优点:转化率为80%,产物长度比较长,为495bp.能够很到限度的利用所选序列的,在5’端没有A或者多聚A结构,引物的Tm值的差距小于5,GC含量在45%到55%之间。
pcr引物设计操作流程
PCR引物设计是PCR技术中非常重要的一步,引物的设计质量直接影响到PCR反应的效果和结果。
下面是PCR引物设计的操作流程:
1. 确定目标序列:首先需要确定要扩增的目标序列,这个序列可以是基因、DNA片段或RNA序列等。
2. 选择引物设计工具:选择合适的引物设计工具,比如NCBI Primer-BLAST、Primer3等在线工具或者专业的引物设计软件。
3. 设定引物设计参数:根据实验需求设定引物设计的参数,比如引物长度、GC含量、Tm值等。
4. 引物设计:根据目标序列和设定的参数,使用引物设计工具进行引物设计。
通常需要设计一对引物,一个用于扩增目标序列的前端,一个用于扩增目标序列的后端。
5. 引物评估:对设计出的引物进行评估,包括检查引物的特异性、二聚性、自身互补性等。
6. 引物合成:将设计好的引物提交给引物合成公司进行合成。
通常引物的长度在18-25个碱基对之间。
7. PCR反应:将合成好的引物与DNA模板、PCR反应缓冲液、
DNA聚合酶等混合,进行PCR反应。
根据引物的设计,可以选择不同的PCR条件进行扩增。
8. PCR产物分析:对PCR反应产物进行分析,比如琼脂糖凝胶电泳、测序等,确认扩增的目标序列是否正确。
总的来说,PCR引物设计是PCR技术中至关重要的一步,需要仔细设计和评估引物,确保PCR反应的准确性和可靠性。
通过以上的操作流程,可以有效地设计出高质量的引物,为PCR实验的成功提供保障。
PCR引物流程设计详解PCR(Polymerase Chain Reaction)引物流程设计是在进行PCR反应过程中引物的设计。
PCR反应是一种体外的DNA复制技术,可在短时间内扩增特定DNA序列。
引物在PCR反应中起到了至关重要的作用,因此设计合适的引物是成功进行PCR反应的关键。
1.目标序列选择:首先需要明确PCR反应的目标序列,即要扩增的特定DNA序列。
选定目标序列后,需要使用相应的软件分析该序列的特性,如GC含量、碱基组成、互补性等。
这些特性将有助于引物的设计和优化。
2. 引物长度:引物的长度通常在18-30bp之间。
较短的引物能提高PCR反应的特异性,但较长的引物能提高PCR反应的特异性和效率。
引物长度不宜超过30bp,以免在PCR反应过程中产生副产物。
3. 引物序列设计:PCR反应通常需要设计两个引物,一个称为前向引物(forward primer),另一个称为反向引物(reverse primer)。
两个引物应该在目标序列两侧的互补区域上设计,以确保引物能够结合在目标序列的两端。
为了提高特异性,引物的3'端应尽可能与目标序列互补,而5'端则可根据需要进行一定的修改,如添加限制性酶切位点、引入Tm值调整等。
4.引物Tm值计算:Tm值可用于估计引物与目标序列结合的稳定性。
Tm值是引物在PCR反应中的解链温度,通常在50-60°C之间。
使用软件计算引物的Tm值时需要考虑引物的长度、碱基组成和浓度等因素,确保引物的Tm值相近。
5.引物特异性检验:根据引物设计的序列,使用引物设计软件进行特异性检验,确保引物只结合在目标序列上而不结合在其他非特定序列上。
特异性检验可通过引物序列的BLAST分析和二聚体结构预测等方法进行。
6.引物修饰:在一些情况下,可以根据需要对引物进行特定的修饰,以增强PCR反应的效果。
常见的修饰方法包括添加引物标记(如荧光标记)、引物末端修饰(如磷酸化)等。
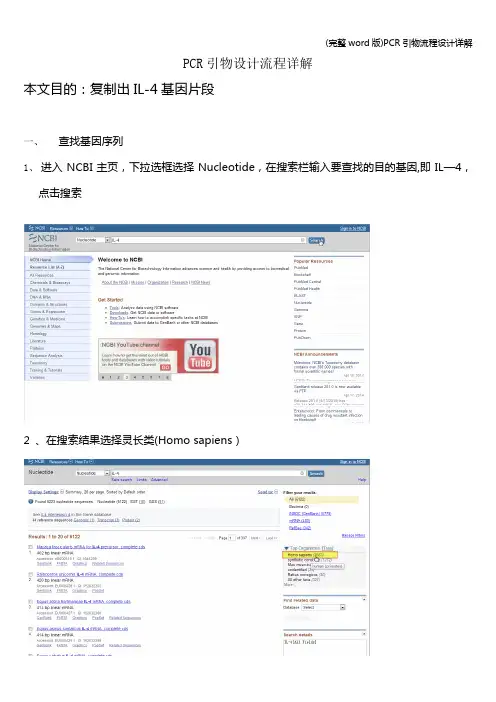
PCR引物设计流程详解
本文目的:复制出IL-4基因片段
一、查找基因序列
1、进入NCBI主页,下拉选框选择Nucleotide,在搜索栏输入要查找的目的
基因,即IL-4,点击搜索
2 、在搜索结果选择灵长类(Homo sapiens)
2、在灵长类IL-4基因中选择需要的mRNA序列
3、查看基因的相关信息
外显子区域
CDs区域
4、点击FASTA格式,并将序列保存到文档
二、使用primer premier 5.0设计引物
1、建立新文件,将所得的序列复制进输入框内
2、点击搜索按钮,搜索引物
3、设置引物设计参数(因为在之前查找基因序列的时候获知,外显子区域分别
为:1-200、201-248、249-425、426-618,又知在引物设计时引物位置最好跨越一个内含子,PCR产物长度通常为100-150bp,故设定上游引物位置为201-248,下游引物位置为249-425,产物长度为100-150bp)
4、确认条件后,显示搜索结果
4、双击选中得分最高的引物查看引物情况
(上图为上游引物情况,下图为下游引物情况)
5、将设计的上下游引物复制出来,保存到文档中
三、使用oligo 6.0对设计的引物进行评价
1、建立新文件,将从cnki上获得的cDNA复制进输入框,并点击accept接收
2、接收后显示出该序列的相关信息
3、点击edit按钮录入用primer设计的上游引物,每一次输入新数据后都需要
点击accept按钮接收
4、同理,录入下游引物
5、分析上下游引物二聚体形成情况
6、分析上下游引物发卡形成情况
7、分析上下游引物GC%
8、检测上下游引物与PCR模板其它位置错配情况
9、分析PCR整体情况
四、引物特异性检验(primer blast)
1、进入NCBI主页,并选择blast
2、选择primer blast
3、在输入框内输入模板序列和上下游引物,并设定对比数据库,点击get
primer进行对比
4、查看blast结果
Blast 结果显示,尽管IL-4与其它基因有相似区,但是引物的3’端没有完全互补。
故设计的引物能特异性的扩增出目的基因。