手把手教你设计引物
- 格式:docx
- 大小:600.44 KB
- 文档页数:7

设计引物的方法
首先,确定目标序列是进行引物设计的第一步。
目标序列的选
择应该考虑到所需扩增的DNA片段的长度和特点,以及引物的位置
和方向。
在确定了目标序列之后,就可以根据目标序列的特点来选
择合适的引物设计方法了。
一种常用的引物设计方法是基于序列比对的方法。
通过对目标
序列与相关物种的序列进行比对,可以找到共同的保守区域,从而
设计出具有较高特异性的引物。
这种方法适用于目标序列与已知序
列高度保守的情况,可以有效地提高引物的特异性和扩增效率。
另一种常用的引物设计方法是基于引物特性的方法。
在这种方
法中,可以根据引物的特性来设计引物,例如引物的长度、GC含量、熔解温度等。
通过调整这些引物特性,可以提高引物的特异性和扩
增效率。
同时,还可以利用一些引物设计软件来辅助设计引物,这
些软件可以根据一定的算法和模型来预测引物的性能,提高引物设
计的准确性和效率。
除了以上介绍的两种方法外,还有一些其他的引物设计方法,
例如基于结构的方法、基于引物库的方法等。
这些方法都有各自的
优缺点,可以根据实际情况来选择合适的方法进行引物设计。
总的来说,设计引物的方法是一个复杂而又重要的过程。
在进行引物设计时,需要考虑到目标序列的特点、引物的特性以及实验条件等多个因素。
通过选择合适的引物设计方法,可以设计出具有较高特异性和扩增效率的引物,从而提高PCR扩增和测序结果的准确性和可靠性。
希望本文介绍的引物设计方法能够对研究人员在实验中有所帮助。

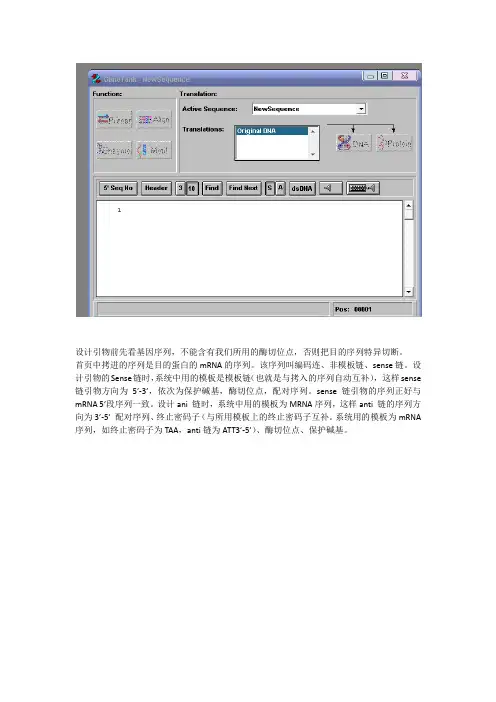
设计引物前先看基因序列,不能含有我们所用的酶切位点,否则把目的序列特异切断。
首页中拷进的序列是目的蛋白的mRNA的序列。
该序列叫编码连、非模板链、sense链。
设计引物的Sense链时,系统中用的模板是模板链(也就是与拷入的序列自动互补),这样sense 链引物方向为5’-3’,依次为保护碱基,酶切位点,配对序列。
sense链引物的序列正好与mRNA 5’段序列一致。
设计ani 链时,系统中用的模板为MRNA序列,这样anti 链的序列方向为3’-5’配对序列、终止密码子(与所用模板上的终止密码子互补。
系统用的模板为mRNA 序列,如终止密码子为TAA,anti链为ATT3’-5’)、酶切位点、保护碱基。

PCR使用说明引物设计技巧PCR(聚合酶链反应)是一种常用的分子生物学技术,可用于扩增DNA片段以及进行基因分型、疾病诊断和DNA克隆等应用。
在PCR实验中,引物的设计是非常关键的步骤之一,合理的引物设计可以确保PCR反应的特异性和高效性。
以下是一些PCR引物设计的技巧和原则。
1.引物长度:引物长度应该在18到30个核苷酸对之间,一般来说,较短的引物可以提高反应的特异性,但也容易导致非特异性扩增。
较长的引物可以提高特异性,但也会降低PCR反应的效率。
2.引物的碱基组成:引物的G+C含量应在40%到60%之间,避免过高或过低的含量,以确保引物的熔解温度适中。
3.引物之间的互补性:引物之间不应有任何互补性,以避免引物之间的杂交和产生非特异性扩增。
4. 引物的熔解温度:引物的熔解温度(Tm)应该相近,通常设计为60℃至70℃之间。
可以使用一些在线工具来计算引物的Tm,例如NCBI的Primer-BLAST。
5.引物的位点选择:引物应该选择在目标序列上独特的位点,避免引物在其他不需要扩增的区域上产生扩增。
可以使用序列比对工具,如BLAST,来确定引物的特异性。
6.引物的末端设计:引物的末端应该避免酶切位点,以防止引物被酶切和降解。
此外,末端的碱基对的GC含量应保持平衡,以确保引物的稳定性。
7.引物的序列结构:引物的序列中应避免重复和倒序重复的碱基序列,因为这些序列容易形成引物间的二级结构和非特异性扩增。
8.引物的交叉反应:引物的序列应该经过认真筛选,避免与其他非目标序列发生交叉反应。
在引物设计前,可以先使用基因序列比对工具,如BLAST,来检查引物是否会与其他区域发生交叉反应。
9.引物的引导方向:引物的引导方向应与目标序列的末端互补,以确保正确的扩增方向。
总而言之,PCR引物的设计应遵循特异性、高效性和可重复性的原则。
合理设计的引物对PCR实验的成功至关重要,可以提高扩增产物的特异性和产量,并避免非特异性扩增和交叉反应的发生。

引物设计的详细步骤详细步骤如下:步骤一:了解引物设计的基本原理引物设计是指为特定的DNA序列设计一对合适的引物,以便在PCR反应中扩增目标DNA序列。
引物是PCR反应的关键组成部分,引物的选择和设计对于PCR扩增的成功率和特异性非常重要。
因此,了解引物设计的基本原理对于有效设计合适的引物至关重要。
步骤二:确定PCR反应的目标序列在设计引物之前,我们需要确定PCR反应的目标序列,即我们需要扩增的DNA区域。
这个目标序列可以是已知的基因序列,也可以是未知的区域。
确定目标序列后,我们可以继续设计引物。
步骤三:确定引物的一些基本参数在设计引物之前,我们需要确定一些基本的参数,以便帮助我们选择合适的引物。
这些参数包括引物的长度、GC含量、Tm值以及避免二聚体形成等。
引物长度:通常来说,引物的长度应在18-25个核苷酸之间。
过长的引物可能导致不特异的扩增产物的形成,而过短的引物则可能导致低扩增效率。
GC含量:引物的GC含量对于引物的稳定性和特异性有影响。
在正常情况下,引物的GC含量应在40%-60%之间。
Tm值:引物的Tm值是指引物在PCR反应中的解离温度。
Tm值过低可能导致非特异的扩增产物的形成,而Tm值过高则可能导致低扩增效率。
避免二聚体形成:在设计引物时,我们还需要考虑引物之间的互补性以及避免引物形成二聚体。
引物之间的互补性可能导致引物形成二聚体,从而降低PCR反应的效率和特异性。
步骤四:选择合适的引物设计工具目前有很多在线引物设计工具可供选择,例如NCBI Primer-BLAST、OligoAnalyzer等。
这些工具可以根据输入的目标序列帮助我们快速选择合适的引物。
此外,还可以使用一些商业引物设计软件,如Primer Premier等。
步骤五:进行引物特异性分析设计好引物后,我们需要进行引物特异性分析,确保引物只扩增目标序列而不扩增其他非特异性产物。
这可以通过BLAST或其他相似性工具来完成。
特异性分析的目的是排除可能存在的非特异性扩增产物,以确保PCR反应的准确性和特异性。
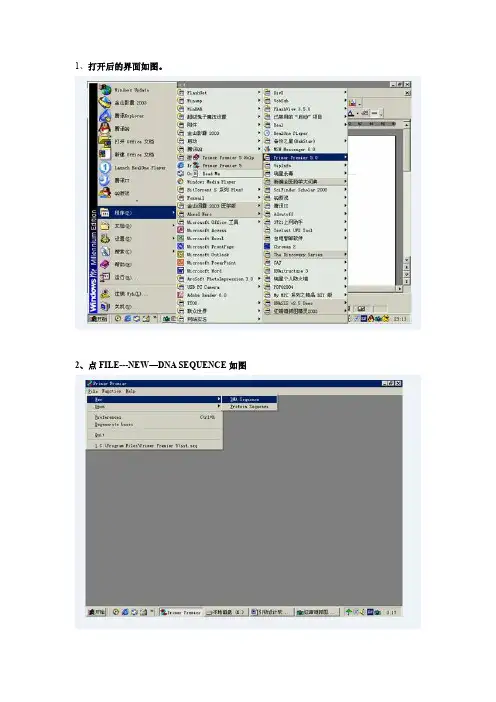
1、打开后的界面如图。
2、点FILE---NEW—DNA SEQUENCE如图3、输入目的基因片段,可以复制后用ctrl+V键拷贝到栏内,后应加数个N以备后续设计时加酶切位点及保护碱基,如图所示。
输入目的基因片段,可以复制后用ctrl+V键拷贝到栏内,后应加数个N以备后续设计时加酶切位点及保护碱基,如图所示。
4、此主题相关图片如下:选中enzyme图标,将所选质粒上的多克隆酶切位点加入左栏此主题相关图片如下:选中OK键,5、分析目的基因中所含的酶切位点,选插入位点时就应排除这些酶此主题相关图片如下:6、选中primer图标,点S图标,edit primer,开始设计正义链。
此主题相关图片如下:7、软件默认引物为二五个碱基此主题相关图片如下8、可将鼠标点在设计框的3端从右向左删除7-9个碱基,保留16-18个配对即可此主题相关图片如下:9、在引物的5端加入选好的酶切位点并在其左侧加3个保护碱基,入该图加入HIND III 酶切位点及TTA保护碱基,完成后点analyze,认为可以后点OK。
此主题相关图片如下:10、选中左上角A图标,用鼠标拉动滑块将待选引物放至目的基因末端。
11、从3端删除7-9个碱基同正义链。
此主题相关图片如下:12、将酶切位点加在5端,应将产品目录所示的酶切位点序列从右至左加入(注意不要加反)如图加入BamH I酶切位点及CGC3个保护碱基。
完成后点analyze,认为可以后点OK。
此主题相关图片如下:13、最后分析结果如图,反义链的FALSE PRIMING可以不考虑,RATING表示引物评分也可以不考虑,主要看Tm值正义链和反义链相差不应超过3度。
GC含量不应超过60%此主题相关图片如下:14、该软件有个缺点,不能保存分析结果,只能选择打印此主题相关图片如下:15、如果设计RT-PCR检测的引物就如下所示,同上输入目的基因片段,选SEARCH图标,选择参数,一般选PCR primers---both—100至250个碱基,引物长短20+/-2,search parametere 中的参数可以不选,为默认设置。

引物设计——手把手教新手引物设计是指在实验室中用于特定目的的DNA序列,用于扩增特定的DNA片段。
引物设计在分子生物学研究和遗传工程方面起着至关重要的作用。
对于新手来说,如何设计合适的引物是一个基础且重要的技能。
下面将以一种扩增人类基因的引物设计为例,手把手地教新手进行引物设计。
首先,我们需要确定扩增的目标基因。
假设我们的目标基因是人类TNF-α基因。
TNF-α是一种调节免疫反应的细胞因子,与一些疾病如炎症性疾病、自身免疫性疾病等密切相关。
因此,深入研究TNF-α基因的功能和表达是非常重要的。
第二步是获取TNF-α基因的序列。
我们可以在公共数据库如NCBI (National Center for Biotechnology Information)中人类TNF-α基因的序列。
在NCBI的网站上,可以输入关键词“human TNF-alpha gene sequence”,并选择合适的结果进行。
我们可以找到人类TNF-α基因的RefSeq基因组序列。
然后,我们需要对所获得的基因序列进行分析,以确定扩增的目标区域。
在TNF-α基因中,选择合适长度的外显子或启动子区域作为目标区域进行扩增。
在选择时,需要考虑该区域的特异性和扩增效率。
接下来,我们需要使用一些在线工具来进行引物设计。
有许多免费的引物设计工具可供选择,如Primer3、NCBI Primer-BLAST等。
在这里,我们将使用Primer3进行引物设计。
在Primer3的网站上,可以将TNF-α基因的目标区域序列粘贴到“SEQUENCE INPUT”输入框中。
然后,可以设置一些参数,如引物长度、Tm值、GC含量等。
对于引物长度,通常选择18-25个核苷酸;Tm值通常在55-65°C之间;GC含量通常在40-60%之间。
设置好参数之后,点击“PICK PRIMERS”按钮,即可获得计算结果。
得到计算结果后,将得到一对引物的序列和其他相关信息,如引物的Tm值、GC含量、特异性等。

设计引物的方法
首先,我们需要明确引物的特性。
引物是一种短链的DNA或RNA序列,通常用于PCR扩增、实时荧光定量PCR、测序等实验中。
因此,引物的选择需要考虑
到引物的长度、GC含量、配对温度等特性。
一般来说,引物的长度应在18-25个
碱基对之间,GC含量应在40%-60%之间,配对温度应在50-65摄氏度之间。
这些
特性的选择将直接影响到引物的扩增效率和特异性。
其次,针对目标序列的特点,我们需要选择合适的引物设计方法。
对于已知的
序列,我们可以利用计算机软件进行引物设计,如Primer3、Beacon Designer等。
这些软件可以根据用户输入的序列信息,自动给出符合要求的引物设计方案。
对于未知序列,我们可以采用引物库筛选的方法,从引物库中挑选符合要求的引物进行实验。
此外,实验的具体要求也是引物设计的考量因素之一。
在进行引物设计时,我
们需要考虑到实验的目的、样本的特点、实验条件等因素。
比如,在进行实时荧光定量PCR实验时,我们需要选择特定的引物和探针,以确保实验的准确性和灵敏度。
在进行测序实验时,我们需要选择特定的引物,以确保测序结果的可靠性和准确性。
综上所述,设计引物的方法需要考虑引物的特性、目标序列的特点以及实验的
具体要求。
在进行引物设计时,我们可以利用计算机软件进行设计,也可以采用引物库筛选的方法。
同时,我们需要根据实验的具体要求,选择合适的引物设计方案。
希望以上内容能够帮助大家更好地进行引物设计,提高实验的效率和准确性。

设计引物的方法引物设计是分子生物学和遗传学研究中的重要环节,它的质量直接影响到PCR 扩增、基因克隆和序列分析等实验的结果。
因此,设计引物的方法显得尤为重要。
在进行引物设计时,需要考虑引物的长度、GC含量、配对特异性、副产物形成和二聚体等因素,以下将详细介绍设计引物的方法。
首先,引物的长度是影响引物特异性和PCR扩增效率的重要因素。
通常,引物的长度应在18-25个碱基对之间,过短的引物可能导致特异性不足,而过长的引物则可能影响PCR扩增效率。
因此,在设计引物时,需要根据目标序列的特点合理确定引物的长度。
其次,GC含量对引物的特异性和稳定性也有重要影响。
一般来说,引物的GC 含量应在40%-60%之间,过高或过低的GC含量都可能导致引物的特异性不足。
因此,在设计引物时,需要通过计算目标序列的GC含量,并合理调整引物的序列,以保证引物的特异性和稳定性。
此外,引物的配对特异性也是设计引物时需要考虑的重要因素。
在目标序列中,需要避免引物与非特异靶序列的配对,因此在设计引物时,需要通过比对目标序列和相关非靶序列,以保证引物的特异性。
另外,引物的副产物形成和二聚体的形成也是设计引物时需要注意的问题。
副产物形成可能导致PCR扩增产物的杂交和非特异性扩增,而引物的二聚体可能导致引物的特异性和扩增效率下降。
因此,在设计引物时,需要通过计算引物的热力学特性,以避免副产物形成和二聚体的形成。
综上所述,设计引物的方法需要综合考虑引物的长度、GC含量、配对特异性、副产物形成和二聚体等因素。
通过合理设计引物,可以保证PCR扩增的特异性和效率,从而确保实验结果的准确性和可靠性。
在实际操作中,可以借助生物信息学工具和引物设计软件,以辅助设计高质量的引物。
希望本文介绍的引物设计方法对您有所帮助。

定量pcr引物设计的详细步骤宝子,来给你唠唠定量PCR引物设计的步骤哈。
一、确定目的基因序列。
你得先知道你要研究的目的基因是啥呀。
这就好比你要找一个小伙伴,你得先知道他长啥样。
你可以去一些基因数据库,像NCBI这种超厉害的地方,找到你要的那个基因的序列。
这个序列就像是这个小伙伴的身份证号码,是独一无二的哦。
二、引物设计软件选择。
有好多好用的引物设计软件呢。
比如说Primer Premier,这个就像一个贴心的小助手。
你把目的基因序列输进去,它就能开始帮你设计引物啦。
还有Beacon Designer也很不错哦。
这些软件就像是魔法棒,能在基因的海洋里给你捞出合适的引物来。
三、引物设计参数设置。
这一步很关键哦。
一般来说呢,引物的长度大概在18 - 25个碱基左右就好啦。
太短了就像小短腿,不太稳;太长了又像大长脚,容易出问题。
还有引物的GC含量,最好在40% - 60%之间。
这就像是一个黄金比例,能让引物和模板结合得更牢。
另外,引物自身不能有太多互补的地方,不然它自己就抱成一团,不跟模板好好玩啦。
四、特异性检查。
设计好引物之后,可不能就这么不管了。
得检查一下它的特异性呢。
这就像是给引物做个忠诚度测试。
你可以用BLAST这个工具,把你设计的引物序列放进去,看看它是不是只和你的目的基因结合,要是和其他乱七八糟的基因也有结合,那可不行,就像找错小伙伴啦。
五、引物合成。
如果前面的步骤都没问题啦,那就要把引物合成出来。
这时候就可以找专门的公司啦,就像把设计图交给工匠,让他们做出真正的成品。
合成好的引物拿回来,就可以开始你的定量PCR之旅啦。
宝子,按照这些步骤来,设计定量PCR引物就不是啥难事啦。
加油哦! 。

一、引物设计st ep by step1、在NCBI上搜索到目的基因,找到该基因的mRNA,在CDS选项中,找到编码区所在位置,在下面的or igin中,Copy该编码序列作为软件查询序列的候选对象。
2、用Prime r Premie r5搜索引物①打开Prim er Premie r5,点击File-New-DNA sequen ce,出现输入序列窗口,Copy目的序列在输入框内(选择As),此窗口内,序列也可以直接翻译成蛋白。
点击Prim er,进入引物窗口。
②此窗口可以链接到“引物搜索”、“引物编辑”以及“搜索结果”选项,点击Sear ch按钮,进入引物搜索框,选择“PCRprimer s”,“Pairs”,设定搜索区域和引物长度和产物长度。
在Searc h Parame ters里面,可以设定相应参数。
一般若无特殊需要,参数选择默认即可,但产物长度可以适当变化,因为100~200bp的产物电泳跑得较散,所以可以选择300~500bp.③点击OK,软件即开始自动搜索引物,搜索完成后,会自动跳出结果窗口,搜索结果默认按照评分(Rating)排序,点击其中任一个搜索结果,可以在“引物窗口”中,显示出该引物的综合情况,包括上游引物和下游引物的序列和位置,引物的各种信息等。
④对于引物的序列,可以简单查看一下,避免出现下列情况:3’不要出现连续的3个碱基相连的情况,比如GGG或CCC,否则容易引起错配。
此窗口中需要着重查看的包括:Tm应该在55~70度之间,GC%应该在45%~55%间,上游引物和下游引物的T m值最好不要相差太多,大概在2度以下较好。
该窗口的最下面列出了两条引物的二级结构信息,包括,发卡,二聚体,引物间交叉二聚体和错误引发位置。
手把手教你设计引物(图文并茂)不知不觉几年下来自己也快毕业了,感谢丁香园这些年来的帮助。
没有什么可回报的东西,就发个帖教教新人如何设计引物吧,尽量做到手把手的教,图文并茂。
引物设计的帖子不少,以前很多战友会推荐Oligo、PrimerPremier、DNA man等等软件。
这些软件设计完最后还是要去BLAST比对下,所以我教大家一种易懂实用的在线设计方法,觉得好的话请投个票。
就以人的PTEN基因为例,首先你要找到他的基因序列,如果你要用的是cDNA,就找它的mRNA序列。
如果你要做的是DNA,就找DNA的序列。
以cDNA为例,普遍的一种方法是上PUBMED中GENE栏搜索找到cDNA那栏,但PUBMED导出序列不太方便,我介绍个网站/index.html1. 输入目的基因,进入2.在左侧栏选择TRANSCRIPT,选择后进入3. 选择PTEN-001中的TRANSCRIPT进入,点击左侧cDNA4. 然后点击CONFIGURE THIS PAGE进入设置你要显示的内容5. 除了第一栏SHOWEXONS选择YES外,其他的都选择NO,然后取个名字保存SAVE CONFIGURATION AS6. 然后在左侧栏点击DOWNLOADVIEW AS RTF可下载你要的cDNA序列,这个文件可以用WORD打开,不同的颜色代表一个外显子间断下载后打开的WORD7. 然后根据可以根据你感兴趣的序列设计引物了,比如我在分别在第6和第7外显子分别设计上下游引物。
选取并复制第6和第7外显子序列8. 登陆/tools/primer-blast/,粘贴这段序列,设置好RANGE和PCR产物的大小,然后在下面点击GET PRIMERS,可以在线设计并比对引物9.最后选择一个比较特异性的引物,条带大小要尽量单一,其他的基因序列尽量不要比对到。
引物设计不求⼈!⼿把⼿教你设计RT-qPCR引物!RT-qPCR 全称 Real-time polymerase chain reaction, 即实时聚合酶链锁反应,是⼀种在DNA扩增反应中,以萤光染⾊剂检测每次聚合酶链锁反应(PCR)循环后产物总量的⽅法。
OK~这⾥对RT-qPCR的原理,应⽤及数据分析就不再赘述了,因为今天我们主要讲的是如何设计出好的PCR引物。
相信科研⼩伙伴们都已经⽤过Pubmed来设计引物了,基本就是 “傻⽠式操作”就解决了!但这⾥还需提醒⼩伙伴们两点:1. 需跨越⾄少⼀个外显⼦连接(⼀般我们⽤来做RT的RNA都是通过Trizol抽提法分离出来的,为了避免基因组的DNA也有可能被扩增出来,就要⾄少跨越⾄少⼀个exon-exon junction 了)。
2. 不要忘记选对“物种”!(这⾥以⼈种⽰例)但是Pubmed设计出来的引物⼀般不尽如⼈意,⽐如说引物之间存在极⼤的互补性,3’端的互补重叠,形成引物⼆聚体等等。
因为⽤Pubmed设计引物本⾝能设置的参数条件就很少,所以它才是最简单的。
所以,今天我要给⼤家介绍两款实⽤的软件,Primer Premier 6.0 和 Oligo 7.0。
今天主要讲讲第⼀款,⾄于第⼆款,改天再聊啦!当然,这两款软件,要想⽤全功能版本,都是需要付费的哦!不过...,我们伟⼤的祖国有强⼤的某宝,这⾥就不多说了哈。
好了,我们进⼊今天的正题……(说了这么多废话,终于要进⼊正题了!!!)⾸先,我们选⼀个基因举例来说,PPARA,⾄于为什么会选这个基因呢?因为啊,PPAR⽬前是肿瘤学研究中的⼀个“明星基因”,它的全称是Peroxisome proliferator-activated receptor, 即过氧化物酶体增殖物激活型受体,⽬前发现有三种亚型,PPAR a,PPARbeta/delta,PPAR r。
好吧…….听着好复杂的样⼦啊!总之,管它到底是什么呢,下⾯来说说它的引物如何设计。
qPCRprimerdesign(手把手教你设计qPCR引物)Step-by-Step Guide to Designing qPCR PrimersBy Marisa Fernández-Cachón - 6th February, 2013 Primer design is a very important step while setting up your qPCR assay. If your primers anneal poorly or to more than one sequence, this can significantly impact the quality and reliability of your results. The good news is that primers are cheap, so you can test several different pairs and choose the best pair to use in your experiments. The bad news is that primer testing requires time and patience, so the sooner you get a pair of primers working, the better. I like to use the NCBI tool Primer BLAST to design primers for qPCR. Here are the main steps to design primers using this free program: Go to the Pubmed gene database and search for your gene of interest. You can filter by species in the right corner of the screen. Click on the gene of interest and scroll down until you find the NCBI Reference Sequence (RefSeq) of your gene (e.g. “NM_203483”).Click there and in the next screen you can see on the right corner of the screen a link to “Pick primers”.Primer ParametersPCR product/amplicon size: For efficient amplification in real-time RT-PCR, primers should be designed so that the size of the amplicon is <200 bp.Number of primers to return: Up to you, but 10 won’t take too long to calculate and will give you plenty of options to choose from.Melting temperature: as a rule, aim for a minimum of 57°C and a maximum of 63°C; the ideal melting temperature is 60°C (with a maximum difference of 3°C in the Tm’s of the twoprimers).Exon/intron selectionTo avoid amplification of contaminating genomic DNA, design primers or probes so that one half hybridizes to the 3′ end of one exon and the other half to the 5′ end of the adjacent exon. To do this, simply select “Primer must span an exon-exon junction”. You don’t ne ed to change the other settings.Primer pair specificity checking parameters: Leave the default settings. The program will use then the Refseq mRNA sequence from the organism that you selected in the screen before to calculate the primers.Checking the output screenThe primers should end with a C or G residue, because T and A residues can bind more easily to DNA in a non-specific way.Optimal primers also have a GC content of around 50-60% to ensure maximum product stability.Regarding self complementarity, the lower the better, to decrease the possibility of primer-dimer formation. Ideally the primer will have a near random mix of nucleotides.Now you can pick the best two or three primers and test them. Good luck!。
PCR引物流程设计详解PCR(Polymerase Chain Reaction)引物流程设计是在进行PCR反应过程中引物的设计。
PCR反应是一种体外的DNA复制技术,可在短时间内扩增特定DNA序列。
引物在PCR反应中起到了至关重要的作用,因此设计合适的引物是成功进行PCR反应的关键。
1.目标序列选择:首先需要明确PCR反应的目标序列,即要扩增的特定DNA序列。
选定目标序列后,需要使用相应的软件分析该序列的特性,如GC含量、碱基组成、互补性等。
这些特性将有助于引物的设计和优化。
2. 引物长度:引物的长度通常在18-30bp之间。
较短的引物能提高PCR反应的特异性,但较长的引物能提高PCR反应的特异性和效率。
引物长度不宜超过30bp,以免在PCR反应过程中产生副产物。
3. 引物序列设计:PCR反应通常需要设计两个引物,一个称为前向引物(forward primer),另一个称为反向引物(reverse primer)。
两个引物应该在目标序列两侧的互补区域上设计,以确保引物能够结合在目标序列的两端。
为了提高特异性,引物的3'端应尽可能与目标序列互补,而5'端则可根据需要进行一定的修改,如添加限制性酶切位点、引入Tm值调整等。
4.引物Tm值计算:Tm值可用于估计引物与目标序列结合的稳定性。
Tm值是引物在PCR反应中的解链温度,通常在50-60°C之间。
使用软件计算引物的Tm值时需要考虑引物的长度、碱基组成和浓度等因素,确保引物的Tm值相近。
5.引物特异性检验:根据引物设计的序列,使用引物设计软件进行特异性检验,确保引物只结合在目标序列上而不结合在其他非特定序列上。
特异性检验可通过引物序列的BLAST分析和二聚体结构预测等方法进行。
6.引物修饰:在一些情况下,可以根据需要对引物进行特定的修饰,以增强PCR反应的效果。
常见的修饰方法包括添加引物标记(如荧光标记)、引物末端修饰(如磷酸化)等。
怎样设计PCR引物的方法引言PCR(Polymerase Chain Reaction,聚合酶链反应)是一种常用的分子生物学技术,常用于DNA或RNA的扩增和定量分析。
而PCR引物是PCR反应中的重要组成部分,它们的设计质量直接影响PCR反应的准确性和灵敏度。
本文将介绍如何设计PCR引物的方法。
引物选择在设计PCR引物之前,首先需要选择需要扩增的目标序列。
一般而言,PCR引物应该满足以下几个要求:1.引物应该与目标序列的两端相互作用,因此需要选择目标序列的两个互补区域作为引物的引导序列。
2.引物长度通常为15-30个碱基对,过短的引物可能导致非特异性扩增,过长的引物则可能导致扩增效率降低。
3.引物的GC含量应在40%-60%之间,过高或过低的GC含量都可能导致引物的特异性下降。
4.引物之间的互补度应尽量避免,以避免产生二聚体或复杂结构。
引物设计工具为了方便设计PCR引物,可以借助一些在线引物设计工具。
以下是一些常用的引物设计工具介绍:1.PrimerQuest: PrimerQuest是IDT(Integrated DNA Technologies)提供的一款免费在线引物设计工具。
用户只需要输入目标序列,该工具将自动设计一对合适的引物,并提供引物质量评估和特异性分析。
2.Primer3: Primer3是一款常用的免费引物设计工具,它可以根据用户提供的目标序列和设计参数,自动设计出符合要求的引物。
3.NCBI Primer-BLAST: NCBI Primer-BLAST是由美国国家生物技术信息中心(NCBI)提供的引物设计工具。
用户可以输入目标序列和其他相关参数,该工具会从NCBI数据库中寻找合适的引物。
4.OligoAnalyzer: OligoAnalyzer是Thermo Fisher Scientific提供的在线引物分析工具。
它可以评估引物的物理和化学性质,同时还能够检测引物之间的二聚体和复杂结构形成情况。
引物设计图⽂教程。
⼀顿操作猛如虎,⼀看战绩0-5。
尽管引物设计是PCR实验成功的前提,但是蛮多⼩伙伴的实战经验稍显不⾜。
今⼉就向⼤家介绍2种引物设计⽅法。
This is the dividing line.引物设计尽管是⽼⽣常谈了,但是⼩编还是想先把引物设计的原则放出来(其实是复制粘贴啦,哪哪都有)。
1、引物应⽤核酸系列保守区设计并具有特异性。
最好位于编码区5\'端的300-400bp区域内,可以⽤DNAMAN,Alignment软件看看结果。
2、不能形成2级结构。
3、引物长度⼀般在17-25bp之间,上下游引物不能相差太⼤。
4、G+C含量在40-60%之间,45-55%最佳。
5、碱基要随机分布,尽量均匀。
6、引物⾃⾝不能有连续4个碱基的互补。
7、引物之间不能有连续4个碱基的互补。
8、引物5‘端可以修饰。
9、3’端不可修饰,⽽且要避开AT,GC富集的区域,避开T/C,A/G连续结构(2-3个)。
10、引物3\'端要避开密码⼦的第三位。
11、引物整体设计⾃由能分布5\'端⼤于3‘端。
12、定量产物长度80-150bp最好,最长是300bp。
原则忒多啦,⼩伙伴们,先别晕,看下⽅。
⽅法⼀:PrimerBank法⽹址:打开以上⽹址,进⼊PrimerBank⽹站界⾯,如下图个⼈觉得这是最便捷的引物获取⽅法,该⽹站可以⾮常迅速地检索到已经他⼈验证的引物序列及相关反应条件。
图中第⼀个⽅框,⼀般选择NCBI Gene ID;第⼆个⽅框选择你所关注的物种,常⽤的就是Mouse和Human了,如果为其它物种,则选择All Species即可;第三个⽅框中需要输⼊你关注的基因ID号码。
如何获取基因ID呢,这⾥就需要打开NCBI⽹站,选择Gene,并输⼊你的⽬的基因如p53 homo,选择第⼀个,可见其ID号为7157。
如下图所⽰。
然后将ID号7157输⼊上⽅PrimerBank⽹站的For text框中,并选择种属为⼈,点击Submit后,跳转页⾯如下。
手把手教你设计引物(图文并茂)
关键词:引物设计2014-05-14 10:33来源:丁香园点击次数:9911
不知不觉几年下来自己也快毕业了,感谢丁香园这些年来的帮助。
没有什么可回报的东西,就发个帖教教新人如何设计引物吧,尽量做到手把手的教,图文并茂。
引物设计的帖子不少,以前很多战友会推荐Oligo、PrimerPremier、DNA man等等软件。
这些软件设计完最后还是要去BLAST比对下,所以我教大家一种易懂实用的在线设计方法,觉得好的话请投个票。
就以人的PTEN基因为例,首先你要找到他的基因序列,如果你要用的是cDNA,就找它的mRNA序列。
如果你要做的是DNA,就找DNA的序列。
以cDNA为例,普遍的一种方法是上PUBMED中GENE栏搜索找到cDNA那栏,但PUBMED 导出序列不太方便,我介绍个网站 /index.html
1. 输入目的基因,进入
2.在左侧栏选择TRANSCRIPT,选择后进入
3. 选择PTEN-001中的TRANSCRIPT进入,点击左侧cDNA
4. 然后点击CONFIGURE THIS PAGE进入设置你要显示的内容
5. 除了第一栏SHOWEXONS选择YES外,其他的都选择NO,然后取个名字保存SAVE CONFIGURATION AS
6. 然后在左侧栏点击DOWNLOADVIEW AS RTF可下载你要的cDNA序列,这个文件可以用WORD打开,不同的颜色代表一个外显子间断
下载后打开的WORD
7. 然后根据可以根据你感兴趣的序列设计引物了,比如我在分别在第6和第7外显子分别设计上下游引物。
选取并复制第6和第7外显子序列
8. 登陆 /tools/primer-blast/,粘贴这段序列,设置好RANGE和PCR产物的大小,然后在下面点击GET PRIMERS,可以在线设计并比对引物
9.最后选择一个比较特异性的引物,条带大小要尽量单一,其他的基因序列尽量不要比对到。