水稻突变体介绍及鉴定(很详细)
- 格式:doc
- 大小:585.50 KB
- 文档页数:9



水稻粒型基因水稻作为世界上最重要的粮食作物之一,在全球粮食安全和农业经济中扮演着重要的角色。
水稻的形态特征中,粒型是农民和育种者关注的重点之一。
粒型的改善可以对水稻产量和品质产生显著影响。
而这一特征的遗传基础主要是由水稻的粒型基因所决定的。
一、粒型的分类水稻的粒型通常分为长粒、中粒和短粒三类。
长粒水稻种子长度超过7毫米,宽度较窄,比较瘦长。
中粒水稻种子长短在5-7毫米之间,宽度中等。
短粒水稻种子长度不到5毫米,较为短胖。
这三种粒型的特点对水稻的种植和加工具有重要影响。
二、粒型基因的发现和研究在过去几十年中,科学家们通过对水稻粒型的观察和研究,已经发现了一些与粒型紧密相关的基因。
其中最有代表性的是粒形因子1基因(GS3)。
该基因位于水稻第三染色体上,对于水稻粒型的控制有着显著影响。
不同突变型的GS3基因会导致粒型的变化。
具体来说,短粒突变型与长粒型相比,GS3基因的功能发生了改变,抑制了水稻胚乳细胞的纵向扩张,从而导致了粒型的改变。
除GS3基因外,还有一些其他与粒型相关的基因也被科学家们发现。
例如,GS5、GL3.1、GW2等基因,它们分别参与了水稻产量、种子长宽比和粒重等方面的调控。
通过研究这些粒型基因,可以更好地了解水稻形态特征的形成机制,为水稻的育种和生产提供科学依据。
三、粒型基因的利用和应用粒型基因的发现为水稻育种带来了巨大的潜力。
通过控制特定粒型基因的表达和功能,可以实现对水稻粒型的调控和改良。
例如,在水稻育种中,人工选择长粒突变体可以通过改善米质来提高米饭的品质。
长粒水稻通常有较高的胶质含量,米粒吸水性好,煮熟后更容易吸收调味品的味道。
对于出口大米和高端餐饮市场来说,长粒水稻是更受欢迎的选择。
此外,根据粒型基因的遗传规律,通过杂交育种和基因编辑等技术手段,可以将不同粒型的优良基因进行组合,加速培育出更加理想的水稻品种。
这些品种不仅能够适应不同环境条件下的种植,还能够满足消费者对粒型多样性的需求。


张琪,刘爽,胡艳娟,等.水稻OsRIP 基因家族的全基因组鉴定及其表达分析[J].沈阳农业大学学报,2023,54(4):385-395.沈阳农业大学学报,2023,54(4):385-395Journal ofShenyang Agricultural University http ://DOI:10.3969/j.issn.1000-1700.2023.04.001收稿日期:2023-06-09基金项目:科技部国家重点研发计划项目(2017YFD0300107);国家自然科学基金项目(32070642)第一作者:张琪(1994-),女,博士研究生,从事水稻分子生物学研究,E-mail :******************* 通信作者:王晓雪(1963-),女,博士,教授,博士生导师,从事水稻分子生物学研究,E-mail :***************.cn水稻OsRIP 基因家族的全基因组鉴定及其表达分析张琪,刘爽,胡艳娟,王晓雪(沈阳农业大学水稻研究所,沈阳110161)摘要:水稻(Oryza sativa )中RIP 基因家族编码典型的E3泛素连接酶,在生长发育、逆境响应过程中发挥重要作用。
利用水稻基因组数据,采用生物信息学方法鉴定了水稻中的RIP 基因家族成员,鉴定到6个RIP 家族基因,命名为OsRIP1~6,其编码区长度在906~1086bp 之间,分布在5条染色体上,内含子数量为2~3个。
水稻中RIP 基因被分为3个亚家族,每个亚家族的内含子数量、基因结构、motif 基本一致。
蛋白结构域分析结果表明,在水稻中,RIP 家族的所有成员均含有SINA 和RING 结构域。
共线性分析显示,水稻与玉米存在最多的共线对,与拟南芥不存在共线对。
对启动子顺式作用元件分析发现,RIP 基因启动子区有多个非生物胁迫响应元件、光响应元件及激素响应元件。
另外,还分析RIP 基因家族在不同组织器官、不同发育时期及昼夜节律的表达模式,结果表明在根、茎及花器官中表达水平较高;OsRIP6在播种后83d 表达水平最高,而其他成员在播种后48~69d 的表达处于较高水平;昼夜表达的分析结果显示,在光照后10h 该基因家族成员的表达水平较高。


项目类型学科一级门学科二级类项目来源项目人数项目时段指导教师项目组其他成员:项目研究内容摘要1、对水稻中一个突变体YJS的鉴定与分离。
YJS突变体是水稻品种93-11经过辐照诱变得到,大田表现为剑叶鞘松散,最上节间弯曲下垂。
2、对引起该表型的YJS基因进行精细定位在已有的初定位的基础上,利用YJS与水稻品种N22进行杂交,在海南种植F2群体并取得极端个体,运用图位克隆的方法,对YJS进行进一步的精细定位。
立论依据(包括项目的研究意义、现状分析,并附主要参考文献及出处)研究意义、现状分析:水稻是主要的粮食作物之一,全世界近一半的人口以大米为主食。
随着水稻全基因组测序的完成,水稻已经成为遗传学和生物学研究的模式植物。
对于育种工作来讲,理想株型是他们的目标。
早在20世纪30年代,Heath和Boysen Jenson等指出叶片的姿态和数量是决定物质生产差异的重要因素,实际上提出了作物的株型问题,(Boysen-Jenson, 1932; Heath, 1938)。
水稻株型是指在群体条件下,个体植株的茎、桑、叶、穗的整体形态性状的配置。
理想株型是育种家建立的优化配置,指在群体条件下,使植株的形态尤其是叶片始终处于最佳姿势,能充分利用特定生态条件下的光能,从而使生物学产量和经济系数均达到尽可能高的水平。
水稻的株型涉及到叶片形态、株高、分蘖情况、穗部形态等(陈温福与徐正进, 1989; 晏静, 2010)。
稻的叶是由叶片和叶鞘组成的,在其交界的部位有叶耳和叶舌。
叶鞘紧紧包裹着茎秆,一方面承受着穗子的重量,使茎秆更加强壮。
另一方面在孕穗过程中如果叶鞘包裹不紧,有可能致使病害的发生,(曹慧娟, 1992)。
叶鞘包裹茎秆是否紧密,对水稻的产量有直接重要的影响。
对于水稻产量而言,最上节间的形态尤为重要,其长度直接决定了穗子的建成。
(Zhu et al., 2006)本研究诣在克隆到一个引起水稻叶鞘和最上节间畸形基因YJS,将有助于水稻株体形态建成机理的探明以及围绕有关性状展开的一系列生理生态学和产量性状等研究的进行,从而有助于水稻育种家在育种过程中通过生物设计对叶型和株型进行改良,从而实现优化高效的育种目的。



水稻类病变突变体性状及分子机理研究进展焦然;徐娜;胡娟;宋周琳;胡佳青;饶玉春;王跃星【期刊名称】《中国水稻科学》【年(卷),期】2018(032)003【摘要】类病变突变体是植物在无显著逆境或损伤情况下以及未受到病原物危害时叶片上自然形成的类似病斑的一类突变体.水稻类病变突变体的产生常伴有抗病性的增强,其中涉及到防御相关基因的表达.相关研究主要集中于各种突变体的定位和克隆以及突变体的抗病性和抗病机制.综述了水稻类病变突变基因的性状及抗病性的研究进展,特别是对水稻类病变突变体形成原因、遗传特性、调控途径进行了梳理,并提出对水稻抗病育种的展望,以期为进一步分析类病变突变体的各种机制奠定理论基础,并为水稻育种提供参考.【总页数】11页(P285-295)【作者】焦然;徐娜;胡娟;宋周琳;胡佳青;饶玉春;王跃星【作者单位】浙江师范大学化学与生命科学学院, 浙江金华 321004;浙江师范大学化学与生命科学学院, 浙江金华 321004;浙江师范大学化学与生命科学学院, 浙江金华 321004;浙江师范大学化学与生命科学学院, 浙江金华 321004;浙江师范大学化学与生命科学学院, 浙江金华 321004;浙江师范大学化学与生命科学学院, 浙江金华 321004;中国水稻研究所水稻生物学国家重点实验室, 杭州 310006;中国水稻研究所水稻生物学国家重点实验室, 杭州 310006【正文语种】中文【中图分类】Q343.5;S511.032【相关文献】1.水稻类病变突变体中抗病相关基因的研究进展 [J], 程晨;王晶;原文霞;李冬月;杨勇;严成其;陈剑平2.水稻类病变突变体spl5的抗病及其机理分析 [J], 傅淑芳;陈析丰;郝亮;孙青芝;金杨;马伯军3.水稻类病变突变体及其基因的研究进展 [J], 王海杰;秦月月;董娜;赵力;李文英;李秀兰4.水稻抗倒伏性状的分子机理研究进展 [J], 刘畅;李来庚5.水稻spl5突变体类病变受光诱导的机理分析 [J], 刘辉;袁俊杰;傅小进;陈析丰;马伯军因版权原因,仅展示原文概要,查看原文内容请购买。
颜静宛,陈子强,周淑芬,等.利用CRISPR/Cas9系统创制水稻品种GW2基因的突变体[J].江苏农业科学,2024,52(3):73-78.doi:10.15889/j.issn.1002-1302.2024.03.011利用CRISPR/Cas9系统创制水稻品种GW2基因的突变体颜静宛,陈子强,周淑芬,王 锋(福建省农业科学院生物技术研究所/福建省农业遗传工程重点实验室,福建福州350003) 摘要:培育具有育种价值的GW2基因编辑的水稻优异新品种在水稻育种中具有重要意义,利用CRISPR/Cas9基因编辑技术,以生产上广泛推广应用的13份水稻品种为材料,对粒质量基因(GW2)进行定向性状改良,通过农杆菌转化创制出一批无T-DNA元件的水稻非转基因GW2突变纯合株系。
结果表明:13份T0代水稻转基因中,有28.0%~59.1%植株的GW2基因发生了突变,纯合突变株数量占总突变株数量的35.0%,双等位突变株数量占总突变株数量的14.2%,杂合突变株数量占总突变株数量的50.8%。
此外,不同水稻品种发生的突变类型也略有不同。
对13份T2代非转基因水稻GW2突变纯合株进行千粒质量性状的考种分析。
与对应的野生型亲本品种相比,纯合突变水稻植株的千粒质量显著提高10.81%~58.22%。
本研究结果极大地丰富了GW2的突变类型,为不同水稻品种的高产稳产创造了重要的种质资源,同时也为利用基因编辑提高水稻产量提供了有价值的育种信息。
关键词:水稻;CRISPR/Cas9;基因编辑;粒质量;GW2基因;突变 中图分类号:Q344+.14;S511.01 文献标志码:A 文章编号:1002-1302(2024)03-0073-06收稿日期:2023-04-08基金项目:福建省科技计划———省属公益类科研院所基本科研专项(编号:2020R1027008);福建省农业高质量发展超越“5511”协同创新工程(编号:XTCXGC2021002)。
3BIOTECHWORLD 生物技术世界1 综述生物信息学(Bioinformatics )是一门综合性的前沿科学,是信息科学的迅速发展在生物学和计算机科学的综合性应用[1]。
它是一门与信息科学、计算机科学、数学、物理学和生物学等学科相互渗透的交叉学科[2]。
生物信息学的研究重点由一般的计算生物学逐步渗入基因组学(Genomics)和蛋白组学(Proteomics)两方面, 即从核酸和蛋白质序列出发, 分析序列中表达的结构与功能的生物信息[3]。
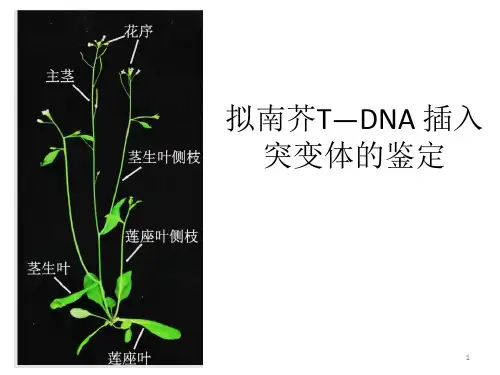
拟南芥属于被子植物门,双子叶植物纲。
拟南芥的优点是植株小,结子多,拟南芥的基因组是目前已知植物基因组中最小的而水稻是属于被子植物门,单子叶植物纲[4]。
水稻所结子实即稻谷,去壳后称为大米。
属于直接经济作物,世界近一半的人口以大米为主食,大米是人类重要的主食之一。
在被子植物中,花粉壁是雄性配子体表面包裹的一层致密物质。
在抵御各种环境压力或微生物的侵袭以及授粉时细胞的识别等方面具有重要作用。
花粉壁由花粉内壁和花粉外壁所组成[5]。
近年来在模式植物拟南芥中,人们克隆了许多与花粉外壁形成相关的基因,这些基因的突变往往会导致植株雄性不育的表型。
这些基因涉及到绒毡层发育的调控、孢粉素的生物合成与运输,以及胼胝质壁和初生外壁的形成[6]。
拟南芥CYP704B1基因是植物细胞色素P450基因家族中的成员之一[7]。
拟南芥CYP704B1基因是合成催化中长链脂肪酸的—羟基化的酶它参与花粉外壁的合成有利于花粉的育性[8]。
目前有关于拟南芥CYP704B1基因的研究有国外的Anna等人2009年的研究揭示了CYP704B1基因在拟南芥花粉形成中的作用,介绍了该基因的功能。
CYP704B1属于植物细胞色素P450蛋白,在花粉外壁长链脂肪酸合成过程中起还原酶的作用,该基因在在花药发育过程中表达强烈,该基因突变体表现为花粉外壁的破坏,造成花粉不育,所说该基因是花粉外壁发育过程中不可缺少的功能基因。
RMD水稻突变体信息及基因型鉴定1.背景介绍:突变体对于遗传学研究有着重要作用,随着拟南芥和水稻等物种全基因组测序的开展,人类积累了前所未有的基因序列信息,为了弄清这些基因序列的生物学信息,寻找该基因区段序列发生变异的突变体是阐释基因功能最直接最有效的方法。
植物在自然的环境条件下也会产生突变性状,早期普通正向遗传学研究往往通过寻找与某种生物学特性相关的突变体来发掘或定位某个特定基因。
为配合植物功能基因组研究高通量的策略,构建水稻等物种的大型突变体库已成为必然,借助水稻全基因组测序信息、通过反向遗传学的手段大规模地筛选突变体库,理论上可以获得基因组中任一基因的突变体,最终实现阐释基因功能的目的。
2.原理:2.1农杆菌介导的T-DNA 插入农杆菌是寄主范围非常广泛的土壤杆菌,它能通过伤口侵染植物导致冠瘿瘤和毛状根的发生。
1974从根癌农杆菌中分离出一种与肿瘤诱导相关的质粒,称为致瘤质粒(Tumor-inducing plasmid),简称Ti 质粒。
Ti 质粒上存在一段DNA,能够转移并整合到植物基因组中,称为Transferred DNA,简称T-DNA。
研究发现,T-DNA 两端存在非常保守的同向重复的25bp 序列,分别称为左边界(LB)和右边界(RB)。
T-DNA 的转移只与边界序列相关,尤其是RB,而与T-DNA区段的其它基因或序列无关。
我们将T-DNA 区段上的致瘤基因和其它无关序列去掉,利用其转移的特性,实现农杆菌介导的T-DNA 转入水稻愈伤,从而构建水稻突变体库。
大量研究表明,农杆菌T-DNA 整合到植物基因组中的位置是随机的,并且整合到植物基因组中的T-DNA 能稳定遗传。
由于插入到植物基因组中的T-DNA 区段序列已知,这样随机插入到植物基因组中的T-DNA类似于给植物基因“贴”了一个序列标签。
我们利用这个标签,通过各类PCR技术最终可以获取其插入的位点。
2.2 水稻Tos17 反转录转座子创造水稻突变体的另一种方法是利用植物的反转录转座子,它们是以DNA→RNA→DNA 的方式进行转座,在水稻上已发现大约40 种长未端重复的反转录转座子,它们是Tos1-Tos32,RIRE1-RIRE8,其中5 类被证明是有转座活性的,分别是Tos10、Tos17、Tos19、Tos25 和Tos27。
这些反转录转座子只有在组织培养条件下才具备转座活性,其中Tos17 的转座活性最强,容易插入到富含基因的区域,因此可以直接用于创造插入失活的突变体库。
利用含有Tos17 插入的水稻突变体库,可以进行突变性状的筛选,T os17 反转录转座子正成为水稻功能基因组研究的一个有力工具。
由于Tos17 反转录转座子为水稻内源的转座子,不需要进行转基因的过程,而且平均每株含有8 个Tos17 个拷贝,在正常情况下能够稳定遗传,因此Tos17 转座子突变体库是水稻功能基因组研究的一个有用资源。
但也有研究表明,Tos17 在转座过程中存在几类转座热点,它容易插入到与抗病相关基因及蛋白激酶基因中。
同时,Tos17 插入突变体库中观察到的突变体大约只有10%的突变是真正由于Tos17插入造成,另外的突变可能是由于组织培养过程中体细胞变异造成的,也有可能是组织培养激活了其它类的转座子发生转座所致。
3.水稻突变体库3.1 水稻突变体库功能由于T-DNA 在植物基因组中能稳定遗传,且拷贝数较低,对于突变体的遗传分析较容易。
加之T-DNA 序列已知,一旦发现某个突变性状与T-DNA 共分离,则采用Tail-PCR、Inverse-PCR 或质粒拯救等方法很容易分离T-DNA 侧翼植物基因组序列。
欲寻找某个基因的突变体,可以设计相应引物,采用Pool-PCR 的方法高通量地筛选突变体库。
另外,通过构建T-DNA 侧翼序列数据库,也可以大规模地寻找基因的突变体。
采用Tail-PCR 的方法分离T-DNA 插入突变体库的侧翼序列,有助于将T-DNA 插入位点在染色体上定位,寻找已知基因不同区段的突变体。
此外,在T-DNA 区段构建Gene trap、Enhance trap 等元件后,还可通过检测报告基因来反映靶位点基因的表达模式。
3.2 遗传转化体系建立农杆菌介导的转化主要依据Hiei(1994)的水稻转化方法并有所改动。
简单流程如下:菌株活化↓愈伤诱导→继代→预培养→侵染→共培养→抗性愈伤筛选→分化成苗→生根→炼苗移栽粳稻品种愈伤组织诱导/继代、预培养/共培养及筛选培养基用N6 培养基,分化和生根用MS 培养基(其成分见附录1)。
3.3 突变体库转化植株的编号为了便于突变体库产生的所有信息如植株种子、T-DNA 侧翼序列、报告基因的表达模式等的统一管理,本研究中水稻突变体库的管理参照统一的数据库管理和编号方法进行,如编号“01Z11AB90”,“01”表示该突变体产生的年份为2001 年;“Z11”表示转化的野生型亲本为中花11 号,中花15 的编号则为“Z15”,“AB”表示不同英文字的组合,代表不同的区号;“90”为阿拉伯数字编号,表示同一小区内的顺序号,编号范围为“01-96”。
3.4 水稻突变体发芽:由于突变体发芽率会普遍降低,我们建议发芽的方法是:首先取10-20粒突变体种子,采用常规催芽的方法,若最后突变体发芽率高于60%的,即可将余下的种子进行常规催芽。
如果发芽率偏低,我们建议您采用组培发芽(具体方法见附录2)。
4.突变体基因型鉴定4.1 载体介绍构建水稻T-DNA 突变体库我们一共用过3个载体:pFX ,pSMR 和pEGFP 。
它一样。
T-DNA 为图中红线左侧。
载体 报告基因 pFX GUS&GFP pSMR GUSpEGFP GFP 因此,无论是哪个载体,鉴定基因型的步骤都是一样的。
载体左端测序引物:NTLB5 5’ AAT CCA GAT CCC CCG AAT TA 3’载体右端测序引物:PFRB4 5’ TGC AGG TTC TCT CCA AAT 3’ 4.2 基因型检测的原理如下:对每个插入位点基因设计一对跨T-DNA 或Tos17的基因组引物-Sense primer (S )和Antisense primer (AS ),通过这两条基因特异引物(S 和AS )与一条载体上已的引物(primer A 或primer B )进行配对扩增,就能确定后代的基因型(见下图)。
1) 如果T-DNA 或Tos17在某位点的插入是纯合的,当用基因组上一条引物(S 或AS )和载体引物进行配对扩增时,能够得到目的条带。
但是由于T-DNA 或Tos17的插入使两条基因组引物(S 和AS )间距太大,所以用两条基因组引物S 和AS 不能扩增出条带;2) 如果该位点没有T-DNA 或Tos17插入,当用两条基因组引物(S 和AS )扩增时能够得到目标条带,但用基因组上一条引物(S 或AS )和载体引物配对扩增时不能得到目标条带;3) 如果T-DNA 或Tos17在某位点的插入是杂合的,则用两条基因组引物(S 和AS )以及一条基因组引物(S 或AS )和载体引物的配对扩增都能得到目标条带。
pFX-E24.2-15R 17741 bpAmp CAM RESISTANCE HYG(R)GFP EGFP BoGUSCAT-1 Intron Catalase intron T BORDER (L)T-BORDER (R)pVS1 StapBR322 bom site GAL4/VP16pUC ori F1 oriPOLY A SITE CAMV35S6xUAS pLAC pVS1-REP pBR322 ori4.2 引物选择农杆菌T-DNA整合到植物基因组中的位置是随机的,可能出现正向插入和反向插入两种情况,同时我们分离侧翼序列时根据使用引物的不同,会分离到T-DNA左端侧翼序列和右端侧翼序列。
当我们拿到感兴趣的突变体的编号后,如何选择合适的引物组合来进行基因型的鉴定呢?第一步,检查T-DNA的插入方向具体方法是:将突变体的侧翼序列在NCBI上进行比对,比对结果中会出现一个strand值(如下图红框显示):strand=Plus/ Plus,代表我们分离到的侧翼序列与基因组正义链匹配;Strand=Plus/Minus,代表我们分离到的侧翼序列与基因组反义链匹配;但是,侧翼序列在基因组上的正反向并不能代表T-DNA在基因组上的的插入方向,我们还需要通过分析当初分离侧翼序列时使用的是T-DNA的左端还是右端的引物,来确定T-DNA在基因组上的的插入方向。
第二步,确定侧翼序列是在T-DNA左端还是右端具体方法是:我们的突变体有统一的编号,前缀表示分离的方法等(具体见RMD网站详细介绍),后缀显示的是分离侧翼序列时所使用的引物(见下图红框,测序时即可使用该引物)。
如果我们使用引物NTLB5,代表分离得到的是T-DNA 的左端序列,如果我们使用引物PFRB4,代表分离得到的是T-DNA的右端序列,第三步,T-DNA在基因组上的的插入方向根据第一步和第二步的结论,我们即可确定T-DNA在基因组上的的插入方向,并选择合适的检测基因型的引物(结论见表一)。
分离侧翼序列引物strand=Plus/ Plus Strand=Plus/MinusNTLB5分离反向插入正向插入PFRB4分离正向插入反向插入T-DNA正向插入到基因组中情况:T-DNA反向插入到基因组中情况:第四步,确定引物组合根据第三步的结论,我们可以选择引物组合进行基因型鉴定(见表二)。
表二:分离侧翼序列的引物T-DNA正向插入T-DNA反向插入NTLB5 S+AS/S+NTLB5 S+AS/AS+NTLB5 PFRB4 S+AS/AS+PFRB4 S+AS/S+PFRB4 4.3 举例说明鉴定基因型的步骤:1)用突变体的侧翼序列(可在/查找)和水稻基因组比对(推荐NCBI),找到T-DNA插入位点。
以03Z11AA66为例,其侧翼序列为:>Sequence1|Possible location: Chromosome 3 (by Zhang Jian)CGTCC GCAATGTACTCACCCGGCCTAGCA TACGACCTGCTGCCTGTCTTGAGAACGCC ACGAAGCGGCAGCCTGCATGTCGTTTACG AGGAGCCCTTAGATTCCCCTCCAGAATTA GGAGTACACGCCCTGTGTTGGTGTCGATC AATCGATAATGTAGATTGATCGCAATCGA GATGGGTGTTGATGAGCAGGATCATATCC ATCCTCCTCTTGGGGGTTTCTCTGCAGGC TCCTGACAGACAATGAATGTGTGCACATG ATCAATGCAGATGCGTGCAGCTATCGAAA TCTTGGTTGGTACTTGTCCTGGATGGCTT GATTAGATTGTTAATACCAAAACACATTT TTTCGCTCATACTGATATACGAGTAGTAG TAGGACTTCCTCGTGAGCTCTCTTGAATTCAT CTCACTCGACGA用该侧翼序列在NCBI上做BLAST,结果如右图:由BLAST结果可知以下信息:1.T-DNA插入为点为第三染色体5013391处,2.该家系strand=Plus/ Plus,3.分离侧翼序列的引物为NTLB5(TTL _03Z11AA66 _NTLB5)将2和3结果对应表一得知T-DNA是反向插入在基因组中。