U6和H1启动子序列——Sequences mouse and human U6 promoter and human H1 promoter
- 格式:docx
- 大小:12.25 KB
- 文档页数:1

启动子概述启动子是DNA链上一段能与RNA聚合酶结合并能起始mRNA合成的序列,它是基因表达不可缺少的重要调控序列。
启动子是一段位于结构基因5’-端上游区的DNA序列,能活化RNA聚合酶,使之与模板DNA准确地结合,并具有转录起始的特异性。
基因的特异性转录取决于酶与启动子能否有效地形成二元复合物。
启动子分三类:启动子Ⅰ、启动子Ⅱ、启动子Ⅲ.只有启动子Ⅱ指导mRNA的转录。
真核生物启动子Ⅱ由两大部分组成:上游元件(upstream element)和启动子核心(core promoter)。
上游元件与转录的效率有关;启动子核心包括3部分:TATA盒、起始子(initinator)及下游元件(downstream element)。
TATA盒为转录调控因子包括各种调节蛋白的结合区,与转录起始位点的精确选择及转录有关,起始子是转录起始所必须,下游元件作用尚不清楚。
原核生物启动子区范围较小,包括TATAAT区(Pribnow区)及其上游的TTGACA区。
启动子是一段提供RNA聚合酶识别和结合位点的DNA序列,位于基因上游。
启动子具有如下特征:1序列特异性。
在启动子的DNA序列中,通常含有几个保守的序列框,序列框中碱基的变化会导致转录启动活性的改变。
2方向性。
启动子是一种有方向性的顺式调控元件,有单向启动子和双向启动子两类。
3位置特性。
启动子一般位于所启动转录基因的上游或基因内的前端。
处于基因的下4种属特异性。
原核生物的不同种、属,真核生物的不同组织都具有不同类型的启动没有启动子,基因就不能转录。
原核生物启动子是由两段彼此分开且又高度保守的核苷酸序列组成,对mRNA的合成极为重要。
启动子区域:(1)Pribnow盒,位于转录起始位点上游5—10bp,一般由6~8个碱基组成,富含A和T,故又称为TATA盒或—10区。
启动子来源不同,Pribnow盒的碱基顺序稍有变化。
(2)—35区,位于转录起始位点上游35bp处,故称—35区,一般由10个碱基组成。
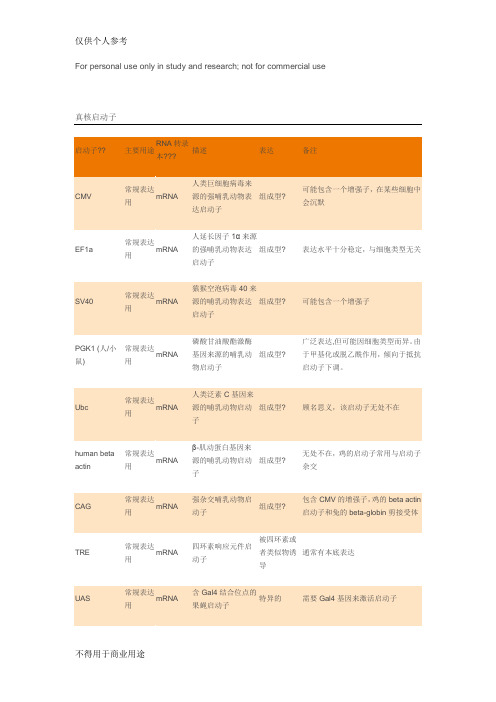
For personal use only in study and research; not for commercial use真核启动子EF1a常规表达用mRNA人延长因子1α来源的强哺乳动物表达启动子组成型? 表达水平十分稳定,与细胞类型无关PGK1 (人/小鼠)常规表达用mRNA磷酸甘油酸酯激酶基因来源的哺乳动物启动子组成型? 广泛表达,但可能因细胞类型而异。
由于甲基化或脱乙酰作用,倾向于抵抗启动子下调。
human beta actin常规表达用mRNAβ-肌动蛋白基因来源的哺乳动物启动子组成型? 无处不在,鸡的启动子常用与启动子杂交TRE常规表达用mRNA四环素响应元件启动子被四环素或者类似物诱导通常有本底表达Ac5常规表达用mRNA果蝇Actin 5c 基因来源的强昆虫启动子组成型? 果蝇表达系统的常用启动子CaMKIIa光遗传学基因表达mRNA?Ca2+/钙调蛋白依赖的蛋白激酶 II 启动子特异的 用于中枢神经系统/神经元表达。
受到钙和钙调蛋白调节。
TEF1常规表达用mRNA酵母转录延伸因子启动子组成型? 与哺乳动物的EF1a 启动子类似ADH1常规表达用mRNA乙醇脱氢酶I 的酵母启动子被乙醇抑制全长版本很强,促进高表达。
截短启动子是组成型的,表达较低。
Ubi常规表达用mRNA玉米泛素基因的植物启动子组成型? 在植物中促进高表达U6小RNA 表达shRNA 来源于人U6小核启动子组成型? 小鼠U6也使用,但效率略差。
常用的原核表达系统启动子T7lac 高水平基因表达T7噬菌体来源的启动子加上lac操纵子几乎没有本底表达,需要T7 RNA聚合酶,受到lac操纵子的控制,可以被IPTG诱导。
常用与pET载体,受到lac操纵子的严格调控araBAD 常规表达用阿拉伯糖代谢操纵子的启动子阿拉伯糖诱导弱,常用与pBAD载体。
适合于快速调控和低的本底表达lac 常规表达用Lac操纵子来源的启动子可以被IPTG或者乳糖诱导在常规的大肠杆菌中,lacI阻遏蛋白表达量不高,仅能满足细胞自身的lac操纵子,无法应付多拷贝的质粒的需求,导致非诱导条件下较高地表达,为了让表达系统严谨调控产物表达,能过量表达lacI阻遏蛋白的lacIq 突变菌株常被选为Lac/Tac/trc表达系统的表达菌株。

分生考点Copyright by 孙倩1.顺式作用元件(cis-acting elements): 存在于基因内外,与基因表达调控相关、能够被基因调控蛋白特异性识别和结合的特定的DNA序列称为顺式作用元件。
2.启动子(promoter):真核基因的启动子指的是RNA聚合酶识别、结合的基因转录调控区中启动基因转录的一段特异DNA序列,包含一组转录调控功能组件,其中每一个功能组件的DNA序列约7~20 bp。
3.典型的启动子核心序列(core sequences)是在转录起始位点上游25~35 bp处,有一保守的TATA序列,被称为TATA盒(TATA box),真核细胞的TATA盒多为TATAAAA序列。
TA TA盒与原核细胞的启动子一样,对RNA聚合酶II的转录起始位点起定位作用。
4. 有一些编码蛋白质基因不含TA TA盒或起始子,多在起始位点上游约100bp内含有20~50个核苷酸的CG序列,被称做CpG岛(CpG island)。
此种基因可有多个转录起始点,可产生含不同5’末端的mRNA。
这些基因大多为低转录基因,编码中间代谢酶的管家基因。
5.启动子上游元件(promoter-proximal elements, 或upstream promoter elements)是一些位于TATA盒上游的DNA序列,与调节蛋白结合,调节通用转录因子与TATA盒的结合、RNA聚合酶与启动子的结合,以及转录起始复合物的形成,从而决定基因的转录效率与专一性。
常见的序列是CAA T盒和GC盒。
6.一些真核细胞基因含有另一种启动子元件,称为起始子(initiator,Inr),决定启动子的强度。
7.增强子(enhancer):是能够结合特异基因调节蛋白,促进邻近或远隔特定基因表达的DNA 序列。
在酵母中,被称为上游活化序列(upstream activator sequences, UASs)。
增强子的作用通常与其所处的位置和方向无关。

真核生物启动子由于真核生物中有三种不同的RNA聚合酶,因此也有三种不同的启动子,其中以启动子Ⅱ最为复杂,它和原核的启动子有很多不同:(1)有多种元件:TATA框,GC框,CATT框,OCT等;(2)结构不恒定。
有的有多种框盒如组蛋白H2B;有的只有TATA框和GC框,如SV40早期转录蛋白,(3)它们的位置、序列、距离和方向都不完全相同,(4)有的有远距离的调控元件存在,如增强子;(5)这些元件常常起到控制转录效率和选择起始位点的作用;(6)不直接和RNA pol 结合。
转录时先和其它转录激活因子相结合,再和聚合酶结合。
(一)Ⅱ类基因的启动子和调控区Ⅱ类基因的启动子由核心元件和上游元件组成。
核心元件包括TATA框和转录起始位点附近的启始子(initiator,Inr)。
在起始点一般没有同源序列,但mRNA的第一个碱基倾向A,另一侧翼由Py组成(在原核启动子的CAT起始序列也有这种情况),称为起始子(initiator),一般由PY2CAPY5构成,位于-3~+5,可能提供RNA pol Ⅱ识别。
无论TATA是否存在,Inr对于启动子的强度和起始位点的选择都是十分重要的。
现已分离纯化了与Inr特异结合的蛋白质因子。
1.核心元件TATA框合又称Hogness框,Goldberg-Hogness框,俚语称为金砖(Goldbrick),其一致序列是:T85A97T93A85A63A83A50,常在起始位点的上游-25左右,相当于原核的-10序列。
但-10是不可缺少的,而真核启动中也有的缺乏TATA框。
其作用是:(1) 选择正确的转录起始位点,保证精确起始,故也称为选择子(selector),当有的基因缺少TATA框时,可能由Inr来替代它的这一作用,如鼠的脱氨核苷转移酶(Tdt)基因就没有TATA框,但有17bp的Inr;(2) 影响转录的速率。
TATA框的8bp的保守序列一般都是由A.T对组成,少数情况在其中的两个位点上由G.C对取代了A.T,可见它是较容易打开。

翻译控制中使用的启动子与终止子序列翻译是蛋白质合成的一个重要环节。
RNA分子在翻译时,需要识别启动子和终止子序列,才能正常进行翻译。
启动子和终止子序列的识别是由蛋白质因子完成的,这些因子的结构和功能非常复杂,与翻译的效率和准确性密切相关。
启动子和终止子序列的作用启动子序列是指RNA分子上的一个序列,它是翻译起始的位置,也叫做起始密码子。
终止子序列则是指RNA分子上的一个序列,它是翻译终止的位置,也叫做终止密码子。
翻译需要识别启动子和终止子序列,才能确定翻译的区间,从而正常进行蛋白质的合成。
启动子和终止子序列的识别RNA分子的翻译需要调控因子的参与,这些调控因子可以帮助RNA分子上的识别因子识别起始密码子和终止密码子。
启动子和终止子序列的识别是复杂的过程,牵涉到多种机制和因素的参与。
1.启动子序列的识别在翻译开始之前,启动子序列的位置需要被识别因子确定。
激活蛋白质复合物(APC)是识别启动子序列的一种蛋白质复合物。
APC由多种蛋白质组成,它可以与RNA分子上的启动子序列结合,从而引起翻译的开始。
此外,转录起始因子(TF)也是识别启动子序列的关键蛋白质。
TF可以识别特定的启动子序列,从而在翻译过程中调节起始区域的翻译。
另外一些蛋白质调控因子也可以影响启动子序列的识别。
例如,代表性因子(RP)在翻译起始区域的具体位置上对启动子的识别起到关键作用。
代表性因子的存在可以将起始区域上的翻译定位更准确地控制在起始密码子位置,从而促进RNA分子的翻译。
2.终止子序列的识别终止子序列的识别是翻译过程的另一个关键环节。
终止子序列被识别后,翻译就会自然停止,并且避免蛋白质的过度合成。
终止子序列的识别由一种专门的蛋白质因子完成,称为终止因子(TS)。
终止因子可以识别RNA分子上的特定终止序列,从而引发翻译的停止。
TS的存在对于保证翻译精准程度非常重要。
启动子和终止子序列的调节除了识别,启动子和终止子序列的识别还受到很多其他因素的调节。

u6基因序列
u6基因序列是一种非编码RNA,其主要功能是作为RNA聚合酶III的转录因子,参与核糖体RNA和tRNA的转录。
该基因序列的长度约为100个核苷酸,其中包括一个特定的TATA盒序列和一个保守的结构域。
u6基因序列在真核生物中广泛存在,并在细胞的基因表达调控中发挥着重要作用。
此外,u6基因序列也是许多RNAi技术中的基本元素,通过与特定的小分子RNA结合,可以实现对基因的靶向沉默。
因此,研究u6基因序列的特性和功能对于深入理解细胞基因表达调控机制、开发新的RNAi技术和治疗基因相关疾病具有重要意义。
- 1 -。
U6启动子是一种常用于基因工程中的启动子序列。
启动子是一段DNA序列,位于基因的上游区域,用于调控基因的转录过程。
U6启动子是一种特殊的启动子,用于驱动RNA聚合酶III(RNA polymerase III)转录U6小核RNA(U6 snRNA)。
U6 snRNA是一种小分子RNA,参与到剪接反应中,帮助形成成熟的mRNA分子。
U6启动子的作用是在细胞中产生足够的U6 snRNA,以维持正常的剪接反应。
U6启动子的原理是通过与转录因子相互作用,使得RNA polymerase III能够结合到启动子上,并开始转录过程。
转录因子是一类蛋白质,能够与启动子序列特定的DNA序列结合,从而招募RNA polymerase III到启动子上。
一旦RNA polymerase III结合到U6启动子上,它会开始沿着DNA模板链合成U6 snRNA的互补RNA链。
这个过程会一直进行,直到RNA polymerase III到达终止信号,停止转录。
总之,U6启动子通过与转录因子相互作用,使得RNA polymerase III能够结合到启动子上,并开始转录U6 snRNA。
这样就能够产生足够的U6 snRNA,参与到剪接反应中,维持
正常的基因表达。
一、名词解释:转录:是指以DNA为模板,在依赖于DNA的RNA聚和酶催化下,以4中NTP(ATP、CTP、GTP和UTP)为原料,合成RNA的过程。
转录单位 (transcription unit):从启动子到终止子的序列 (转录起始点)。
模板链(template strand):又称反义链, 指与转录物互补的DNA链(极性方向3’→5’)。
编码链:又称有义链, 指不作模板的DNA单链(极性方向5’→3’)。
hnRNA:核内不均一RNA,是存在于真核细胞核中的不稳定,大小不均一的一组高分子RNA的总称。
转录的极性:转录的效率与转录单位的位置有关。
转录起始:RNA聚合酶与DNA转录启动子结合形成有功能的转录起始复合物的过程。
启动子(Promoters):指DNA分子上被RNA聚合酶、转录调节因子等识别并结合形成转录起始复合物的区域。
核心启动子:RNA聚合酶能够直接识别并结合的启动子。
RNA聚合酶:是催化以DNA为模板(template)、三磷酸核糖核苷为底物、通过磷酸二酯键而聚合的合成RNA的酶。
C端结构域(CTD):RNApolⅡ的大亚基中有 C 末端结构域。
CTD中含一保守氨基酸序列的多个重复Tyr-Ser p-Pro-Thr p-Ser p-Pro-Ser p C端重复七肽。
沉默子(silencer):沉默子能够同反式因子结合从而阻断增强子及反式激活因子作用并最终抑制该基因的转录活性的真核基因中的一种特殊的序列。
增强子(enhancer):是一类正调控元件,能够从转录起始位点的上游或下游数千个碱基处来激活转录。
绝缘子(insulater):阻断增强子或沉默子的DNA序列。
上游:转录起点上游的序列,是调控区,与转录的方向相反。
下游:转录起点下游的区域,是编码区,与转录的方向一致。
转录起点:+1位点,RNA聚合酶的转录起始位点,起始NTP多为ATP或GTP。
转录泡:在转录时RNA聚合酶Ⅱ(RNAPⅡ)与DNA模板结合,会形成一个泡状结构,成为转录泡。
CRISPR/Cas9-based genome editing technologyA robust CRISPR/Cas9 vector system for multiplex targeting ofgenomic sites in monocot and dicot plants亚热带农业生物资源保护与利用国家重点实验室华南农业大学生命科学学院刘耀光课题组(**************.cn )1. pYLCRISPR/Cas9多靶点载体介绍CRISPR/Cas9是新近发展的基因组编辑技术(图1)。
CRISPR/Cas9切割靶序列仅需要single-guide RNA (sgRNA)以及由sgRNA 引导的Cas9蛋白,比锌指核酸酶(ZFNs ),TALENs 更加简便,高效,因而成为基因组编辑工具的首选。
我们利用CRISPR/Cas9技术可方便地进行多重靶向的特征,构建了一套用于单子叶和双子叶植物的多靶点CRISPR/Cas9基因打靶载体系统。
本套载体将Cas9蛋白表达盒整合到双元载体上,用于装载多个sgRNA 表达盒的多克隆位点Bsa I 位于靠近双元载体RB 位置。
sgRNA 表达盒元件设在中间质粒载体上,利用酶切连接和PCR 方法拼装好,再利用Golden Gate 或Gibson Assembly 克隆方法组装到双元载体上。
5’ NNNNNNNNNNNN GNNNNNNNNNNNNNNNNNNN Target SitePAM NNNNNNNNNNNN-3’3’ NNNNNNNNNNNN NCCNNNNNNNNNNNN-5’NNNNNNNNNNNNNNNNNNNN 5’-G/A NNNNNNNNNNNNNNNNNNNN GUUUUAGAGCUAG A A CGAUA GAAAACUAUUGCCUGAUCGGAAUAAAAUU Cas9nuclease Cleavage site genomesequenceRuvC-like domain HNH domain CUUGAAAAAGUGGCACCGA G CGUGGCU UUUUU-3’NGGFigure 1. A working model of the CRISPR/Cas9 system. The Cas9-sgRNA complex locates to the target site to cleave the DNA to produce double strand break (DSB).2.CRISPR/Cas9载体与sgRNA载体图谱2.1CRISPR/Cas9双元载体本套载体系统的双元载体骨架为pCAMBIA-1300 (ACCESSION: AF234296),Cas9p为本实验室设计合成的植物优化密码子基因,它模拟了禾本科植物基因具有5’端GC含量较高的特征(Figure 2)。