网上生物信息学教程
- 格式:docx
- 大小:21.13 KB
- 文档页数:9

生物信息学软件的使用教程与数据分析生物信息学是一门结合生物学和计算机科学的学科,通过利用计算机科学和统计学的方法来研究生物学中的大规模生物分子数据。
在生物研究中,大量的生物信息数据被产生,如基因组测序数据、蛋白质结构数据、转录组数据等,这些数据的分析对于理解生物过程和疾病发生机制至关重要。
生物信息学软件是专门用于处理和分析这些生物信息数据的工具。
本文将介绍一些常见的生物信息学软件的使用教程和数据分析方法。
1. BLAST(Basic Local Alignment Search Tool):BLAST是最常用的序列比对工具之一,用于在数据库中寻找类似序列或通过序列相似性比对两个或多个序列。
BLAST可以用于查找一个给定的序列是否存在于一个已知的数据库中,也可用于快速比较两个序列的相似性,并寻找具有高度相似性的区域。
在使用BLAST时,首先需要选择合适的数据库,然后输入待比对的序列,设置相似性阈值和其他参数,最后运行BLAST程序并分析结果。
2. NCBI(National Center for Biotechnology Information)工具:NCBI提供了许多生物信息学工具,如BLAST、Entrez等。
Entrez是一个可检索多种生物信息学数据库的工具,包括GenBank(存储核酸序列)、PubMed(存储科学文献摘要与索引)、Protein(蛋白质序列数据库)等。
通过使用NCBI提供的工具,可以比对和分析大量的生物序列和相关的生物信息。
使用NCBI工具时,可以通过访问NCBI网站或使用命令行工具来查询和分析数据。
3. R和Bioconductor:R是一种用于统计计算和数据可视化的自由软件环境,而Bioconductor是一个在R环境中为生物学研究提供的开源生物信息学软件包。
R和Bioconductor提供了丰富的统计和生物信息学分析方法,可用于分析基因表达数据、基因组测序数据、蛋白质结构数据等。

生物信息学自学顺序一、了解生物信息学的基本概念和应用领域生物信息学是将计算机科学、统计学和生物学知识相结合,用于处理和分析生物学数据的交叉学科。
它在基因组学、蛋白质组学、转录组学等领域发挥着重要作用。
二、学习生物学基础知识生物信息学的理论基础是生物学知识,因此在开始学习生物信息学之前,需要掌握一些基本的生物学概念和知识,包括细胞结构与功能、遗传学原理、基因表达调控等内容。
三、学习计算机科学和编程基础知识生物信息学需要运用计算机科学和编程技术进行数据处理和分析,因此学习计算机科学和编程基础知识是必要的。
包括学习编程语言(如Python、R)、算法与数据结构、数据库管理等内容。
四、学习生物信息学常用工具和软件生物信息学常用的工具和软件包括BLAST、NCBI、Bioconductor、UCSC Genome Browser等,学习使用这些工具和软件可以帮助我们进行基因序列比对、基因功能注释、基因表达分析等。
五、学习生物信息学常用数据库和资源生物信息学的研究依赖于大量的生物学数据库和资源,包括基因组数据库(如GenBank、Ensembl)、蛋白质数据库(如UniProt)、代谢通路数据库(如KEGG)等。
了解并熟练使用这些数据库和资源对于生物信息学的学习和研究非常重要。
六、学习基因组学和序列分析基因组学是生物信息学的重要分支领域,通过学习基因组学的基本概念和方法,可以了解基因组的组成、结构和功能。
同时,学习序列分析的方法和技巧,可以进行DNA、RNA和蛋白质序列的比对、搜索、注释等分析。
七、学习蛋白质结构预测和分析蛋白质结构预测是生物信息学中的重要研究方向,通过学习蛋白质结构预测的方法和工具,可以对蛋白质的结构进行模拟和预测。
此外,学习蛋白质结构的功能和相互作用分析,可以揭示蛋白质的生物学功能和分子机制。
八、学习转录组学和表达谱分析转录组学研究基因在特定条件下的表达情况,通过学习转录组学的方法和技术,可以了解基因表达的调控机制和影响因素。

生物信息学技术的使用教程与基因分析生物信息学是一门综合性学科,它将计算机科学、数学和生物学相结合,研究生物序列氨基酸序列、DNA序列和RNA序列等大规模数据的分析和解读。
生物信息学技术在基因分析、基因组学、蛋白质组学等领域发挥着重要作用。
本文将介绍生物信息学技术的使用教程和基因分析的基本方法。
一、生物信息学技术的使用教程1. 数据采集与预处理在进行生物信息学分析之前,首先需要采集所需的数据。
数据的来源可以包括公共数据库(如NCBI、EBI等)、实验室测序数据和文献中的相关数据等。
采集到的数据往往需要进行预处理,包括数据清洗、去除低质量序列、低质量碱基等,以保证后续分析的准确性和可靠性。
2. 序列比对与注释序列比对是将所研究的序列与已知的序列进行比较,找出两者之间的相似性和差异性。
常用的比对方法有BLAST、BWA、Bowtie等。
注释则是对比对结果进行解读,给出序列的功能、结构和表达等信息。
注释工具包括NCBI的GenBank、Ensembl、GeneCards等。
3. 基因组组装与注释基因组组装是将高通量测序生成的序列数据进行拼接,恢复出物种的全基因组结构。
基因组注释是对组装得到的基因组序列进行功能注释和结构注释。
组装工具包括SOAPdenovo、Velvet、ABySS等,注释工具包括Glimmer、RepeatMasker、GeneMark、Augustus等。
4. 转录组分析与差异表达基因筛选转录组分析是对一种生物体中所有基因的转录活动进行定性和定量研究。
差异表达基因筛选是找出在不同样品之间表达量差异显著的基因。
常用的转录组分析工具包括Cufflinks、DESeq、edgeR等,差异表达基因筛选工具包括Limma、SAM、DEGseq等。
二、基因分析的方法与应用1. 基因结构预测基因结构预测是预测DNA序列中基因的位置和边界,并预测该基因编码的蛋白质的结构和功能。
常用的工具有Genscan、Augustus、GENSCAN 等。

生物信息学分析工具的使用教程导言:在生物学领域中,随着高通量测序技术的快速发展,生物信息学分析工具的应用变得越来越重要。
这些工具能够帮助研究人员进行基因组、转录组、蛋白质组等大规模数据的分析和解释。
本文将为您介绍几种常用的生物信息学工具,并提供详细的使用指南。
一、BLAST(基因序列比对工具)BLAST(Basic Local Alignment Search Tool)是最常用的生物信息学工具之一,用于比对基因或蛋白质序列中的相似性。
以下是使用BLAST的步骤:1. 打开NCBI网站的BLAST页面,并选择适当的BLAST程序(如BLASTn、BLASTp等)。
2. 将查询序列粘贴到"Enter Query Sequence"框中,或者上传一个FASTA格式的文件。
3. 选择适当的数据库,如"nr"(非冗余序列数据库)或"refseq_rna"(已注释的RNA序列数据库)。
4. 设置相似性阈值、期望值和其他参数。
5. 点击"BLAST"按钮开始比对。
6. 结果页面会显示比对结果的列表和详细信息,包括匹配上的序列、相似性得分等。
二、DESeq2(差异表达基因分析工具)DESeq2是一种用于差异表达基因分析的R包。
以下是使用DESeq2的步骤:1. 安装R语言和DESeq2包。
2. 将基因表达矩阵导入R环境中,并进行预处理(如去除低表达基因)。
3. 根据实验设计设置条件和组别。
4. 进行差异分析,计算基因的表达差异和显著性。
5. 可视化差异表达基因的结果,如绘制散点图、MA图、热图等。
三、GSEA(基因集富集分析工具)GSEA(Gene Set Enrichment Analysis)是一种基于基因集的富集分析方法,用于识别与特定性状或实验条件相关的生物学功能。
以下是使用GSEA的步骤:1. 准备基因表达矩阵和相关的分组信息。

生物信息学分析平台的使用教程与数据挖掘生物信息学是将信息科学和生物学相结合的交叉学科领域,它利用计算机和统计学等工具来管理、解释和分析生物学数据。
生物信息学分析平台是为帮助生物学家处理和分析大规模生物学数据而设计的软件工具。
本文将介绍生物信息学分析平台的使用教程,并探讨如何利用数据挖掘技术在生物学研究中发现新的知识。
一、生物信息学分析平台的基本功能生物信息学分析平台通常提供一系列工具和算法,用于处理和分析生物学数据,包括测序数据、基因表达数据、蛋白质结构数据等。
常见的生物信息学分析平台有NCBI、UCSC、Ensembl等。
1. 数据查询和检索:生物信息学分析平台允许用户通过关键词、ID号或其他属性来查询和检索生物学数据库中的数据。
用户可以根据自己的研究目的来选择合适的数据库,如基因组数据库、蛋白质数据库等。
2. 数据处理和分析:生物信息学分析平台提供各种工具和算法,用于处理和分析生物学数据。
常见的功能包括质量控制、序列比对、基因表达定量、蛋白质互作预测等。
用户可以根据自己的研究问题选择合适的工具和算法进行分析。
3. 数据可视化和结果解释:生物信息学分析平台通常提供数据可视化工具,用于将分析结果以图表或图形的形式展示出来。
这有助于用户理解和解释分析结果,并从中提取有意义的信息。
二、生物信息学分析平台的使用教程以下是一般性的生物信息学分析平台使用教程,具体操作可能因平台而异,仅供参考。
1. 注册账户和登录平台:生物信息学分析平台通常需要用户注册账户后进行登录,以便保存用户的分析结果和设置。
2. 数据查询和检索:在平台的搜索栏中输入关键词、ID号或其他属性,选择合适的数据库,点击搜索按钮进行查询和检索。
3. 数据下载和导入:根据查询结果选择需要的数据,并下载到本地计算机。
下载的文件可能是文本文件、FASTA格式文件等。
将数据导入到生物信息学分析平台中,准备进行后续的数据处理和分析。
4. 数据质量控制:对导入的数据进行质量控制,去除低质量的序列或数据点。

斑马鱼中酪氨酸激酶JAK2b 的生物信息操作步骤:一.检索序列斑马鱼是模式生物,酪氨酸激酶是信号通路中的重要蛋白,而且研究的也比较清楚,所以准备酪氨酸激酶的基因,通过NCBI 找到斑马鱼中JAK 2b 的序列信息。
在NCBI 首页/进行搜索,输入protein tyrosine kinase 搜索,查找到斑马鱼中的酪氨酸激酶JAK2b 的蛋白序列信息,截图如下:同时根据此页面的信息,获得其基因的序列号,查得其mRNA序列信息如下:得到该蛋白的氨基酸序列为(得到mRNA序列过长,不在这里予以显示):MDMETA VCEPHYQNGDVHHEDPCPKQPTVLKIHLYHSSRSDADSSPICYPPGEYITEQLIIHAAKESGVSP VYCSLFGL YKEDEA VWLSPNHVLHLDETANESLLFRMRYYFPGWYSCGTSRAYRYGVTKGSDSPVLDD YVMSYLFAQWRSDFVNGWVKLSGSHENQEECLGMA VLDMMRTAKEKDCSPLDIYNDISYKSFLPKDM RERIQDYHFVTRKRIRYRFRKVVQQLSQCRVTARDLKLKYLISLESLDNGFYTERFQVKEPSTEQVTIVVS ADRGIQWCHERHKDTQSEDLLQTYCDFAEVVDISIRQASKDGTNMNRIVSIDRQDGTPLELVFSTSMQA LSFVSLIDGYYRLTTDAHHYLCKDVAPPYLLEAIESYCHGPISMDFAISKLRKSGNQKGLFVLRCSPKDY NKYFLTLAFGGYGNVEYKHCLITRSESGNYNLSGTKKSFRSLQDLLKCYQKETVKSDGIVFQFSKCCSPR PKEKSSLLVCRSHRGLEVPLSSSLQRHNISQMVFHKIKKEDLELGESQGQGTFTKIFKGIRKEQGDYGETH KTEVIVKVLDKAHRNYSESFFEAASMMSQLTHKHLVLTYGICVCGDENIMVQEYVKFGSLDTYLKKNK SSVSVNILWKLEVAKQLAWAML YLEEKSLAHGNVCAKNILLIREEDRALGNTPFIKLSDPGISITVLPREIL VERIPWVPPECILDPKNLSLATDKWSFGTTLWEICSGGEQPLANMDNSKKHLFYENHHQLPAPKWTELA NLINSCMDYEPTFRPSFKAIIRDLNSLFCPDYEIVKESDIMPSRAAASIFNTGTFKNNEPVQFEERHLIFLQQ LGKGNFGSVEMCRYDPLQDNTGEVV A VKKLQHSTTEHIRDFEREIEILKSLQHENIVKYKGVCYGAGRRNLRLVMEYLPYGSLRDYLNKNRDRIDHQKLVHYASQICKGMEYLATKRYIHRDLATRNILVESECRVKIG DFGLTKVLPQDKEYYKVKEPGESPIFWHAPESLTESKFSVASDVWSFGVVL YELFTYSDKLCSPPTVFLS MVGGDKQGQTIVYHLIELLKRGNRLPQPMGCPTEMFEIMQECWDNDPSLRPNFKELALRVDLIRDSSDA DRYTQVPE二. 同源比对用NCBI 的blastp 工具,对该蛋白进行比对。


生物信息学技术的使用教程与分析步骤解析生物信息学是生物学领域的重要分支,它应用于基因组学、转录组学、蛋白质组学等领域的研究中。
在当前的大数据时代,生物信息学技术的发展为解决生物科学研究中的复杂问题提供了便利和支持。
本文将为您介绍生物信息学技术的使用教程与分析步骤解析。
一、生物信息学技术的使用教程生物信息学技术的使用过程包括数据获取、数据处理和数据分析等步骤。
下面将详细介绍这些步骤的内容及相关工具的选择。
1. 数据获取生物信息学研究常用的数据主要来源于公共数据库,如NCBI、ENA、GenBank等。
在获取数据时,需要根据研究需求选择合适的数据库,并确定所需的数据类型,如基因组、转录组或蛋白质组等。
此外,需要掌握相应的搜索和下载技巧,如使用关键词、过滤条件和下载工具等。
2. 数据处理数据处理是将原始数据转化为可分析的格式,通常包括数据清洗、格式转换和数据预处理等步骤。
为了提高数据质量,需要对原始数据进行去噪、去冗余、去重复等处理,并将数据转换为常用的格式,如FASTA、GFF、BAM等。
此外,还需要进行数据预处理,如基因组组装、序列比对和变异检测等。
3. 数据分析数据分析是生物信息学研究的核心内容,主要涉及序列分析、结构分析和功能分析等方面。
在序列分析方面,常见的技术包括序列比对、序列聚类和序列比较等。
在结构分析方面,可以利用已知的结构数据进行比对和模拟,以预测蛋白质的结构和功能。
而在功能分析方面,可以运用基于GO注释的功能富集分析和基于KEGG数据库的代谢通路分析等方法来揭示基因和蛋白质的功能。
二、分析步骤解析在进行生物信息学研究时,需要经过一系列的分析步骤来获取有意义的结果。
下面将介绍常见的分析步骤及其解析。
1. 基因组组装基因组组装是将高通量测序生成的reads拼接成完整的基因组序列的过程。
该步骤的关键是选择合适的组装工具,如SOAPdenovo、Velvet和SPAdes等,并根据测序产出的数据类型,如illumina、PacBio或OXFORD NANOPORE等来制定合适的参数设置。

生物信息学技术的使用教程生物信息学技术是一门综合性学科,将计算机科学、统计学和生物学等多个学科的研究方法和技术应用于生物研究中。
它的出现和发展,使得科学家们能够更好地处理和分析海量的生物数据,从而推动了生物科学的快速发展。
本文将介绍几个常用的生物信息学技术,并提供相应的使用教程。
1. 基因序列分析技术基因序列分析是生物信息学中最基础和常用的技术之一。
它可以帮助科学家理解基因的结构和功能。
常用的基因序列分析方法有:1.1 BLAST(Basic Local Alignment Search Tool)分析BLAST是一种用于比对和比较核酸或蛋白质序列的工具。
它可以根据已知序列找到与之相似的新序列,从而预测新序列的功能和结构。
BLAST分析的基本步骤包括:选择合适的数据库和比对算法、设置参数、上传待比对的序列文件并运行BLAST程序、分析结果并进行解释。
详细的BLAST教程可以参考NCBI官方网站提供的帮助文档。
1.2 基因组装基因组装是将大量碎片化的DNA序列重新组装成完整的基因组或染色体的过程。
常用的基因组装软件有SOAPdenovo、Velvet和SPAdes等。
进行基因组装时的主要步骤包括:去除低质量序列、拼接碎片、填补插入缺失、纠错错误序列等。
具体操作可以参考相关软件的使用手册或文档。
2. 蛋白质结构预测技术蛋白质结构预测是指通过计算方法预测蛋白质的三维结构。
蛋白质的结构对于了解其功能和与其他生物分子的相互作用非常重要。
下面介绍两种常用的蛋白质结构预测技术:2.1 蛋白质序列比对蛋白质序列比对是通过比对已知结构的蛋白质序列与未知结构的蛋白质序列来推断其结构。
常用的蛋白质序列比对工具有Clustal Omega和MAFFT等。
使用这些工具进行蛋白质序列比对的步骤一般包括:输入蛋白质序列、选择比对算法、运行程序,最后分析和解释比对结果。
2.2 蛋白质结构模建蛋白质结构模建是通过计算方法构建未知结构的蛋白质的三维模型。

生物信息入门课程生物信息学是生物学与计算机科学的交叉学科,它利用计算机技术和数学方法来解决生物学中的问题,例如基因组序列分析、蛋白质结构预测、基因表达分析等。
以下是一个入门生物信息学的课程大纲,涵盖了基本的概念、工具和技术:第一部分:生物学基础细胞生物学基础细胞结构与功能基因表达调控分子生物学基础DNA、RNA、蛋白质的结构和功能基因组和蛋白质组的概念第二部分:生物信息学基础生物信息学概述生物信息学的定义和应用生物信息学在生命科学研究中的角色数据库和资源基因组数据库:GenBank、ENSEMBL、UCSC Genome Browser等蛋白质数据库:UniProt、PDB等生物信息学工具和软件:BLAST、FASTA、EMBOSS等第三部分:序列分析DNA序列分析序列比对:全局比对、局部比对寻找开放阅读框(ORF)和基因预测蛋白质序列分析蛋白质序列比对和分类蛋白质结构预测和功能预测第四部分:基因组学和转录组学基因组学基因组结构和组织基因组序列比较和进化分析转录组学mRNA测序技术(RNA-Seq)概述转录组数据分析流程:数据预处理、差异表达分析、功能注释等第五部分:蛋白质组学和结构生物信息学蛋白质组学蛋白质组学概述蛋白质相互作用和功能预测结构生物信息学蛋白质结构预测方法蛋白质结构分析和模拟第六部分:实验设计和数据可视化实验设计生物信息学实验设计的原则和方法实验设计中的统计学原理数据可视化生物信息学数据可视化工具和技术数据可视化在生物信息学中的应用案例第七部分:实践项目与案例研究实践项目学生可以选择一个生物信息学项目进行实践,例如基因序列分析、转录组数据分析等案例研究针对真实生物学研究问题的案例研究,学生通过解决问题来应用所学的生物信息学知识和技能这样的课程可以帮助学生建立起对生物信息学的基本理解,并掌握一些常用的生物信息学工具和技术,为进一步深入学习和研究打下良好的基础。
同时,通过实践项目和案例研究,学生可以将所学知识应用到实际问题中,提升解决生物学问题的能力。
科研实验中的生物信息学工具使用教程生物信息学是将数学、统计学和计算机科学应用于生物学研究的交叉学科。
在现代科研中,生物信息学工具已经成为了生物学实验和研究的重要组成部分。
本文将介绍几种常用的生物信息学工具,并提供详细的使用教程。
1. BLAST(Basic Local Alignment Search Tool)BLAST是生物信息学领域中最常见的工具之一,用于在数据库中快速比较DNA或蛋白质序列的相似性。
以下是使用BLAST进行基本比对的步骤:(1)打开NCBI网站,并进入BLAST页面。
(2)选择“nucleotide”或“protein”,取决于你要比对的序列类型。
(3)复制粘贴或上传你要比对的序列。
(4)选择合适的数据库进行搜索,如“nr”(非冗余数据库)。
(5)点击“BLAST”按钮,等待搜索结果。
BLAST会为你提供一个比对报告,其中包含了与你的查询序列相似的序列列表。
2. EMBOSS(European Molecular Biology Open Software Suite)EMBOSS是一个开源的生物信息学软件包,提供了一系列用于序列分析和比对的工具。
以下是使用EMBOSS进行序列分析的步骤:(1)打开EMBOSS软件(可以下载并安装在你的计算机上)。
(2)选择合适的工具,如“water”(Smith-Waterman比对算法)。
(3)输入查询序列和数据库序列。
(4)设置相关参数,如匹配分数和距离惩罚。
(5)点击“Run”按钮,等待分析结果。
EMBOSS将为你提供一个比对报告,并给出一些统计数据,如匹配分数和最佳比对。
3. R/BioconductorR是一种统计软件和编程语言,Bioconductor是R语言的一个生物信息学扩展包,提供了丰富的生物信息学工具和分析方法。
以下是使用R/Bioconductor进行基因表达分析的步骤:(1)打开R软件并加载Bioconductor包。
生物信息学软件的基本使用方法介绍生物信息学是研究生物学中大规模数据的获取、存储、管理、分析和解释的学科。
为了能够有效地处理这些复杂的生物数据,生物信息学研究者使用了许多专门设计的软件工具。
本文将介绍几种常见的生物信息学软件,并提供基本的使用方法。
1. BLAST(Basic Local Alignment Search Tool):BLAST是一种用于基因序列比对和相似性搜索的软件工具。
它能够找到在数据库中与输入序列相似的序列,并计算它们之间的相似度分数。
使用BLAST时,首先需要选择要比对的数据库,如NCBI的nr数据库。
然后,将待比对的序列输入到BLAST中,并选择合适的算法和参数,最后点击运行按钮即可得到比对结果。
2. ClustalW:ClustalW是一种常用的多序列比对软件。
它能够将多个序列对齐,并生成比对结果。
使用ClustalW 时,首先需要输入要比对的序列。
可以通过手动输入、从文件中导入或从数据库中获取序列。
然后,选择合适的比对算法和参数,并点击运行按钮。
在比对结果中,会显示相似性分数矩阵和序列的对齐信息。
3. FASTA:FASTA是一种用于快速比对和搜索序列相似性的工具。
它使用一种快速的搜索算法,能够在大型数据库中快速找到与输入序列相似的序列。
使用FASTA时,需要将待比对的序列输入到软件中,并选择匹配的算法和搜索参数。
运行后,软件会生成相似序列的列表和相似性评分。
4. R:R是一种统计分析软件,也被广泛用于生物信息学领域。
它提供了丰富的函数和库供生物信息学研究者使用,用于数据处理、统计分析和可视化。
使用R时,可以通过命令行或脚本编写代码来执行各种操作。
例如,可以使用R中的Bioconductor库进行基因表达数据的分析和可视化。
5. IGV(Integrative Genomics Viewer):IGV是一种用于基因组数据可视化的软件工具。
它能够显示基因组位置上的测序深度、SNP、CNV等信息,并支持交互式操作和注释查看。
生物信息学分析工具的操作指南与使用技巧近年来,随着生物学研究的向深度学习和大数据方向转变,生物信息学分析工具越来越重要。
这些工具能够处理和解读庞大的生物信息数据,从而提供对基因、蛋白质和其他生物分子功能的深入了解。
为了帮助研究者更好地应用这些工具,本文将提供生物信息学分析工具的操作指南与使用技巧。
一、 BLASTBLAST(Basic Local Alignment Search Tool)是最常用的生物信息学工具之一,用于比对基因或蛋白质序列并寻找相似性。
以下是使用BLAST的操作指南:1. 登录NCBI(National Center for Biotechnology Information)网站,选择"BLAST"选项卡。
2. 选择合适的BLAST程序,如nucleotide BLAST(用于比对核苷酸序列)或protein BLAST(用于比对蛋白质序列)。
3. 输入待比对的序列或上传序列文件。
4. 选择适当的数据库进行比对。
例如,对于人类基因,可以选择"Human genome"数据库。
5. 调整BLAST参数,如期望阈值(E-value)和比对长度,以优化结果。
6. 提交任务并等待结果。
BLAST将返回比对结果和相似性分数。
使用技巧:- 选择正确的数据库,以确保比对结果具有生物学相关性。
- 调整参数以满足特定的研究需求,如提高灵敏度或选择严格的相似性阈值。
- 分析比对结果时,关注较高的BLAST分数和较低的E-value,以确定最相关的序列。
二、DNA序列编辑器DNA序列编辑器是生物信息学研究中常用的工具,用于编辑、操作和分析DNA序列。
以下是使用DNA序列编辑器的操作指南:1. 下载和安装合适的DNA序列编辑器,如ApE(A plasmid Editor)或SnapGene。
2. 打开编辑器并创建新项目。
3. 在序列窗口中输入或粘贴DNA序列。
生物信息学软件使用指南第一章:生物信息学简介在进入生物信息学软件的具体使用指南之前,我们先来简要介绍一下生物信息学的概念和应用领域。
生物信息学是通过计算机科学和统计学的方法,对生物学数据进行收集、存储、管理、分析和解释的学科。
其应用领域包括基因组学、蛋白质组学、转录组学和代谢组学等。
第二章:常用生物信息学软件1. BLAST: BLAST(Basic Local Alignment Search Tool)是一种常用的序列比对工具,可以用于比对已知序列和未知序列之间的相似性。
使用BLAST,可以将一个未知序列与已知数据库中的序列进行比对,并找到最相关的序列。
2. CLC Genomics Workbench: CLC Genomics Workbench是一种强大的基因组信息分析软件,可用于测序数据处理、基因组组装、蛋白质结构预测等多项分析任务。
它提供了丰富的工具和算法,使用户能够快速、准确地分析和解释生物学数据。
3. R: R是一种广泛应用于生物信息学和统计学领域的编程语言和环境。
它提供了丰富的数据处理、统计分析和可视化功能,可以用于从基因表达数据、蛋白质互作网数据等大规模数据中提取有用信息。
第三章:生物序列分析软件1. SeqKit: SeqKit是一款简单易用的生物序列处理工具,可用于处理常见的DNA、RNA和蛋白质序列。
它提供了丰富的序列分析和格式转换功能,如序列比对、物种分类、碱基组成分析等。
2. MEME Suite: MEME Suite是一套用于序列模因分析的工具集合,可以用于鉴定和分析DNA、RNA和蛋白质序列中的隐含模式。
它提供了多个模因分析算法,并支持可视化显示结果。
3. HMMER: HMMER是一种用于序列比对和搜寻的软件包,支持隐马尔可夫模型(Hidden Markov Model)的应用。
它可以进行蛋白质序列比对、域搜索、蛋白质结构预测等多项功能。
第四章:结构生物信息学软件1. PyMOL: PyMOL是一款用于分析和可视化分子结构的软件。
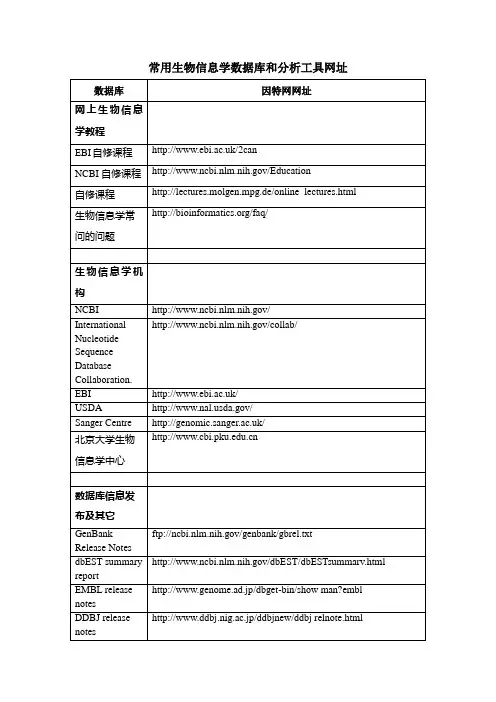
网上生物信息学教程EMBL biocomputing tutorials/Embnetut/Gcg/index.html Plant genome dababase tutorial/pgdic生物信息学机NCBI/International Nucleotide Sequence Database Collaboration. /collab/EBI/USDA/Sanger Centre/北京大学生物信息学中心数据库信息发布及其它GenBank Release Notesftp:///genbank/gbrel.txtdbEST summary report/dbEST/dbESTsummarv.html EMBL release noteshttp://www.bio.unizh.ch/db/docu.html?data=emrel Eukaryotic promoter database release noteshttp://www.genome.ad.jp/dbget/dbget2.htmlKEGG release noteshttp://www.genome.jp/kegg/docs/relnote.html核苷酸数据库GenBank/dbEST/dbEST/index.htmldbSTS/dbSTS/index.htmldbGSS/dbGSS/index.htmlGenome (NCBI)/entrez/query.fcgi?db=Genome dbSNP/SNP/HTGS/HTGS/UniGene/UniGene/EMBL核苷酸数据库/emblGenome (EBI)/genomes/向EMBL数据库提交序列/embl/Submission/webin.htmlDDBJhttp://www.ddbj.nig.ac.jp/启动子数据库Eukaryotic promoter databasehttp://www.epd.isb-sib.ch/转录因子数据库FRANSFAC/pub/databases.htmlTFD基因分类数据库Gene Ontology (GO)蛋白质数据库SWISS-PROT或TrEMBL/swissprot/http://www.expasy.ch/sprot/PIR/pir/PRFhttp://www.prf.or.jp/PDBSTRhttp://www.genome.ad.jp/dbget-bin/www_bfind?pdbstr Prositehttp://www.expasy.ch/sprot/prosite.html结构数据库PDB/pdbNDB/NDB/ndb.html/DNA-Binding Protein Database/NDB/structure-finder/dnabind/index.html NMR Nucleic Acids Database/NDB/structure-finder/nmr/index.html Protein Plus Database/NDB/structure-finder/protein/index.html Swiss 3Dimagehttp://www.expasy.ch/sw3d/SCOP/scop/CATH/bsm/cath/酶、代谢和调控路径数据库KEGGhttp://www.genome.ad.jp/kegg/Enzyme Nomenclature Databasehttp://expasy.hcuge.ch/sprot/enzyme.htmlProtein Kinase Resource (PKR)/kinases/LIGANDhttp://www.genome.ad.jp/dbget/ligand.htmlWIT/WIT/EcoCyc/ecocyc/UM-BBD/umbbd/多种代谢路径数据库/stc-95/ResTools/biotools/biotools8.html基因调控路径数据库(TRANSPATH)http://transfac.gbf.de基因组数据库禾本科比较基因组GrainGeneBotanical Databases/botanicaldatabase.htmBotanical Data/calflora/batanical.html日本水稻基因组(RGP)http://rgp.dna.affrc.go.jp水稻物理图谱/projects/rice/fpc华大水稻基因组框架图欧洲水稻测序(第12染色体)s.frMaize genomeBarley genome/Research/barley/nabgmp.htm Forage grasses genomes//Topics/Species/Grasses/ Triticum genomes/index.shtmlArabidopsis genomeSoyBasehttp://129.186.26.94Alfalfa genomeCotton genomeGlycine max genome/PlantGDB/glycine_max.html /PlantGDBC. elegans genome藻类(Chlamydomonas)基因组/chlamy_genome粘菌(Dictyostelium)基因组Animal genomes (ArkDB)FlyBase/Mouse Genome Informatics/Saccharomyces Genome Database/多种基因组数据库/GenomeWeb文献数据库PubMed/PubMed/OMIM/Omim/Agricola/ag98/Rice Genetics Newsletter/newsletters/rice_genetics Proceedings of the National Academy of Sciences USA (PNAS) 关键词为基础的数据库检索Entrez/Entrez/Entrez Nucleotide Sequence Search/Entrez/nucleotide.htmlEntrez Protein Sequence Search/Entrez/protein.htmlSequence Retrieval System, Singapore.sg:80/srs5/Sequence Retrieval System, US:80/srs/srscSequence Retrieval System, UK/GetEntry Nucleotide & Protein Sequence Searchhttp://ftp2.ddbj.nig.ac.jp:8000/getstart-e.htmlDatabase Search with Key Wordshttp://ftp2.ddbj.nig.ac.jp:8080/dbsearch-e-new.htmlDBGET/LinkDBhttp://www.genome.ad.jp/dbget/dbget2.html序列为基础的数据库检索BLAST/BLAST/FASTA/fasta33/index.htmlBLITZ/bicsw/SSearchrs.fr/bin/ssearch-guess.cgiElectronic PCR/STS/Proteome analysis/proteome/多序列分析Clustal multiple sequence alignment:9331/multi-align/Options/clustalw. html BCM:9331/multi-align/multi-align.htmlEBI ClustalW analysis系谱分析PAUP/PAUP/EBI ClustalW analysisGCG package/PHYLIP/phylip.htmlMEGA/METREE/imegHennig86/~mes/hennig/software.htmlGAMBIT/mcdbio/Faculty/Lake/Research/Programs/ MacClade/macclade/macclade.html Phylogenetic analysis/stc-95/ResTools/biotools/biotools2.html ClustalXftp://ftp-igbmc.u-strasbg.fr/pub/ClustalXMEGA基因结构预测分析GENSCAN/GENSCAN.htmlhttp://bioweb.pasteur.fr/seqanal/interfaces/genscan-simple.html http://bioweb.pasteur.frGeneFinder/gf/gf.shtml/nucleo.htmlGene Feature Searches:9331/Grail/Grail-1.3/GrailEXP/grailexp/GeneMark/GeneMark/eukhmm.cgi/GeneMark/hmmchoice.html Veil/labs/compbio/veil.htmlAAT/aat.htmlGENEIDhttp://www.imim.es/GeneIdentification/Geneid/geneid_input.html Genlang/~sdong/genlang_home.html GeneParser/~eesnyder/GeneParser.htmlGlimmer/labs/compbio/glimmer.htmlMZEF/genefinderProcrustes/software/procrustes/基因分类GO Annotator/gofigure蛋白质结构预测分析Expasyhttp://www.expasy.ch/CBShttp://www.cbs.dtu.dkPredicting protein secondary structure:9331/pssprediction/pssp.html Predicting protein 3D Structureshttp://dove.embl-heidelberg.de/3D/Predicting protein structures:9331/seq-search/struc-predict.html其它分析工具和软件Putative DNA Sequencing Errors Checkhttp://www.bork.embl-heidelberg.de/Frame/MatInspectorhttp://www.gsf.de/cgi-bin/matsearch.plFastMhttp://www.gsf.de/cgi-bin/fastm.plWeb Signal Scanhttp://www.dna.affrc.go.jp/htdocs/sigscan/signal.htmlBCM Search Launcher:9331/seq-util/seq-util.html Webcutter/cutter/cut2.htmlTranslate DNA to proteinhttp://www.expasy.ch/tools/dna.htmlABIMhttp://www-biol.univ-mrs.fr/english/logligne.htmlsequence motifs:Pfam/Pfam//ProDomhttp://protein.toulouse.inra.fr/prodom.htmlPRINTS/bsm/dbbrowser/PRINTS/其它多种数据库、分析工具和生物信息学机构/stc-95/Restools/biotools多种数据库和分析工具/Tools/Comparative sequence analysishttp://www.bork.embl-heidelberg.de/功能基因组分析Transcription profiling technologies/ncicgap/expression_tech_info.html Protocols for cDNA array technology/pbrown/array.htmlData management and analysis of gene expression arrays/DIR/LCG/15k/HTML/Examples of commercially available filter arrays: GeneFiltersTM (Research Genetics)Gene Discovery Arrays (Genome Systems)AtlasTM Arrays (CLONTECH)资料来源:新浪博客(一抹新绿)/sll888。