第四章-DNA序列分析
- 格式:ppt
- 大小:1.67 MB
- 文档页数:74

生物信息学方法在特定基因调控区识别和分析中的应用随着生命科学的发展,生物信息学方法在分子生物学研究中扮演着越来越重要的角色。
特定基因调控区(gene regulatory region)是指位于基因的上游或下游区域,包含了各种调控元件和转录因子结合位点,是基因表达调控的重要部分。
通过识别和分析这些调控区,可以深入了解基因表达的调控机制,并有助于发现新的治疗和预防疾病的方法。
本文将介绍生物信息学方法在特定基因调控区识别和分析中的应用。
1. DNA序列分析DNA序列分析是指对调控区DNA序列进行计算机处理,以识别其中包含的调控元件和转录因子结合位点,并预测它们对基因表达的影响。
这一过程可以借助许多生物信息学工具实现,如MEME和Weeder等。
这些工具可以进行模式识别和序列比较,从而发现DNA序列中的共同模式和保守序列。
2. ATAC-Seq技术ATAC-Seq技术是一种基于开放染色质的测序方法,用于研究特定细胞类型中基因调控区的开放度。
该技术可以利用转座酶插入开放染色质区域,然后通过PCR扩增和测序来分析这些区域的DNA序列。
通过露出的DNA序列,可以确定基因调控区的开放状态,并预测转录因子的结合位点。
3. CHIP-Seq技术CHIP-Seq技术是一种高通量测序方法,用于鉴定某种转录因子与调控区DNA 结合的位点及其相应的上游基因。
该技术利用可特异地识别转录因子的抗体,将与之结合的DNA序列片段分离出来,并通过测序来鉴定所结合的基因区域。
通过CHIP-Seq技术可以全面地鉴定基因的上游区域和下游区域中的转录因子结合位点,从而为研究基因调控提供基础数据。
4. Hi-C技术Hi-C技术是一种全基因组3D染色质拓扑结构的测序方法,可以用于分析基因调控区的空间结构和相互作用。
通过该方法,可以同时测定两个DNA序列片段之间的空间距离和它们之间的相互作用,从而构建基因组范围的联系图。
利用这一联系图,可以了解基因调控区在三维空间中的位置及其与其他基因区域的互动,从而发现新的调控元件。
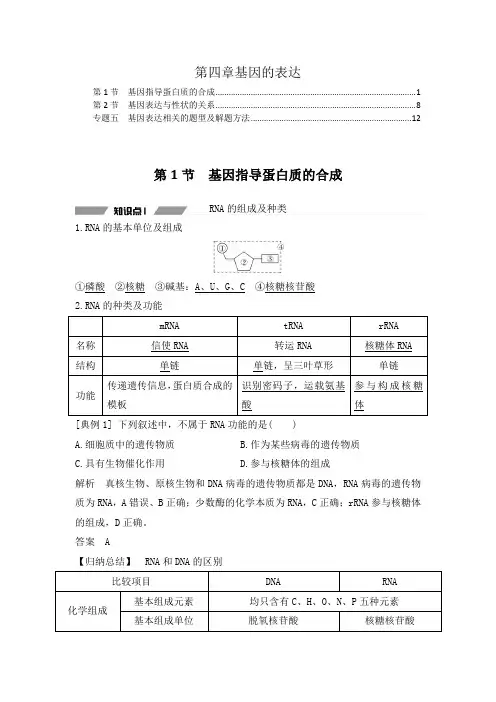
第四章基因的表达第1节 基因指导蛋白质的合成 ........................................................................................... 1 第2节 基因表达与性状的关系 ........................................................................................... 8 专题五 基因表达相关的题型及解题方法 . (12)第1节 基因指导蛋白质的合成RNA 的组成及种类1.RNA 的基本单位及组成①磷酸 ②核糖 ③碱基:A 、U 、G 、C ④核糖核苷酸 2.RNA 的种类及功能 mRNA tRNA rRNA 名称 信使RNA 转运RNA 核糖体RNA 结构 单链单链,呈三叶草形单链功能传递遗传信息,蛋白质合成的模板识别密码子,运载氨基酸参与构成核糖体[典例1] 下列叙述中,不属于RNA 功能的是( ) A.细胞质中的遗传物质 B.作为某些病毒的遗传物质 C.具有生物催化作用D.参与核糖体的组成解析 真核生物、原核生物和DNA 病毒的遗传物质都是DNA ,RNA 病毒的遗传物质为RNA ,A 错误、B 正确;少数酶的化学本质为RNA ,C 正确;rRNA 参与核糖体的组成,D 正确。
答案 A【归纳总结】 RNA 和DNA 的区别比较项目DNARNA化学组成基本组成元素 均只含有C 、H 、O 、N 、P 五种元素 基本组成单位脱氧核苷酸核糖核苷酸碱基A、G、C、T A、G、C、U五碳糖脱氧核糖核糖无机酸磷酸磷酸空间结构规则的双螺旋结构通常呈单链结构【归纳】DNA与RNA的判定方法(1)根据五碳糖种类判定:若核酸分子中含核糖,一定为RNA;含脱氧核糖,一定为DNA。
(2)根据含氮碱基判定:含T的核酸一定是DNA;含U的核酸一定是RNA。



名词解释:第一章基因组遗传图(连锁图):指基因或DNA标记在染色体上的相对位置与遗传距离。
单位是厘摩cM (基因或DNA片段在染色体交换过程中分离的频率)。
物理图:以已知核苷酸序列的DNA片段(序列标签位点,sequence-tagged site, STS)为“路标”,以碱基对作为基本测量单位(图距)的基因组图。
转录图:以EST(expressed sequence tag ,表达序列标签)为标记,根据转录顺序的位置和距离绘制的图谱。
EST:通过从cDNA文库中随机挑选的克隆进行测序所获得的部分cDNA的5'或3'端序列称为表达序列标签(EST),一般长300-500 bp左右。
序列图(分子水平的物理图):序列图是指整个人类基因组的核苷酸序列图,也是最详尽的物理图。
既包括可转录序列,也包括非转录序列,是转录序列、调节序列和功能未知序列的总和。
基因:合成有功能的蛋白质或RNA所必需的全部DNA序列,即一个基因不仅包括编码蛋白质或RNA的核酸序列,还应包括为保证转录所必需的调控序列。
基因组(genome):生物所具有的携带遗传信息的遗传物质的总和。
基因组学(genomics):涉及基因组作图、测序和整个基因组功能分析的一门学科。
C值:单倍体基因组的DNA总量,一个特定种属具有特征C值C值矛盾(C value paradox):指一个有机体的C值和其编码能力缺乏相关性。
单一序列:基因组中单拷贝的DNA序列。
重复序列:基因组中多拷贝的DNA序列。
复杂性(complexity):基因组中不同序列的DNA总长。
高度重复序列(highly repetitive sequence):重复片段的长度单位在几个到几百个碱基对(base pair,bp)之间(一般不超过200 bp),串联重复频率很高(可达106以上),高度重复后形成的这类重复顺序称为高度重复顺序。
中度重复序列(intermediate repetitive sequence ):重复长度300~7000 bp不等,重复次数在102~105左右。

第四章遗传物质——基因和染色体第一节核被膜与核孔复合体细胞核的结构:在固定和染色的细胞中,可观察到细胞有下列结构:核被膜、染色质、核仁、核液(质)四部分。
一、核被膜(nuclear envelope)亦称核膜(nuclear membrane),由此使遗传物质DNA与细胞质分开。
电镜下证实为双层单位膜呈同心性排列。
除两膜之间有间隙外,膜上还有些特化结构。
所以,认为核被膜含义深刻,包括内容多,并执行重要的生理功能。
(一)核被膜结构1 外层核被膜(ONE)(外核膜)膜厚 6.5—7.5nm,相邻细胞质的一面常有核糖体附着,并有时与内质网(RER)相连,因此显得粗糙不平。
2 内层核被膜(INE)(内核膜):膜厚度基本同ONE,膜上无核糖体附着,显得比ONE 平滑。
但在其内表面常附有酸性蛋白质分子的聚合物组成的纤维网状结构(密电子物质),称纤维层(fibrous Lamina)或核纤层(nuclear lamina),又有内致密层之称。
其厚度约在10—20nm(30—160nm),是位于细胞内核膜下的纤维蛋白或纤维蛋白网络。
3 核周隙(perinuclear space)又有核围腔或核围池之称。
指两膜之间的空隙,宽约20—40nm(10—50nm),内充满液态无定形物质(蛋白质、酶类、脂蛋白、分泌蛋白、组蛋白等),它是核质之间活跃的物质交换渠道(有些部位直接与ER或Golgi池相通)。
4 核孔(nuclear pore)核膜并不完全连续,在许多部位,核膜内外两层常彼此融合,形成环状孔道,称为核孔,它们是核质之间的重要通道。
(二)核被膜的主要功能核孔复合体可以看作是一种特殊的跨膜运输蛋白复合体,并且是一个双功能、双向性的亲水性核质交换通道。
双功能表现在它有两种运输方式:被动扩散与主动运输;双向性表现在既介导蛋白质的入核转运,又介导RNA、核糖核蛋白颗粒(RNP)的出核转运。
1、构成核、质之间的天然选择性屏障避免生命活动的彼此干扰,保护DNA不受细胞骨架运动所产生的机械力的损伤2、核质之间的物质交换与信息交流1)通过核孔复合体的被动扩散——小分子物质的转运:核孔复合体作为被动扩散的亲水通道,其有效直径为9~10nm,有的可达12.5nm,即离子、小分子(相对分子质量在60KD以下)以及直径在10nm以下的物质原则上可以自由通过。

第9章_DNA序列分析DNA序列分析是指对DNA序列进行系统性研究和分析的过程。
DNA序列是生物体内的遗传信息的载体,对于了解基因功能、生物演化、疾病发生机制等具有重要意义。
本章将介绍DNA序列分析的方法和应用。
DNA序列分析的方法包括序列比对、基因预测、遗传变异检测和进化分析等。
序列比对是将已知DNA序列与未知序列进行对比,寻找相似之处,从而推断未知序列的功能。
常用的序列比对工具有BLAST、Bowtie等。
基因预测是利用生物信息学方法预测未知DNA序列中的基因位置和功能。
常用的基因预测工具有GeneMark、Glimmer等。
遗传变异检测是通过比较不同个体之间的DNA序列差异,寻找与疾病相关的遗传变异。
进化分析是利用DNA序列比较不同物种之间的遗传差异,推断它们的亲缘关系和演化过程。
常用的进化分析方法有多序列比对、系统发育树构建等。
DNA序列分析在生物学研究和应用领域具有广泛的应用。
在基础研究方面,DNA序列分析可以帮助研究人员了解基因的功能和调控机制。
通过比对不同物种之间的DNA序列,可以揭示物种的进化关系和演化过程。
在医学研究方面,DNA序列分析可以用于疾病的诊断和预测。
通过检测DNA序列中的遗传变异,可以发现与疾病相关的基因突变,并为疾病的治疗和预防提供理论基础。
在农业研究方面,DNA序列分析可以应用于作物和畜禽的遗传改良。
通过分析作物和畜禽的DNA序列,可以挖掘有益基因和导育改良品种,提高农作物和畜禽的产量和品质。
随着高通量测序技术的发展,DNA序列分析在研究领域的应用也得到了大幅度的提升。
高通量测序技术可以快速、准确地获取大量的DNA序列信息,为DNA序列分析提供了更为丰富的数据。
同时,也为DNA序列分析提供了更多的挑战,如序列比对的速度和精度、大规模数据的储存和分析等。
因此,进一步研发和改良DNA序列分析的方法和工具,提高分析效率和准确性,将是今后的研究重点。
综上所述,DNA序列分析是一项重要的生物信息学研究方法,具有广泛的应用前景。

DNA测序技术原理:基因组中的碱基序列分析DNA测序是分析基因组中的碱基序列的技术,它的原理基于化学、生物学和计算机科学的多学科知识。
以下是DNA测序技术的基本原理:1. 样本准备:DNA测序的第一步是准备DNA样本。
样本可以来自生物体的细胞,可以是整个基因组的DNA,也可以是特定基因的DNA。
2. DNA复制:DNA样本中的DNA链被复制,以产生更多的DNA。
这通常通过PCR (聚合酶链式反应)或其他放大技术来完成。
3. DNA片段化:复制的DNA链被切割成短片段,通常长度在几百到几千碱基对之间。
4. 测序反应:使用测序反应来确定每个DNA片段的碱基序列。
目前有多种不同的测序技术,包括Sanger测序、Next-Generation Sequencing(NGS)等。
5. Sanger测序原理:反应体系: Sanger测序使用一种特殊的DNA聚合酶、DNA引物、四种不同的荧光标记的二进制核苷酸和可终止DNA链合成的二进制核苷酸(dideoxynucleotide)。
合成终止:在DNA合成的过程中,如果在新合成链上加入了带有荧光标记的dideoxynucleotide,DNA链的生长就会终止。
分离与检测:反应产物通过凝胶电泳分离,然后使用荧光检测器检测荧光标记的终止核苷酸,从而确定DNA链的碱基序列。
6. Next-Generation Sequencing(NGS)原理:并行测序: NGS技术允许同时测序许多DNA片段,通过并行处理大量的DNA序列信息。
荧光标记: DNA片段被标记,然后通过光学或电化学方法进行测量。
数据分析:通过计算机进行大规模的数据分析,将碱基序列的信息还原出来。
7. 数据分析与装配:通过计算机对得到的测序数据进行分析,将碱基序列还原成原始DNA序列。
这包括去除杂音、纠正测序错误和对碱基进行准确的排序。
8. 结果解读:最后,通过生物信息学工具和数据库比对,对DNA序列进行解读,找到基因、调查变异或识别其他生物学信息。

第七章 DNA序列分析DNA的一级结构决定了基因的功能,欲想解释基因的生物学含义,首先必须知道其DNA 顺序。
因此DNA序列分析(DNA sequencing)是分子遗传学中一项既重要又基本的课题。
1986年由美国学者提出的,目前正在实施的人类基因组计划(human genome project),则是要通过对人类基因组3×109bp全序列的序列分析和人类基因的染色体图谱制定达到了解其结构,认识其功能,即从分子遗传学水平来认识人类自身的结构和功能特征的目的。
核酸的核苷酸序列测定方法已经过近20年的发展,因而测序的具体方法五花八门、种类繁多。
但是究其所依据的基本原理,不外乎Sanger的核酸链合成终止法及Maxam和Gilbert的化学降解法两大类。
虽然原理不同,但这两种方法都同样生成互相独立的若干组带放射性标记的寡核苷酸,每组寡核苷酸都有固定的起点,但却随机终止于特定的一种或多种残基上。
由于DNA链上每一个碱基出现在可变终止端的机会均等,因而上述每一组产物都是一些寡核苷酸的混合物,这些寡核苷酸的长度由某一种特定碱基在原DNA片段上的位置所决定。
然后在可以区分长度仅相差一个核苷酸的不同DNA分子的条件下,对各组寡核苷酸进行电泳分析,只要把几组寡核苷酸加样于测序凝胶中若干个相邻的泳道之上,即可从凝胶的放射自显影片上直接读出DNA上的核苷酸顺序。
以下分别介绍。
1、Sanger的双脱氧链终止法这是1977年由英国剑桥大学分子生物学实验室的生物化学家Sanger(桑格)等人发明的,是一种简单快速的DNA序列分析法,利用DNA聚合酶和双脱氧链终止物测定DNA核苷酸序列。
它的基本原理是:利用DNA聚合酶的两种酶促反应的能力。
第一是,DNA聚合酶能够利用单链的DNA作模板,准确地催化合成出DNA互补链。
实际上这是DNA在体外进行的复制过程。
第二是,DNA聚合酶能够利用2′,3′-双脱氧核苷三磷酸作底物,使之掺入到寡核苷酸链(由几个核苷酸组成的核苷酸链叫做寡核苷酸链)的3′末端,从而终止DNA链的生长。
第一章绪言生物信息学的主要信息载体:DNA和蛋白质生物主要的遗传物质DNA生物的物质基础蛋白质一、生物信息学概述1、定义生物信息学(Bioinformatics)是生命科学、现代信息科学、数学、物理学以及化学等多个学科交叉结合形成的一门学科,是利用信息技术和数学方法对生命科学研究中的生物学数据进行存储、检索和分析的科学。
2、特点⁕以计算机为主要工具,以大量生物数据库和分析软件为基础⁕依赖于Internet⁕为人类揭示生命的奥秘提供了一条新的途径二、生物信息学的发展前基因组时代——生物数据库的建立、检索工具的开发、DNA和蛋白质序列分析、全局和局部的序列对位排列基因组时代——基因寻找和识别、网络数据库系统的建立、交互界面的开发后基因组时代——大规模基因组分析、蛋白质组分析三、生物信息学应用基础研究和教学:分子生物学研究的重要手段之一;生命科学的教学药物开发:新药筛选、药靶设计、分子药理学研究疾病诊断:利用疑难病症的病原DNA序列诊断疾病;遗传病的筛查其他:环境监测;食品安全检测;海关检测第二章数据库及其检索生物信息学数据库的建立及定义生物信息数据库:生物分子数据、分子结构结构及功能等实验证据一级数据库是直接来源于实验室获得的数据,即DNA和蛋白质数据库(X)在生物信息学中数据库查询是指对数据库中的注释信息进行基于关键词匹配查找,而数据库检索是指通过特定的序列相似性比对算法,在核酸或蛋白质序列数据库中获得序列信息(√)一、数据库定义数据库(database)是一类用于存储和管理数据的计算机文档,是统一管理的相关数据的集合,其存储形式有利于数据信息的检索与调用。
数据库的每一条记录(record),也可以称为条目(entry),包含了多个描述某一类型数据特性或属性的字段(field),如基因名、来源物种、序列的创建日期等;值(value)则是指每条记录中某个字段的具体内容。
二、生物信息数据库的分类(1)按照数据来源一级数据库:数据直接来源于实验获得的原始数据,只经过简单的归类整理和注释二级数据库:对原始生物分子数据进行整理、分类的结果,是在一级数据库、实验数据和理论分析的基础上针对特定的应用目标而建立的。
绪论1.20世纪50年代,Waston和Crick提出了DNA双螺旋结构,标志着现代分子生物学的兴起。
2.从广义上来讲,应用到临床的分子标志物包括基因组DNA、各种RNA、蛋白质和各种代谢物,目前,临床分子生物学检验的靶标主要以核酸(DNA或RNA)为主.第一章:核酸与分子标志物1.分子标志物:可以反映机体生理、病理状态的核酸、蛋白质、代谢产物等生物分子,是生物标志物的一种类型。
核酸分子标志物是分子生物学检验的主要内容,包括基因突变、DNA多态性、基因组DNA片段、RNA和循环核酸等多种形式。
2.DNA的组成:是一种高分子化合物,其基本单位是脱氧核苷酸,每个核苷酸由磷酸、脱氧核糖和含氮碱基3部分组成。
3。
DNA与RNA的区别:2位脱氢。
4。
DNA的结构:①DNA一级结构:各种核苷酸单体沿多核苷酸链排列的顺序,表明该DNA分子的化学构成。
②DNA二级结构:双螺旋结构,DNA双螺旋的直径为2nm,一圈螺旋含10个碱基对,每一圈螺距为3.4nm,每个碱基的旋转角度为36度.维持DNA结构稳定的力量主要是碱基对之间的堆积力,碱基对之间的氢键也起着重要的作用.③DNA三级结构:DNA双螺旋进一步盘曲形成的更加复杂的结构。
5.核小体的形成(真核生物染色体包装过程):在核小体中,DNA盘绕组蛋白八聚体核心,使DNA缩短为原来的1/7;6个核小体形成螺丝管,缩短为1/6;核小体彼此相连成串珠状染色质细丝,螺旋化形成染色质纤维,进一步折叠成染色单体.6.DNA双螺旋结构的变异:右手螺旋A—DNA、C-DNA、D-DNA、E—DNA、H—DNA、L—DNA、P-DNA,左手螺旋Z —DNA7.RNA主要分为三类:tRNA、rRNA、mRNA 还有一些小型RNA:反义RNA、微小RNA(microRNA,miRNA是一类内源性的具有调控功能的非编码RNA。
)8.真核mRNA与原核mRNA的区别(简答题)原核mRNA结构简单,为多顺反子,编码序列之间有间隔序列,原核生物mRNA中没有修饰碱基。