DNA指纹图谱分析实验
- 格式:doc
- 大小:120.00 KB
- 文档页数:4


DNA指纹图谱法的基本操作:从生物样品中提取DNA(DNA一般都有部分的降解),可运用PCR 技术扩增出高可变位点(如VNTR系统,串联重复的小卫星DNA等)或者完整的基因组DNA,然后将扩增出的DNA酶切成DNA片断,经琼脂糖凝胶电泳,按分子量大小分离后,转移至尼龙滤膜上,然后将已标记的小卫星DNA探针与膜上具有互补碱基序列的DNA片段杂交,用放射自显影便可获得DNA指纹图谱。
琼脂糖凝胶电泳是分离,鉴定和纯化DNA片段的常规方法。
利用低浓度的荧光嵌入染料-溴化乙锭进行染色,可确定DNA在凝胶中的位置。
如有必要,还可以从凝胶中回收DNA条带,用于各种克隆操作。
琼脂糖凝胶的分辨能力要比聚丙烯酰胺凝胶低,但其分离范围较广。
用各种浓度的琼脂糖凝胶可以分离长度为200bp至近50kbp的DNA。
长度100kb或更大的DNA,可以通过电场方向呈周期性变化的脉冲电场凝胶电泳进行分离。
在基因工程的常规操作中,琼脂糖凝胶电泳应用最为广泛。
它通常采用水平电泳装置,在强度和方向恒定的电场下进行电泳。
DNA分子在凝胶缓冲液(一般为碱性)中带负电荷,在电场中由负极向正极迁移。
DNA分子迁移的速率受分子大小,构象。
电场强度和方向,碱基组成,温度和嵌入染料等因素的影响。
三. 实验材料和试剂1. DNA样品2.化学试剂和溶液(1)DNA样品反应缓冲液:100mM Tris,200mM NaCl,20mM MgCl2,2mM DTT,pH 8.0 (2)EcoRⅠ限制性内切酶(3)PstⅠ限制性内切酶(4)电泳缓冲液(50×TAE)Tris 242g冰醋酸57.1mlEDTA(0.5mol/L pH 8.0)100ml使用时用蒸馏水稀释50倍。
(5)样品缓冲液(DNA sample loading dye)0.25%溴酚蓝0.25%二甲苯青40%(W/V)蔗糖(6)溴化乙锭(EB)10mg/ml(7)琼脂糖agarose(电泳级)(8)DNA分子量标记物:Lambda HindⅢDNA markers3. 仪器设备和消耗品电泳仪、电泳槽、样品梳、微波炉、水浴锅、移液器(10μl,200μl,1000μl)、离心管、一次性枪头(200μl,1000μl)。



DNA指纹的遗传分析【实验原理】“DNA指纹”是指可以利用DNA差异来进行与传统指纹分析相似的身份识别。
DNA指纹是以DNA的多态性为基础,而卫星DNA的发现则是其最重要的奠基石。
卫星DNA是由一短序列(即重复单位或核心序列)多次重复而成,因此也有人称其为可变数目串联重复序列(variable numbers of tandem reprat,VNTR),在人类基因组中存在多种由不同重复单位组成的卫星DNA,重复单位的碱基序列在不同个体中具有高度的保守性,而卫星DNA 的多态性则来源于重复单位的重复次数不同,并形成了众多的等位基因。
列如,人类1号染色体上的VNTR D1S80,核心序列由16个核苷酸组成,拷贝数在14~41个之间,已知29种不同的等位基因。
1984年Jefferys等人首次将分离的人源小卫星DNA用作基因探针,同人类核DNA的酶切片段杂交,产生了由10多条带组成的杂交图谱,不同个体杂交图谱上带的位置就像指纹一样因人而异,因而Jefferys等人称之为DNA 指纹图谱。
产生DNA指纹图谱的过程叫做DNA指纹分析,目前包括PCR、RFLP(限制性内切酶酶切片段长度多态性)和RAPD(随机扩增多态性DNA)等方法。
DNA指纹图谱的基本特点:(1)多位点性:基因组中某些位点的小卫星重复单位含有相同或相似的核心序列。
在一定的杂交条件下,一个小卫星探针可以同时与十几个甚至几十个小卫星位点上的等位基因杂交。
(2)高变异性:DNA指纹图谱反应的是多个位点上的等位基因的特征,具有很高的变异性。
发现两个无血缘关系个体具有相同DNA指纹图谱的概率仅为5×10-19,因此,除了同卵双胞胎,几乎不可能有两个人的DNA指纹图谱完全相同。
(3)稳定的遗传性:DNA指纹图谱中的谱带能够稳定遗传,杂合带遵守孟德尔遗传规律。
子代DNA指纹图谱中产生与双亲都不同的新带的概率(基因突变)仅在0.001~0.004之间。

DNA指纹图谱分析实验一. 实验目的1. 掌握DNA指纹图谱技术的概念、原理和基本操作过程2. 学习DNA的限制性酶切的基本技术3. 掌握琼脂糖凝胶电泳的基本操作技术,学习利用琼脂糖凝胶电泳测定DNA片段的长度,并能对实验结果进行分析。
二. 实验原理1984年英国莱斯特大学的遗传学家Jefferys及其合作者首次将分离的人源小卫星DNA 用作基因探针,同人体核DNA的酶切片段杂交,获得了由多个位点上的等位基因组成的长度不等的杂交带图纹,这种图纹极少有两个人完全相同,故称为"DNA指纹",意思是它同人的指纹一样是每个人所特有的。
DNA指纹的图像在X光胶片中呈一系列条纹,很像商品上的条形码。
DNA指纹图谱,开创了检测DNA多态性(生物的不同个体或不同种群在DNA结构上存在着差异)的多种多样的手段,如RFLP(限制性内切酶酶切片段长度多态性)分析、串联重复序列分析、RAPD(随机扩增多态性DNA)分析等等。
各种分析方法均以DNA的多态性为基础,产生具有高度个体特异性的DNA指纹图谱,由于DNA指纹图谱具有高度的变异性和稳定的遗传性,且仍按简单的孟德尔方式遗传,成为目前最具吸引力的遗传标记。
DNA指纹具有下述特点:1.高度的特异性:研究表明,两个随机个体具有相同DNA图形的概率仅3×10-11;如果同时用两种探针进行比较,两个个体完全相同的概率小于5×10-19。
全世界人口约50亿,即5×109。
因此,除非是同卵双生子女,否则几乎不可能有两个人的DNA指纹的图形完全相同。
2.稳定的遗传性:DNA是人的遗传物质,其特征是由父母遗传的。
分析发现,DNA?指纹图谱中几乎每一条带纹都能在其双亲之一的图谱中找到,这种带纹符合经典的孟德尔遗传规律,即双方的特征平均传递50%给子代。
3.体细胞稳定性:即同一个人的不同组织如血液、?肌肉、毛发、精液等产生的DNA指纹图形完全一致。

样品前处理:1.冷冻保存:-80℃组织应在离体30min内,快速取材,放入专用冷冻管中,并编上号,登记在册。
1.从组织提取基因组DNA一、材料外囊、外膜、“外膜”附近肝组织、正常肝组织(多组标本)二、设备移液管、高速冷冻离心机、台式离心机、水浴锅。
三、试剂1、分离缓冲液:10mmol/L Tris·Cl pH7.4, 10mmol/L NaCl, 25mmol/L EDTA。
2、其它试剂:10% SDS,蛋白酶 K (20mg/ml或粉剂),乙醚,酚:氯仿:异戊醇(25:24:1),无水乙醇及70%乙醇,5mol/L NaCl,3mol/L NaAc,TE。
四、操作步骤:1. 切取组织5g左右,剔除结缔组织,吸水纸吸干血液,剪碎放入研钵(越细越好)。
2. 倒入液氮,磨成粉末,加10ml分离缓冲液。
3. 加1ml 10% SDS, 混匀,此时样品变得很粘稠。
4. 加50ul或1mg 蛋白酶 K, 37℃保温1-2小时, 直到组织完全解体。
5. 加1ml 5mol/L NaCl, 混匀,5000rpm离心数秒钟。
6.取上清液于新离心管,用等体积酚:氯仿:异戊醇(25:24:1)抽提。
待分层后,3000rpm离心 5分钟。
7. 取上层水相至干净离心管, 加2倍体积乙醚抽提(在通风情况下操作)。
8. 移去上层乙醚,保留下层水相。
9. 加1/10 体积3mol/L NaAc, 及2倍体积无水乙醇颠倒混合沉淀DNA。
室温下静止10-20分钟,DNA沉淀形成白色絮状物。
10. 用玻棒钩出DNA沉淀,70%乙醇中漂洗后,在吸水纸上吸干,溶解于1ml TE 中,-20℃保存。
11. 如果DNA溶液中有不溶解颗粒,可在5000rpm短暂离心,取上清; 如要除去其中的RNA,可加5μl RNaseA(10μg/μl), 37℃保温30分钟, 用酚抽提后, 按步骤9-10重沉淀DNA。
基因组DNA的检测(即定性及定量分析)上述方法得到的DNA一般可以用作Southern, RFLP、PCR等分析。

DNA指纹图谱技术在土壤微生物多样性研究中的应用pace等于1986年首次利用rrna基因确定环境样品中的微生物,通过对5s rrna基因的序列分析来研究微生物的生态和进化。
该方法很快被用于微生物多样性研究领域。
由于5s rrna基因相对较小,携带信息相对较少,因而揭示微生物群落多样性的能力有限。
相比之下,随后开展的16s rrna基因序列分析为微生物多样性研究提供了更多信息,且效率更高。
目前16s rrna基因序列分析已广泛应用于微生物多样性的研究。
16s rrna基因序列分析是主要基于已建立的微生物16s rrna基因序列数据库,用以确定细菌的系统发育关系,并使序列探针用于识别未知菌成为可能。
当然序列探针的确定也可根据分子标记方法,如rapd方法进行。
利用16s rrna基因序列研究微生物多样性可采用不同的策略。
目前一般常用以下几种方法。
一、变性梯度凝胶电泳(dgge)和温度梯度凝胶电泳(tgge )1993年muyzer等将dgge技术引入环境微生物学多样性的研究。
随后该技术被广泛用于比较不同生态系统中的微生物群落的多样性及监测特定微生物种群的动态变化,dgge和tgge的原理是根据含有不同序列的dna片段(16s rrna基因扩增产物)在具有变性剂梯度或温度梯度的凝胶上由于其解链行为的不同而导致迁移率的不同,部分解链的dna片段在凝胶中的迁移速率低于完全螺旋形式的dna分子,从而含有不同碱基序列的dna片段就得到有效分离。
结合pcr扩增标记基因或其转录物(rrna和mrna)的dgge方法能直接显示微生物群落中优势组成成分。
由于它可同时对多个样品进行分析,使之非常适合研究微生物群落的时空变化,而且可以通过对酶切条带进行序列分析,或通过与独特的探针杂交鉴定群落组成,可以方便地了解环境被干扰后的微生物群落变化或某种指示微生物的命运。
oliver等用dgge方法考察了农田微生物的变化情况,认为环境变化对农田微生物的多样性有很大影响。

生物学导论(工科平台)实验报告
实验名称:DNA指纹图谱
学生姓名:罗磊
学生学号:516021910409
指导教师:何丽明
实验时间:2016 年10 月14 日
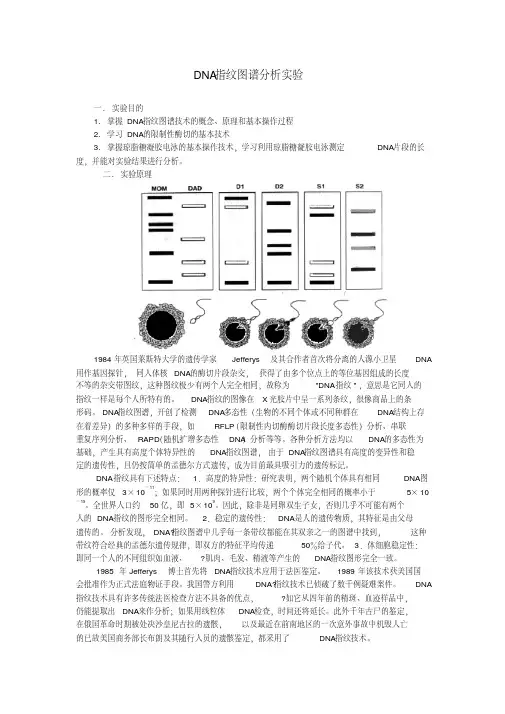
1、插入小组DNA指纹图谱电泳结果照片,进行分析,找出罪犯(50分)。
第二列为犯罪现场DNA样
品的电泳图,通过对比第三
组图片和第二组一样,因此
第三组DNA的主人为罪犯。
2、评价分析实验中的关键步骤(30分)。
a)制胶时拔出梳子应该垂直拔出。
b)DNA 样品应先离心。
电泳液应没过凝胶2mm。
c)点样时应注意不能戳破点样孔。
d)电解时不可将电极接反。
3、 DNA分子在电泳缓冲液中带正电荷还是负电荷?在电场中的运动方向如何(20分)?
负电荷,向正极移动。

家养鸟类DNA指纹图谱的建立和遗传学分析随着人们对自然科学的研究越来越深入,遗传学作为一门新兴的学科已经受到越来越多人的重视。
家养鸟类的DNA指纹图谱的建立和遗传学分析也成为了人们的研究重点。
一、DNA指纹图谱的建立DNA指纹是一种基于DNA序列的个体识别技术。
在建立DNA指纹图谱时,首先需要提取鸟类的DNA样本,可以采用血液、羽毛或口腔粘液等样品提取方式。
提取好的DNA样本需要进行PCR扩增以获得足够数量的DNA。
PCR扩增的产物会在凝胶电泳中形成DNA带,根据DNA带的大小和数量,可以建立DNA指纹图谱,并记录下每只鸟类的DNA指纹。
二、家养鸟类DNA指纹图谱的遗传学分析家养鸟类的DNA指纹图谱能够为遗传学分析提供重要的依据。
首先可以利用DNA指纹图谱判断家养鸟类的亲缘关系,如子代与亲本之间的亲缘关系、同一亲本子代之间的亲缘关系等。
此外,还可以分析鸟类的种群结构和群体历史。
通过比对不同个体之间的DNA指纹图谱,可以发现遗传多样性和基因频率等信息,从而研究鸟类的遗传演化、进化和分化等问题。
三、家养鸟类DNA指纹辨识技术的应用前景家养鸟类DNA指纹辨识技术的应用前景十分广泛。
首先,可以用于宠物鸟类的识别和管理,帮助养鸟人员快速精准地辨认不同的宠物鸟类。
其次,家养鸟类DNA指纹辨识技术还可以用于繁殖管理。
通过对配种鸟类的DNA指纹进行分析,可以评估配种的情况和效果,为繁殖管理提供科学依据。
四、家养鸟类DNA指纹图谱建立及分析中需要注意的问题在家养鸟类DNA指纹图谱的建立和遗传学分析过程中,需要注意以下几个问题。
首先是鸟类样本的选择问题。
要保证DNA样本的质量和数量,选择合适的样品非常重要。
其次是PCR扩增反应体系的优化问题,这是影响扩增效果和质量的重要因素。
还需要掌握准确的分析方法,如如何分辨不同长度的DNA带和如何验证DNA指纹图谱的可重复性等。
总之,家养鸟类DNA指纹图谱的建立和遗传学分析在研究鸟类遗传特征、品种鉴定、繁殖管理等方面都具有重要作用,并且应用前景广阔。
利用DNA分子指纹图谱技术检测小麦粉中害虫的研究鲁玉杰;袁园;刘凤杰;王争艳;何向楠【摘要】为开发快速检测我国成品粮中害虫的技术,研究了DNA分子指纹图谱技术检测小麦粉中主要害虫的碎片和虫卵的含量.结果表明,通过对害虫的延长因子EF1基因片段引物的筛选和PCR条件的选择,确定了有效地检测出来小麦粉中的赤拟谷盗、杂拟谷盗和锯谷盗的成虫碎片、幼虫和卵通用引物和PCR扩增条件.通过改良的PCR技术可在4h内快速检测杂拟谷盗、锯谷盗的卵、幼虫和成虫碎片.最低检出限为可检测出小麦粉中0.05%(m/m)卵和幼虫、1%成虫的碎片,灵敏度超过了国际标准.DNA指纹图谱中电泳条带亮度(y)与被检测的不同害虫碎片含量(X)成正相关,回归方程为Y=858.36X+ 74.540相关系数R为0.961 9.利用该技术检测随机取样的小麦粉中成虫的碎片,检测的准确率最高可达80.56%.结果说明了利用DNA分子指纹图谱技术可快速检测出小麦粉中害虫的碎片、幼虫和卵含量,并可预测小麦粉中害虫的感染程度.本研究为开发快速小麦粉中害虫的快速检测技术具有重要的参考意义.【期刊名称】《中国粮油学报》【年(卷),期】2018(033)011【总页数】6页(P105-110)【关键词】DNA分子指纹图谱;害虫碎片;赤拟谷盗;杂拟谷盗;小麦粉;最低检测限【作者】鲁玉杰;袁园;刘凤杰;王争艳;何向楠【作者单位】河南工业大学粮油食品学院;河南省小麦储藏协同创新中心,郑州450001;河南工业大学粮油食品学院;河南省小麦储藏协同创新中心,郑州450001;河南工业大学粮油食品学院;河南省小麦储藏协同创新中心,郑州450001;河南工业大学粮油食品学院;河南省小麦储藏协同创新中心,郑州450001;河南工业大学粮油食品学院;河南省小麦储藏协同创新中心,郑州450001【正文语种】中文【中图分类】TS201.6;Q969.98小麦粉中害虫碎片的数量是面粉质量等级的划分的重要指标。
第1篇一、实验背景指纹,作为人类生物识别的重要标志,自1890年由英国生物学家弗朗西斯·高尔顿提出以来,一直被广泛应用于身份识别、法医学鉴定等领域。
指纹的遗传性使其成为研究人类遗传变异的重要工具。
本实验旨在通过指纹遗传分析,探究指纹形成的基本原理及其遗传规律。
二、实验目的1. 了解指纹的基本结构及其遗传特征;2. 掌握指纹遗传分析的基本方法;3. 分析指纹遗传规律,为法医学鉴定、亲子鉴定等领域提供理论依据。
三、实验原理指纹的形成与遗传密切相关。
指纹是由皮肤嵴和沟组成的,其遗传模式遵循孟德尔遗传规律。
指纹遗传分析主要基于以下几个方面:1. 指纹的基本结构:指纹由嵴和沟组成,嵴和沟的排列组合形成不同的指纹类型。
2. 指纹的遗传方式:指纹的遗传方式遵循孟德尔遗传规律,表现为常染色体显性遗传。
3. 指纹的变异:指纹存在多种变异类型,如弓形纹、箕形纹和旋形纹等。
四、实验材料与仪器1. 实验材料:指纹样本、DNA提取试剂盒、PCR试剂盒、凝胶成像系统等。
2. 实验仪器:DNA提取仪、PCR仪、电泳仪、凝胶成像系统等。
五、实验步骤1. 指纹采集:使用铅笔在复印纸上画出10个格子,用于贴印取的指纹。
将采集到的指纹样本贴在格子上,并标注姓名和样本编号。
2. DNA提取:按照DNA提取试剂盒说明书,提取指纹样本中的DNA。
3. PCR扩增:设计特异性引物,针对指纹相关基因进行PCR扩增。
4. 电泳分析:将PCR产物进行琼脂糖凝胶电泳,观察指纹条带。
5. 结果分析:根据指纹条带,分析指纹类型及其遗传规律。
六、实验结果与分析1. 指纹类型:根据指纹条带,本实验共检测出三种指纹类型:弓形纹、箕形纹和旋形纹。
2. 指纹遗传规律:通过分析指纹类型,发现指纹遗传符合孟德尔遗传规律,表现为常染色体显性遗传。
3. 指纹变异:本实验中,指纹变异类型包括指纹脊数、指纹类型等。
七、实验结论1. 指纹遗传分析是一种有效的研究人类遗传变异的方法。
桂花树苗的DNA指纹图谱构建与分析桂花(Osmanthus fragrans)是我国传统的重要观赏和经济植物之一。
它以其独特的香气和美丽的花朵而闻名,广泛种植于中国南方地区。
随着人们对桂花树苗质量的关注,研究桂花苗的遗传多样性和亲缘关系变得越来越重要。
DNA指纹图谱是研究植物亲缘关系和遗传多样性的有效工具之一。
本文将介绍桂花树苗的DNA指纹图谱构建和分析的方法。
1. 样品收集和DNA提取在进行DNA指纹图谱构建之前,我们首先需要收集桂花树苗的样品并提取其DNA。
可以选择健康的树苗叶片或芽为样品。
使用商用的DNA提取试剂盒,按照生产商的说明书提取DNA。
确保提取的DNA质量良好,以获得可靠的分析结果。
2. 样品DNA浓度和纯度的检测使用分光光度计或荧光定量仪测量DNA的浓度和纯度。
确保DNA的浓度在50-100 ng/μL之间,并且纯度(A260/A280比值)在1.8-2.0之间。
如果浓度或纯度不符合要求,可以进行适当的稀释或纯化处理。
3. PCR反应设置根据所选择的DNA标记和PCR扩增方法,设置PCR反应体系。
选择适当的引物和反应条件,并优化PCR反应的温度梯度以提高扩增效率和特异性。
确保每个样品都进行三个独立反应以消除偶然误差。
4. PCR扩增反应将设置好的PCR反应体系加入样品DNA,并进行PCR扩增反应。
在热循环仪中进行PCR反应,包括初始变性(94℃,3-5分钟),循环变性(94℃,30秒)、退火与延伸(退火温度和时间根据引物设计确定,一般为55-65℃,30秒-2分钟),最终延伸(72℃,5-10分钟),最后保持(4-10℃)。
5. PCR产物检测和分析将PCR扩增产物通过琼脂糖凝胶电泳进行检测和分析。
在1.5%琼脂糖凝胶上进行电泳,运行电泳约30分钟,以检测PCR扩增产物的大小和数量。
使用DNA分子量标记物作为参考,根据PCR扩增产物的迁移距离和大小进行分析。
6. 数据处理和分析根据PCR产物的大小,构建PCR指纹图谱。
DNA指纹图谱分析实验
一. 实验目的
1. 掌握DNA指纹图谱技术的概念、原理和基本操作过程
2. 学习DNA的限制性酶切的基本技术
3. 掌握琼脂糖凝胶电泳的基本操作技术,学习利用琼脂糖凝胶电泳测定DNA片段的长度,并能对实验结果进行分析。
二. 实验原理
1984年英国莱斯特大学的遗传学家Jefferys及其合作者首次将分离的人源小卫星DNA 用作基因探针,同人体核DNA的酶切片段杂交,获得了由多个位点上的等位基因组成的长度不等的杂交带图纹,这种图纹极少有两个人完全相同,故称为"DNA指纹",意思是它同人的指纹一样是每个人所特有的。
DNA指纹的图像在X光胶片中呈一系列条纹,很像商品上的条形码。
DNA指纹图谱,开创了检测DNA多态性(生物的不同个体或不同种群在DNA结构上存在着差异)的多种多样的手段,如RFLP(限制性内切酶酶切片段长度多态性)分析、串联重复序列分析、RAPD(随机扩增多态性DNA)分析等等。
各种分析方法均以DNA的多态性为基础,产生具有高度个体特异性的DNA指纹图谱,由于DNA指纹图谱具有高度的变异性和稳定的遗传性,且仍按简单的孟德尔方式遗传,成为目前最具吸引力的遗传标记。
DNA指纹具有下述特点:1.高度的特异性:研究表明,两个随机个体具有相同DNA图形的概率仅3×10-11;如果同时用两种探针进行比较,两个个体完全相同的概率小于5×10-19。
全世界人口约50亿,即5×109。
因此,除非是同卵双生子女,否则几乎不可能有两个人的DNA指纹的图形完全相同。
2.稳定的遗传性:DNA是人的遗传物质,其特征是由父母遗传的。
分析发现,DNA•指纹图谱中几乎每一条带纹都能在其双亲之一的图谱中找到,这种带纹符合经典的孟德尔遗传规律,即双方的特征平均传递50%给子代。
3.体细胞稳定性:即同一个人的不同组织如血液、•肌肉、毛发、精液等产生的DNA指纹图形完全一致。
1985年Jefferys博士首先将DNA指纹技术应用于法医鉴定。
1989年该技术获美国国会批准作为正式法庭物证手段。
我国警方利用DNA•指纹技术已侦破了数千例疑难案件。
DNA 指纹技术具有许多传统法医检查方法不具备的优点,•如它从四年前的精斑、血迹样品中,仍能提取出DNA来作分析;如果用线粒体DNA检查,时间还将延长。
此外千年古尸的鉴定,在俄国革命时期被处决沙皇尼古拉的遗骸,以及最近在前南地区的一次意外事故中机毁人亡的已故美国商务部长布朗及其随行人员的遗骸鉴定,都采用了DNA指纹技术。
此外,它在人类医学中被用于个体鉴别、确定亲缘关系、医学诊断及寻找与疾病连锁的遗传标记;在动物进化学中可用于探明动物种群的起源及进化过程;在物种分类中,可用于区分不同物种,也有区分同一物种不同品系的潜力。
在作物的基因定位及育种上也有非常广泛的应用。
DNA指纹图谱法的基本操作:从生物样品中提取DNA(DNA一般都有部分的降解),可运用PCR技术扩增出高可变位点(如VNTR系统,串联重复的小卫星DNA等)或者完整的基因组DNA,然后将扩增出的DNA酶切成DNA片断,经琼脂糖凝胶电泳,按分子量大小分离后,转移至尼龙滤膜上,然后将已标记的小卫星DNA探针与膜上具有互补碱基序列的DNA片段杂交,用放射自显影便可获得DNA指纹图谱。
琼脂糖凝胶电泳是分离,鉴定和纯化DNA片段的常规方法。
利用低浓度的荧光嵌入染料-溴化乙锭进行染色,可确定DNA在凝胶中的位置。
如有必要,还可以从凝胶中回收DNA 条带,用于各种克隆操作。
琼脂糖凝胶的分辨能力要比聚丙烯酰胺凝胶低,但其分离范围较广。
用各种浓度的琼脂糖凝胶可以分离长度为200bp至近50kbp的DNA。
长度100kb或更大的DNA,可以通过电场方向呈周期性变化的脉冲电场凝胶电泳进行分离。
在基因工程的常规操作中,琼脂糖凝胶电泳应用最为广泛。
它通常采用水平电泳装置,在强度和方向恒定的电场下进行电泳。
DNA分子在凝胶缓冲液(一般为碱性)中带负电荷,在电场中由负极向正极迁移。
DNA分子迁移的速率受分子大小,构象。
电场强度和方向,碱基组成,温度和嵌入染料等因素的影响。
三. 实验材料和试剂
1. DNA样品:
犯罪现场DNA样品(CS)、嫌疑犯1 DNA样品(S1)、嫌疑犯2 DNA样品(S2)、嫌疑犯3 DNA样品(S3)、嫌疑犯4 DNA样品(S4)、嫌疑犯5 DNA样品(S5)
2.化学试剂和溶液
(1)DNA样品反应缓冲液:100mM Tris, 200mM NaCl, 20mM MgCl2, 2mM DTT, pH 8.0。
(2)EcoRⅠ限制性内切酶
(3)PstⅠ限制性内切酶
(4)电泳缓冲液:242g Tris,57.1ml冰醋酸,100ml EDTA(0.5mol/L pH 8.0),使
用时用蒸馏水稀释50倍。
(5)样品缓冲液:0.25%溴酚蓝,0.25%二甲苯青,40%(W/V)蔗糖
(6)溴化乙锭(EB):10mg/ml
(7)琼脂糖agarose(电泳级)
(8)DNA分子量标记物:Lambda Hin dⅢ DNA markers
3.仪器设备和消耗品
电泳仪、电泳槽、样品梳、微波炉、水浴锅、移液器(10μl,200μl,1000μl)、离心管、一次性枪头(200μl,1000μl)。
四. 实验步骤
1. DNA样品的制备
采集生物检测样本,在弱碱和螯合剂存在条件下进行组织匀浆,溶解细胞或细胞核膜;利用阴离子去垢剂和蛋白酶,在37℃孵化数小时,消化蛋白质分离DNA;使用有机溶剂如苯酚、氯仿等除去残余蛋白质,萃取DNA;用乙醇或某些盐类从溶液中沉淀DNA。
由于一般采集的样本中的DNA都有不同程度的降解,采用PCR技术扩增出完整的基因组DNA或者特定的高可变位点,以此制备出的DNA样品备用。
2. DNA样品的酶切反应
设置DNA样品的双酶切反应,按下列顺序加样(体积:μl):
反应管对照管样品DNA 10 10
反应缓冲液(10×) 2 2
双蒸水 6 8
Eco RⅠ10
PstⅠ 1 0
总体积 20 20
加完反应液,温和混匀,置于37℃水浴中反应1小时,取出备用。
3.酶切产物的琼脂糖电泳
(1)在100ml电泳缓冲液(1TAE或0.5TBE)中加入1g琼脂糖,加热熔化,注意观察,当心煮沸的液体,溢出!。
当凝胶冷却至60℃左右时加入5ul溴化乙锭溶液(终浓度为1ug/ml),充分混匀。
(2)先用透明胶带封固胶托边缘,放好梳子,然后再倒入凝胶(凝胶厚度在5mm左右)。
(3)在凝胶完全凝固后(室温放置30-45分钟),小心移去梳子和透明胶带,将凝胶放入电泳槽中,加入电泳缓冲液(液面超过胶带约2-3mm)。
(4)取已制备好的酶切DNA样品,加入1/5样品缓冲液,充分混匀。
用移液器将样品小心地加入点样孔。
在不同的点样孔中,分别加入DNA分子量标记物,对照以及酶切DNA 样品各5-10 l。
(5)盖上电泳槽,打开电源并调节电压(通常用50-100伏),电泳40-60分钟(注意:DNA样品从负极向正极泳动)。
4. 结果观察与分析
关闭电源,取出凝胶,在紫外灯下观察DNA的迁移位置,并讨论实验结果。
判断CS与哪一个DNA样品是同一个样品,找出罪犯。
五. 注意事项
(1)酶切时,应尽量减少反应中的加水量以使反应体系减到最小。
但要确保酶体积不超过反应总体积的十分之一,否则限制酶活性将受到甘油的抑制。
(2)进行酶切消化时,将除酶以外的所有反应成分加入后再从冰箱中取出酶,并应放
置于冰上。
每次取酶时都应换一个无菌吸头。
操作要尽可能快,用完后立即将酶放回冰箱。
(3)溴化乙锭是一种强烈的致癌物质,使用时必须带手套。
实验结束后,含溴化乙锭的凝胶要进行净化处理。