Miseq数据分析
- 格式:pdf
- 大小:559.72 KB
- 文档页数:4

miseqfgx法医基因组二代测序原理一、引言法医基因组二代测序(miseqfgx)是一种重要的生物技术,广泛应用于法医鉴定、遗传学研究等领域。
本文将介绍法医基因组二代测序的原理、实验流程及其在法医学中的应用。
二、原理法医基因组二代测序的基本原理是基于高通量技术。
其基本步骤包括:DNA 提取、模板制备、测序反应、数据解析等。
首先,从样本中提取出DNA,将其打断成小片段后加入接头,再进行PCR扩增。
接下来,通过高通量测序仪对经过处理的DNA模板进行测序,得到大量的序列数据。
最后,通过生物信息学分析,将这些序列数据转化为基因组信息,从而实现对样本的鉴定。
三、实验流程1. 样本采集:收集含有DNA的样本,如血液、精液、毛发等。
2. DNA提取:对样本进行提取,得到较为纯净的DNA。
3. 模板制备:将DNA打断成小片段,并加入接头。
接头的目的是为了稳定片段并增加序列信息。
4. 测序反应:将带有接头的DNA片段加入测序仪进行测序,得到序列数据。
5. 数据解析:对测序仪得到的原始数据进行处理,包括去除噪音、拼接序列、注释基因等步骤,以获得基因组信息。
四、应用法医基因组二代测序在法医学中的应用主要体现在以下几个方面:1. 亲子鉴定:通过比较样本和疑似父亲的基因组信息,可以确定是否存在亲子关系。
2. 遗传疾病研究:通过对患病家系的基因组信息进行分析,可以研究遗传疾病的发病机制和基因变异。
3. 法医案件分析:通过比较犯罪现场的生物样本和嫌疑人的基因组信息,可以进行个体识别和种属鉴定。
4. 种群遗传学研究:通过对不同群体基因组信息的分析,可以研究种群的遗传结构和生活史。
五、结论法医基因组二代测序作为一种重要的生物技术,具有高通量、高灵敏度、高精度等优点,在法医鉴定、遗传学研究等领域发挥着越来越重要的作用。
随着技术的不断进步,法医基因组二代测序将在更多领域得到应用,为人类健康和科学发展做出更大的贡献。
六、参考文献(此处省略参考文献)七、致谢感谢各位读者对法医基因组二代测序的支持和关注,希望能为大家提供有益的信息和帮助。

Miseq 介绍测序原理MiSeq测序系统采⽤Illumina成熟的TruSeq边合成边测序技术,它是唯⼀⼀台在单个仪器上整合了扩增、测序和数据分析的新⼀代测序仪,每次运⾏最多能产⽣超过7 Gb的数据。
MiSeq系统特有全新的射流结构,能使试剂循环时间缩短5倍。
⾰命性的流程和⽆可⽐拟的准确性,这让MiSeq成为快速⾼效的测序平台,适合⼴泛的应⽤。
MiSeq以Illumina的边合成边测序技术为基础,通过专利的可逆终⽌试剂⽅法对数百万个⽚段同时进⾏⼤规模平⾏测序。
当每个dNTP加⼊时,对荧光标记的终⽌⼦成像,随后切割,允许下⼀个碱基的掺⼊。
由于每个测序循环中四种可逆终⽌⼦结合的dNTP都存在,所以⾃然竞争让掺⼊偏差最⼩化。
根据每个循环的荧光信号测定直接检出碱基,与其他技术相⽐⼤⼤降低了原始错误率,实现了可靠的碱基检出。
实验流程MiSeq采⽤简单快捷的新型测序流程,制备⽂库的DNA起始量可低⾄50 ng,时间不到2⼩时。
⽂库制备完成后,簇⽣成和测序都在MiSeq上完成,最后在内置的仪器计算机上开展数据分析,从质量打分的碱基检出到突变检出和⽐对,不到2⼩时即可完成。
通过直观的触摸屏界⾯开展简单的仪器操作,即插即⽤的试剂带有RFID追踪,具有⾃动化的便利。
⼩巧、⼀体化的MiSeq平台整合了簇⽣成,末端配对测序和完整的数据分析,不再需要辅助性设备,节省了宝贵的实验室台⾯空间。
技术优势1.最短的实验周期:从样品制备开始只需要8⼩时即可得到超过7G的⾼质量测序结果。
2.最便捷的样品制备流程:只需50 ng DNA模板,2⼩时的⾃动化操作就可以完成全部样品制备⼯作。
3.最精确的测序原理:MiSeq沿⽤GA和HiSeq平台的边合成边测序的化学原理,数千篇⾼质量的国际期刊⽂章是对Illumina NGS平台测序结果可信度最有⼒的证明。
5.最完整的整合功能:MiSeq可以独⽴完成簇⽣成、双向测序及数据分析⼯作,让经费和实验室空间不再成为NGS研究的障碍。

MiSeq TM Dx使用手册MiSeqDx 使用手册文件号1000000039320 v04 | 1 2020年12月Material # 20030132引言本文档及其内容是Illumina,Inc.及其附属公司(“Illumina”)所有,并且仅供与所述产品相关的合同约定的客户使用,无其他用途。
未经Illumina事先书面同意,本文件及其内容不得以其他方式传播、披露或转载和/或用于任何其他目的。
Illumina不会通过本文件对专利、商标、版权或普通法权利或任何第三方类似的权利传达任何许可。
本文档中的说明必须由合格且经过适当培训的人员严格明确地遵循,从而确保本文所述产品的正确和安全使用。
在使用此类产品前,必须全面阅读并理解本文档的所有内容。
未能完全阅读并明确地遵守本文包含的所有说明可能会对产品造成损害,对人员造成伤害,包括用户或其他人员的损害以及对其他财产的损害,并失去该产品的保修。
由于本文所述产品(包括其中的任何部分或软件)的不当使用,ILLUMINA不承担任何责任。
© 2017 Illumina, Inc. 版权所有。
Illumina, MiSeq TM Dx,南瓜橙颜色和流动底纹设计是Illumina, Inc.和/或其在美国和/或其他国家/地区附属公司的商标。
所有其他名称、徽标和其他商标均为其各自所有者的财产。
MiSeqDx 使用手册文件号1000000039320 v04 | 2 2020年12月Material # 20030132修订历史MiSeqDx 使用手册文件号1000000039320 v04 | 3 2020年12月Material # 20030132目录引言 (2)修订历史 (3)目录 (4)第一章概述 (6)产品名称 (6)预期用途 (6)产品结构及组成 (6)第二章设备安装及环境要求 (14)运输和储存 (14)运输和安装 (14)实验室要求 (15)电气要求 (17)环境要求 (18)网络要求 (18)用户自备耗材和设备 (19)第三章工作原理 (21)工作原理 (21)需要但不提供的设备和材料 (21)第四章性能指标 (22)仪器技术指标 (22)仪器使用期限 (23)产品合规性和监管声明 (23)第五章运行操作 (26)P ART וL OCAL R UN M ANAGER(本地运行管理) (26)本地运行管理 (26)登陆信息管理 (26)操作界面概述 (27)管理设置和任务 (31)工作流程概述 (37)P ART װM I S EQ O PERATING S OFTWARE (M I S EQ操作软件) (41)启动和开机 (41)MiSeqDx 使用手册文件号1000000039320 v04 | 42020年12月Material # 20030132测序运行 (43)结果分析 (54)质量控制 (55)文件夹管理 (55)MOS软件界面图标 (56)第六章局限性和注意事项 (58)使用局限性 (58)警告和注意事项 (58)第七章危害及标志 (60)安全考虑和标志 (60)通用标志 (62)第八章设备维护 (63)维护频率 (63)维护清洗 (63)待机清洗 (65)关机步骤 (67)重启步骤 (68)所需磁盘空间 (68)杀毒软件 (68)第九章故障排除 (69)附件 (78)MiSeqDx 使用手册文件号1000000039320 v04 | 52020年12月Material # 20030132第一章概述产品名称中文名称:基因测序仪英文名称:MiSeq TM Dx Instrument型号:MiSeq TM Dx预期用途用于体外诊断。

Illumina MiSeq高通量测序分析核桃内生细菌多样性陈泽斌;李冰;王定康;余磊;徐胜光;任禛;靳松;张永福;彭声静【摘要】In this study, the species abundance and alpha diversity of walnut endophytic bacteria were analyzed by Illumina MiSeq high-throughput sequencing of the 16S rDNA-V4 region. Softwares such as Uparse, Flash, and Qiime were employed to sort and calculate the number of sequences and operational taxonomic units ( OTUs) . The numbers of effective sequences and OTUs for each sample were 63183 and 103, respectively. The rarefaction curves showed that adequate sampling was achieved, and the number of OTUs was close to saturation. Majority of the endophytic bacteria belonged to Sphingomonas ( 27. 27%) , Halomonas ( 27. 27%) and Agrobacterium ( 45. 45%) which were therefore the dominant bacterial families in walnut. Illumi-na MiSeq high-throughput sequencing technology provided more accurate and scientific data resources for the study of endophytic bacteria.%应用Illumina MiSeq高通量测序技术测定核桃内生细菌的16S rDNA-V4变异区序列,使用Uparse等软件整理和统计样品序列数目和操作分类单元( OTUs)数量,分析内生细菌的丰度和a-多样性。



MiSeq平台相关技术MiSeq平台简介MiSeq系统是Illumina公司推出的测序平台,测序原理和HiSeq基本相同,即基于DNA 单分子簇的边合成边测序技术和可逆终止化学反应原理。
其拥有不同测序读长模式,测序质量可与HiSeq相媲美。
性能参数如下截图所示。
图 1 MiSeq测序性能参数目前可以支持500-600bp的读长,堪与罗氏454平台读长媲美,但是通量却远高于454平台,2*250读长一个run可产出7-9G左右数据量,2*300读长一个run可产出12G左右数据。
MiSeq测序平台同时拥有较长的读长和较高的数据量产出的特点,是HiSeq和454都无法比拟的优势,是应用于分子生态和基因组测序的最佳平台。
相对于HiSeq系统,具有更长读长,可使拼装结果更加准确完整。
相对于454平台,高数据量产出,加大了测序深度和覆盖度,使测序得到的变异信息更加准确。
技术特点■读长及通量优势:读长优势可得到更准确拼装结果,通量高,PE250高达9G通量,PE300高达15G的通量;■样本制备便捷,试验周期短:最低50ng DNA模板,短时间几个小时的自动化操作就可以完成全部样品制备工作。
■最精确的测序原理: 沿用GA和HiSeq平台的边合成边测序的化学原理■最完整的整合功能:独立完成簇生成、双向测序及数据分析工作。
■数据产出均一性好:对每个样品的DNA量进行严格定量,提高了数据产出的均一性。
适用项目■宏基因组16S RDNA测序:美国Earth Microbiome Project已将该方法作为16S RDNA测序与环境群落结构多样性研究的标准方法,已有多名权威学者的研究成果在ISME J、Cell、Nature 和等顶尖期刊上发表。
■转录组测序:通过该平台测序可相对于Illumina HiSeq平台,可获得更长更准确的转录本拼装信息。
■微生物基因组(重)测序:Illumina MiSeq非常适用于对微生物基因组(重)测序,可得到更准确基因拼装结果的同时可大量准确检出SNP和indel信息。
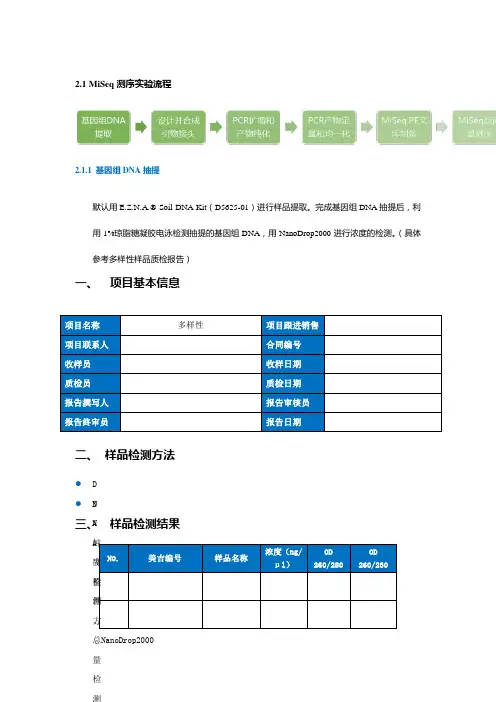
2.1 MiSeq 测序实验流程2.1.1 基因组DNA 抽提默认用E.Z.N.A.® Soil DNA Kit (D5625-01)进行样品提取。
完成基因组DNA 抽提后,利用1%琼脂糖凝胶电泳检测抽提的基因组DNA ,用NanoDrop2000进行浓度的检测。
(具体参考多样性样品质检报告)一、 项目基本信息二、 样品检测方法● D NA 纯度检测方 NanoDrop2000 ● D N A 完整性、总量检三、 样品检测结果 N0. 美吉编号样品名称浓度(ng/μl ) OD 260/280OD 260/230项目名称 多样性 项目跟进销售 项目联系人 合同编号 收样员 收样日期 质检员 质检日期 报告撰写人 报告审核员 报告终审员报告日期胶图(取2μl样品跑胶):2.1.2 PCR扩增按指定测序区域,合成带有barcode的特异引物。
为保证后续数据分析的准确性及可靠性,需满足两个条件,1) 尽可能使用低循环数扩增;2) 保证每个样本扩增的循环数一致。
随机选取具有代表性的样本进行预实验,确保在最低循环数中使绝大多数样本能够扩增出浓度合适的产物。
PCR 采用TransGen AP221-02:TransStart Fastpfu DNA Polymerase;PCR仪:ABI GeneAmp® 9700型;全部样本按照正式实验条件进行,每个样本3个重复,将同一样本的PCR产物混合后用2%琼脂糖凝胶电泳检测,使用AxyPrepDNA凝胶回收试剂盒(AXYGEN公司)切胶回收PCR产物,Tris_HCl洗脱;2%琼脂糖电泳检测。
(具体参照PCR实验报告)PCR扩增报告1.项目基本信息2. PCR预实验2.1引物设计2.2 PCR扩增条件为了保证后续数据分析的准确性及可靠性,需要两个条件,首先是尽可能使用低循环数扩增;其次保证每个样品扩增的循环数统一。
为了达到以上目的,我方首先随机选取10个样品进行预实验,以确保在最低的循环数中绝大多数的样品能够扩增出浓度合适的产物,为所有样品的正式试验做充分准备。

一、数据读出(通过“fasta”文件生成“classification”和“txt”文件)1、下载Java:for64位。
2、cmd进入DOS界面,进入数据所在的文件夹,逐个分析并命名数据,见下行。
Java-Xmx4g-jar..\rdp_classifier_2.6\dist\classifier.jarclassify-c0.8-ffi lterbyconf-h hier_output.txt–o samplename.classification.txt input.f asta注意:刚开始时输入“cd..”(cd空格加两点)即退回上一级目录,直到回到C盘,fasta原始数据也必须放在C盘。
手打指令,适用本机。
3、用Excel打开目标文件txt文本,“筛选”,选择不同的分类单位进行数据整理和分析。
Class:纲Domain:域Family:科Genus:属Order:目Phylum:门Kingdom:界Species:种二、删除chloroplast(叶绿体)1、将原始文件(“fasta”和“classification”文件)拷贝至与程序“mothur”相同的目录下;2、找到后缀名为“H1.classification”的数据原文件(以样品H1为例),用Excel打开;3、选中“Class”对应的物种列,“筛选”,在下拉框中勾掉物种“chloroplast(叶绿体,非细菌)”,“确定”;复制第一列到粘贴板;4、新建“H1.accnos”的txt文件,将第一列(物种序列)粘贴,保存、退出;将后缀名改为“.accnos”(窗口界面“组织”、文件夹和搜索选项、查看、勾掉“隐藏已知文件类型的扩展名”);5、打开程序“mothur”,输入:get.seqs(accnos=H1.accnos,fasta=H1.fasta),回车,即从原始的物种序列中选出了去除chloroplast以外的新序列,系统会自动生成一个新的fasta文件“H1.pick.fasta”。

MiSeq系统指南文档号15027617v06CHS材料号20000262ILLUMINA所有2021年1月MiSeq系统指南本文档及其内容归Illumina,Inc.及其附属公司(以下简称“Illumina”)所有,并且仅供其客户用于与本文档内所述产品用途相关的合同用途,不得用于其他任何目的。
若事先未获得Illumina的书面许可,不得出于任何其他目的使用或分发本文档及其内容,以及/或者以其他任何方式对其进行传播、披露或复制。
Illumina不通过本文档向第三方授权其任何专利、商标、所有权或普通法权利或类似权利。
必须由具备资质且受过相关培训的人员严格明确遵照本文档中的说明操作,以确保本文档中所述产品的使用适当且安全。
在使用此类产品之前,相关人员必须通读并理解本文档中的所有内容。
未能完整阅读并明确遵守本文档中包含的所有说明可能会导致产品损坏、对用户或其他人员造成人身伤害以及对其他财产造成损害,并且将导致产品适用的保证失效。
对于由不当使用本文档中描述的产品(包括其部件或软件)引起的任何后果,ILLUMINA概不承担任何责任。
©2021Illumina,Inc.保留所有权利。
所有商标均为Illumina,Inc.或其各自所有者的财产。
有关特定的商标信息,请参见/company/legal.html。
修订历史记录目录第1章概述1简介1更多资源1组件2 MiSeq概念4系统软件4二级分析选项5 Sequencing Analysis Viewer6所需磁盘空间6 MiSeq试剂盒概述7第2章入门9启动MiSeq9自定义系统设置9配置BaseSpace更新的通知10设置电子邮件首选项10设置默认的文件夹位置10耗材11第3章测序12简介12运行持续时间12簇生成13测序13分析13解冻试剂夹盒13检查试剂夹盒13变性和稀释文库14装入样品文库14使用MCS设置运行14清洁流动槽16装入流动槽17装入试剂18启动运行20监控运行21执行运行后清洗23第4章维护27维护频率27 VeriSeq PGS工作流程的维护频率27执行维护清洗28执行备用清洗30管理文件31MiSeq系统指南软件更新33关闭仪器33附录A故障诊断34简介34用于故障诊断的打包日志34执行系统检查35暂停或停止运行35手动提起试剂夹盒吸管36解决运行设置错误37解决RFID读取失败37执行液量测试38测量预期的清洗量38解决试剂冷却器温度错误38解决Local Run Manager分析错误39配置系统设置39附录B输出文件和文件夹40运行文件夹40 MiSeqOutput文件夹内容40 RTA文件夹和文件41索引43技术协助46第1章概述简介1更多资源1组件2 MiSeq概念4系统软件4二级分析选项5 Sequencing Analysis Viewer6所需磁盘空间6 MiSeq试剂盒概述7简介Illumina®MiSeq™系统结合了经证明的Sequencing by Synthesis(SBS)技术和创新的工作流程,让您只需八小时就能从DNA完成数据分析。

高通量基因测序系统美国illumina公司Miseq技术参数1.仪器功能:可开展快速经济高效的第一代测序应用,包括扩增产物测序和克隆检查等;也可以开展新一代基因组测序应用,如高度多重PCR扩增子测序,小基因组重测序,de novo测序,小RNA测序,文库质控和16s细菌宏基因组研究等;2.技术规格*2.1一轮测序反应生成的有效数据量最高可达15Gb(即150亿个碱基);*2.2一轮测序反应生成的可读测序片段序列最高可达2500万条;*2.3一轮测序反应(最高完成15Gb数据量)的所用的全部试剂和耗材成本不高于2000美元;一轮测序反应的过程包括样本文库克隆扩增、测序和数据分析;*2.4单端读取序列的有效数据读长可达300个碱基;双端读取序列的有效数据读长可达2×300个碱基。
2.5测序质量标准为Q30(即每个碱基的准确度为99.9%),并且75%以上的碱基比例达到Q30;2.6 样本文库克隆扩增原理为桥式PCR法;2.7 每100碱基测序的碱基缺失率<0.001;2.8具备多重SNP分析功能,可在一轮实验中同时检测384个样品,对384×1536=589824个SNP的DNA片段进行测序比对分析;2.9可精确读取≥12个的连续单个重复碱基(如AAAAAAAAAAAAAAAA);2.10具备云储存功能,并具有公共交流平台,方便与业内专家共同分析与合作。
2.11测序系统主机内置服务器,主机同时具备样本文库克隆扩增、测序和数据分析功能;无需再配置额外的样本文库克隆扩增和分析服务器;2.12仪器同时具备并免费提供以下软件分析功能:全基因组重测序、定向重测序、De novo 测序、序列特异DNA结合蛋白的ChIP-Seq、组蛋白修饰和表观遗传标记的ChIP-Seq、16s宏基因组学研究、mRNA测序、基于标签的基因表达、小RNA测序、降解DNA样品的测序、末端配对mRNA测序、DNA印迹和等位基因特异的表达、CNV-Seq:用测序测定拷贝数变异研究。
24㊀2020Vol.46No.24(Total 420)DOI:10.13995/ki.11-1802/ts.024560引用格式:宋思家,黄子彧,林莹莹,等.基于Illumina MiSeq 高通量测序技术分析不同保质期鸡蛋干的微生物群落多样性[J].食品与发酵工业,2020,46(24):24-30.SONG Sijia,HUANG Ziyu,LIN Yingying,et al.Microbial community diversity in egg curds with different shelf life revealed by Illumina MiSeq sequencing[J].Food and Fermentation Industries,2020,46(24):24-30.基于Illumina MiSeq 高通量测序技术分析不同保质期鸡蛋干的微生物群落多样性宋思家1,黄子彧2,林莹莹1,郭慧媛1∗1(中国农业大学食品科学与营养工程学院,精准营养与食品质量重点实验室,教育部功能乳品重点实验室,北京,100083)2(中国农业大学食品质量安全北京实验室,北京,100083)摘㊀要㊀为探究造成鸡蛋干腐败变质的核心优势微生物及造成鸡蛋干保质期差异的微生物种类,为鸡蛋干的防腐保鲜提供理论依据,采用Illumina MiSeq 高通量测序技术对保质期不同的3组腐败变质鸡蛋干的菌群组成进行分析㊂结果表明,3组鸡蛋干样品在门水平的优势菌群均为厚壁菌门,该菌门在15d 组㊁22d 组和29d 组的丰度占比分别为96.73%㊁87.55%和70.94%;在属水平,15d 组㊁22d 组和29d 组中相对丰度最高的菌属均为芽孢杆菌属,其在各组中的占比分别为33.14%㊁47.38%和68.85%,因此,芽孢杆菌属是鸡蛋干的核心致腐微生物㊂此外,造成鸡蛋干保质期差异的菌属包括乳杆菌属和狭义梭菌属,它们可加速鸡蛋干的腐败变质,是鸡蛋干保质期的限制因素,应在杀菌防腐环节重点关注㊂关键词㊀鸡蛋干;Illumina MiSeq 测序;微生物多样性;芽孢杆菌属;乳杆菌属;狭义梭菌属Microbial community diversity in egg curds with different shelf liferevealed by Illumina MiSeq sequencingSONG Sijia 1,HUANG Ziyu 2,LIN Yingying 1,GUO Huiyuan 1∗1(Key Laboratory of Precision Nutrition and Food Quality,Key Laboratory of Functional Dairy (Ministry of Education),College of Food Science and Nutritional Engineering,China Agricultural University,Beijing 100083,China)2(Beijing Laboratory for FoodQuality and Safety,China Agricultural University,Beijing 100083,China)ABSTRACT ㊀To investigate the dominant bacteria that caused the deterioration of egg curd and the mi-crobial species that caused the difference in shelf life of egg curd,Illumina MiSeq sequencing was ap-plied to analyze the microbial community diversity in three groups of spoiled egg curd with different shelf life.The results indicated that Firmicutes were the dominant phylum in these three groups of egg curd,and the abundances of Firmicutes in the 15d group,22d group and 29d group were 96.73%,87.55%and 70.94%,respectively.At the genus level,the most abundant bacteria in the 15d group,22d group,and 29d group were Bacillus ,which accounted for 33.14%,47.38%,and 68.85%in each group,respectively.Therefore,Bacillus was the most dominant spoilage bacteria in egg curd.In addi-tion,the dominant spoilage bacteria also included Lactobacillus and Clostridium _sensu _stricto _13,which can accelerate the deterioration of egg curd.They were the main bacteria that caused the shelf life differ-ence of egg curd and should be paid close attention to in the sterilization process.Key words ㊀egg curd;Illumina MiSeq sequencing;microbial community diversity;Bacillus ;Lactobacil-lus ;Clostridium _sensu _stricto _13第一作者:博士研究生(郭慧媛副教授为通讯作者,E-mail:guohuiyuan@)㊀㊀基金项目:国家重点研发计划课题(2018YFD0400305)收稿日期:2020-05-26,改回日期:2020-09-02㊀㊀鸡蛋干是中国市面上常见的休闲蛋制品,因其营养丰富㊁风味独特而深受消费者喜爱㊂依据GB 2749 2015‘食品安全国家标准蛋与蛋制品“内容,在保质期内的鸡蛋干在感官方面应具有产品正常的色泽㊁滋味㊁气味,无异味,且具有产品正常的形状㊁形态㊁无酸败㊁霉变等;在微生物限量方面,其菌落总数应<104CFU/g,大肠菌群应<10CFU/g[1]㊂目前在实际生产过程中,鸡蛋干的保质期为9个月㊂鸡蛋干的生产原料以全蛋液或蛋清液为主,在加工制作过程中极易受到微生物污染而导致产品腐败变质[2]㊂相关调查研究表明,预包装鸡蛋干即便经过高压蒸汽灭菌后也可能出现胀包㊁汤汁浑浊㊁有异味的变质现象,尤其在夏秋高温季节,鸡蛋干发生腐败变质的概率更高[3]㊂最近数十年,鸡蛋干的市场在不断扩大,因此对鸡蛋干的防腐保鲜开展研究愈显重要㊂而明确鸡蛋干的优势致腐微生物,是防腐的首要关键㊂然而目前对于鸡蛋干的腐败微生物群落多样性的报道十分有限,贺燕等[3]从腐败鸡蛋干中分离鉴定得到2株芽孢杆菌,袁先铃等[4]在腐败鸡蛋干中检测到彭氏变形菌和奇异变形菌㊂因此,开展鸡蛋干中腐败微生物群落多样性的研究具有重要意义㊂对于微生物群落多样性的研究,长期以来主要是基于‘伯杰细菌鉴定手册“,对微生物菌落进行分离㊁纯化,进而通过形态学观察和一系列复杂的生理生化实验进行鉴定㊂这种传统方法不但耗费时间和精力,而且无法对微生物进行精确鉴定,不能检测到微生物群落多样性的真正概貌[5-6]㊂因为有些微生物的培养条件非常苛刻,甚至不可培养,常规方法难以检测到不可培养的微生物㊂近年来,在微生物群落多样性研究中已经越来越倾向于采用Illumina MiSeq高通量测序技术[7-10]㊂高通量测序技术也称 下一代测序技术 ,可同时测序数百万个核酸分子,根据16S rDNA基因序列分析特定样品中微生物群落的构成情况[11-12]㊂目前高通量测序技术已成为监测食品贮藏过程中微生物群落演替㊁分析发酵及腐败食品微生物群落结构等食品微生物多样性研究的首选方法[11]㊂本研究以保质期不同的腐败变质鸡蛋干为研究对象,基于Illumina Miseq测序平台对其16S rDNA V3~V4进行扩增子测序分析,研究其微生物群落结构组成和相对丰度,探究造成鸡蛋干腐败变质的核心优势微生物以及造成鸡蛋干保质期差异的微生物种类,为靶向抑制鸡蛋干的腐败变质提供理论依据㊂1㊀材料与方法1.1㊀材料与试剂鸡蛋,北京德青源农业科技股份有限公司;细菌基因组DNA提取试剂盒,美国Omega科技公司;Axy Prep DNA凝胶回收试剂盒,美国Axygen公司;2ˑTaq PCR MasterMix㊁DNA Marker,天根生化科技(北京)有限公司㊂1.2㊀仪器与设备5418R高速离心机,德国Eppendorf公司;拍击式均质机,法国Interscience公司;RS-400型真空包装机,北京日上公司;T100Thermal Cycler聚合酶链式反应(polymerase chain reaction,PCR)仪㊁PowerPac Basic型电泳仪,美国Bio-Rad公司;ChampGel5000增强型全自动凝胶成像仪,北京赛智创业科技有限公司;Illumina MiSeq测序仪,美国Illumina公司㊂1.3㊀试验方法1.3.1㊀样品制备鸡蛋干的制作工艺参照参考文献[13],具体为取预处理蛋液于模具中,水浴加热使其中心温度达到80ħ,并维持20min,待其冷却收缩后脱模㊂将鸡蛋干样品真空包装后,进行90ħ㊁30min灭菌,于25ħ保存直至变质㊂预实验发现,对于使用不同批次原料㊁于不同的时间点制作的鸡蛋干样品,即使其加工工艺完全一致,样品的保质期也会有较大差异㊂为探究不同保质期的鸡蛋干样品中菌群结构的差异,采用上述加工工艺,分别使用不同批次原料,于不同的时间点制作了多批样品,从其中选取贮藏15d㊁22d和29d腐败变质的3批鸡蛋干产品,取样保存于-80ħ冰箱待测㊂将3批鸡蛋干样品分别编号为15d㊁22d 和29d,每组样品设置3个平行㊂1.3.2㊀细菌总DNA的提取参考高乾坤等[14]的方法,对样品进行处理后,采用美国Omega公司的细菌基因组DNA提取试剂盒,提取细菌总DNA,提取步骤参照试剂盒产品说明书,并进行1%(质量分数)的琼脂糖凝胶电泳,以检测提取效果㊂1.3.3㊀PCR扩增及高通量测序以检验合格的基因组DNA为模板,扩增细菌16S rDNA基因V3~V4区㊂正向引物为338F(5ᶄ-ACTCCTACGGGAGGCAGCA-3ᶄ),反向引物为806R (5ᶄ-GGACTACHVGGGTWTCTAAT-3ᶄ)㊂所得PCR产物经2%(质量分数)琼脂糖凝胶电泳检测扩增效果,2020年第46卷第24期(总第420期)25㊀26㊀2020Vol.46No.24(Total 420)然后将样品进行纯化并定量检测㊂将检测合格的样品送至上海美吉生物医药科技有限公司进行Illumina MiSeq 高通量测序㊂1.4㊀数据分析对MiSeq 测序得到的双端序列数据进行处理,首先将成对的reads 拼接成一条序列,同时对reads 的质量和拼接的效果进行质控过滤㊁得到有效序列,并校正序列方向[15]㊂基于操作分类单元(operational taxonomic units,OTU)聚类分析结果,研究腐败变质鸡蛋干样品中微生物的多样性,通过Alpha 多样性检测单样本测序深度,并分析微生物群落的丰度和多样性[16]㊂基于分类学信息,在各个分类水平上进行群落结构统计,综合讨论保质期不同的鸡蛋干样品中微生物群落结构组成和相对丰度㊂2㊀结果与分析2.1㊀测序数据统计与OTU 分析采用Illumina Miseq 高通量测序获得了3种不同保质期鸡蛋干样品的原始序列数据后,经Fastp 及FLASH 软件优化数据后得到了9个样本共529384条有效序列,序列平均长度为409.70~429.92bp,主要集中在401~440bp(图1-a)㊂稀释曲线可反映在高通量测序过程中的样品取样深度,因此是评价测序数据量是否全面覆盖微生物群落的常用指标[14]㊂为了探究鸡蛋干样品的测序数据量是否科学,从测序获得的所有原始数据中随机抽取数据,并统计其物种多样性指数,得到稀释曲线(图1-b 和图1-c)㊂结果表明,随着数据量的增加,OTU 曲线和Shannon-Wiener 曲线的趋势已经逐渐平缓,即再增加数据量a -样品序列长度分布图;b -OTU 曲线;c -Shannon-Wiener 曲线图1㊀样品序列长度分布图㊁OTU 曲线和Shannon-Wiener 曲线Fig.1㊀Length distribution profile of trimed sequences,OTU curves and Shannon-Wiener curves对于发现新物种也无显著影响㊂样本的OTU 覆盖度已经基本饱和,更多的数据量对发现新OTU 的边际贡献很小,这个结果说明测序数据量足够大,可以反映样本中绝大多数的微生物多样性信息[17]㊂2.2㊀Alpha 多样性分析根据97%相似性水平下的OTU 信息,采用Alpha 多样性指标的Shannon㊁Simpson㊁ACE㊁Chao1及Cov-erage 指数对鸡蛋干样品微生物物种的丰富度和多样性进行评估[14]㊂如表1所示,3组样品的Coverage指数都达到了0.999以上,即样品文库的覆盖率足够高,有未被检出序列的可能性较小,表明本次试验所建文库能够较准确地反映出待测鸡蛋干的微生物多样性情况㊂Chao1㊁ACE 指数用于计算菌群丰度,二者的值越大,说明样品的群落丰富度越高;Shannon㊁Simpson 指数用于计算菌群多样性指数,Shannon 的值越大,说明样品群落多样性越高,而Simpson 指数值越大,则说明其群落多样性越低[18]㊂22d 组的鸡蛋干样品ACE㊁Chao1及Shannon 指数最高,Simpson 值最低,这说明该组样品的细菌群落丰富度及多样性高于其他2组样品,可能由于该组样品的原料中及加工过程中污染的细菌种类最多;29d 组鸡蛋干样品的ACE㊁Chao1及Shannon 指数均为3组中最小,这说明了该组样品中细菌群落丰度及多样性最低,可能由于该组样品的原料中及加工过程中受到的污染程度较低㊂综上,经Alpha 多样性指数统计分析可知,鸡蛋干样品的物种多样性由高到低依次为22d 组㊁15d 组和29d 组㊂此结果显示,相比于污染的微生物物种的多样性,鸡蛋干所污染微生物的具体种类可能与鸡蛋干的保质期相关性更大,这与孙同辉等[11]的报道相一致㊂2.3㊀OTU 的Veen 分析基于样品总OTU 数作Veen 图,利用Venn 图可以展示多组样品间共有和每组样品特有的OTU 数目,并直观展示样品间OTU 的重叠情况[19]㊂如图22020年第46卷第24期(总第420期)27㊀所示,15d 组样品与22d 组样品共有的OTU 数107个,15d 组与29d 组共有的OTU 数131个,22d 组与29d 组共有的OTU 数92个,3组样本共有的OTU 数为81个,占各样本OTU 数比例较小,而各样本特有的OTU 数在113~259,表明各样本之间的菌群结构差异性较大㊂表1㊀Alpha 多样性指数Table 1㊀Alpha diversity indexes样品名称Shannon 值Simpson 值ACE 值Chao1值Coverage 值15d 0.370.91120.69122.720.999722d 1.640.52138.52139.080.999829d0.340.8585.1682.550.9997图2㊀不同保质期样品中微生物物种间OUT 分析图Fig.2㊀OUT analysis diagram of microbial species insamples with different shelf life2.4㊀不同保质期鸡蛋干样品细菌群落结构分析2.4.1㊀基于门水平的细菌菌群结构分析在门水平上,3组鸡蛋干样品的群落组成如图3所示㊂15d 组的鸡蛋干样品总细菌组成以厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)为主,相对丰度分别为96.73%和1.96%㊂除此之外还包括少量的放线菌门(Actinobacteria )和拟杆菌门(Bacte-roidetes)微生物㊂而22d 组的鸡蛋干样品菌群结构以厚壁菌门㊁变形菌门和拟杆菌门为主,相对丰度分别为87.55%㊁9.41%和1.32%,同时还含有少量的放线菌门微生物㊂29d 组的鸡蛋干样品中微生物以厚壁菌门(70.94%)㊁放线菌门(27.81%)和变形菌门(1.09%)为主,还含有少量的拟杆菌门微生物㊂以上结果表明,在3组不同保质期的样品中,厚壁菌门的丰度均为最高,可见厚壁菌门是腐败变质鸡蛋干中的优势微生物㊂近年来许多研究表明,厚壁菌门微生物是高蛋白食品中的优势腐败菌,在乳制品㊁肉制品及水产品中都十分常见[18-21],这与本研究的结果一致㊂图3㊀基于门分类水平上的样品菌群分布图Fig.3㊀Bacterial distribution pattern at the phylum level2.4.2㊀基于科水平的细菌菌群结构分析基于科分类水平的3组鸡蛋干样品的微生物群落结构如图4所示㊂测序结果表明,15d 组的鸡蛋干样品中类芽孢杆菌科(Paenibacillaceae)所占比例最大,为47.43%,其次为芽孢杆菌科(Bacillaceae)和梭菌科(Clostridiaceae_1),所占比例分别为33.14%和14.96%,还有乳杆菌科(Lactobacillaceae)㊁伯克氏菌科(Burkholderiaceae)㊁链球菌科(Streptococcaceae)㊁假单胞菌科(Pseudomonadaceae)等相对丰度较低的菌种,说明鸡蛋干在制作和包装过程中污染微生物的种类复杂,导致样品菌落结构呈现多样性㊂22d 组的鸡蛋干样品菌群结构以芽孢杆菌科㊁乳杆菌科㊁链球菌科㊁伯克氏菌科为主,相对丰度分别为47.39%㊁29.11%和3.76%㊁3.29%,除此之外还包括少量的红杆菌科(Rhodobacteraceae)㊁假单胞菌科㊁类芽孢杆菌科微生物㊂29d 组鸡蛋干样品中的菌群在科水平上主要有芽孢杆菌科(68.85%)和小单孢菌科(Mi-cromonosporaceae,27.60%),还含有少量的假单胞菌科和伯克氏菌科微生物㊂以上结果表明,不同保质期鸡蛋干样品所含微生物种类和数量有明显差异,如相比于其他2组,29d 组样品中却未检出乳杆菌科微生物,可能由于该组样品的原料中及加工过程中污染的乳杆菌科较少㊂29d 组鸡蛋干样品的保质期最长,其微生物多样性低于其余2组,说明样品中污染的微生物种类和数量与鸡蛋干的保质期密切相关㊂22d 组样品微生物多样性最高,但此组保质期不是最短,可能由于样品中含有丰度较高的乳杆菌科微生物[22-23],对其他微生物产生拮抗作用㊁抑制其生长繁殖,相对延长了鸡蛋干保质期㊂此外,在15d 组样品中检测出梭菌科微生物,而其他2组中均未检出,梭菌科中有些菌种为厌氧菌,可在真空包装鸡蛋干中迅速生长繁殖,这可能是本组鸡蛋干保质期远小于其他2组的原因㊂3组不同保质期的样品中,均含有芽孢28㊀2020Vol.46No.24(Total 420)杆菌科微生物,且丰度较高,表明芽孢杆菌科是腐败变质鸡蛋干中的核心优势微生物㊂图4㊀基于科分类水平上的样品菌群分布图Fig.4㊀Bacterial distribution pattern at the family level2.4.3㊀基于属水平的细菌菌群结构分析选取在鸡蛋干样品丰度排名前15的属,根据其在每组鸡蛋干样品中的丰度信息聚类绘制成热图(图5-a)㊂热图可通过颜色变化直观地反映出各组鸡蛋干样品群落组成的相似性和差异性,更便于发现在各组样品中分布较多或较少的菌属[10]㊂图中颜色越偏砖红色,表明样品中所含该菌属比例越高,颜色越偏深蓝色,比例则越低㊂如图5-a 所示,在属水平,22d 组和29d 组聚为一类,15d 组单独聚为一类,说明22d 组和29d 组菌群整体结构相似度较高,与15d 组相似度较低,表明鸡蛋干样品的保质期与其菌群结构相关性较大㊂从图5-b 各样品菌群柱状分布图中可以看出,在属水平上,15d 组优势菌群主要是芽孢杆菌属(Bacillus ,33.14%)㊁短芽孢杆菌属(Brevibacillus ,30.96%)㊁类芽孢杆菌属(Paenibacillus ,16.46%)和狭义梭菌属(Clostridium _sensu _stricto _13,14.86%)㊂在优势菌群中,芽孢杆菌属所占比例最大,它属于需氧或兼性厌氧菌,此属中大部分细菌为腐生菌,广泛分布于自然环境中㊂该菌属可形成芽孢,对不良环境条件有很强的抵抗力,不易杀灭㊂贺燕等[3]在腐败鸡蛋干中也有检测到芽孢杆菌属微生物㊂短芽孢杆菌属为需氧或兼性厌氧菌,近年来相关报道多集中于该属工程菌株的研究,鲜见有研究发现其在食品腐败中的作用[24-25]㊂类芽孢杆菌属为需氧或兼性厌氧菌,近年来许多研究表明,其为多种乳制品和即食食品的腐败微生物[26-27]㊂狭义梭菌属广泛分布于土壤㊁下水污泥㊁人和其他哺乳动物的肠道等环境中,是乳制品和肉制品中常见的腐败微生物[20,28-29]㊂22d 组优势菌群主要是芽孢杆菌属(47.38%)㊁乳杆菌属(Lactobacillus ,28.26%)和链球菌属(Strep-tococcus ,3.57%)㊂乳杆菌属为兼性厌氧菌,广泛分布于自然界中,污染乳制品㊁肉制品㊁水产品等高蛋白食品后可引起腐败变质㊂链球菌属为兼性厌氧菌,该属中有少数种为腐生菌,污染食品后可引起腐败变质[29]㊂a -样品菌群相对丰度热图;b -样品菌群相对丰度柱形图图5㊀基于属分类水平上的样品菌群相对丰度热图和柱形图Fig.5㊀Heat map and stacked-column of relative abundances ofbacterial communities at the genus level29d 组优势菌属主要是芽孢杆菌属(68.85%)和小单孢菌属(Micromonospora ,27.60%)㊂小单孢菌属为好氧或微好氧的腐生菌,是土壤和水域的常见微生物群体[30],其在真空包装鸡蛋干中占比达到27.60%,可能由于鸡蛋干中初始污染小单孢菌属较多,且微好氧微生物能在较低的氧分压下生长,鸡蛋干虽然经过真空包装,但仍无法将样品中氧气完全排出,因此小单孢菌属中部分微好氧菌种可以生长㊂以上结果表明,在3组不同保质期的样品中,芽孢杆菌属的相对丰度均为最高,可见芽孢杆菌属是鸡蛋干的核心致腐微生物㊂保质期最长的29d组样品中优势菌属为芽孢杆菌属和小单孢菌属,其中小单孢菌属为好氧或微好氧菌,在真空包装鸡蛋干中生长缓慢,而芽孢杆菌属中部分菌种为兼性厌氧菌,可在真空包装条件下良好生长,因此芽孢杆菌属应为限制此组样品保质期的微生物㊂而相比于29d组,22d组样品中还含有较高丰度的乳杆菌属,这可能是造成本组鸡蛋干保质期<29d的原因㊂此外,狭义梭菌属仅在保质期为15d的鸡蛋干样品中检测到,它属于厌氧菌,因此在真空包装鸡蛋干中的生长速度大于非厌氧菌,可快速分解鸡蛋干中营养物质造成腐败变质,这可能是15d组鸡蛋干保质期远低于其他2组的原因㊂3㊀结论本研究以保质期不同的3组腐败变质鸡蛋干为研究对象,采用Illumina Miseq高通量测序技术分析样品中菌群组成情况㊂结果表明,3组腐败变质鸡蛋干的微生物的多样性以及丰度存在较大差异,而总体来看,厚壁菌门为腐败变质鸡蛋干的优势菌门,芽孢杆菌科为优势菌科㊂在属水平,芽孢杆菌属在3组样品中的相对丰度均为最高,是鸡蛋干的核心致腐微生物㊂此外,造成鸡蛋干保质期差异的菌属包括乳杆菌属和狭义梭菌属,它们可加速鸡蛋干的腐败变质,是鸡蛋干保质期的限制因素,应在杀菌防腐环节重点关注㊂高通量测序技术检测不同保质期鸡蛋干样品的微生物组成,不仅可以分析鸡蛋干腐败过程中的核心优势菌群,还可以高效快速地分析样品中微生物种类的差异,为今后鸡蛋干质量安全监测及靶向研究鸡蛋干防腐保鲜技术提供理论依据㊂参考文献[1]㊀GB2749 2015食品安全国家标准蛋与蛋制品[S].北京:中国标准出版社,2017.GB2749 2015National standards of food safety eggs and egg products[S].Beijing:China Standards Press,2017.[2]㊀薛艳蓉,茂文,赵瑞生,等.鸡蛋在储存过程中微生物及蛋品质的变化研究[J].畜牧与饲料科学,2017,38(8):67-69;72.XUE Y R,MAO W,ZHAO R S,et al.Dynamic changes of microbio-logical and qualitative features of chicken eggs during storage process [J].Animal Husbandry and Feed Science,2017,38(8):67-69;72.[3]㊀贺燕,谭纯良,李娜,等.复配防腐剂抑制鸡蛋干腐败菌效果研究[J].食品与机械,2019,35(11):159-164.HE Y,TAN C L,LI N,et al.Study on the effect of compound pre-servatives on inhibiting spoilage of egg curd[J].Food and Machinery, 2019,35(11):159-164.[4]㊀袁先铃,黄丹,潘明.腐败鸡蛋干中主要微生物的分离纯化及鉴定[J].食品研究与开发,2012,33(7):57-59.YUAN X L,HAUNG D,PAN M.Purification and identification of microbes in corruption dried eggcurd[J].Food Research and Devel-opment,2012,33(7):57-59.[5]㊀HANDELSMAN J.Metagenomics:Application of genomics to uncul-tured microorganisms[J].Microbiology and Molecular Biology Re-views,2004,68(4):669-685.[6]㊀吴燕燕,钱茜茜,李来好,等.基于Illumina MiSeq技术分析腌干鱼加工过程中微生物群落多样性[J].食品科学,2017,38(12):1-8.WU Y Y,QIAN Q Q,LI L H,et al.Microbial community diversity in dried-salted fish during processing revealed by Illumina MiSeq se-quencing[J].Food Science,2017,38(12):1-8.[7]㊀POOŁKA J,REBECCHI A,PISACANE V,et al.Bacterial diversityin typical Italian salami at different ripening stages as revealed by high-throughput sequencing of16S rRNA amplicons[J].Food Mi-crobiology,2015,46(4):342-356.[8]㊀ZHANG J B,LI Y,LIU X C,et al.Characterization of the microbialcomposition and quality of lightly salted grass carp(Ctenopharyng-odon idellus)fillets with vacuum or modified atmosphere packaging [J].International Journal of Food Microbiology,2019,293(6):87-93.[9]㊀ZANG J H,XU Y S,XIA W S,et al.Dynamics and diversity of mi-crobial community succession during fermentation of Suan yu,a Chi-nese traditional fermented fish,determined by high throughput se-quencing[J].Food Research International,2018,111(9):565-573.[10]㊀戴玲瑛,齐曼婷,王立梅,等.虾酱中间产物细菌群落结构及多样性[J].食品科学,2018,39(22):173-178.DAI L Y,QI M T,WANG L M,et al.Bacterial community struc-ture and diversity in unsterilized shrimp sauce[J].Food Science,2018,39(22):173-178.[11]㊀孙同辉,陈洁,李雪琴,等.杀菌方式对年糕微生物多样性及品质的影响[J/OL].食品科学,2020./kc-ms/detail/11.2206.TS.20200109.0934.012.html.SUN T H,CHEN J,LI X Q,et al.Effect of sterilization method onmicrobial diversity and quality of rice cake[J/OL].Food Science,2020./kcms/detail/11.2206.TS.20200109.0934.012.html.[12]㊀SOON W W,HARIHARAN M,SNYDER M P.High-throughput se-quencing for biology and medicine[J].Molecular Systems Biology,2013,9(1):640.[13]㊀张伯男,樊晓盼,马俪珍,等.风味鸡蛋干的加工工艺研究[J].食品研究与开发,2018,39(11):101-105.ZHANG B N,FAN X P,MA L Z,et al.Study on processing tech-nology of flavor egg curd[J].Food Research and Development,2018,39(11):101-105.[14]㊀高乾坤,焦琳舒,杜贺超,等.高通量测序分析不同产地带鱼冷藏时微生物群落多样性[J].食品科学,2018,39(18):127-132.GAO Q K,JIAO L S,DU H C,et al.High-throughput sequencingdifferent waters during chilled storage[J].Food Science,2018,39(18):127-132.[15]㊀ANDREEVSKAYA M,JOHANSSON P,LAINE P,et al.Genome2020年第46卷第24期(总第420期)29㊀30㊀2020Vol.46No.24(Total 420)sequence and transcriptome analysis of meat-spoilage-associated lactic acid bacterium Lactococcus piscium MKFS47[J].Applied andEnvironmental Microbiology,2015,81(11):3800-3811.[16]㊀LU X M,LU P Z.Characterization of bacterial communities in sedi-ments receiving various wastewater effluents with high-throughput se-quencing analysis[J].Microbial Ecology,2014,67(3):612-623.[17]㊀江杨阳.小龙虾低温贮藏品质变化规律及其腐败菌鉴定[D].杭州:浙江大学,2019.JIANG Y Y.Studies on the quality changes of red swamp crayfish and its specific spoilage organisms during low temperature storage[D].Hangzhou:Zhejiang University,2019.[18]㊀马岩石,姜明,李慧,等.基于高通量测序技术分析东北豆酱的微生物多样性[J].食品工业科技,2020,41(12);100-105.MA Y S,JIANG M,LI H,et al.Analysis of microbial diversity of northeast soy sauce based on high-throughput sequencing technolo-gy[J].Science and Technology of Food Industry,2020,41(12):100-105.[19]㊀李春生,王悦齐,李来好,等.传统鱼露发酵过程中细菌群落演替及对其挥发性风味形成的影响分析[J].食品科学,2018,39(24):85-92.LI C S,WANG Y Q,LI L H,et al.Bacterial community succes-sion during fermentation of Chinese fish sauce and its effect on for-mation of volatile flavor components[J].Food Science,2018,39(24):85-92.[20]㊀毛海萍,袁开,金仁耀,等.基于传统分离培养和高通量测序分析市售咸鳓鱼中微生物多样性[J].食品研究与开发,2019,40(21):193-201.MAO H P,YUAN K,JIN R Y,et al.Study of microbial diversity in commercial salted Chinese herring using culture isolation and high-throughput sequencing[J].Food Research and Development,2019,40(21):193-201.[21]㊀李伟程,侯强川,于洁,等.传统发酵乳制品中微生物多样性研究[J].食品工业科技,2018,39(1):131-136.LI W C,HOU Q C,YU J,et al.Study on the diversity of microbi-al in traditional fermented dairy products[J].Science and Technol-ogy of Food Industry,2018,39(1):131-136.[22]㊀孙群,易华西,张冬,等.潜在益生菌的筛选及抑菌功能特性评价[J].食品科技,2019,44(12):1-5.SUN Q,YI H X,ZHANG D,et al.Screening of potential probiot-ics and evaluation of antibacterial function characteristics[J].FoodScience and Technology,2019,44(12):1-5.[23]㊀王玉文,刘英华.乳酸菌的生物拮抗作用[J].中国乳品工业,2008,36(2):55-57.WANG Y W,LIU Y H.State antagonism about lactic acid bacteria [J].China Dairy Industry,2008,36(2):55-57.[24]㊀赵乃昕,苑广盈.医学细菌名称及分类鉴定[M].济南:山东大学出版社,2013.ZHAO N X,YUAN G and Classification of Medical Bac-teria[M].Jinan:Shandong University Press,2013.[25]㊀邹亮,吴敬,陈晟.重组蔗糖异构酶在短短芽孢杆菌中的表达及发酵优化[J].食品与生物技术学报,2019,38(1):22-28.ZOU L,WU J,CHEN S.Expression and fermentation optimization of recombinant sucrose isomerase in Brevibacillus choshinensis [J].Journal of Food Science and Biotechnology,2019,38(1):22-28.[26]㊀HELMOND M,NIEROP GROOT M N,HERMIEN B V D V.Characterization of four Paenibacillus species isolated from pasteur-ized,chilled ready-to-eat meals[J].International Journal of FoodMicrobiology,2017,252(13):35-41.[27]㊀TRMCIC A,MARTIN N H,BOOR K J,et al.A standard bacterialisolate set for research on contemporary dairy spoilage[J].Journal of Dairy Science,2015,98(8):5806-5817.[28]㊀AKHTAR S,PAREDES-SABJA D,TORRES J A,et al.Strategyto inactivate Clostridium perfringens spores in meat products [J].Food Microbiology,2009,26(3):272-277.[29]㊀何国庆,贾英民,丁立孝.食品微生物学[M].北京:中国农业大学出版社,2009.HE G Q,JIA Y M,DING L X.Food Microbiology[M].Beijing:China Agricultural University Press,2009.[30]㊀杨苏声.细菌分类学[M].北京:中国农业大学出版社,1997.YANG S S.Bacterial Taxonomy [M ].Beijing:China AgriculturalUniversity Press,1997.。
第 32 卷 第 7 期Vol.32,No.796-1082023 年 7 月草业学报ACTA PRATACULTURAE SINICA 张振粉, 黄荣, 李向阳, 等. 基于Illumina MiSeq 高通量测序的燕麦种带细菌多样性及功能分析. 草业学报, 2023, 32(7): 96−108.ZHANG Zhen -fen , HUANG Rong , LI Xiang -yang , et al . Seed -borne bacterial diversity of oat and functional analysis based on Illumina MiSeq high -throughput sequencing. Acta Prataculturae Sinica , 2023, 32(7): 96−108.基于Illumina MiSeq 高通量测序的燕麦种带细菌多样性及功能分析张振粉1,2*,黄荣1,2,李向阳1,2,姚博1,2,赵桂琴1,2(1.甘肃农业大学草业学院,甘肃 兰州 730070;2.草业生态系统教育部重点实验室,中-美草地畜牧业可持续发展研究中心,甘肃 兰州 730070)摘要:采用高通量测序法,分析 7 份来自不同地区的燕麦种带细菌群落的细菌丰度、Alpha 多样性、Beta 多样性和物种组成差异,并采用 PICRUSt 和FAPROTAX 功能预测的方法分析了各种带细菌间的丰度差异。
结果显示:1)7份燕麦中共获得5187个细菌可操作分类单元(OTUs ),主要归属于33个门、180个目和435个属。
2)基于OTUs 的丰度及注释信息,7份燕麦种带细菌群落中变形菌门、绿弯菌门、酸杆菌门、放线菌门和蓝菌门为优势菌门,立克次氏体目和鞘脂单胞菌目为优势菌目,鞘氨醇单胞菌属、Solibacter 和假节杆菌属为优势菌属。
3) Alpha 多样性和Beta多样性表明不同燕麦品种的细菌群落多样性和群落结构具有较大差异,其中 LXY -5 具有最大的Alpha 多样性。
基于Miseq测序技术分析脑缺血对大鼠肠道菌群多样性的相
关作用
张慧楠;孙雅涵;高枫;罗文哲;张慧明
【期刊名称】《黑龙江医药科学》
【年(卷),期】2022(45)5
【摘要】目的:研究脑缺血对肠道菌多样性的相关作用。
方法:将6只身体状况相似的雄性大鼠随机分为3组:对照组、双侧颈动脉结扎3d组、双侧颈动脉结扎7d组,每组2只,待实验操作结束后分别取其粪便,多次进行实验并基于Miseq方法测序分析。
结果:肠道微生物测序合理,脑缺血可导致大鼠OTUS组成变化与Alpha水平多样性的失调,其样品中厚壁菌门等丰度降低,而拟杆菌、变形杆菌及一些未分类菌群数目上升,且随结扎天数越多,其变化更为明显,且差异有统计学意义(P<0.05)。
结论:脑缺血导致肠道菌群的丰富度和多样性发生变化,这种变化可使有益菌多样性及丰度下降,有害菌及未分类的细菌多样性及丰度上升。
【总页数】2页(P88-89)
【作者】张慧楠;孙雅涵;高枫;罗文哲;张慧明
【作者单位】佳木斯大学临床医学院;佳木斯大学基础医学院
【正文语种】中文
【中图分类】R-33
【相关文献】
1.应用Illumina-MiSeq高通量测序技术分析脱氧雪腐镰刀菌烯醇对小鼠肠道菌群的影响
2.基于16S rRNA测序技术初步探讨多囊卵巢综合征大鼠肠道菌群多样性
3.基于Illumina Miseq测序技术分析剁辣椒产地预加工过程中菌群变化
4.基于16S rDNA测序技术分析利福平致大鼠肝损伤中肠道菌群变化特征
5.基于Illumina MiSeq测序技术分析大鲵肉冷藏过程中微生物菌群演替规律
因版权原因,仅展示原文概要,查看原文内容请购买。
采用 Illumina MiSeq测序技术分析断奶幼兔盲肠微生物群落的多样性许宇静;张煜坤;沈雪梅;李晶;徐秀容【期刊名称】《动物营养学报》【年(卷),期】2015(027)009【摘要】本试验旨在采用Illumina MiSeq测序技术分析断奶幼兔盲肠微生物群落的多样性. 选取30窝同日出生、胎次相同和平均窝重相近的健康新生仔兔,于14 日龄开始补饲,断奶当日( 30日龄)从30窝仔兔中选取48只体重相近的健康幼兔作为试验兔,饲喂不添加抗生素的基础饲粮. 分别于断奶当日及断奶后1、2和3周随机选取6只健康幼兔屠宰,无菌采集盲肠内容物,从中选取3个样本提取微生物基因组总DNA,采用Illumina MiSeq测序技术检测其中的微生物群落多样性. 结果显示:1 )健康幼兔盲肠微生物群落α 多样性指数指标( ACE 值、Chao1 值、Shannnon值)随日龄的增长有升高的趋势(P<0.10). 2)不同日龄健康幼兔盲肠微生物各菌门的相对丰度差异不显著( P>0.05) ,且均主要由厚壁菌门、拟杆菌门、变形菌门和疣微菌门组成;在科水平上,厚壁菌门主要由瘤胃球菌科和毛螺旋菌科组成,拟杆菌门主要由拟杆菌科和紫单胞菌科组成;在属水平上,拟杆菌属、Barnesiella、Akkermansia、普雷沃氏菌属和4种未知菌属为优势菌属;拟杆菌属和Parabacteroides在断奶当天的相对丰度较高,且与其他日龄组差异显著( P<0.05).3)健康幼兔盲肠中均存在乳杆菌属,且相对丰度在各日龄组间差异不显著( P>0.05). 由此得出,断奶时健康幼兔盲肠微生物区系已基本建立,相对丰度最大的优势菌属为拟杆菌属,但断奶后随着日龄的增长,微生物组成趋于复杂.%The current study was conducted to evaluate the caecal microbiota diversity of weaningyoung rabbits by using Illumina Miseq sequencing technology. Thirty litters of newborn healthy rabbits with the same birthday and parity and similar average body weight were used, and forty-eight healthy rabbits with similar body weight were selected on the day of weaning ( 30 days of age) . No antibiotics were used in the basal diet and all rabbits were fed restrictedly from 14 days of age to the end of the trial. Six healthy young rabbits were randomly se-lected and slaughtered on the day of weaning and 1, 2 and 3 weeks after weaning, respectively. Three samples of caecal content at each sampling time were collected for extracting the microbial genomic total DNA. Micro-biota diversity of caecal contents was evaluated using Illumina Miseq sequencing technology. The results showed as follows:1) the alpha diversity indexes ( ACE value, Chao1 value and Shannnon value) of caecal microbiota of healthy young rabbits were tended to rise with age increasing ( P<0.10) . 2) The caecal microbi-ota of healthy young rabbits at different age was mainly consisted of Firmicutes, Bacteroidetes, Proteobacteria and Verrucomicrobia at the phylum level, and their relative abundances were not significantly affected by age( P>0.05) . Ruminococcaceae and Lachnospiraceae were the dominant families in Fimicutes, and Bacteroidetes was predominantly composed by Bacteroidaceae and Porphyromonadaceae. Bacteroides, Barnesiella, Akker-mansia , Prevotella and four unclassified genus were the common dominant genera at the genera level. The rela-tive abundances of Bacteroides and Parabacteroides on the day of weaning were higher which significantly higher than those of other ages (P<0.05). 3) Lactobacillusexisted in the caecum of healthy young rabbits, and its relative abundance was not significantly changed by age ( P>0.05) . Base on the results, it is concluded that the caecal microflora has been basically established and the relative abundance of Bacteroides is the highest at weaning, but microbial components tend to be complex after weaning with age increasing.【总页数】10页(P2793-2802)【作者】许宇静;张煜坤;沈雪梅;李晶;徐秀容【作者单位】西北农林科技大学动物科技学院,杨凌 712100;西北农林科技大学动物科技学院,杨凌 712100;西北农林科技大学动物科技学院,杨凌 712100;西北农林科技大学动物科技学院,杨凌 712100;西北农林科技大学动物科技学院,杨凌712100【正文语种】中文【中图分类】S826【相关文献】1.应用Illumina MiSeq高通量测序技术分析饲粮添加丁酸钠对蛋雏鸡盲肠微生物的影响 [J], 张李荣;杨海明2.应用Illumina MiSeq高通量测序技术分析饲粮添加乳双歧杆菌对蛋雏鸡盲肠微生物的影响 [J], 张李荣;杨海明;龚道清;肖霞;唐徐华;王志跃3.基于Illumina MiSeq高通量测序技术分析不同保质期鸡蛋干的微生物群落多样性 [J], 宋思家;黄子彧;林莹莹;郭慧媛4.应用Illumina MiSeq高通量测序技术分析来凤地区腌制大头菜叶中微生物多样性 [J], 朱俊喆;邓晓茜;胡晓淞;李云捷;赵慧君5.基于Illumina MiSeq高通量测序分析清香型白酒酒糟微生物群落多样性 [J], 罗爱国;郗鑫瑞;郑同庆;杨兆艳;田艳花;马晓丽;赵红梅因版权原因,仅展示原文概要,查看原文内容请购买。
MiSeq System Highlights• Exceptional Data QualityHighest-quality data demonstrated through peer-reviewed,scientific comparison• Simple and Intuitive Instrument WorkflowHighly automated system features a simple,easy-to-use instrument interface• Fastest Turnaround TimeMost rapid sequencing and variant detection fortime-critical studies• Extensive Suite of ApplicationsAdjustable read length and flow cell options provide ultimateflexibility across a broad range of applicationsIntroductionThe MiSeq System offers the first end-to-end sequencing solution, integrating cluster generation, amplification, sequencing, and data analysis into a single instrument. Its small footprint—approximately2 square feet—fits easily into virtually any laboratory environment (Figure 1). The MiSeq System leverages Illumina sequencing by synthesis technology (SBS), the most widely used, next-generation sequencing chemistry. With over 750 publications to date, the MiSeq System is the ideal platform for rapid and cost-effective genetic analysis.Exceptional Data QualityIllumina SBS chemistry is the most widely adopted next-generation sequencing technology. Exceptional data quality is achieved by SBS chemistry: a proprietary, reversible terminator-based method that detects single bases as they are incorporated into massively parallel DNA strands. Fluorescent terminator dyes are imaged as each dNTP is added and then cleaved to allow incorporation of the next base. With all 4 reversible, terminator-bound dNTPs present during each cycle, natural competition minimizes incorporation bias. Base calls are made directly from signal intensity measurements during each cycle, greatly reducing raw error rates compared to other technologies.1–5 The result is highly accurate base-by-base sequencing that virtually eliminates sequence context-specific errors, even within repetitive sequence regions or homopolymers. Illumina sequencing deliversthe highest yield of error-free data for the most sensitive or complex sequencing samples (Figure 3).Simple and Intuitive Instrument WorkflowThe MiSeq System offers straightforward, easy-to-follow instrument control software. Perform simple instrument operations with an intuitive touch screen interface, use plug-and-play reagent cartridges with RFID tracking, consult on-screen video tutorials, and enjoy step-by-step guides throughout each sequencing workflow.All MiSeq Systems include onboard data analyis and access to BaseSpace®— the Illumina genomic analysis platform. BaseSpace provides real-time data uploading, simple data analysis tools, internet-based run monitoring, and a secure, scalable storage solution. A suite of data analysis tools, and a growing list of third-party BaseSpace Apps, empowers researchers to perform their own informatics. BaseSpace also enables fast and easy data sharing with colleagues orcustomers. To learn more, visit /basespace.Fast Turnaround TimeFor results in hours rather than days, the combination of rapid library preparation and the MiSeq System delivers a simple, accelerated turnaround time (Figure 2). Prepare your sequencing library in just 90 minutes with Nextera® library prep reagents, then move to automated clonal amplification, sequencing, and quality-scored base calling in as little as 4 hours on the MiSeq instrument. Sequence alignment can be completed directly on the onboard instrument computer with MiSeq Reporter software or through the BaseSpace platform within 3 hours.MiSeq® SystemFocused power. Speed and simplicity for targeted resequencing and small-genome sequencing.Figure 1: MiSeq SystemThe compact MiSeq System is well suited for rapid, cost-effective next-generation sequencing.Extensive Suite of ApplicationsExplore an ever increasing range of sequencing applications. With faster turnaround time and simplified workflows, the MiSeq System offers a cost-effective alternative to capillary electrophoresis (CE) for applications such as targeted resequencing, clone checking, and amplicon sequencing. Optimized analysis workflows are also available for small genome sequencing, 16S metagenomics, RNA sequencing, HLA sequencing, forensics, preimplantation genetic screening (PGS), and preimplantation genetic diagnosis (PGD), as well as highly multiplexed applications such as TruSeq® Custom Amplicon and TruSeq Custom Enrichment. Adjustable read lengths, flow cell options, and choice of single or paired-end reads allow unprecedented flexibility for matching data output to a broad range of experimental needs. MiSeq System SpecificationsInstrument ConfigurationRFID tracking for consumablesMiSeq Control SoftwareMiSeq Reporter SoftwareInstrument Control Computer (Internal)*Base Unit: Intel Core i7-2710QE 2.10 GHz CPUMemory: 16 GB RAMHard Drive: 750 GBOperating System: Windows 7 embedded standard*Computer specifications are subject to change.Operating EnvironmentTemperature: 22°C ± 3°CHumidity: Noncondensing 20%–80%Altitude: Less than 2,000 m (6,500 ft)Air Quality: Pollution degree rating of IIVentilation: Maximum of 1,364 BTU/hFor Indoor Use OnlyLight Emitting Diode (LED)530 nm, 660 nmDimensionsW×D×H: 68.6 cm × 56.5 cm × 52.3 cm (27.0 in × 22.2 in × 20.6 in) Weight: 57.2 kg (126 lbs)Crated Weight: 93.6 kg (206 lbs)Power Requirements100–240V AC @ 50/60Hz, 10A, 400 WRadio Frequency Identifier (RFID)Frequency: 13.56 MHzPower: 100 mWProduct Safety and ComplianceNRTL certified IEC 61010-1CE markedFCC/IC approvedLearn MoreTo learn more about the next revolution in desktop sequencing, visit: /miseqReferences1. Junemann S, Sedlazeck FJ, Prior K, Albersmeier A, John U, Kalinowski J, etal. Updating benchtop sequencing performance comparison. Nat Biotechnol . 2013;31:294-296.2. Ross MG, Russ C, Costello M, Hollinger A, Lennon NJ, Hegarty R, et al.Characterizing and measuring bias in sequence data. Gen Biol . 2013;14:R51.3. Loman NJ, Misra RV , Dallman TJ, Constantinidou C, Gharbia SE, Wain J,et al. Performance comparison of benchtop high-throughput sequencing platforms.Nat Biotechnol . 2012;30:434-439.4. Quail MA, Smith M, Coupland P , Otto TD, Harris SR, Connor TR, et al. A taleof three next generation sequencing platforms: comparison of Ion Torrent, Pacific Biosciences and Illumina MiSeq sequencers. BMC Genomics . 2012;13:341.5. Liu L, Li Y , Li S, Hu N, He Y , Pong R, et al. Comparison of Next-Generationsequencing systems.J Biomed Biotechnol . 2012;2012:251364.Ordering InformationInstrument Name Catalog No.MiSeq SystemSY-410-1003Illumina • 1.800.809.4566 toll-free (U.S.) • +1.858.202.4566 tel • techsupport@ • For Research Use Only. Not for use in diagnostic procedures.© 2015 Illumina, Inc. All rights reserved. Illumina, BaseSpace, MiSeq, Nextera, TruSeq, and the pumpkin orange color are trade-marks or registered trademarks of Illumina, Inc and/or its affiliates in the U.S. and/or other countries.Pub. No. 770-2011-001 Current as of 17 May 2016。