无乳链球菌的检测试剂盒、检测方法与相关技术
- 格式:pdf
- 大小:114.44 KB
- 文档页数:5

A群链球菌抗原检测试剂盒(胶体金法)使用说明书【产品名称】通用名称:A群链球菌抗原检测试剂盒(胶体金法)英文名称:Strep A Test【包装规格】25人份/盒。
【预期用途】A群链球菌抗原检测试剂盒(胶体金法)是一种快速免疫层析试验,用于定性检测咽拭子标本中的A群化脓性链球菌抗原。
A群链球菌是引起咽炎的主要病原体。
对该致病原做出准确诊断是正确治疗这种疾病的重要条件。
链球菌感染后要选用抗生素治疗,如果未经治疗,可能会引起诸如风湿热之类的严重后遗症1。
检测A群链球菌感染的传统方法是将咽拭子标本培养24-48小时后,证实β溶血菌落是A群链球菌2。
阳性结果可通过观察羊血琼脂平板上微生物对杆菌肽的敏感性而获得3,4。
基于碳水化合物抗原群特异性可对A群链球菌做出免疫学诊断5。
A群链球菌抗原检测试剂盒(胶体金法)使用的是免疫层析方法,所用抗体对A群链球菌是特异的。
试验设计可对抗原在试剂上进行提取,没有转移步骤,标本完全包含在检测卡中,结果6分钟可得。
【检验原理】A群链球菌抗原检测试剂盒(胶体金法)是一种检测咽拭子标本中A群化脓性链球菌抗原的薄膜免疫层析试验。
一条吸附到硝酸纤维素膜上的兔抗A群链球菌抗体带,作为样本线,另一条吸附到同一膜上的兔抗种属抗体,作为对照线。
结合有可视粒子的兔抗A群链球菌抗体和种属抗体干燥结合到惰性纤维支持物上,形成的结合物垫与带条带的薄膜结合在一起构成检测条。
检测条和一个放咽拭子的小孔位于书形检测卡的两侧。
试验时,将咽拭子插入检测卡中,从滴瓶中加入提取试剂,将咽拭子转三圈。
温育1分钟后,将检测卡闭合,使提取的标本与检测条接触。
A群链球菌抗原被固定的A群链球菌抗体捕获,并与结合物抗体结合。
固定的抗种属抗体捕获另一可视结合物,阳性结果5分钟内出现,5分钟读数时的阴性结果表明不存在A群链球菌抗原。
试验结果可用紫红色线的存在与否来解释。
阳性结果会出现样本线和对照线两条线,阴性结果只出现一条对照线,其他检测结果表示试验无效。

停乳链球杆菌检测标准一、引言停乳链球菌是一种重要的动物和人类致病菌,可引起各种感染性疾病,如乳腺炎、败血症和肺炎等。
为了有效控制和预防停乳链球菌的传播和感染,建立一套准确的检测标准至关重要。
本篇文章将详细介绍停乳链球菌的检测标准,包括检测方法的建立、样本采集与处理、检测方法验证以及检测标准的应用。
二、停乳链球菌概述停乳链球菌是一种革兰氏阳性球菌,属于链球菌属。
与乳链球菌相比,停乳链球菌对牛、羊等家畜以及人类具有更强的致病性。
其传播途径主要包括直接接触感染部位或污染物,也可通过飞沫传播。
停乳链球菌感染可引起急性炎症反应,导致高热、咳嗽等症状,严重时可引发败血症和死亡。
三、检测方法的建立建立停乳链球菌的检测方法主要包括培养法、免疫学方法和分子生物学方法等。
其中,培养法是最常用的方法,通过分离培养停乳链球菌并对菌落进行鉴定,可以准确诊断感染。
免疫学方法包括血清学检测和抗原检测,具有较高的特异性和敏感性。
分子生物学方法利用基因探针和PCR技术等手段进行检测,具有快速、准确和灵敏度高的特点。
四、样本采集与处理在采集样本时,应根据感染部位的不同选择相应的样本类型,如血液、痰液、分泌物等。
采集后的样本应尽快送至实验室进行检测,以免延误最佳检测时机。
在处理样本时,应遵循无菌操作原则,避免交叉污染。
样本需要进行预处理、增菌和分离培养等步骤,以便进一步进行鉴定和检测。
五、检测方法验证为了确保检测方法的准确性和可靠性,需要进行充分的验证。
验证内容包括方法的特异性、敏感性、重复性和临床符合率等。
特异性是指检测方法对停乳链球菌的识别能力,确保不与其他菌种发生交叉反应;敏感性是指检测方法能够检出低浓度停乳链球菌的能力;重复性是指在不同时间、不同实验条件下,相同样本检测结果的稳定性;临床符合率是指检测结果与临床诊断的一致性。
通过以上验证指标的综合评估,可以确定检测方法的可靠性,为实际应用提供依据。
六、检测标准的应用应用停乳链球菌的检测标准时,需要考虑实验室条件、试剂和设备的选择以及实验操作流程的规范。


doi: 10.7541/2017.100鱼源无乳链球菌的血清型及分子分型研究张雨薇1耿毅1余泽辉1汪开毓1陈德芳2向正刚1李亚军1郭坤宁1牟维豪1(1. 四川农业大学动物医学院, 温江 611130; 2. 四川农业大学水产系, 温江 611130)摘要: 为了解不同鱼源无乳链球菌(Streptococcus agalactiae)分子分型特征, 分析菌株之间的相关性和同源性,研究在采用S. agalactiae特异性cfb基因对分离菌株鉴定的基础上, 对26株不同鱼源S. agalactiae进行了荚膜多糖血清型(CPS)多重PCR鉴定, 同时采用多位点序列分型(MLST)和脉冲场凝胶电泳(PFGE)进行分子特征比较与同源性分析。
结果显示, 26株菌CPS型存在Ia (n=22)和III型(n=4)两种类型, 其中黄河裸裂尻鱼源和罗非鱼源菌株均为Ia血清型, 齐口裂腹鱼源菌株存在Ia (n=2)和III (n=4)型两种CPS型; 多位点序列分型共得到3种STs序列型(ST-891、ST-103、ST-19), 其中黄河裸裂尻鱼源和罗非鱼源菌株均为新的序列型ST-891, 齐口裂腹鱼源菌株存在ST-103和ST-19两种STs型; PFGE聚类分析可分为16个PFGE谱型(A-P), 其中优势带型为P型(n=9)。
相同荚膜多糖血清型—MLST分型菌株在PFGE带型中呈现高度聚集。
CPS分型与MLST分型、PFGE分型有很好的相关性, 而CPS型、STs序列型、PFGE带型与宿主的来源没有明显的相关性。
不同鱼源S.agalactiae分子特征的相似性, 提示其存在交叉感染的可能性, 而齐口裂腹鱼源S. agalactiae分子特征的多样性, 提示其存在遗传变异的情况。
关键词: 无乳链球菌; 荚膜多糖血清分型; 多位点序列分型; 脉冲场凝胶电泳中图分类号: S941.41 文献标识码: A 文章编号: 1000-3207(2017)04-0800-07无乳链球菌(Streptococcus agalactiae), 属于链球菌科(Streptococcace), 链球菌属(Streptococcus),也称B群链球菌(Group B Streptococcus, GBS), 是兼性厌氧G+的链状球菌。
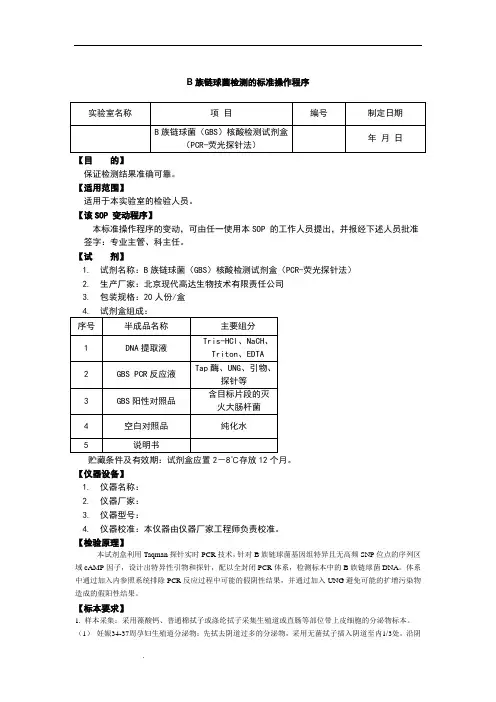
B族链球菌检测的标准操作程序【目的】保证检测结果准确可靠。
【适用范围】适用于本实验室的检验人员。
【该SOP 变动程序】本标准操作程序的变动,可由任一使用本SOP 的工作人员提出,并报经下述人员批准签字:专业主管、科主任。
【试剂】1.试剂名称:B族链球菌(GBS)核酸检测试剂盒(PCR-荧光探针法)2.生产厂家:北京现代高达生物技术有限责任公司3.包装规格:20人份/盒贮藏条件及有效期:试剂盒应置2-8℃存放12个月。
【仪器设备】1.仪器名称:2.仪器厂家:3.仪器型号:4.仪器校准:本仪器由仪器厂家工程师负责校准。
【检验原理】本试剂盒利用Taqman探针实时PCR技术,针对B族链球菌基因组特异且无高频SNP位点的序列区域cAMP因子,设计出特异性引物和探针,配以全封闭PCR体系,检测标本中的B族链球菌DNA。
体系中通过加入内参照系统排除PCR反应过程中可能的假阴性结果,并通过加入UNG避免可能的扩增污染物造成的假阳性结果。
【标本要求】1. 样本采集:采用藻酸钙、普通棉拭子或涤纶拭子采集生殖道或直肠等部位带上皮细胞的分泌物标本。
(1)妊娠34-37周孕妇生殖道分泌物:先拭去阴道过多的分泌物,采用无菌拭子插入阴道至内1/3处,沿阴道壁轻轻旋转取得分泌物,将采集的拭子置于无菌管中,密闭送检;(2)妊娠34-37周孕妇直肠分泌物:将拭子插入肛门,在肛门括约肌以上约2.5cm处,沿肠壁轻轻旋转取得标本,将采集的拭子置于无菌管中,密闭送检;(3)样本一经送检,则应尽快送至检测实验室,运输应符合当地法规。
2. 标本存放:待测样本在2-8℃保存不应超过24小时;-18℃以下保存不超过4天。
样本的冻融次数:实验数据证明,样本冻融6次以内无影响,但应尽量避免冻融。
3. 样本的运输条件:冷冻(干冰运输)4天内无影响,冷藏(冰袋运输)2天内无影响。
4. 含血样本无法正常检测应避免。
5. 研究显示,常用栓剂药物“保妇康栓”会对试剂盒检测造成较大影响,因此取样前应避免该药品的使用。

乳酸链球菌素检测标准
1. 范围
本标准规定了乳酸链球菌素的检测方法、检测流程、检测结果判定、检测报告及注意事项。
本标准适用于乳酸链球菌素的检测。
2. 规范性引用文件
下列文件对于本文件的应用是必不可少的。
凡是注日期的引用文件,仅注日期的版本适用于本文件。
凡是不注日期的引用文件,其最新版本(包括所有的修改单)适用于本文件。
GB 4789.15 食品卫生微生物学检验霉菌和酵母计数
3. 术语和定义
下列术语和定义适用于本文件。
3.1 乳酸链球菌素nisin
由乳酸链球菌产生的一种天然生物活性物质,是一种高效、无残留、无毒的食品添加剂,主要用于延长食品的保质期。
4. 检测方法
4.1 试剂和材料
4.1.1 nisin标准品:纯度≥98%。
4.1.2 培养基:平板计数琼脂培养基。
4.1.3 试剂:无菌生理盐水。
4.2 仪器和设备
4.2.1 无菌操作台:使用前需经过灭菌处理。
4.2.2 电子天平:感量为0.01g。
4.2.3 显微镜:用于观察菌落形态。
4.2.4 培养箱:温度可调,用于细菌的培养。
4.3 样品处理
4.3.1 将待测样品按照无菌操作要求进行处理,获取无菌的液体样品。
4.3.2 将无菌的液体样品进行稀释,制备成适合测定的浓度。
4.4 检测步骤
4.4.1 将nisin标准品用无菌生理盐水配制成一定浓度的nisin溶液。
4.4.2 将nisin溶液与培养基混合均匀,制备成含nisin的培养基。


牛无乳链球菌的危害、检测方法及防治夏良华【期刊名称】《中国畜牧兽医文摘》【年(卷),期】2016(032)008【总页数】1页(P51)【作者】夏良华【作者单位】内蒙古赤峰市巴林左旗碧流台镇动物防疫站,内蒙古赤峰 025458【正文语种】中文无乳链球菌是引起奶牛乳腺炎的主要病原菌之一。
乳腺炎是严重危害畜群生产的一类疾病,它虽不能造成大批的畜群死亡,却是影响全世界奶牛产业最重大的经济问题之一,奶牛乳腺炎的发生直接影响着牛奶质量,从而严重地威胁着人类的健康。
马保臣等的研究表明,在无乳链球菌的16SrRNA和23SrRNA之间的区域设计3对引物,建立了检测无乳链球菌的多重PCR方法。
结果表明,利用多重PCR培养法对无乳链球菌的检测与传统的细菌培养法的检测的结果基本一致,敏感性差异不显著,用PCR方法鉴定细菌,选取的扩增片段是保守的,但在不同种的水平上是有一定差异的。
贾玉萍等建立了巢式PCR检测无乳链球菌的方法,她们利用了链球菌属16SrRNA 基因的保守区设计通用引物作为链球菌属的阳性控制,在保守区内的可变区设计无乳链球菌的特异的引物。
通过DNA提取、引物设计与合成、PCR扩增、敏感性实验、无乳链球菌检测等一系列步骤,得出扩增后均产生明显的目的条带,用含无乳链球菌101、102和103CFU/ml的奶样过夜培养后,通用引物和特异引物PCR扩增结果均为阳性,这充分说明了此方法具有较高的灵敏性和特异性。
无乳链球菌分离培养所需的条件比较严格,操作不当就会造成假阴性的发生而妨碍治疗,上述的方法可以解决这些问题,利用链球菌属的16SrRNA基因,在保守区设计出通用引物可以更好地进行检测,从而正确的指导临床用药。
另一方面,无乳链球菌感染的初期不引起明显的变化,而用此方法经过夜培养后,可检测到1CFU/ml的链球菌,这对于早期预防和监测都具有十分重要的意义。
目前,临床上多用常规的CAMP法来检测无乳链球菌,这样不仅费时费力,而且还不能满足临床快速检测的需要。


收稿日期:2009-03-25基金项目:科技部国际合作项目(2006DFA32380);天津市农业科学院院偿基金项目(05011)作者简介:王 永(1977-),男,天津人,助理岩究员,硕士,主要从事农产品安全质量分子检测技术研究。
通讯作者:程 奕(1963-),女,安徽歙县人,研究员,硕士,主要从事农产品质量检测技术研究。
LAMP 检测无乳链球菌方法的建立及应用王 永1,赵 新1,景海春2,兰青阔1,朱 珠1,程 奕5(1.天津市农业科学院中心实验室,天津 300381;2.Plant Pathogen Interactions Division ,R othamsted Research ,A L52JQ UK ) 摘要:以无乳链球菌纤连蛋白fbs 基因为主要研究对象,采取环介导等温扩增技术(Loop 2Mediated Is othermal Ampli 2fication ,LAMP ),针对fbs 基因的6个区域设计4条特异性引物,利用一种链置换DNA 聚合酶(Bst DNA polymerase )在63℃保温1h ,通过荧光显色即可完成对无乳链球菌的检测工作。
结果显示,LAMP 方法能够特异性检测fbs 基因,其检测灵敏度是常规PCR 方法的100倍,并与实时荧光定量PCR 方法相当。
所建立的针对无乳链球菌fbs 基因的LAMP 检测方法具有高度的特异性及稳定性,结果可靠,非常适合无乳链球菌的快速检测。
关键词:环介导等温扩增技术;无乳链球菌;fbsB ;检测中图分类号:S852.61 文献标识码:A 文章编号:1000-7091(2009)05-0234-05Development and Application of Loop 2Mediated Isothermal Amplification forDetection of Streptococcus agalactiaeWANG Y ong 1,ZHAO X in 1,J I NG Hai 2chun 2,LAN Qing 2kuo 1,ZHU Zhu 1,CHE NG Y i 1(1.Central Lab of T ianjin Academy of Agriculture Sciences ,T ianjin 300381,China ;2.Plant Pathogen Interactions Division ,R othamsted Research ,A L52JQ ,UK)Abstract :T o develop a technique for detecting the fbsB gene of Streptococcus agalactiae using a novel DNA am plifi 2cation method designated Loop 2Mediated Is othermal Am plification (LAMP ).The detection assay is based on the loop 2me 2diated is othermal am plification (LAMP )reaction.The fbsB gene was am plified by a set of four specially primers that rec 2ognize six distinct sequences of the target.The am plification can be obtained in 1h by incubating all of the reagents in a single tube with Bst DNA polymerase at 63℃.Results showed that the developed LAMP assay dem onstrated an exception 2ally higher than the conventional PCR.This assay results was found to be 100times m ore sensitive than the PCR assay ,and consistent with the results of real 2time PCR.Our results clearly dem onstrate that the LAMP 2based assay is a sensitive and reliable means for the detection of Streptococcus agalactiae .K ey w ords :Loop 2mediated is othermal am plification (LAMP );Streptococcus agalactiae ;fbsB ;Detection Streptococcus agalactiae is a highly contagious ,obli 2gate bacterium of the bovine mammary gland.Its presence is frequently ass ociated with high s omatic cell counts in milk and decreased milk yields [1,2].It is considered one of the major causes of economic losses to dairy producers without a control program.The diagnosis of Streptococcusagalactiae is typically performed by cultural is olation andserological examinations ,but these methods are time con 2suming and not appropriate for rapid detection in laborato 2ry.Rapid and sensitive laboratory diagnostic methods are required if early treatment for disease is to be initiated.PCR has ev olved as one or the m ost specific and sensitive method for detecting pathogenic microorganisms report 2ed [3-6].Recently ,Real 2time PCR was developed as a powerful tool and its applications of a real 2time PCR to microorganisms have been reported [7-10].In spite of specificity and sensitivity of PCR and real 2time PCR ,these methods are not widely used in the detectionof华北农学报・2009,24(5):2342238Streptococcus agalactiae for economic reas ons.A sensitive and specific method for DNA am plifica 2tion method was developed by Notomi et al.and termedloop 2mediated is othermal am plification (LAMP )[11].LAMP method relies on auto 2cycling strand displacement DNA synthesis by a Bst DNA polymerase.This method em ploys a set of four specifically designed primers that recognize six different sequences on the targeted DNA and am plifies DNA under is othermal conditions.M oreover ,LAMP method requires sim ple incubators ,such as a water bath or block heater.In reaction process ,a large am ount of am plification products are produced with stem 2loop DNA structures.Visual inspections of the LAMP am plified product was done by using intercalating dyes like SY BRG reen I ,which bind to the double 2stranded DNA and pro 2duce a green color [12].In this study ,we have developed a highly sensitive and specific detecting method for Strepto 2coccus agalactiae .LAMP method have been designed andcom pared with PCR and real 2time PCR.1 Materials and methods1.1 B acterial strainsA total of 12pathogens were used to determine thespecificity of LAMP detection (T able 1).A Streptococcusagalactiae reference strain ATCC 9925was applied to de 2termine the sensitivity of LAMP ,PCR and real 2time PCR assay.T ab.1 B acterial strains used in this study and results for PCR,real 2time PCR and LAMPS peciesS train N o.Detection of fbsB byPCR Real 2time PCRLAMP Streptococcus agalactiaeAT CC 9925+++(positive control )AT CC 624+++Staphylococcus aureus AT CC 6538---Streptococcus bovisAT CC 9809---Streptococcus dysgalactiae AT CC 9926---Streptococcus lactis AT CC 7963---E scherichia coli AT CC 11229---Bacillus cereus AT CC 14579---Listeria innocuaAT CC 33090---P seudomonas aeruginosa AT CC 27853---Salmonella enterica sub sp.AT CC 13076---Bacillus subtilis sub sp.AT CC 6051--- N ote :+.P ositive ;-.Negative.1.2 DNA extraction and preparationDNA was extracted from an overnight culture of the organism by boiling for 10min followed by centrifugation at 14000g for 3min.One microliters of the supernatant was used as the DNA tem plate [13].F or the sensitivity test ,an overnight culture of Streptococcus agalactiae ATCC 9925was diluted with sterile water ,ranging from 101to 107CFU/m L.DNA was extracted as described above.Anegative control was performed using sterile water instead of the DNA tem plate.1.3 Oligonucleotide primersPrimer design for the LAMP method is illustrated in Fig.1.A set of four primers com prising tw o outer and tw o inner primers was designed.The tw o outer primers aredesignated the forward outer primer (F3)and the backward outer primer (B3).The tw o inner primers are designatedT o find specific nucleotide sequences o f Streptococcus agalactiae ,a multiple alignment was determined with analyses of various fbsB sequences (Y P 329577,NP 687847,NP 735300,from G eneBank data base ).The sequences are Y P 329577.Fig.1 Nucleotide sequences of targets for primers in the LAMP assay on Streptococcus agalactiae fbsB5期王 永等:LAMP 检测无乳链球菌方法的建立及应用235 the forward inner primer (FIP )and the backward innerprimer (BIP ).The specific primers for Streptococcus a 2galactiae were designated against the fibronectin 2bindingprotein gene sequences (G ene Bank accession numbers :Y P 329577).The outer primers (F3and B3)were used as the PCR and real 2time PCR primer pair.All primers sequences are listed in T ab.1.T ab.2 LAMP PrimersPrimerSequence (5’-3’)F3G C AAACTT CTG T CC AAC AG B3CT AAAG CTTT CT C AAC AT C AG AFIP G TG TG CTG C ATT AAT CT CCT CTTTTTTG CT CCGG TT C AAT C AG TT BIP TG CT ATTT CGG CG T AT AAAT C AAC ATTTT AG T ATT AATG AG CG TGG T C A F2G CT CCGG TT C AAT C AG TTF1c (sequence complementary to F1)G TG TG CTG C ATT AAT CT CCT CTT B2AG T ATT AATG AG CG TGG T C AB1c (sequence complementary to B1)TG CT ATTT CGG CG T AT AAAT C AAC A1.4 LAMP reactionThe LAMP reaction was carried out on a scale of 25μL reaction mixture containing 40pm ol of each inner primer FIP and BIP ,5pm ol each of F3and B3,8U of theBst DNA polymerase large fragment (New England Bio 2labs Inc.,Beverly ,Mass.),1.4mm ol/L concentrations of deoxynucleoside triphosphates ,0.8m ol/L Betaine (Sig 2m a 2Aldrich C o.Steinheim ,G ermany ),20mm ol/L Tris 2HCl (pH 8.8),10mm ol/L K Cl ,10mm ol/L (NH 4)2S O 4,8mm ol/L MgS O 4,0.1%T ween20,and 2μL tem plate DNA.The mixture was incubated at 63℃for 60min and then heated at 80℃for 2min to terminate the reaction.By observation of the LAMP reaction tube after adding 2μL 1000×SY BR G reen I Nucleic acid gel stain ,it was noted whether the color turned green ,indicating that the reaction is positive ,and the color remains orange ,indicat 2ing that the negative reaction.F or controlling visual in 2spection ,all the am plified products were viewed in a 2%agarose gel and documented using a gel imaging system.1.5 Sensitivity of the LAMP assayThe detection limit of the LAMP assay was estimated by testing a ten fold serial dilution of the Streptococcus a 2galactiae (strain No.ATCC 9925).The result was com 2pared with the detection limit of PCR and real 2time PCR.1.6 PCR assayPCR was performed in a 10μL reaction mixture con 2sisting of 5μL of G o T aq ○RG reen Master Mix (PromegaC orporation ,Madis on ,US A ),0.5μm ol/L concentrations of each primer ,and 1μL of tem plate DNA.PCR was per 2formed with a thermal cycler (Applied Biosystems ,C A ,US A )for 35cycles.Each cycle consisted of 1min at 95℃,30s at 55℃,and 1min at 72℃for the fbsB gene.Sam ples (5μL )of the PCR am plification products weresubjected to electrophoresis on a 2%agarose gel.1.7 R eal 2time assayReal 2time PCR reactions were performed in a 102μL reaction v olume containing :5μL of Fast SY BR ○RG reen Master Mix (Applied Biosystems ,C A ,US A ),015μm ol/L concentrations of each primer ,and 1μL of tem 2plate DNA.Real 2time PCR reactions were executed in a StepOnePlus T M Real 2T ime PCR System (Applied Biosys 2tems ,C A ,US A )with a tem perature profile of 95℃for 10min ,followed by 40cycles at 95℃for 3s and 60℃for 30s.Sam ples (5μL )of the PCR am plification products were subjected to electrophoresis on a 2%agarose gel.2 Results and AnalysisThe LAMP assay produced ladder 2like patterns on the gel LAMP products were detected only with the fbsB gene of Streptococcus agalactiae .P ositive LAMP tube turned green after 2μL of 1000×SY BR G reen I Nucleic acid gel stain due to the presence of LAMP products ,while the negative tube remained orange in color due to the absence of LAMP products (Fig.3).C om parative analysis of sensitivity of detection by LAMP ,PCR and real 2time PCR was carried out using 102fold serial dilutions of Streptococcus agalactiae .The detec 2tion limit for the LAMP reaction was found tobe 10CFU/m L for a 60min reaction as well as in electroph oretic analysis.F or the PCR assay ,the detection limit was 103CFU/m L in 90min.F or the real 2time PCR assay ,the de 2tection limit was 10CFU/m L in 45min (T ab.3and Fig.2).The specificity of LAMP reaction was shown in (Fig.4).Reaction products were detected only whenStreptococcus agalactiae DNA was presented.There were236 华 北 农 学 报24卷no am plification products detected with other pathogen.T ab.3 Comp arative sensitivities of the LAMP,PCR and real 2time PCR methodsMethodsBacteria number/(CFU/m L )1061051041031021010PCR++++---Real 2time PCR ++++++-LAMP++++++-a.Sensitivity of electrophoretic analysis of LAMP 2am plified products ;b.Sensi 2tivity of electrophoretic analysis of PCR products ;c.Sensitivity of real 2time PCR for the detection for S treptococcus agalactiae.M.D L2000;1.106CFU/m L ;2.105CFU/m L ;3.104CFU/m L ;4.103CFU/m L ;5.102CFU/m L ;6.10CFU/m L ;7.0CFU/m L.Fig.2 Comp arative sensitivity of LAMP,PCR and real 2timePCR for detection fbsB gene using serial dilutions ofStreptococcusagalactiae1.Negative LAMP reaction remained orange ;2.P ositive LAMP reaction turned green.Fig.3 Visu al detection of LAMP productsusing SYBR G reen IstainM.D L2000;1.Streptococcus agalactiae (ATCC 9925);2.Streptococcus a 2galactiae (ATCC 624);3.Staphylococcus aureus (ATCC 6538);4.Streptococ 2cus bovis (ATCC9809);5.Streptococcus dysgalactiae (ATCC9926);6.Strepto 2coccus lactis (ATCC7963);7.E scherichia coli (ATCC11229);8.Bacillus cereus (ATCC14579);9.Listeria innocua (ATCC330;0);10.P seudomonas aeruginosa (ATCC27853);11.Salmonella enterica subsp.(ATCC1J3076);12.Bacillus subtilis subsp.(ATCC6071).All the products were elec 2trophoresed on a 2%agarose gels and stained with ethidium bromide.Fig.4 Specificity of LAMP primers detectingStreptococcus agalactiae3 Discussion and conclusionUntil recently ,PCR and real 2time PCR have been found to be useful technique for Streptococcus agalacti 2ae[14-16].H owever ,PCR and real 2time PCR are technical 2ly demanding and require a relatively long time and need a high 2precision thermal cycler for a com plete detection.In this study ,a novel method called LAMP was used as a detection technique for Streptococcus agalactiae .By con 2trast ,the LAMP assay reported here is advantageous owing to its sim ple operation ,rapid reaction ,and easy detection.M oreover ,LAMP only requires sim ple reaction equipment ;it can be performed using a regular laboratory bath or heatblock that provides a constant tem perature of 63℃[17,18].The LAMP assay used for detection is highly sensi 2tive ,as it detects 10CFU/m L of Streptococcus agalactiae .In the present study ,the assay specifically am plified onlyStreptococcus agalactiae and no cross 2reactivity was ob 2served with other bacterial species.Our findings regarding high specificity are in agreement with previous reports of other LAMP methods [19-20].A 1002fold increase in sensi 2tivity was seen in LAMP ,com pared to PCR ,where the PCR was 103CFU/m L.We found that the LAMP assay has the same sensitivity as real 2time PCR assay.The LAMP method described in this study represents a very sensitive ,specific ,and rapid alternative mthod to PCR assay for the detection of Streptococcus agalactiae .We recommend that this technique be applied routinely to check milk ,s o that bacterium 2carrying cow can be found during the early in fenction stages.References :[1] Danielle D ,Evelyne G,Jean M G,et al .Serotypes of Strepto 2coccus agalactiae cultured from dairy milk samples in Qu bec[J ].Can Vet J ,2003,44(3):217-220.[2] Andersen H J ,Pedersen L H ,Aarestrup F M ,et al .Evaluationof the surveillance program of Streptococcus agalactiae in dan 2ish dairy herds[J ].J Dairy Sci ,2003,86:1233-1239.[3] Lampel K A ,Orlandi P A ,K ornegay L.Improved templatepreparation for PCR 2based assays for detection of food 2borne5期王 永等:LAMP 检测无乳链球菌方法的建立及应用237 bacterial pathogens [J ].Appl Environ Microbiol ,2000,60:4539-4542.[4] Lim Y H ,Hirose K,I zumiya H ,et al .Multiplex polymerasechain reaction assay for selective detection of Salmonella en 2terica Serovar T yphimurium [J ].Jpn J In fect Dis ,2003,56:151-155.[5] K awasaki S ,H orikoshi N ,Okada Y,et al .Multiplex PCR forsimultaneous detection of Salmonella spp.,Listeria monocyto 2genes ,and E scherichia coli O157:H7in meat samples [J ].JF ood Prot ,2005,68:551-556.[6] Arthur T M ,Bosilevac J M ,N ou X ,et al .Evaluation of cultureand PCR based detection methods for E scherichia coli O157:H7in inoculated ground beef[J ].J F ood Prot ,2005,68:1566-1574.[7] Cummings K L ,T arleton R L.Rapid quantitation of Try 2panosoma cruzi in host tissue by real 2time PCR [J ].M olBiochem Parasitol ,2003l129:53-59.[8] Y in L ,X iaoning C ,X ia Z ,et al .Real time PCR using T aqMan(and SY BR G reen for detection of Enterobacter sakazakii in in fant0formula[J ].Journal of Microbiological Methods ,2006,65:21-31.[9] Jens D ,Melanie S ,K nut K.Real 2T ime polymerase chain reac 2tion in trans fusion medicine :applicaty ons for detektion of bac 2terial contamination in blood products [J ].T rans fusion Medicine Reviews ,2007,21(3):237-254.[10] Justin O ”G,Sara S B ,Majella M ,et al .Rapid real 2timePCR detection of Listeria monocytogenes in enriched food samples based on the ssr A gene ,a novel diagnostic target [J ].F ood Microbiology ,2008,25:75-84.[11] N otomi T ,Okayama H ,Masubuchi H ,et al .Loop 2mediatedis othermal amplification of DNA [J ].Nucleic Acids Res ,2000,28:e63.[12] Parida M M ,Santhosh S R ,Dash P K,et al .Developmentand evaluation of reverse transcription loop mediated is other 2mal amplification assay for rapid and real 2time detection of Japanese Encephalitis virus[J ].Journal of Clinical Microbi 2ology ,2006,44(11):4172-4178.[13] K arami A ,Ranjbar R ,Ahmadi Z ,et al .Rapid detection ofdifferent serovares of Salmonella entrica by multiplex PCR [J ].Iranian J Publ Health ,2007,36(2):38-42.[14] Danbing K,Christian M ,Fran ois J P ,et al .Development ofconventional and real 2time PCR assays for the rapid detec 2tion of group B streptococci[J ].Clinical Chemistry ,2004,6:324-331.[15] Martinez G,Harel J ,G ottschalk M.S pecific detection byPCR of Streptococcus agalactiae in milk[J ].Can J Vet Res ,2001,65(1):68-72.[16] G illespie B E ,Oliver S P.S imultaneous detection of mastitispathogens ,staphylococcus aureus ,streptococcus uberis ,and streptococcus agalactiae by multiplex real 2time polymerase chain reaction[J ].J Dairy Sci ,2005,88:3510-3518.[17] M ori Y,Nagamine K,T omita N ,et al .Detection of loop 2me 2diated is othermal amplification reaction by turbidity derived from magnesium pyrophosphate formation[J ].Biochem Bio 2phys Res C ommun ,2001,289:150-154.[18] Nagamine K,Hase T ,N otomi T.Accelerated reaction byloop 2mediated is othermal amplification using loop primers [J ].M ol Cell Probes ,2002,16:223-229.[19] Y ukiko H K,Manabu Y,T adashi K,et al .Loop 2mediatedis othermal amplification for the rapid detection of Salmonel 2la [J ].FE MS Microbiology Letters ,2005,253:155-161.[20] Y ano A ,Ishimaru R ,Rie H.Rapid and sensitive detection ofheat 2labile I and heat stable I enterotoxin genes of enterotox 2igenic E scherichia coli by loop 2mediated ishothermal amplifi 2cation[J ].Journal of Microbiological Methods ,2007,68:414-420.238 华 北 农 学 报24卷。
本技术公开了一种无乳链球菌的检测试剂盒及其检测方法。
该试剂盒包括用于稀释奶液样品的无菌生理盐水、用于菌体裂解的裂解缓冲液,该试剂盒还包括:用于将溶液中残余的乳蛋白和脂类物质溶解在溶液中的洗涤液,用于吸附在分散的二氧化硅的载体上的核酸脱离在溶液中的洗脱液,以及,吸附体系;其中,所述吸附体系为重悬有包埋性单分散二氧化硅磁性微球的裂解缓冲液。
本技术通过上述检测试剂盒提取含有待检生物材料的核酸,再通过PCR检测反应体系对上述核酸进行PCR扩增,对扩增产物进行电泳观察,当220bp处出现条带则判定为阳性样。
本技术能直接从奶液中提取无乳链球菌的核酸,检测效率更高、假阳性率也更低。
权利要求书1.一种无乳链球菌的检测试剂盒,该试剂盒包括用于稀释奶液样品的无菌生理盐水、用于菌体裂解的裂解缓冲液,其特征在于,该试剂盒还包括:用于将溶液中残余的乳蛋白和脂类物质溶解在溶液中的洗涤液,用于吸附在分散的二氧化硅的载体上的核酸脱离在溶液中的洗脱液,以及,吸附体系;其中,所述吸附体系为重悬有包埋性单分散二氧化硅磁性微球的裂解缓冲液。
2.如权利要求1所述的无乳链球菌的检测试剂盒,其特征在于,所述洗涤液包括浓度均为25%的纯乙醇和异丙醇、浓度为100mmol/L的NaCl、浓度为10mmol/L、pH为8.0的Tris-HCl;所述洗脱液包括浓度为10mmol/L的Tris-HCl、浓度为1mmol/L的EDTA。
3.如权利要求1所述的无乳链球菌的检测试剂盒,其特征在于,所述裂解缓冲液中的guanidine thiocyanate浓度为3mol/L、EDTA的浓度为20mmol/L、Tris-HCl的pH为6.8、浓度为10mmol/L、Triton X100浓度为40mg/mL、DL-dithiothreitol的浓度为10mg/mL。
4.如权利要求1所述的无乳链球菌的检测试剂盒,其特征在于,所述无菌生理盐水中氯化钠的浓度为0.9%wt。
5.一种无乳链球菌的检测方法,其特征在于,该方法包括以下步骤:(1)通过上述权利要求1~4任一项所述的检测试剂盒提取含有待检生物材料的核酸;(2)通过PCR检测反应体系对上述核酸进行PCR扩增,对扩增产物进行电泳观察。
6.如权利要求5所述的无乳链球菌的检测方法,其特征在于,在步骤(1)中,所述提取包括以下步骤:A、往牛奶离心后所得的沉淀中加入裂解缓冲液和吸附体系,重悬、静置、离心、弃上清;B、加入裂解缓冲液混匀、离心、弃上清,重复该操作一次;C、加入洗涤液混匀、离心、弃上清,重复该过程一次;D、加入100%乙醇溶液混匀、离心、弃上清,在温箱中干燥管底沉淀10min;E、加入洗脱液,重悬沉淀,在65℃水浴加热,离心,取上清。
7.如权利要求6所述的无乳链球菌的检测方法,其特征在于,在步骤A中,所述牛奶用无菌生理盐水将牛奶样品稀释、离心、弃上清,重复该过程一次。
8.如权利要求5所述的无乳链球菌的检测方法,其特征在于,在步骤(2)中,所述PCR检测反应体系中无乳链球菌上下游引物为:无乳链球菌上游引物F:5’-AGAGTTTGATCCTGGCTCAG-3’无乳链球菌下游引物R:5-ACCAACATGTGTTAATTACTC-3’。
9.如权利要求8所述的无乳链球菌的检测方法,其特征在于,所述PCR检测反应体系是25μL反应体系:Green Taq Mix 12.5μL,dd H2O 8.5μL,无乳链球菌上下游引物各1μL,待检测菌液2μL;该PCR反应体系的反应条件为:94℃预变性5min,94℃变性1min,53℃退火1min,72℃延伸1min,共30个循环,最后72℃延伸10min。
技术说明书一种无乳链球菌的检测试剂盒、检测方法技术领域本技术属于病原微生物检测领域,尤其涉及一种无乳链球菌的检测试剂盒,以及通过上述检测试剂盒直接从奶液中检测无乳链球菌的检测方法。
背景技术奶牛乳房炎是奶牛的多发病,也是引起奶牛业发展的重要疾病之一,能引起产奶量及其品质下降,造成严重的经济损失。
常见的致病菌有金黄色葡萄球菌、大肠杆菌、无乳链球菌、停乳链球菌、乳房链球菌等。
无乳链球菌是牛群中乳腺炎的重要传染性病原菌,对规模饲养的奶牛,其危害是相当严重的。
据报道,无乳链球菌可引起新生儿败血症、脑膜炎、呼吸道和妇女生殖感染等。
在检测上,传统乳房炎病原菌的检测是通过细菌培养、生化反应或是免疫学诊断来确定,虽然较为准确,但由于需专人操作、周期长,成本高等因素,难以满足临床检验需求。
近年来,随着分子生物学的发展,PCR技术在检测致病菌方面得以广泛应用,并且展现出其快速、特异的优势和前景。
国内外对无乳链球菌和金黄色葡萄球菌的检测方法已有一定的相关研究基础。
他们利用已知无乳链球菌16sRNA序列设计特异性引物,通过PCR 方法鉴定,然而对于病原微生物的分子检测是建立在目标DNA扩增基础上的,因此,高效提取DNA致病细菌是一个重要的步骤,这就限定了PCR鉴定技术的推广应用。
技术内容本技术的目的在于提供一种能够准确、高效地直接从奶液中检测无乳链球菌的检测试剂盒。
本技术的再一目的在于提供上述检测试剂盒直接从包括奶液在内的乳液中直接检测无乳链球菌的方法。
本技术是这样实现的,一种无乳链球菌的检测试剂盒,该试剂盒包括用于稀释奶液样品的无菌生理盐水、用于菌体裂解的裂解缓冲液,该试剂盒还包括:用于将溶液中残余的乳蛋白和脂类物质溶解在溶液中的洗涤液,用于吸附在分散的二氧化硅的载体上的核酸脱离在溶液中的洗脱液,以及,吸附体系;其中,所述吸附体系为重悬有包埋性单分散二氧化硅磁性微球的裂解缓冲液。
优选地,所述洗涤液包括浓度均为25%的纯乙醇和异丙醇、浓度为100mmol/L的NaCl、浓度为10mmol/L、pH为8.0的Tris-HCl;所述洗脱液包括浓度为10mmol/L的Tris-HCl、浓度为1mmol/L的EDTA。
优选地,所述裂解缓冲液中的guanidine thiocyanate浓度为3mol/L、EDTA的浓度为20mmol/L、Tris-HCl的pH为6.8、浓度为10mmol/L、Triton X100浓度为40mg/mL、DL-dithiothreitol的浓度为10mg/mL。
优选地,所述无菌生理盐水中氯化钠的浓度为0.9%wt。
本技术进一步公开了一种无乳链球菌的检测方法,该方法包括以下步骤:(1)通过上述检测试剂盒提取含有待检生物材料的核酸;(2)通过PCR检测反应体系对上述核酸进行PCR扩增,对扩增产物进行电泳观察。
优选地,在步骤(1)中,所述提取包括以下步骤:A、往牛奶离心后所得的沉淀中加入裂解缓冲液和吸附体系,重悬、静置、离心、弃上清;B、加入裂解缓冲液混匀、离心、弃上清,重复该操作一次;C、加入洗涤液混匀、离心、弃上清,重复该过程一次;D、加入100%乙醇溶液混匀、离心、弃上清,在温箱中干燥管底沉淀10min;E、加入洗脱液,重悬沉淀,在65℃水浴加热,离心,取上清。
优选地,在步骤A中,所述牛奶用无菌生理盐水将牛奶样品稀释、离心、弃上清,重复该过程一次。
优选地,在步骤(2)中,所述PCR检测反应体系中无乳链球菌上下游引物为:无乳链球菌上游引物F:5’-AGAGTTTGATCCTGGCTCAG-3’无乳链球菌下游引物R:5-ACCAACATGTGTTAATTACTC-3’。
优选地,所述PCR检测反应体系是25μL反应体系:Green Taq Mix 12.5μL,dd H2O8.5μL,无乳链球菌上下游引物各1μL,待检测菌液2μL(即上述步骤E中所获得的上清2μL);该PCR反应体系的反应条件为:94℃预变性5min,94℃变性1min,53℃退火1min,72℃延伸1min,共30个循环,最后72℃延伸10min。
在步骤(2)中,电泳观察结果中,若220bp处出现条带则判定为阳性样;否则为阴性样。
相比于现有技术的缺点和不足,本技术具有以下有益效果:本技术检测试剂盒能直接从奶液中提取无乳链球菌的核酸,相较于传统细菌检测24小时培养期来说,大大缩短了提取时间,提高了检测效率;此外,直接利用奶液检测,即使奶液只含有微量的病原菌也可检出,这样高度的敏感性很大程度上提高了隐形乳房炎和乳房炎早期奶液检出率,最大限度地降低了假阴性情况的发生。
附图说明图1是本技术试剂盒提取无乳链球菌的特异性检验结果;其中,泳道1~5和8~12孔分别为停乳链球菌、乳房链球菌、大肠杆菌、产酸克雷伯菌、液化沙雷氏菌,泳道6为无乳链球菌,泳道7为阴性对照;泳道M为DL2000plus DNA Marker,图中箭头指向核酸大小为220bp。
具体实施方式为了使本技术的目的、技术方案及优点更加清楚明白,以下结合附图及实施例,对本技术进行进一步详细说明。
应当理解,此处所描述的具体实施例仅仅用以解释本技术,并不用于限定本技术。
实施例1(1)无菌生理盐水(NaCl 0.9%)将牛奶样品稀释后,离心,弃上清。
重复上述过程一次;(2)在沉淀中加入裂解缓冲液和的吸附体系,重悬于无菌生理盐水中。
在室温放置5分钟。
离心,弃上清;(3)再次加入裂解缓冲液并混匀。
离心,弃上清,重复此过程一次;(4)加入洗涤液并混匀。
离心,弃上清,重复此过程一次;(5)加入100%乙醇溶液并混匀。
离心并弃上清。
在温箱中干燥管底沉淀10min;(6)加入洗脱液,重悬沉淀,在65°水浴加热后,离心,将上清转移到新EP管中,室温放置5min,该新EP管中所获得的上清即为待检测菌液。
实施例2通过PCR检测反应体系对上述核酸进行PCR扩增,对扩增产物进行电泳观察。
在本实施例中,PCR检测反应体系是25μL反应体系:Green Taq Mix 12.5μL,ddH2O 8.5μL,无乳链球菌上下游引物各1μL,待检测菌液2μL;该PCR反应体系的反应条件为:94℃预变性5min,94℃变性1min,53℃退火1min,72℃延伸1min,共30个循环,最后72℃延伸10min;所述PCR检测反应体系中无乳链球菌上下游引物为:无乳链球菌上游引物F:5’-AGAGTTTGATCCTGGCTCAG-3’无乳链球菌下游引物R:5-ACCAACATGTGTTAATTACTC-3’。
其中,选择上述实施例1制备得到的待检测菌液为对象,并选取含有停乳链球菌(1×103CFU/mL)、乳房链球菌(1×103CFU/mL)、大肠杆菌(1×103CFU/mL)、产酸克雷伯菌(1×103CFU/mL)、液化沙雷氏菌(1×103CFU/mL)的菌液为参照,通过上述PCR检测反应体系分别进行PCR反应,并将反应结果进行跑胶电泳,结果如图1所示,其中,泳道1~5和8~12孔分别为停乳链球菌、乳房链球菌、大肠杆菌、产酸克雷伯菌、液化沙雷氏菌,泳道6为无乳链球菌,泳道7为阴性对照;泳道M为DL2000plus DNA Marker,图中箭头指向核酸大小为220bp。