Chimera图像教程:密度显示
- 格式:doc
- 大小:301.50 KB
- 文档页数:3
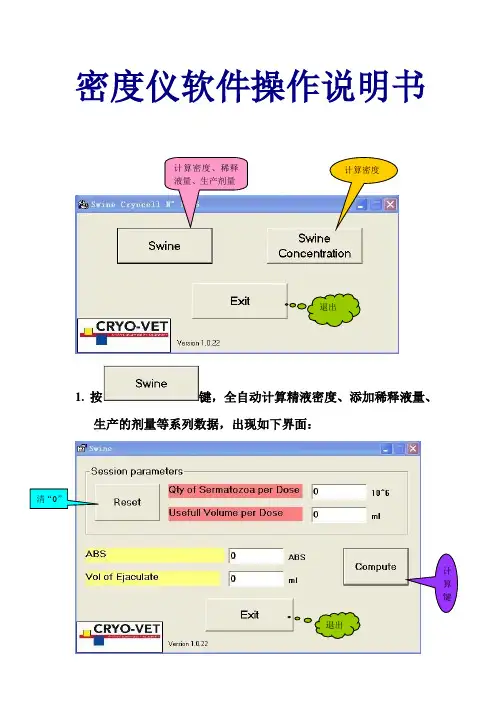
密度仪软件操作说明书
1. 按键,全自动计算精液密度、添加稀释液量、
生产的剂量等系列数据,出现如下界面:
退出
退出
①
:每剂的精子含量(20亿-35亿)
:每剂精液量(40ml-80ml)
②
:
密度仪直接显示的数据(测出的吸光度)
:公猪的总射精量(200ml-500ml)
设定:每剂精子含量为:30亿
每剂精液量为:80ml
密度仪吸光率为:0.96ABS
公猪总射精量为:220ml
退出
2.
按
键,可直接读取精子密度(每毫升),
出现如下界面:
:
密度仪直接显示的数据(测出的吸光度)
退出
设定密度仪直接显示的数据(测出的吸光度)----0.96,输入到空格内,
机器自动计算每毫升的精子密度:497*106 个
/ml
注意:在使用密度仪测量的时候,比色皿内需要加:
1.生理盐水(生理盐水的量为:2400µL);
2.原精(原精量为:100µL);
3.比例为1:25。

2021.13科学技术创新一种基于Chimera 软件的分子动力学模拟方法马学婧*李俊甫宋立立张兆英王悦孙琳琳(沧州师范学院生命科学学院,河北沧州061001)Chimera 由美国加州大学旧金山分校的生物计算可视化和信息学中心开发,并得到了美国国立卫生研究院的部分支持[1]。
作为一个可高度延伸的程序,它可应用于密度图、超分子组装、序列比对、对接结果、轨迹和构象集合,也可以生成高质量图像和动画[2-4]。
Chimera 的官方网站(/chimera/)上可以查阅快速入门、用户手册、命令行索引和一些教程与视频,还可以观赏用该软件制作的图片和动画。
在下载页面,它还提供了Windows ,Mac 和Linux 三个版本供学术、政府和非营利机构和个人使用者免费下载使用。
Chimera 功能强大,界面友好,可帮助没有编程基础的教师和科研人员利用其提供的多种模块化工具进行简单的分子动力学模拟,并且可用于多种操作系统,无需购买昂贵的服务器也可以满足基本的分析需求。
因此,本文将对基于Chimera 软件的分子动力学模拟方法进行阐述。
1蛋白质结构准备1.1蛋白质结构可视化首先打开一个已经下载的蛋白质结构文件(后缀为.PDB )或选择Fetch by ID ,输入蛋白质结构的ID 调入蛋白质结构文件。
在Presets 中根据需要选择展示模式。
在Select 中,Chain 选项可以选择蛋白质复合物不同的链;Chemistry 选项可以根据化学性质,如元素、功能基团和原子轨道对蛋白质进行选择;Residue 选项可以对20种标准氨基酸进行选择,也可以按照性质对某一类氨基酸进行选择;Structure 选项可以对蛋白质的骨架、离子、配体、核酸和特定的二级结构等进行选择。
选择完毕后,可应用Actions 中的工具对选定的对象进行编辑,如:Atoms/Bonds 选项可以隐藏或显示原子或键,设置显示模式为棍型、球棍型等;Ribbon 选项可以隐藏或显示蛋白质的某一部分,还可以改变带状模式的类型;Surface 选项可以隐藏或显示蛋白质的表面,并设置显示模式和透明度;Color 选项可以对选中的对象进行上色;Label 选项可以对原子、氨基酸残基进行标注,且可自主选择标注的格式。

顺序和结构教程本教程介绍使用Multalign Viewer处理顺序排列和相关结构。
数据选自enolase 家族:The enolase superfamily: a general strategy for enzyme-catalyzed abstraction of the alpha-protons of carboxylic acids.Babbitt PC, Hasson MS, Wedekind JE, Palmer DR, Barrett WC, Reed GH, Rayment I, Ringe D, Kenyon GL, Gerlt JA. Biochemistry. 1996 Dec 24;35(51):16489-501.接下来下载顺序排列文件super8.msf到一个方便的位置。
本顺序排列包括含有8个enolase超家族成员的barrel域 domains。
Windows/Mac系统,点击chimera图标;UNIX系统,从系统提示符开启Chimera :unix: chimera一个splash屏幕将会出现,几秒后被Chimera主窗口代替。
选择File... Open,然后寻找并打开super8.msf. 开启一个sequence alignment file自动开始了Multalign Viewer,用它显示排列。
Multalign Viewer有其独立的参数设置,包括几个控制显示。
从排列窗口菜单,选择Preferences... Appearance调整Multiple alignments的line-wrapping behavior, font,和color scheme到你的要求。
关闭preferences对话框。
改变顺序敞口的尺寸和放置使其均可见。
如果顺序窗口变得在任何点都模糊,可以从Chimera Tools菜单增加。
(Tools... MAV -super8.msf... Raise).Multalign Viewer sequence window序列名称显示于左边,Consensus sequence和Conservation histogram显示于顺序的上面。

顺序和结构教程本教程介绍使用Multalign Viewer处理顺序排列和相关结构。
数据选自enolase 家族:The enolase superfamily: a general strategy for enzyme-catalyzed abstraction of the alpha-protons of carboxylic acids.Babbitt PC, Hasson MS, Wedekind JE, Palmer DR, Barrett WC, Reed GH, Rayment I, Ringe D, Kenyon GL, Gerlt JA. Biochemistry. 1996 Dec 24;35(51):16489-501.接下来下载顺序排列文件super8.msf到一个方便的位置。
本顺序排列包括含有8个enolase超家族成员的barrel域 domains。
Windows/Mac系统,点击chimera图标;UNIX系统,从系统提示符开启Chimera :unix: chimera一个splash屏幕将会出现,几秒后被Chimera主窗口代替。
选择File... Open,然后寻找并打开super8.msf. 开启一个sequence alignment file自动开始了Multalign Viewer,用它显示排列。
Multalign Viewer有其独立的参数设置,包括几个控制显示。
从排列窗口菜单,选择Preferences... Appearance调整Multiple alignments的line-wrapping behavior, font,和color scheme到你的要求。
关闭preferences对话框。
改变顺序敞口的尺寸和放置使其均可见。
如果顺序窗口变得在任何点都模糊,可以从Chimera Tools菜单增加。
(Tools... MAV -super8.msf... Raise).Multalign Viewer sequence window序列名称显示于左边,Consensus sequence和Conservation histogram显示于顺序的上面。


熊猫分部密度制图目录The Third Lesson ..................................................... 错误!未定义书签。
Ex3 熊猫分部密度制图.. (2)操作步骤: (2)1 (2)2 (2)3 (3)4 (4)5 (5)6 (6)7 (7)8 (8)Ex4 GDP 区域分布图的生成与对比 (10)操作步骤 (10)(1)IDW 插值法 (10)(2)Spline 内插法 (13)(3)IDW 与Spline 对比分析 (18)Ex5提取山顶点、凹陷点 (23)操作步骤 (23)Ex3 熊猫分部密度制图操作步骤:1运行ArcMap,加载Spatial Analyst 模块,如果Spatial Analyst 模块未能激活,点击Tools 菜单下的Extensions ,选择Spatial Analyst,点击Close 按钮。
在Options 中的General 页面中在设置默认工作路径,此处假定为“F:\罗荣霖\the third lesson\Ex3\Result”2熊猫的生存必须满足一定的槽域范围。
选择熊猫活动足迹数据图层,点击Spatial Analyst 模块的下拉箭头,选择Distance 并点击Allocation ,设置参数如图所示;点击OK,生成熊猫槽域范围图3选择FP 数据层,点击鼠标右键并选择Open Attribute Table 命令,打开FB 属性表。
点击FB 属性表右下角Option 按钮的下拉箭头,选择Export 命令,导出FB 属性数据表,参数设置如图所示,点击确定按钮;当出现提示是否需要加载该数据表,选择“是(Y )”4选择熊猫活动足迹数据图层(XMPoint),点击鼠标右键,选择Joinand Relates 子菜单下的Join ,弹出数据连接对话,参数设置如图所示,点击OK 按钮,完成熊猫采样数据与槽域范围数据的连接。

地理国情监测云平台/
密度分析可以对某个现象的已知量进行处理,然后将这些量分散到整个地表上,依据是在每个位置测量到的量和这些测量量所在位置的空间关系。
为什么要对地图进行密度分析?
密度表面可以显示出点要素或线要素较为集中的地方。
例如,每个城镇都可能有一个点值,这个点值表示该镇的人口总数,但是您想更多地了解人口随地区的分布情况。
由于每个城镇内并非所有人都住在聚居点上,通过计算密度,您可以创建出一个显示整个地表上人口的预测分布状况的表面。
下图给出了一个密度表面的示例。
相加到一起时,像元的人口值将等于原始点图层人口的总和。
密度分析的应用
“密度分析”工具可以将输入点图层的测量量分布到整个地表上,以生成一个连续的表面。
这里有一个密度分析应用的示例,考虑在某特定地区拥有多家店面的连锁零售店。
对于每家店面,管理部门都保存有与顾客有关的销售数字。
管理部门假定顾客根据路程的远近来选择光顾哪家店面。
在本示例中,很自然地就会假定任何一个顾客总是会选择最近的那家店面。
距离最近的店面越远,顾客到那家店面要走的路也就越远。
但是离得比较远的顾客也可能光顾其他店面。
管理部门想研究顾客居住地点的分布状况。
根据这些家店面的销售数字和空间分布情况,管理部门需要将顾客巧妙地分散到整个地表上,以此创建显示顾客分布情况的表面。
要完成这项任务,“密度分析”工具将考虑店面之间的相互关系、光顾每家店面的顾客数量以及需要共享测量量(顾客)的某一部分的像元的数量。
离测量点(即店面)较近的像元占有测量量的比例要高于那些离测量点较远的像元。

ChimeraUCSF Chimera是一个可高度延伸的程序,用于分子结构和相关数据的交互式可视化和分析。
主要包括:密度图,超分子组合,顺序排列,对接结果,轨迹和构象ensemble。
也可以生成高质量图像和动画。
Chimera包括完整的程序说明书和几个教程。
学术,政府和非营利机构和个人使用者可以免费下载。
Chimera由Resource for Biocomputing, Visualization, and Informatics发展,由National Center for Research Resources资助(grant P41-RR001081)教程开始教程 - Menu VersionChimera的很多工作都可以用多种方法完成。
例如:颜色和显示模式(display style)可以用动作菜单(actions)或者输入命令行(commands)。
一般来说, 命令行更详细,功能更强大,而菜单操作则不需要命令行知识和它们的语法规则就可以易于掌握软件特征。
本教程中,在开始教程-命令行 Version中,采用命令行执行的很多相同任务在此使用菜单代替。
为了继续进行下去,请先下载本教程需要的PDB文件到你电脑中的方便位置。
•1zik.pdb - leucine zipper•1d86.pdb - DNA and 纺锤菌素菜单, Part 1 –操作,选择和链Windows/Mac系统, 点击chimera图标; UNIX系统, 从系统提示符开启Chimera:unix: chimera然后,闪现一个欢迎界面,几秒后被Chimera主窗口取代。
如果你喜欢,通过拖拽窗口右下角来改变Chimera的大小。
现在打开一个结构。
从菜单中选择File... Open,用产生的file browser 找出并打开以前下载的(previously downloaded)文件1zik.pdb (文件类型设为all (guess type)或者PDB). 此结构是一个由两个肽链形成的Leucine zipper。

Stata 密度曲线密度曲线是一种通过对数据进行平滑处理来估计概率密度函数的方法。
在 Stata 中,我们可以使用密度曲线来可视化数据的分布情况。
本文将介绍如何使用 Stata 绘制密度曲线,并提供一些实际应用的示例。
1. 密度曲线的概念密度曲线是一种连续概率分布图形,用于描述随机变量的概率密度函数(PDF)。
它是对数据分布进行平滑处理后的估计,通过将数据分布转化为连续的曲线,更直观地展示了数据的分布特征。
2. Stata 绘制密度曲线的基本步骤要在 Stata 中绘制密度曲线,可以按照以下基本步骤进行操作:步骤 1:导入数据首先,我们需要将数据导入到 Stata 中。
可以使用import命令或者通过File菜单中的Import Dataset选项将数据文件导入到 Stata。
步骤 2:生成密度估计使用kdensity命令生成密度估计。
该命令可以指定要绘制密度曲线的变量,并可以选择其他选项来调整曲线的平滑程度。
步骤 3:绘制密度曲线使用twoway命令绘制密度曲线。
可以使用kdensity命令生成的结果作为twoway 命令的参数,指定要绘制的曲线类型和其他绘图选项。
步骤 4:可选的格式调整可以使用graph export命令将绘制好的密度曲线导出为图片文件,或者使用graph save命令将绘图结果保存为 Stata 图形文件以供后续使用。
3. 密度曲线的应用示例下面通过一个实际的数据集来演示如何在 Stata 中绘制密度曲线。
假设我们有一个数据集mydata.dta,其中包含了一组学生的数学考试成绩。
我们想要了解这些成绩的分布情况,并绘制出相应的密度曲线。
首先,我们导入数据集:use mydata.dta然后,使用kdensity命令生成密度估计:kdensity math_score接下来,使用twoway命令绘制密度曲线:twoway kdensity math_score以上命令将在 Stata 的图形窗口中显示出绘制好的密度曲线。

Chimera是一种用于生物信息学的开源软件,可以用于可视化和分析大型生物学数据集。
它支持多种数据格式,包括WRL (Wavefront .obj)格式,这是一种用于描述三维几何形状的格式。
要将Chimera中的数据保存为WRL格式,可以按照以下步骤操作:
1. 打开Chimera软件并加载要保存的数据。
2. 在菜单栏中选择“File”选项,然后选择“Export”。
3. 在弹出的对话框中,选择“OBJ File”选项。
4. 在“File name”字段中输入要保存的文件名和路径。
5. 在“Include”字段中选择要保存的数据类型,例如“Vertices”、“Normals”、“Colors”等。
6. 点击“Save”按钮保存文件。
在保存为WRL格式后,可以使用其他支持该格式的软件打开和编辑该文件。
mathematica densityplot 对称颜色题目: Mathematica密度图的对称颜色导言:Mathematica是一款强大的数学软件,广泛应用于科学计算、数据分析和可视化。
密度图是一种常用的数据可视化方式,用于展示二维数据分布的密度。
在Mathematica中,我们可以通过DensityPlot函数创建和定制密度图。
而在本文中,我们将重点讨论如何使用Mathematica创建具有对称颜色的密度图,以增强可视化效果。
第一步: 密度图基础知识要理解密度图的对称颜色,我们首先需要了解密度图的基本构建。
密度图通常用于可视化二维函数或数据集的分布情况。
在Mathematica中,密度图可以通过DensityPlot函数创建。
DensityPlot函数接受一个函数f(x,y)作为输入,并基于该函数的值在二维坐标系上绘制一个颜色映射。
函数的值越大,对应的颜色越亮;值越小,对应的颜色越暗。
这种颜色映射可以通过ColorFunction参数自定义,从而实现对密度图颜色的定制。
第二步: 对称颜色的定义在研究密度图的对称颜色之前,我们需要明确对称颜色的定义。
对称颜色可以理解为针对一个颜色值,其对应的亮度值在颜色映射中以对称的方式分布。
即,颜色映射的一半亮度值用于表示该颜色的较低亮度值,另一半亮度值用于表示该颜色的较高亮度值。
第三步: 密度图的对称颜色实现在Mathematica中,我们可以通过自定义ColorFunction函数来实现密度图的对称颜色。
下面是一种简单的实现方法:1. 定义对称颜色函数我们首先需要定义一个对称颜色函数。
该函数将根据输入的亮度值返回相应的颜色值,并实现对称分布。
以下是一个示例的对称颜色函数:symmetricColor[x_] := Blend[{{0, Blue}, {1/2, White}, {1, Red}}, x]这个函数使用Blend函数,它接受一个颜色列表和一个分数值,返回对应的颜色值。
染料及染料中间体堆积密度的测定染料及染料中间体的堆积密度是衡量染料稳定性的重要指标之一,其值的高低反映了染料的稳定性和稳定性。
因此,测定染料及染料中间体堆积密度的方法显得尤为重要。
测量染料及染料中间体堆积密度的方法分为两大类:物理方法和化学方法。
物理方法涉及通过量热实验或热分析实验对染料及染料中间体进行测量;化学方法主要是通过利用染料及染料中间体的吸附量来衡量其堆积密度。
物理方法中常用的量热实验测量染料及染料中间体堆积密度的方法大致如下:将样品放入量热实验装置,按照仪器上的要求进行设置,然后测量样品的堆积密度。
其主要步骤为:照表示将样品放入耐高温器具;设定所需的温度及时间;安装温度传感器;记录实验数据;根据数据计算染料及染料中间体的堆积密度。
与量热实验相比,热分析实验更为适用于测定染料及染料中间体的堆积密度,主要步骤如下:将样品放入热分析仪,并进行设定;安装温度传感器;从室温开始分析,按一定的温度递增,直至达到预定的温度;记录分析数据,并计算出染料及染料中间体的堆积密度值。
同时,利用染料及染料中间体的吸附量也可对其堆积密度进行测量。
此类方法是在计算品质中及染料分离相关的应用中采用的,通常利用吸附量的变化以及其所涉及的热学参数,来估计染料及染料中间体的堆积密度。
染料及染料中间体堆积密度的测定是衡量染料特性的重要手段,通过全面的测定及分析,可以有效地改进染料的质量,提高染料的稳定性,并满足用户对染料质量的要求。
同时,测定染料及染料中间体堆积密度还可以为染料分离及品质控制提供重要参考依据。
总之,染料及染料中间体堆积密度的测定是衡量染料特性的重要指标,有助于提高染料的稳定性,改善染料的质量,并为染料分离及品质控制提供重要参考依据。
目前,量热实验、热分析实验以及染料及染料中间体的吸附量都是测定染料及染料中间体堆积密度的有效方法,应用范围广泛,受到人们的青睐。
mathematica densityplot 对称颜色-回复Mathematica是一款功能强大的数学软件,具备制作各种图形和可视化的能力。
其中的DensityPlot函数是一种用于生成二维密度图的工具。
它可以通过对称颜色方案有效地展示数据的分布情况。
在本文中,我将介绍如何使用Mathematica的DensityPlot函数创建一张具有对称颜色的二维密度图。
我们将从介绍对称颜色的概念开始,然后逐步讲解创建图像的过程。
一、什么是对称颜色?对称颜色是指在颜色空间中以某一颜色为中心,向左右两侧对称分布的颜色组合。
这种颜色方案在数据可视化中经常被使用,因为它可以清晰地传达数据的相似性和差异性。
二、创建对称颜色的二维密度图以下是创建对称颜色的二维密度图的步骤:1. 导入Mathematica中的相关包在开始之前,我们需要导入Mathematica中的相关包。
这可以通过执行以下命令完成:Needs["PlotLegends`"]2. 定义数据和密度函数在创建图像之前,我们需要定义用于生成图像的数据和密度函数。
这些数据应包含x和y坐标,并与每个坐标点相关联的密度值。
以下是一个示例数据集及其对应的密度函数定义:data = {{0, 0, 0.2}, {1, 1, 0.5}, {2, 2, 0.8}, {3, 3, 0.3}}; densityFunction[x_, y_] :=Evaluate[PDF[SmoothKernelDistribution[data[[All, {1, 2}]]], {x, y}]]3. 创建对称颜色方案接下来,我们需要创建一个对称颜色方案。
Mathematica提供了一种方便的方法来生成对称颜色,即使用ColorData函数。
以下是一个创建对称颜色方案的例子:colorScheme = ColorData[{"Symmetric", "Reverse"}];在此例中,我们选择了“对称”的颜色方案,并选择了“反转”以生成对称颜色。
图像教程:密度显示背景这些图像显示了蛋白数据库条目2fma 的一部分, Alzheimer's amyloid precursor protein (APP) copper-结合域, 同时它的电子密度图(2fo-fc) 由电子密度服务器提供。
图像 How-T o这里的方法仅仅是一个例子;达到形似的结果通常有很多种路线。
也可以参考: presets , tips on preparing images , and Chimera 网站, 中的Image Gallery 和Guide to Volume Display开启Chimera ,按要求放大窗口。
显示命令行(例如,使用Favorites... CommandLine )。
从蛋白数据库 取回 2fma 结构,然后应用预设值 #2 显示所有的原子和杂原子color-coding:A:B:Command: open 2fmaCommand: preset apply int 2从Electron Density Server取回此结构的密度图。
Command: open edsID:2fmaClick Volume Viewer对话框的小眼睛图标,先隐藏电子密度图。
随时用鼠标按你的要求进行移动和放大操作。
由于一个目的就是显示可替换的构象的例子,通过标签对可替换的构象的残基和原子进行识别。
Command: rlab @.aMet-170 和Glu-183有可替换的构象;选择Met-170 附近的一段残基作为图形。
只显示残基168-170和其相邻残基的骨架。
Command: ~rlabCommand: show :168-170Command: focusCommand: disp :167,171@n,ca,c,o改变到stick表示方法,使棍更细。
Command: rep stickCommand: setattr m stickScale 0.5只选择侧链Tyr-168 和Met-170; 这次选择将会用于把密度显示限定到一个区域。
如何用Chimera生成蛋白3D结构图?之前在《如何用PyMol生成蛋白三维结构图?》一文已对PyMol 的基本操作做过介绍,我们今天介绍一个可免费使用的综合3D建模软件:UCSF Chimera (/chimera)。
Chimera可以用来分子结构数据的可视化(见下图),包括密度图、序列比对、分子对接、分子动力学轨迹等的显示,还可生成高质量的图片和动画。
为了方便大家下载,最新(10月19日)的版本已经上传到了论坛上(包括Mac, Windows),地址:/forum/thread-2891-1-1.html。
下面就开始吧~1结构文件的打开本文用做示例的蛋白的PDB ID为3ZOJ(Fischer et al.,2009)和1d86。
Chimera可以通过两种方式打开文件,一种是知道分子的ID(如这里PDB ID:3ZOJ),可通过File\Fetch by ID 打开结构文件,另一种若本地已有结构文件,可通过File\Open打开本地文件。
打开后的初始效果如下图,peptide backbone主要以ribbon的形式显示,碳原子与模型颜色一致。
对于杂原子,主要是红色表示氧原子,蓝色表示氮原子。
通过Action\Ribbon 或 Atoms/Bonds可自定义选择展示的方式。
2基本的操作Chimera的鼠标操作方式与PyMol相似,左键转动模型,中键平移模型,右键(滚轮)缩放;Ctrl+左键选择,可点选也可框选,点空白区取消选择;Ctrl+Shift+左键,点一次加选,点两次减选。
默认的鼠标操作方式见下图。
上(↑)下(↓)方向键,分别对应扩大和缩小选择的范围,左方(←)向键撤销选择,右方向键(→)反选。
Chimera的Action菜单默认只作用在选中部分,若窗口中没有选中的部分,Action菜单将应用所有的部分。
“选择”在图形处理软件中往往占操作步骤的绝大部分,Chimera有整个Select菜单是关于如何选择的。
matlab 坐标轴密度摘要:1.引言2.Matlab 坐标轴密度概述3.Matlab 坐标轴密度设置方法4.密度对图形显示的影响5.总结正文:Matlab 是一种广泛应用于科学计算和数据分析的编程语言。
在数据分析过程中,我们通常需要绘制图形以便更好地观察数据分布和特征。
而Matlab 中的坐标轴密度设置对于图形的显示效果有着重要的影响。
本文将详细介绍Matlab 坐标轴密度的相关知识。
首先,我们需要了解什么是Matlab 坐标轴密度。
简单来说,坐标轴密度是指在坐标轴上显示数据点的数量。
设置合适的坐标轴密度可以帮助我们更清晰地观察数据分布。
接下来,我们将介绍如何设置Matlab 坐标轴密度。
在Matlab 中,我们可以使用`set`命令来设置坐标轴密度。
具体语法如下:```matlabset(gca, "XDataLimit", [xmin, xmax])set(gca, "YDataLimit", [ymin, ymax])```其中,`gca`表示当前坐标轴对象,`XDataLimit`和`YDataLimit`分别用于设置x 轴和y 轴的数据范围。
通过调整数据范围,我们可以控制坐标轴上的数据点数量。
值得注意的是,坐标轴密度的设置应根据图形显示效果和数据特点进行适当调整。
设置得过高或过低都会导致图形显示效果不佳,甚至无法正确反映数据分布。
总之,Matlab 坐标轴密度在数据可视化中起到了关键作用。
通过合理设置坐标轴密度,我们可以更好地观察和分析数据。
图像教程:密度显示
背景
这些图像显示了蛋白数据库
条目2fma 的一部分,
Alzheimer's amyloid
precursor protein (APP)
copper-结合域, 同时它的
电子密度图(2fo-fc) 由电
子密度服务器提供。
图像 How-To
这里的方法仅仅是一个例
子;达到形似的结果通常有很多种路线。
也可以参考: presets , tips on preparing images , and Chimera 网站,
中的Image Gallery 和Guide to Volume Display 开启Chimera ,按要求放大窗口。
显示命令行(例如,使用Favorites... Command Line )。
从蛋白数据库 取回 2fma 结构,然后应用预设值 #2 显示所有的原子和杂原子color-coding:
Command : open 2fma
Command : preset apply int 2
从 Electron Density Server 取回此结构的密度图。
Command : open edsID:2fma
Click Volume Viewer 对话框的小眼睛图标,先隐藏电子密度图。
随时用鼠标按你的要求进行移动和放大操作。
由于一个目的就是显示可替换的构象的例子,通过标签对可替换的构象的残基和原子进行识别。
Command : rlab @.a
A:
B:
Met-170 和Glu-183有可替换的构象;选择Met-170 附近的一段残基作为图形。
只显示残基168-170和其相邻残基的骨架。
Command: ~rlab
Command: show :168-170
Command: focus
Command: disp :167,171@n,ca,c,o
改变到stick表示方法,使棍更细。
Command: rep stick
Command: setattr m stickScale 0.5
只选择侧链Tyr-168 和Met-170; 这次选择将会用于把密度显示限定到一个区域。
Command: sel :168,170 & without CA/C1'
without CA/C1'部分是进入菜单Select... Structure... side chain/base... without CA/C1'的简洁方法。
所有在Select... Structure下的终端菜单可以用作command-line specifiers。
在Volume Viewer中,
1.点击眼睛图标,使密度显示恢复。
2.从菜单选择Features... Zone,点击出现在对话框中的Zone按钮。
然后,可以取消
选择(例如,在空区通过ctrl-点击鼠标左键),但是区域半径仍然可以调整。
本图中区域半径设置为1.96 Å。
3.柱状图的垂直栏(threshold)代表了轮廓水平;
o要改变水平,用鼠标或者键盘在柱状图下面的level区输入不同值拖拽threshold到左边或者右边。
o要改变Color, 点击柱状图下面的正方形color well,使用Color Editor
o要添加另一个轮廓水平,Ctrl-点击柱状图; Level 和Color的变化应用最近拖拽或者点击最临界的。
4.从菜单选择Features... Surface and Mesh Options显示额外的显示设置。
Figure A的Volume显示设置(除了默认值)
∙Style mesh
∙two contour levels:
1.Level 0.426, Color (RGBA) 0.36 1.0 1.0 0.636
2.Level 2.06, Color (RGBA) 1.0 0.0 1.0 1.0 (magenta)
∙turned on Smooth mesh lines
∙Mesh line thickness 1.5 pixels
Figure B的Volume显示设置(除了默认值)
∙Style surface
∙two contour levels:
1.Level 0.426, Color (RGBA) 0.36 1.0 1.0 0.4
2.Level 2.06, Color (RGBA) 1.0 0.0 1.0 0.6
∙turned on Surface smoothing iterations 2 factor 0.3
使用2D Labels(Tools... Utilities下面) 添加标签文本。
标签对于两个图片想吐。
点击图形窗口开启一个标签,输入内容。
每一个标签可以通过拖拽改变位置,通过点击color well和使用Color Editor可以调整其颜色。
字体可以调到不同的值。
字体的近似度依赖于窗口的大小,用你看起来最好的。
本例中,标签都是白色的,残基标签比可替代的标签大一点点。
后者在两个单词之间输入回车键,以使它们看起来在不同的行。
通过使用File... Save Image,采用默认设置把figures保存为 PNG文件,和Chimera窗口同样的帧数。
如果以后你想需要重新产生相同或者相近的图像或者说明一个不同resolution 值或者改变颜色,保存图片之前或者之后保存(参考File菜单), session在保存时间内会包括position名为session-start. 当你恢复一个session或者移动一些事情时候很有用,但是又想恢复它的最初位置。
Command: reset session-start。