mega使用手册
- 格式:pdf
- 大小:5.97 MB
- 文档页数:62

MEGA使用说明书1. 简介1.1 MEGA的概述1.2 相关术语解释2. 安装与注册2.1 安装包并进行安装2.2 注册新账号或登录现有账号3. 用户界面导览3.. 主要功能区域介绍- 文件管理器:浏览、和文件。
- 联系人列表:查看已添加联系人信息。
- 消息中心:接收系统通知和消息更新。
4.文件操作指南4.. 浏览及搜索文件a) 使用目录树结构快速定位所需文件夹。
b) 利用搜索框输入关键词,筛选出相关文档。
c)利用标签对重要文档进行分类整理。
5.分享与协作a). 分享i)一个公开可访问的,并发送给他人以便共享特定内容。
ii)设置密码保护来限制只能被授权用户打开该。
6.数据同步a). 同步设备间数据i)在不同设备上同时登陆自己的帐户, 可实时获取最新版本资料;ii ) 手动选择需要离线保存到本地端硬盘空间内之个别项目;7.版本历史记录a). 查看文件的修改历史i)查看特定文档在不同时间点上所做过的更改;ii ) 还原到之前某个具体时刻保存下来的旧版;8. 设置与选项8.1 账户设置- 修改密码、邮箱等账户信息。
8.2 高级选项- 自定义和速度限制,以满足网络环境需求。
9.常见问题解答a) 如何恢复误删除或丢失数据?b) 如何增加存储空间容量?c) MEGA是否支持多人同时编辑一个文件?10.附件- 用户手册.pdf11.法律名词及注释i)用户:指使用MEGA服务并注册帐号进行操作和管理资料者。
ii)分享:通过公开可访问URL地址将内容共享给他人。
iii)授权用户: 指被允许浏览、编辑或已经分享出去资源的其他Mega 帐号所有者。
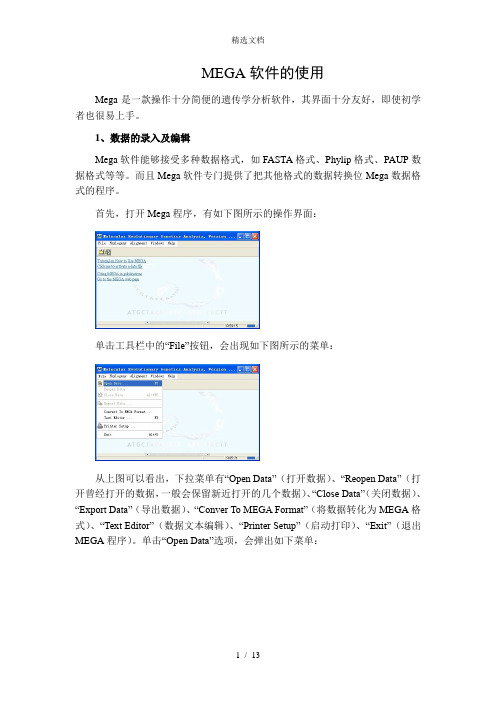
MEGA软件的使用Mega是一款操作十分简便的遗传学分析软件,其界面十分友好,即使初学者也很易上手。
1、数据的录入及编辑Mega软件能够接受多种数据格式,如FASTA格式、Phylip格式、PAUP数据格式等等。
而且Mega软件专门提供了把其他格式的数据转换位Mega数据格式的程序。
首先,打开Mega程序,有如下图所示的操作界面:单击工具栏中的“File”按钮,会出现如下图所示的菜单:从上图可以看出,下拉菜单有“Open Data”(打开数据)、“Reopen Data”(打开曾经打开的数据,一般会保留新近打开的几个数据)、“Close Data”(关闭数据)、“Export Data”(导出数据)、“Conver To MEGA Format”(将数据转化为MEGA格式)、“Text Editor”(数据文本编辑)、“Printer Setup”(启动打印)、“Exit”(退出MEGA程序)。
单击“Open Data”选项,会弹出如下菜单:浏览文件,选择要分析的数据打开,单击“打开”按钮,会弹出如下操作界面:此程序操作界面,提供了三种选择数据选择:Nucleotide Sequences(核苷酸序列)、Protein Sequences(蛋白质序列)、Pairwise Distance(遗传距离矩阵)。
根据输入数据的类型,选择一种,点击“OK”即可。
如果选择“Pairwise Distance”,则操作界面有所不同;如下图所示:根据遗传距离矩阵的类型,如果是下三角矩阵,选择“Lower Left Matrix”即可;如果是上三角矩阵,选择“Upper Right Matrix”即可。
点击“OK”按钮,即可导入数据。
如果是核苷酸数据,则读完之后,会弹出如下对话框:如上图,如果是编码蛋白质的核苷酸序列,则选择“Yes”按钮;如果是不编码蛋白质的核苷酸序列,则点击“No”按钮。
之后,会弹出如下操作窗口:此作界面的名称是“Sequence Data Explorer”,在其最上方是工具栏“Data”、“Display”、“Highlight”等,然后是一些数据处理方式的快捷按钮,在操作界面的左下方是每个序列的名称。

MEGA软件构建系统发育树摘要:以白色念珠菌属下面的十个种的18s RNA 为例,构建系统发育树来说明MEGA 软件的使用方法。
1背景简介1.1 MEGA(分子进化遗传分析)MEGA 的全称是Molecular Evolutionary Genetics Analysis。
MEGA is an integrated tool for automatic and manual sequence alignment, inferring phylogenetic trees, mining web-based databases, estimating rates of molecular evolution, and testing evolutionary hypotheses. MEGA 可用于序列比对、进化树的推断、估计分子进化速度、验证进化假说等。
MEGA 还可以通过网络(NCBI)进行序列的比对和数据的搜索。
最新版本:MEGA 5.1 Beta (软件开发者建议其结果不用于发表文章)建议下载版本:MEGA 5.05 for Windows and Mac OS。
MEGA 5 has been tested on the following Microsoft Windows® operating systems: Windows 95/98, NT, 2000, XP, Vista, version 7, Linux and Mac OS [1].MEGA 5.05 可免费下载,只需输入名字及有效邮箱,下载链接会发送至邮箱,点击可下载。
1.2 系统发育树定义系统发育树(英文:Phylogenetic tree)又称为演化树(evolutionary tree),是表明被认为具有共同祖先的各物种间演化关系的树。
是一种亲缘分支分类方法(cladogram)。
在树中,每个节点代表其各分支的最近共同祖先,而节点间的线段长度对应演化距离(如估计的演化时间)1.3 系统发育树的分类根据有根和无根来区分:树可分为有根树和无根树两类。

mega使用手册Mega使用手册欢迎阅读Mega使用手册。
本文档将详细介绍如何使用Mega以及各项功能的使用方法。
请按照以下章节进行阅读。
1.简介1.1 Mega的概述1.2 Mega的优势1.3 Mega的功能2.账户管理2.1 注册Mega账户2.2 登录和注销账户2.3 密码重置和修改2.4 安全性设置3.文件和3.1 文件3.2 文件3.3 文件夹管理和操作3.4 文件共享和协作4.数据同步与备份4.1 同步文件和文件夹4.2 数据备份和恢复4.3 版本控制和历史记录5.文件搜索和过滤5.1 文件搜索功能5.2 文件过滤和筛选6.安全性和隐私6.1 文件加密和解密6.2 权限管理和共享控制6.3 数据安全和隐私保护7.终端与云端互通7.1 Mega客户端的安装和配置 7.2 云端与本地文件同步7.3 终端与云端文件交互8.其他功能8.1 文件预览和编辑8.2 多设备同步8.3 文件批量处理8.4 容量管理和空间优化附件:本文档所涉及的附件可于Mega官方网站的附件页面。
法律名词及注释:1.Mega:一种云存储和文件同步服务,由Mega Limited提供。
2.账户:用户在Mega上注册的个人或企业账户。
3.文件:用户在Mega上和存储的各种类型的文件。
4.文件夹:用户可以在Mega上创建和管理的文件夹,用于组织和管理文件。
5.文件共享:用户可以通过Mega共享,方便他人访问和文件。
6.数据同步:将本地文件同步到云端,并保持文件的最新状态。
7.数据备份:将云端数据备份到其他存储介质,以避免数据丢失。
8.文件加密:使用加密算法对文件进行加密,以保护文件的安全性和隐私性。
感谢阅读本文档,如有任何问题或困惑,请随时联系我们的客户支持团队。

MEGA使用说明书MEGA使用说明书1、简介MEGA是一款跨平台的云存储和文件共享服务提供商。
它为用户提供了安全、稳定且易于使用的云端存储解决方案。
本文档将详细介绍MEGA的功能和使用方法,以帮助用户快速上手并充分利用该平台。
2、注册和登录2.1 注册账号2.1.1 打开MEGA官方网站2.1.2 注册按钮2.1.3 输入个人信息并创建账号2.2 登录账号2.2.1 打开MEGA官方网站2.2.2 输入注册时所使用的账号和密码2.2.3 登录按钮3、文件和3.1 文件3.1.1 在MEGA界面中找到需要的文件夹3.1.2 按钮3.1.3 选择本地文件并确认3.2 文件3.2.1 在MEGA界面中找到需要的文件3.2.2 右键文件,并选择选项3.2.3 选择本地保存路径并开始4、文件夹管理4.1 创建文件夹4.1.1 在MEGA界面中找到所需文件夹的位置4.1.2 右键文件夹的上级目录,并选择新建文件夹选项 4.1.3 输入文件夹名称并确认创建4.2 移动文件夹4.2.1 在MEGA界面中找到要移动的文件夹4.2.2 右键文件夹,并选择移动选项4.2.3 选择目标位置并确认移动4.3 删除文件夹4.3.1 在MEGA界面中找到要删除的文件夹 4.3.2 右键文件夹,并选择删除选项4.3.3 确认删除操作5、文件共享和权限管理5.1 共享文件5.1.1 在MEGA界面中找到要共享的文件 5.1.2 右键文件,并选择共享选项5.1.3 输入共享和访问权限,并共享5.2 管理共享5.2.1 在MEGA界面中找到需要管理的共享 5.2.2 右键共享,并选择管理选项5.2.3 根据需要修改共享权限或取消共享5.3 接受/拒绝共享5.3.1 在接收到共享的通知邮件中5.3.2 登录MEGA账号5.3.3 根据需求接受或拒绝共享请求6、安全和隐私设置6.1 修改密码6.1.1 登录MEGA账号6.1.2 进入个人设置界面6.1.3 找到密码修改选项并进行修改操作6.2 启用二次验证6.2.1 登录MEGA账号6.2.2 进入个人设置界面6.2.3 找到二次验证选项并进行配置6.3 隐私设置6.3.1 登录MEGA账号6.3.2 进入个人设置界面6.3.3 根据需求修改隐私设置7、恢复和备份7.1 回收站7.1.1 在MEGA界面中找到回收站选项7.1.2 找到需要恢复的文件或文件夹7.1.3 右键文件或文件夹并选择还原选项7.2 历史版本7.2.1 在MEGA界面中找到历史版本选项7.2.2 找到需要恢复的文件7.2.3 右键文件并选择历史版本选项7.2.4 选择要恢复的版本并确认恢复8、常见问题解答9、附件本文档涉及附件:- MEGA用户手册:pdf- MEGA常见问题与解答:docx10、法律名词及注释- MEGA:MEGA是MEGA Limited公司的注册商标,是一家提供云存储和文件共享服务的公司。

MEGA使用说明书MEGA是一款提供云存储服务的应用,它可以帮助用户上传、存储和共享文件。
本文将介绍如何在不同平台上使用MEGA。
第一步:注册帐号在使用MEGA之前,首先需要注册一个帐号。
用户可以通过MEGA官网或者应用程序进行注册。
注册流程1.打开MEGA官网或者应用程序;2.点击“注册”;3.输入电子邮件地址和密码;4.通过电子邮件验证注册帐号。
注意事项1.MEGA仅支持使用电子邮件进行注册,不支持使用手机号注册;2.为了保障帐号的安全,建议使用不易被猜测的密码,并开启双重身份验证。
第二步:下载MEGA应用在注册完帐号后,用户可以下载MEGA应用。
目前MEGA提供了桌面客户端和移动端应用。
用户可以根据自己的需求选择下载。
下载流程下面将介绍如何在不同平台上下载MEGA应用。
Windows1.打开MEGA官网;2.点击“立即下载”;3.在弹出的下载页面中,选择Windows版本;4.下载完后,双击MEGA安装程序,按照提示完成安装。
macOS1.打开MEGA官网;2.点击“立即下载”;3.在弹出的下载页面中,选择macOS版本;4.下载完后,双击DMG文件,将MEGA拖动到应用程序中。
Android1.打开Google Play商店;2.搜索“MEGA”应用;3.点击“安装”;4.下载、安装完成后,打开应用程序。
iOS1.打开App Store;2.搜索“MEGA”应用;3.点击“获取”,输入指纹或密码;4.下载、安装完成后,打开应用程序。
注意事项1.在下载安装应用程序时,需要保障网络通畅,同时保障设备空间充足;2.应用程序可能会因地区、设备、系统等因素而有所不同。
用户在下载安装时需要根据自己的实际情况进行选择。
第三步:上传文件在安装完成MEGA应用后,用户可以开始使用了。
下面将介绍如何上传文件。
上传流程1.打开MEGA应用程序;2.点击“上传”按钮;3.选择要上传的文件;4.点击“上传”。
注意事项1.上传的文件格式应符合MEGA规定,不支持上传非法内容;2.上传的文件大小应符合MEGA规定,如果超过限制,建议将文件分割成较小的部分。
第一步:打开软件下面介绍菜单的使用:Data菜单:Creat a new :创建一个新的数据比对文件,也就是说当我们比对完一组后,想接着比对另一组,那么使用它就可以不用退出直接把数据文件导入;Open :打开先前已经比对并保存好的文件,它包含两个子菜单: retive sequence from file 和saved aligment session ;Close:关闭当前的比对数据文件;Save session :保存当前比对结果,可以给比对的结果一个文件名;Export alignment :将当前的序列比对结果输出到指定文件,有两种输入格式可供选择:MGTA 和FASTA。
DNA sequence :使用它来选择输入的数据 DNA 序列,这里需要说明的是如果你输入的数据是氨基酸序列的话,比对窗口只显示一个标签,若是 DNA 序列的话则显示两个标签,一个是 DNA 序列的,另一个是氨基酸序列的.Protein sequences :选择输入的氨基酸序列,选择后,所以的位点就被当作氨基酸残基位点来对待.Translate/untranslate :只有比对的序列是编码蛋白的 DNA序列的时候才可用。
它可以根据指定的遗传密码表将 DNA 序列翻译成特定的氨基酸序列。
Select genetic code table :使用它将编码蛋白的 DNA 翻译成特定的蛋白序列。
R everse complement :将选择的一整行的 DNA 序列变为与之互补配对碱基序列。
Exit alignment explorer :退出序列比对的资源管理窗口Edit 菜单:使用这个菜单可以对我们的比对序列进行想要的一些编辑工作具体为Undo:撤销上一步操作;Copy:复制;Cut:剪切; Paste:粘贴;这三个操作都可以只针对一个碱基或氨基酸残基也可以是一段甚至是整个序列;Delete:从比对表格中删除一段序列;Delete gaps:去掉序列中的空缺;Insert blank sequence:重新插入一空行;标签和序列都是空的;Insert sequence from file :从已保存的文件中插入新的序列;Select sites :选择一列序列,与点击比对表上方的灰白空格作用类似;Select sequence:选择一行序列,与点击比对表格左侧的标签名作用类似;Select all:全选;Allow base editing :只读保护,只有选择后才能对序列进行编辑操作,否则所以的序列为只读格式,不能进行任何编辑操作。
MEGA软件的使用引言现代分子生物学所积累的数据库(如美国国家生物信息中心建立的GeneBank等)隐含着大量的生物系统学和生物进化的有用信息。
计算机软件是挖掘这些知识宝藏的最有效的工具,而且这些数据库不断快速扩展,信息量十分庞大。
因此,如果没有计算机软件的帮助,我们简直无法开战分子系统学和分子进化方面的研究工作。
同样,这些数据分析方法和软件在古DNA研究中是必不可少的。
因为有着坚实的分子进化和人类遗传学基础,序列比对分析已经成为重构物种和基因家族进化历史,估算分子进化速率、推断基因和基因组进化过程中自然选择力量的强度等的必不可少的方法和手段。
计算机的应用和统计学的介入大大简化这些工作。
在这些背景下,Sudhir Kumar、Koichiro Tamura和Masatonshi Nei 和在上世纪九十年代初就发展了Mega遗传分析软件,并不断改进。
现在公布了3.0版,增添很多新功能,并使软件使用者能在线取得帮助。
Mega(Molecular Evolutionary Genetics Analysis)是一个界面友好、操作简便、功能强大的分子进化遗传分析软件,也是文献中经常用到的分析软件。
尤其是,Mega的新版本对使用界面做了优化,并有改进了许多统计学和遗传学算法,其支持的文件格式很多,而且可以直接从测序图谱中读取序列。
另外,Mega 软件还内嵌了一个Web浏览器,能直接登录NCBI网站。
Mega软件操作起来很方便,其界面与传统的Windows程序界面很像,即使初学者也很易上手。
Mega软件功能十分强大,尤其在计算遗传距离、构建分子系统树方面。
Mega 软件提供多种计算距离的模型,包括Jukes-Cantor距离模型、Kimura距离模型、Equal-input距离模型、Tamura距离模型、HEY距离模型、Tamura-Nei距离模型、General reversible距离模型、无限制距离模型等。
ArduPilot Mega 说明书这里是 ArduPlane wiki,内容包括所有的组装和使用说明。
请使用侧边栏的功能导览。
注意: 如果要找 ArduCopter 的操作说明,请到这里。
? ? ? ? ? ? ? 介绍项目历史项目新闻购买说明书快速入门指南 o APM 2 快速入门指南 o APM 1 快速入门指南设置 o APM2 ? ? ? ? APM 2 板下载及安装 Mission Planner 和飞行软件连接遥控设备首次设置 ? ? o APM 1 ? ? ? ? 组装下载与安装 Mission Planner 及其他飞行软件连接遥控设备首次设置 ? ? 检查传感器逆转舵机和设置普通/升降副翼模式 ? ? ? 飞行 o o APM 开机和校准调整 ArduPlane ? ? ? ? 常用飞机的配置文件 MAVLink 参数说明进阶设定使用地面控制站 ? Mission Planner 使用任务规划器使用硬件开关检查传感器逆转舵机和设置普通/升降副翼模式?HappyKillmore 地面站 QGroundControl ? ? 航点 Widget 参数 Widgeto使用任务规划工具规划和分析任务 ? ? ? ? ? ? ? ? ? 规划航点和任务使用地面站录制和播放任务下载和分析飞行数据配置 PID 使用串口终端与 PC 飞行仿真器交互Python scripting 其他特性增稳模式线控模式自动驾驶模式返航模式(RTL) 盘旋模式o飞行模式 ? ? ? ? ?ooooo ? 模拟 o oo ?使用数据记录器自动起飞和降落启用倒飞设置地理围栏线性飞行的最低高度限制使用 X-Plane 进行半硬件仿真使用 FlightGear 仿真器使用软件再环仿真器可选附件 o 使用无线遥测数据和飞行实时命令 ? APM 1 ? ? ? APM 2 ? ? o ooooooooooo 空速计电压和电流传感器自动襟翼磁力仪简单的相机控制使用游戏游戏杆替代遥控自动相机追踪多副翼通道其他模拟传感器其他 I2C 传感器 3DR Radio Xbee 3DR Radio XbeeOn-screen display (光流传感器)用命令解析器进行高级设置和测试 ? ? 设置飞行模式测试模式 ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? 遥控输入 GPS 输入 IMU 输出陀螺和加速度传感器输入电池输入(可选) 继电器输出航路显示空速计输出(可选) 绝对气压(高度)传感器输出磁力计输出(可选) Xbee 测试(可选) 导出 EEPROM GPS 原始输入日志指南空速计指南 Xbee 指南电压传感器指南高级设置? ?疑难解答 o 疑难解答附录 o oooo 正确的 LED 行为设置 RC 发射器的 6 种模式失效保护功能命令提示符 (CLI) 设置和测试使用 Arduino 编程 ? ? ? o ooooo 设置编程环境使用 Arduino 为 APM 编程下载和使用 APM 软件 ? 设置选项使用 AVR Studio 刻录程序理解状态/诊断串口输出理解偏航距 APM MAV 命令使用调试终端地面站重刷/更新 GPS 固件 ? ? ? uBlox GPS MediaTek GPS 其他 GPS 模块 APM 2 APM 1 ? ? 使用 AVRStudio 为编码器重建/编程使用 Bus Pirate 为编码器编程o更新 PPM encoder 轫体 ? ?ooAPM 硬件技术细节 ArduPilot Mega 主板ArduPilot Mega IMU 传感器板 APM 代码贡献者指南 ? 使用 Git ? ? ? ? ? ? ? ? 逐步引导使用 Git 贡献代码使用 Git 命令行使用 Git 扩展工具使用 TortoiseGit代码规范使用 Eclipse 编译 ArduPilot Mega 使用 make 编译 ArduPilot Mega 使用JTAG 调试 ArduPilot Megao ? ? 词汇表教学图片来源The DIY Drones Dev Team介绍欢迎使用 ArduPlane 操作说明,此说明会告诉你如何将一般的遥控飞机变为自动驾驶的无人机。
MEGA软件的使用引言现代分子生物学所积累的数据库(如美国国家生物信息中心建立的GeneBank等)隐含着大量的生物系统学和生物进化的有用信息。
计算机软件是挖掘这些知识宝藏的最有效的工具,而且这些数据库不断快速扩展,信息量十分庞大。
因此,如果没有计算机软件的帮助,我们简直无法开战分子系统学和分子进化方面的研究工作。
同样,这些数据分析方法和软件在古DNA研究中是必不可少的。
因为有着坚实的分子进化和人类遗传学基础,序列比对分析已经成为重构物种和基因家族进化历史,估算分子进化速率、推断基因和基因组进化过程中自然选择力量的强度等的必不可少的方法和手段。
计算机的应用和统计学的介入大大简化这些工作。
在这些背景下,Sudhir Kumar、Koichiro Tamura和Masatonshi Nei 和在上世纪九十年代初就发展了Mega遗传分析软件,并不断改进。
现在公布了3.0版,增添很多新功能,并使软件使用者能在线取得帮助。
Mega(Molecular Evolutionary Genetics Analysis)是一个界面友好、操作简便、功能强大的分子进化遗传分析软件,也是文献中经常用到的分析软件。
尤其是,Mega的新版本对使用界面做了优化,并有改进了许多统计学和遗传学算法,其支持的文件格式很多,而且可以直接从测序图谱中读取序列。
另外,Mega 软件还内嵌了一个Web浏览器,能直接登录NCBI网站。
Mega软件操作起来很方便,其界面与传统的Windows程序界面很像,即使初学者也很易上手。
Mega软件功能十分强大,尤其在计算遗传距离、构建分子系统树方面。
Mega 软件提供多种计算距离的模型,包括Jukes-Cantor距离模型、Kimura距离模型、Equal-input距离模型、Tamura距离模型、HEY距离模型、Tamura-Nei距离模型、General reversible距离模型、无限制距离模型等。
Mega软件可以计算个体之间的遗传距离,还可估算群体间的遗传差异,及群体间的净遗传距离;而其还可以估算一个群体或整个样本的基因分歧度的大小。
另外Mega还提供了多种构建分子系统树的方法,包括算术平均的不加权对群法(UPGMA,unweighted pair group method with arithmetic mean),邻接法(NJ,Neighbor-Joining),最大简约法(MP,Maximum Parsimony)、最小进化法(ME,Minimum Evolution)等。
在此基础上,Mega软件还提供了对已构建系统树的检验,包括自展法(Bootstrap Method)检验和内部分支检验等。
在对于自然选择方面,Mega软件提供了Codon-Based Z 检验、Codon-Based Fisher`s 原样检验t和Tajima中性检验三种方法。
总之,Mega 软件提供了构建分子系统树,进行系统发育分析各个方面的计算和分析。
本章将以古DNA数据分析为例,介绍Mega软件的基本原理和方法、使用和操作、以及相关结果的分析。
Mega软件包的下载网址为:Mega软件输入数据的格式Mega软件输入数据的格式比较简单,在众多遗传学分析软件中是比较容易制作的一种。
首先,如果输入数据是一般的DNA或RNA序列,则有如下要求:1)文件扩展名以*.meg或*.txt结尾都行;2)输入数据文件,第一行必须有Mega程序所需的特殊标记“#MEGA”;3)“TITLE”位于输入文件的第二行,后边可以跟上一些说明性字符,这些字符在输出结果中会显示出来。
在与“Title”同一行上的字符才有效,而且字符总数不能超过128,超过的也会被忽略。
4)在“#MEGA”和“TITLE”之后,在分析数据之前可以一行或多行的说明性文字。
这些文字可用来说明诸如作者、分析日期、分析目的等信息。
5)在每个数据(或每条序列)的名字之前应该有一个“#”,名字的下一行是具体的序列。
在同一个数据文件里,不能出现数据名相同的序列。
在数据名及具体序列中,空格和TAB是被忽略的。
6)在同一数据文件内,所有序列的长度应该保持一致,否则,程序不能执行。
7)对于DNA或RNA序列,Mega软件能够识别A、T、C、G、U五种字符,缺失字符可以用“?”表示,比对时的空缺位点可以用“—”表示。
下边是一个数据文件示例:Fig其次,如果输入数据是遗传距离矩阵,则要求如下:1)前4点要求同对上述DNA序列的要求相同;2)在每个距离矩阵的名字之前应该有一个“#”,每个名字占一行;先列出距离矩阵的名字,然后再给出距离矩阵;3)距离矩阵有两种形式,下三角和上三角。
下边是一个数据文件示例:下图是距离矩阵的示意图,左边是下三角矩阵,右边是上三角矩阵。
再次,如果数据是测序图谱的形式,直接导入即可。
下图是测序图谱示例:FigMEGA界面及操作Mega是一款操作十分简便的遗传学分析软件,其界面十分友好,即使初学者也很易上手。
1、数据的录入及编辑Mega软件能够接受多种数据格式,如FASTA格式、Phylip格式、PAUP数据格式等等。
而且Mega软件专门提供了把其他格式的数据转换位Mega数据格式的程序。
首先,打开Mega程序,有如下图所示的操作界面:Fig单击工具栏中的“File”按钮,会出现如下图所示的菜单:Fig从上图可以看出,下拉菜单有“Open Data”(打开数据)、“Reopen Data”(打开曾经打开的数据,一般会保留新近打开的几个数据)、“Close Data”(关闭数据)、“Export Data”(导出数据)、“Conver To MEGA Format”(将数据转化为MEGA 格式)、“Text Editor”(数据文本编辑)、“Printer Setup”(启动打印)、“Exit”(退出MEGA程序)。
单击“Open Data”选项,会弹出如下菜单:Fig浏览文件,选择要分析的数据打开,单击“打开”按钮,会弹出如下操作界面:Fig此程序操作界面,提供了三种选择数据选择:Nucleotide Sequences(核苷酸序列)、Protein Sequences(蛋白质序列)、Pairwise Distance(遗传距离矩阵)。
根据输入数据的类型,选择一种,点击“OK”即可。
如果选择“Pairwise Distance”,则操作界面有所不同;如下图所示:Fig根据遗传距离矩阵的类型,如果是下三角矩阵,选择“Lower Left Matrix”即可;如果是上三角矩阵,选择“Upper Right Matrix”即可。
点击“OK”按钮,即可导入数据。
如果是核苷酸数据,则读完之后,会弹出如下对话框:Fig如上图,如果是编码蛋白质的核苷酸序列,则选择“Yes”按钮;如果是不编码蛋白质的核苷酸序列,则点击“No”按钮。
之后,会弹出如下操作窗口:Fig此作界面的名称是“Sequence Data Explorer”,在其最上方是工具栏“Data”、“Display”、“Highlight”等,然后是一些数据处理方式的快捷按钮,在操作界面的左下方是每个序列的名称。
显示序列占了操作界面的绝大部分,与第一个序列相同的核苷酸用“.”表示,发生变异的序列则直接显示。
如果在弹出的对话框中,点击“OK”,即选择输入的数据是编码蛋白质的DNA序列。
那么会再弹出如下对话框:Fig此操作界面提供了多种生物的遗传密码方式的选择,如Vertebrate Mitochondrial(脊椎动物线粒体)、Invertebrate Mitochondrial(非脊椎动物线粒体)、Yeast Mitochondrial(酵母线粒体)等等。
点击此操作界面的“Add”按钮,可以添加密码子表格,其编辑界面如下图所示:Fig通过此操作界面可以创建、修改密码子表格。
点击“OK”按钮可以返回“Select Genetic Code”操作界面。
点击“Select Genetic Code”操作界面的“Delect”按钮,可以删除一个密码子表。
点击“Select Genetic Code”操作界面的“Edit”按钮,可以对已经存在的密码子表格。
其操作界面与“Genetic Code Table”相同。
点击“Select Genetic Code”操作界面的“View”按钮,可以浏览选中的密码子表格。
点击“Select Genetic Code”操作界面的“Statistics”按钮,可以统计密码子表格的一些信息,如每种密码子的频率、同义位点数、非同义位点数等。
点击点击“Select Genetic Code”操作界面的“OK”按钮,会弹出如上图所示的“Sequence Data Explorer”操作界面。
如果点击“Cancel”按钮,也会弹出此操作界面,但是此时会把数据默认为非编码的DNA序列。
单击“Sequence Data Explorer”操作界面工具栏的“Data”按钮,有如下图所示的下拉菜单:Fig下拉菜单有六个选项:“Write Data To File”(将数据转到文件中,利用此选项可以把Mega数据格式的数据转化成其它格式)、“Translate/Untranslate”(是否翻译,这个选项只有所分析的DNA序列是编码序列时才被激活)、“Selcet Genetic Code Table”(选择遗传密码表,这个选项只有所分析的DNA序列是编码序列时才被激活)、“Setup/Selcet Genes&Domains”(选择或设置基因或结构域)、“Setup/Select Taxa&Group”(对数据进行分组)、“Quit Data Viewer”(退出此浏览框)。
单击“Write Data To File”选项,会弹出如下对话框:FigTitle框显示的内容是数据文件中“TITLE”之后的内容。
Description框显示的内容是数据文件中对整体数据描述的内容。
Format选项提供一个下来菜单,通过此下拉菜单可以把数据转化为MEGA 格式、Nexus(PAUP4.0)格式,PHYLIP3.0格式、Nexus(PAUP3.0/MacClade)格式。
Writing site numbers 选项也提供一个下拉菜单,通过此下来菜单可以把给每个核苷酸标序号,“None”为不显示序号,“For each site”为每个位点显示序号,“At the end of line”在每一行行末显示序号。
Missing Data and alignment gaps选项也提供了一个下拉式菜单,这个菜单包括:“Include sites with miss/ambiguous data gaps”(显示缺失位点及模糊位点以及空缺)、“Exclude sites with miss/ambiguous data gaps”(不显示缺失位点及模糊位点以及空缺)、“Exclude sites with miss/ambiguous data only”(仅不显示缺失位点及模糊位点)、“Exclude sites with alignment gaps only”(仅不显示比对是的空缺部分)。