序列分析DNAMAN极简使用方法
- 格式:pdf
- 大小:548.16 KB
- 文档页数:4

查看文章DNAMAN使用说明书(中文)2008年04月16日星期三下午10:50DNAMAN 是一种常用的核酸序列分析软件。
由于它功能强大,使用方便,已成为一种普遍使用的DNA 序列分析工具。
本文以DNAMAN 5.2.9 Demo version 为例,简单介绍其使用方法。
打开DNAMAN,可以看到如下界面:第一栏为主菜单栏。
除了帮助菜单外,有十个常用主菜单,第二栏为工具栏:第三栏为浏览器栏:在浏览器栏下方的工作区左侧,可见Channel 工具条,DNAMAN 提供20 个Channel,(如左所示:)点击Channel 工具条上相应的数字,即可击活相应的Channel。
每个Channel 可以装入一个序列。
将要分析的序列(DNA 序列或氨基酸序列)放入Channel 中可以节约存取序列时间,加快分析速度。
此版本DNAMAN 提供自动载入功能,用户只需激活某个Channel,然后打开一个序列文件,则打开的序列自动载入被激活的Channel 中。
本文以具体使用DNAMAN 的过程为例来说明如何使用DNAMAN 分析序列。
1.将待分析序列装入Channel(1)通过File Open 命令打开待分析序列文件,则打开的序列自动装入默认Channel。
(初始为channel1)可以通过激活不同的channel (例如:channel5)来改变序列装入的Channel。
(2)通过Sequence/Load Sequence 菜单的子菜单打开文件或将选定的部分序列装入Channel 。
通过Sequence/Current Sequence/Analysis Defination 命令打开一个对话框,通过此对话框可以设定序列的性质(DNA 或蛋白质),名称,要分析的片段等参数。
2.以不同形式显示序列通过Sequence//Display Sequence 命令打开对话框,如下图所示:根据不同的需要,可以选择显示不同的序列转换形式。


DNAMAN中文使用说明好不容易找到了一个中文说明,希望可以帮助初学使用DNAMAN的朋友,更快的进入状态,当然现在网上也有汉化版的软件了,但是这个说明还是可以起到很好的帮助作用,与大家分享!DNAMAN 是一种常用的核酸序列分析软件。
由于它功能强大,使用方便,已成为一种普遍使用的DNA 序列分析工具。
本文以DNAMAN 5.2.9 Demo version 为例,简单介绍其使用方法。
打开DNAMAN,可以看到如下界面:第一栏为主菜单栏。
除了帮助菜单外,有十个常用主菜单,第二栏为工具栏:第三栏为浏览器栏:在浏览器栏下方的工作区左侧,可见Channel 工具条,DNAMAN 提供20 个Channel,(如左所示:)点击Channel 工具条上相应的数字,即可击活相应的Channel。
每个Channel 可以装入一个序列。
将要分析的序列(DNA 序列或氨基酸序列)放入Channel 中可以节约存取序列时间,加快分析速度。
此版本DNAMAN 提供自动载入功能,用户只需激活某个Channel,然后打开一个序列文件,则打开的序列自动载入被激活的Channel 中。
本文以具体使用DNAMAN 的过程为例来说明如何使用DNAMAN 分析序列。
1.将待分析序列装入Channel(1)通过File Open 命令打开待分析序列文件,则打开的序列自动装入默认Channel。
(初始为channel1)可以通过激活不同的channel (例如:channel5)来改变序列装入的Channel。
(2)通过Sequence/Load Sequence 菜单的子菜单打开文件或将选定的部分序列装入Channel 。
通过Sequence/Current Sequence/Analysis Defination 命令打开一个对话框,通过此对话框可以设定序列的性质(DNA 或蛋白质),名称,要分析的片段等参数。
2.以不同形式显示序列通过Sequence//Display Sequence 命令打开对话框,如下图所示:根据不同的需要,可以选择显示不同的序列转换形式。



dnaman比对序列结果解读
DNAman是一种常用的生物信息学软件,可以用于比对DNA序列并解读结果。
比对序列结果的解读对于研究人员来说非常重要,因为它提供了有关DNA序列相
似性、突变等信息。
下面我将解读一下比对序列结果:
在DNAman中,比对序列结果通常以多种方式呈现,包括序列比对图、相似
性矩阵、突变分析等。
首先,我们可以通过序列比对图来直观地了解两个或多个DNA序列的相似性和差异性。
比对图中使用不同的符号和颜色表示碱基匹配、插
入或删除等突变类型。
通过比对图,我们可以快速识别出序列间的相似性和差异性。
相似性矩阵是另一种常见的结果展示方式。
它以表格的形式呈现两个或多个序
列间的相似度比较结果。
矩阵中的每个单元格表示对应位置上的碱基匹配得分。
相似性矩阵可以帮助我们快速计算两个序列的相似程度,并可视化揭示出可能的突变类型。
此外,比对序列结果还可以提供详细的突变分析。
它可以标记出序列间的碱基
突变、插入和删除等变异类型,同时计算每种突变类型的频率。
这些详细的突变信息对于研究者来说非常有价值,可以帮助他们了解到底哪些位置上发生了变异,从而进一步研究其对生物体的功能和表型产生的影响。
综上所述,通过DNAman比对序列结果的解读,我们可以获得关于DNA序列
相似性、突变类型和频率等重要信息。
这些信息对于深入研究基因组学、分子进化和病毒变异等领域非常有帮助。
DNAman的强大功能可以帮助科研人员更好地理
解和解释DNA序列比对结果。

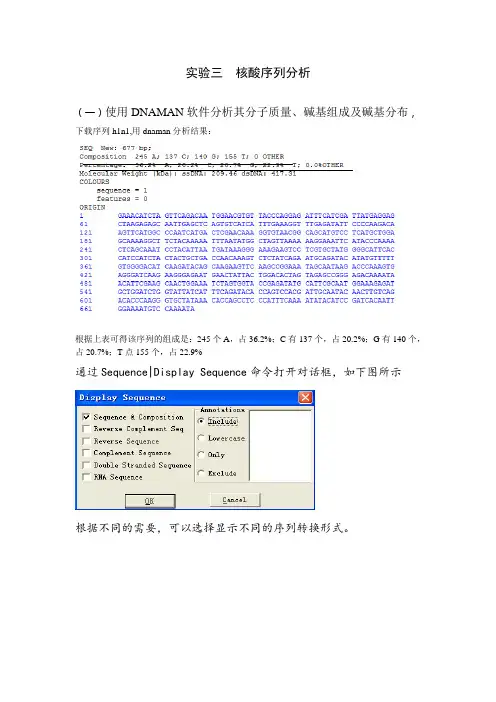
实验三核酸序列分析(一)使用DNAMAN软件分析其分子质量、碱基组成及碱基分布,下载序列h1n1,用dnaman分析结果:根据上表可得该序列的组成是:245个A,占36.2%;C有137个,占20.2%;G有140个,占20.7%;T点155个,占22.9%通过Sequence|Display Sequence命令打开对话框,如下图所示根据不同的需要,可以选择显示不同的序列转换形式。
点击Restriction/Restriction Analysis,选择其中一些参数,可分析当前Channel序列酶切位点。
参数说明如下:Results 分析结果显示其中包括:Show summary(显示概要)Show sites on sequence(在结果中显示酶切位点)Draw restriction map(显示限制性酶切图)Draw restriction pattern(显示限制性酶切模式图)Ignore enzymes with more than(忽略切点多于设定的切点个数的酶)Ignore enzymes with less than(忽略切点少于设定的切点个数的酶)Target DNA (目标DNA特性)circular(环型DNA),dam/dcm methylation(dam/dcm甲基化)all DNA in Sequence Channel(选择此项,在Sequence Channel 中的所有序列将被分析,如果选择了Draw restriction pattern,那么当所有的channel中共有两条DNA时,则只能选择两个酶分析,如果共有三个以上DNA时,则只能用一个酶分析。
限制性酶切分析进行PCR引物设计:构建系统发育树点击左上角按钮,可以从弹出的对话框中选择不同的结果显示特性选项。
点击按钮下的按钮,出现下列选择项:可以通过这些选项,绘制同源关系图(例如Tree|homology tree命令)。
如何用DNAMAN对比核酸序列在生物学的研究中,有一个常用的方法,就是通过比较分析获取有用的信息和知识。
想当年,达尔文爷爷正是研究比较了达尔文雀(galapagos finches)同其它一些物种的形态学特征,从而提出了自然选择学说。
在科技飞速发展的今天,对两个物种进行全基因组序列比较已经不再是一个梦想。
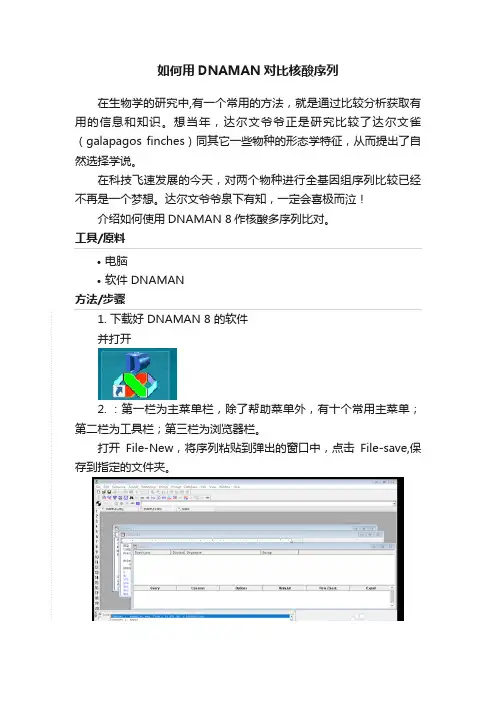
达尔文爷爷泉下有知,一定会喜极而泣!介绍如何使用DNAMAN 8作核酸多序列比对。
工具/原料•电脑•软件 DNAMAN方法/步骤1.下载好 DNAMAN 8 的软件并打开2.:第一栏为主菜单栏,除了帮助菜单外,有十个常用主菜单;第二栏为工具栏;第三栏为浏览器栏。
打开File-New,将序列粘贴到弹出的窗口中,点击File-save,保存到指定的文件夹。
3.END方法/步骤21.将所需比对的序列保存好以后,选中Sequence—Aligment—Multiple aligment sequence进行多序列比较。
2.在弹出的窗口Sequence&Files中加载序列,File、Fold、channel、Database分别表示从文件、文件夹、channel和数据库中获取序列。
勾选窗口中的“DNA”,点击“下一步”。
3.在弹出的窗口Method中,“optimalaligment”最佳比对方式中有四个高大上的选项:Full Alignment(完全比对)、Prosile Aligment(轮廓比对)、 New Swquence on Profile (轮廓上的新序列)、Fast Alignment(快速比对),本文选择了Fast Alignment,并且勾选了Try both strands(尝试使用双链)。
其他项目不需修改,默认就OK,点击“下一步”。
4.“Duang”,结果就出来啦!点击“Option”可以选择要显示的内容。
END方法/步骤31.保存:点击File-output-Graphic(EMF)File,该序列比对的结果窗口可以图片格式保存。

DNAMAN使用说明书DNAMAN 是一种常用的核酸序列分析软件。
由于它功能强大,使用方便,已成为一种普遍使用的DNA 序列分析工具。
本文以DNAMAN Demo version 为例,简单介绍其使用方法。
翻开DNAMAN,可以看到如下界面:第一栏为主菜单栏。
除了帮助菜单外,有十个常用主菜单,第二栏为工具栏:第三栏为阅读器栏:在阅读器栏下方的工作区左侧,可见Channel 工具条,DNAMAN 提供20 个Channel,(如左所示:)点击Channel 工具条上相应的数字,即可击活相应的Channel。
每个Channel 可以装入一个序列。
将要分析的序列〔DNA 序列或氨基酸序列〕放入Channel 中可以节约存取序列时间,加快分析速度。
此版本DNAMAN 提供自动载入功能,用户只需激活某个Channel,然后翻开一个序列文件,那么翻开的序列自动载入被激活的Channel 中。
本文以详细使用DNAMAN 的过程为例来说明如何使用DNAMAN 分析序列。
1.将待分析序列装入Channel〔1〕通过File Open 命令翻开待分析序列文件,那么翻开的序列自动装入默认Channel。
〔初始为channel1〕可以通过激活不同的channel (例如:channel5)来改变序列装入的Channel。
〔2〕通过Sequence/Load Sequence 菜单的子菜单翻开文件或将选定的局部序列装入Channel 。
通过Sequence/Current Sequence/Analysis Defination 命令翻开一个对话框,通过此对话框可以设定序列的性质〔DNA 或蛋白质〕,名称,要分析的片段等参数。
2.以不同形式显示序列通过Sequence择需要的选项,点击按钮,出现:点击按钮,完成操作。
7.画质粒形式图我们常常要用到各种质粒图,无论是制作幻灯片,还是发表文章,常常需要质粒图。
DNAMAN提供强大的绘质粒图功能,能满足我们的需要。
序列分析软件DNAMAN的使用方法简介吕惠平(新疆大学生命科学与技术学院;新疆生物资源基因工程重点实验室,乌鲁木齐,830046) DNAMAN是一种常用的核酸序列分析软件。
由于它功能强大,使用方便,已成为一种普遍使用的DNA序列分析工具。
本文以DNAMAN 5.2.9 Demo version为例,简单介绍其使用方法。
打开DNAMAN,可以看到如下界面:第一栏为主菜单栏。
除了帮助菜单外,有十个常用主菜单,第二栏为工具栏:第三栏为浏览器栏:在浏览器栏下方的工作区左侧,可见Channel工具条,DNAMAN提供20个Channel,(如左所示:)点击Channel工具条上相应的数字,即可击活相应的Channel。
每个Channel可以装入一个序列。
将要分析的序列(DNA 序列或氨基酸序列)放入Channel中可以节约存取序列时间,加快分析速度。
此版本DNAMAN提供自动载入功能,用户只需激活某个Channel,然后打开一个序列文件,则打开的序列自动载入被激活的 Channel中。
本文以具体使用DNAMAN的过程为例来说明如何使用DNAMAN分析序列。
1.将待分析序列装入Channel(1)通过File Open命令打开待分析序列文件,则打开的序列自动装入默认Channel。
(初始为channel1)可以通过激活不同的channel (例如:channel5)来改变序列装入的Channel。
(2)通过Sequence/Load Sequence菜单的子菜单打开文件或将选定的部分序列装入Channel。
通过Sequence/Current Sequence/Analysis Defination命令打开一个对话框,通过此对话框可以设定序列的性质(DNA 或蛋白质),名称,要分析的片段等参数。
2.以不同形式显示序列通过Sequence//Display Sequence命令打开对话框,如下图所示:根据不同的需要,可以选择显示不同的序列转换形式。
dnaman比对序列结果解读摘要:一、简介:Dnaman比对序列的基本概念二、比对结果的解读方法1.匹配度分析2.一致性分析3.变异分析4.基因型分析三、比对结果的实用意义1.在遗传病筛查中的应用2.在基因突变检测中的应用3.在遗传多样性研究中的应用四、结论:Dnaman比对序列在生物技术领域的重要性正文:随着生物信息学的发展,Dnaman比对序列已成为生物学研究中不可或缺的工具。
它通过将目标序列与参考序列进行比对,从而揭示两者之间的相似性和差异性。
比对结果的解读在生物学研究中具有重要意义,可以帮助我们深入了解基因、变异和遗传等方面的问题。
在Dnaman比对序列中,匹配度是指两条序列之间相同或相似的碱基数量。
通过分析匹配度,我们可以了解目标序列与参考序列之间的相似性。
高匹配度表示两条序列具有较高的相似性,可能来源于同一个祖先序列;低匹配度则表示两条序列之间差异较大,可能存在不同的功能或表达方式。
一致性分析是评估比对结果中碱基替换、插入和删除等变异情况的指标。
一致性越高,说明目标序列与参考序列在相应区域越稳定;一致性越低,说明该区域变异较多,可能影响基因功能或表达。
通过对一致性进行分析,我们可以了解目标序列在进化过程中的变异情况,为遗传变异研究提供数据支持。
变异分析是比对结果解读中的重要环节。
通过分析比对序列间的变异,我们可以发现致病基因、种间差异等信息。
在遗传病筛查中,通过比对患者样本与正常人群的序列,可以发现患者序列中的致病性变异;在遗传多样性研究中,对比不同个体或种群的序列,可以揭示遗传变异在不同群体中的分布规律。
基因型分析是比对序列在生物技术领域的重要应用之一。
通过比对不同个体或品种的序列,我们可以确定各品种的基因型,进而分析基因型与表型之间的关系。
这对于分子育种、遗传病诊断等领域具有重要意义。
总之,Dnaman比对序列在生物技术领域具有广泛的应用前景。
通过对比对结果的解读,我们可以深入了解基因、变异和遗传等方面的问题,为生物科学研究和应用提供有力支持。
dnaman比对序列结果解读摘要:1.Dnaman 比对序列结果2.结果解读3.结论正文:在生物信息学领域,Dnaman 是一款广泛使用的DNA 比对工具,通过对比两个或多个DNA 序列,可以揭示它们之间的相似性和差异性。
本文将基于Dnaman 比对序列结果,对其进行解读。
首先,我们来了解Dnaman 比对序列结果。
Dnaman 将输入的DNA 序列进行比对,生成比对结果文件,其中包括了比对过程中所涉及到的所有参数。
这些参数包括比对算法、比对分数、插入、删除、替换等。
通过对这些参数的解读,可以了解序列之间的相似程度以及具体的差异所在。
接下来,我们对比对结果进行解读。
比对分数是衡量两个序列相似性的重要指标,分数越高,表示序列之间的相似性越高。
在Dnaman 的比对结果中,会列出每个位置的比对分数。
通过观察这些分数,可以发现序列间的相似性和差异性。
如果分数较低,说明该位置的序列差异较大,可能是由于插入、删除或替换等操作导致的。
而如果分数较高,则说明该位置序列高度保守,即两者间的序列相似。
在解读过程中,还需要注意插入、删除和替换等操作。
这些操作会导致序列间的差异,进而影响比对结果。
插入是指在参考序列中不存在的核苷酸在比对序列中出现,而删除则是指比对序列中的核苷酸在参考序列中不存在。
替换则是指比对序列中的核苷酸与参考序列中的核苷酸不同。
在解读过程中,需要分析这些操作的分布和性质,以便更好地理解序列间的关系。
根据以上分析,我们可以得出结论:通过对Dnaman 比对序列结果的解读,可以揭示DNA 序列之间的相似性和差异性,以及序列间的关系。
这对于研究基因组结构、功能和进化等方面具有重要意义。
水天2014.11.3编辑
DNAMAN 是美国 Lynnon Biosoft 公司开发的高度集成化的分子生物学综合应用软件,可以
用于多序列比对、PCR 引物设计、限制性酶切分析、质粒绘图、蛋白质分析等,几乎囊括了
所有日常核酸、蛋白质序列的分析工作。
下面是软件界面截图的操作流程以及简单说明,其他的一些功能可以自己摸索,都比较简
单。这个软件很容易找到汉化版,一般都没有功能限制,而且很好的一点就是可以保存所有
的比对和分析结果!!有木有,这一点太赞了。
First.
DNA多序列比对
可以批量载入测序结果以及参考序列
在比对方式的选择上,一般选完全比对和快速比对,其他参数一般默认。其中完
全比对只能同时比对一个方向的测序结果,如果正反向同时进入比对,反向会和
正向从一边开始比对,当然就比对不上了。而快速比对可以正反向批量比对。
生物分析软件DNAMAN简单使用方法
2014年11月3日
9:59
分区ANALYSIS 的第1 页
2.
限制性酶切分析
限制性酶切分析需要首先载入目标序列
分区ANALYSIS 的第2 页
选择合适的酶文件以及想要看到的信息,下一步就OK了
分区ANALYSIS 的第3 页
我的示例序列的分析结果
分区ANALYSIS 的第4 页