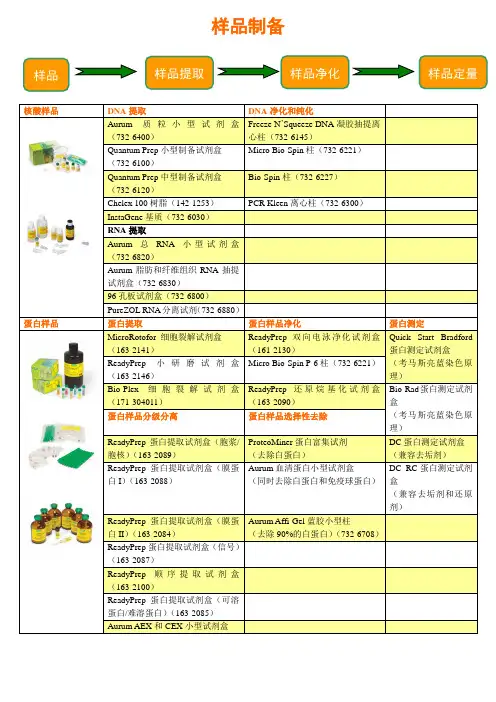
Bio-Plex 核酸偶联操作手册
- 格式:pdf
- 大小:1.08 MB
- 文档页数:13

Biotin偶联蛋白方案目标本方案的目标是将biotin(生物素)与蛋白质偶联,以实现对蛋白质的特异性检测和纯化。
通过该方案,我们希望能够高效、可靠地实现蛋白质的生物素标记,并利用生物素与亲生素-双亲胶体(avidin-biotin system)的高亲和性实现对蛋白质的富集和检测。
实施步骤1. 准备工作在开始实施方案之前,我们需要准备一些必要的试剂和材料: - Biotin-NHS酯:用于将biotin与蛋白质共价结合。
- 目标蛋白质:需要进行偶联的待检测或待纯化的蛋白质样品。
- PBS缓冲液:用于洗涤和稀释样品。
- 硫酸钠:用于调节PBS缓冲液pH值。
2. 生物素与目标蛋白质偶联步骤:1.将目标蛋白质溶解在PBS缓冲液中,确保其浓度在适当范围内(一般为1-10 mg/mL)。
2.准备一定浓度的Biotin-NHS酯溶液,将其加入到目标蛋白质溶液中,使其与蛋白质发生共价结合。
酯化反应需要在碱性条件下进行,因此pH值需调节至8.0-9.0。
3.在室温下轻轻摇晃反应混合物,使其充分反应2-4小时。
4.反应结束后,使用PBS缓冲液洗涤偶联后的蛋白质溶液以去除未反应的Biotin-NHS酯。
3. 检测和纯化偶联后的蛋白质步骤:1.将偶联后的蛋白质样品加入含有亲生素-双亲胶体(如avidin或streptavidin)的柱子或磁珠中。
亲生素-双亲胶体能够与生物素高度特异性结合。
2.在室温下轻轻摇晃混合物,使其充分接触并发生结合反应。
根据需要可延长接触时间(一般为30分钟至数小时)。
3.将柱子或磁珠置于磁架上,使其与上清分离。
上清中的非特异性蛋白质将被去除。
4.使用PBS缓冲液洗涤柱子或磁珠,以去除非特异性结合的蛋白质。
5.使用适当的洗脱缓冲液(如含有高浓度生物素的PBS缓冲液)洗脱目标蛋白质。
洗脱条件需根据具体实验要求进行优化。
预期结果通过本方案,我们预期能够成功地将biotin与目标蛋白质偶联,并利用生物素与亲生素-双亲胶体的高亲和性实现对蛋白质的富集和检测。

BIOG 游离DNA 提取试剂盒操作步骤说明运输条件:常温运输 货 号:51019:50次反应51020:100次反应 储存条件:室温(15-25℃),消化液、DNA Carrier 请于-20℃存放产品特点 试剂盒组成 组分 50次 100次 吸附柱和收集管各50个 各100个 裂解液 18mL 36 mL 洗涤液A 21mL 42 mL 洗涤液B 9 mL 18 mL 洗脱液 3mL 6 mL 消化液 1.2 mL 2.4 mL DNA Carrier 0.24 mL 0.48 mL 说明书1份1份产品介绍BIOG cfDNA Easy Kit 是常州百代生物科技股份有限公司研制的专门用于提取血清、血浆等游离DNA 的试剂盒。
BIOG cfDNA Easy Kit 采用了最新的优质进口离子膜,裂解液和洗脱液经过多次优化,能高效地分离DNA 。
提取得到的DNA 较其它品牌的同类试剂盒产量更大,纯度更高,最大限度地去除了蛋白、色素、脂类等杂质污染,可以直接应用于PCR 、荧光定量PCR 和各种酶切试验等。
操作步骤1. 请自行准备:无水乙醇、1.5mL 离心管。
2. 取出洗涤液,按以下操作:a) 洗涤液A :21mL 加入9mL 无水乙醇;42mL 加入18mL 无水乙醇。
b) 洗涤液B :9mL 加入21mL 无水乙醇;18mL 加入42mL 无水乙醇。
c) 配制好的洗涤液如出现沉淀,可在37℃溶解,摇匀后使用。
3. 取1.5mL 离心管,加入200μL 样本,4μL DNA Carrier 混合均匀,加入300μL 裂解液及20μL 消化液,振荡混匀,56℃水浴10 分钟。
4. 加入1000μL 无水乙醇,轻轻颠倒混匀,如有半透明悬浮物,不影响DNA 的提取与后续实验。
5. 将吸附柱放入收集管内,将760μL 上述溶液转入吸附柱内,静置2分钟,12,000 rpm 4℃离心1 分钟,弃收集管内废液; 6. 将吸附柱放回收集管内,将剩余760μL 溶液转移至吸附柱内,重复步骤5。

CFX96 实时荧光定量PCR仪操作流程及注意事项开始运行仪器打开电脑打开定量PCR仪底座开关启动CFX Manager软件放置样品将PCR反应体系加入到0.2ml低缘八联管,盖上管盖;或加入低缘96孔板,用光学级封膜封好。
注意,必须带一次性塑料手套,不要让手指接触到反应管表面。
将反应管按顺序放入仪器的加热孔中。
设置程序,运行实验定量PCR软件操作基本步骤为:a. 设置热循环程序文件(Protocol Tab) b。
设置反应板文件(Plate Tab). C。
点击“Start Run”键,运行程序热循环程序文件(Protocol Tab)设置指南:点击Edit (编辑)或 Create New(创建新程序).反应板设置文件(Plate Tab)设置指南:选择本次实验所要使用的荧光染料种类;单击样品类型;如要某些反应孔第一荧光染料对应的样品类型为标准品(Standard),点击“Dilution Series”键可设置其标准品浓度及稀释倍数。
点击“Start Run”键。
单击open lid(打开热盖)或Close lid(关闭热盖)放置样品;单击Start Run,保存文件,开始运行程序。
结果分析PCR反应结束后,软件会自动计算标准曲线和Ct值等。
如需进行表达量分析、等位基因分析等,在软件窗口选择相应分析功能.点击右上方的“Report”键,还可输出结果报告单关闭运行仪器实验结束后取出反应管,顺序关闭CFX Manager软件、定量PCR仪电源,关闭电脑.注意!CFX仪器上盖部分为全自动控制,在通电状态,严禁任何人为干涉上盖开启或关闭的行为,此类行为会导致上盖故障,危及仪器使用.CFX Manager软件操作快速指南实验操作程序设置图 1显示实验设置窗口中一个预览的程序.点击Create New,打开程序编辑器创建一个新的程序。
点击Select Existing,通过浏览器加载一个程序或对其进行编辑。

Biomixer II1建立规范标准的Biomixer II 杂交仪操作程序。
2适用于临床分子诊断中心经科主任授权的操作者使用本作业指导书。
3经培训合格后,经授权的检验专业技术人员操作,由科技术主管和质量主管分别负责技术指导和质量监督。
4Biomixer II TM杂交仪是采用芯片杂交清洗系统进行核酸分子杂交检测的仪器设备。
55.1 仪器简介BioMixer TMⅡ芯片杂交仪是博奥生物有限公司根据市场及用户需求开发的新一代芯片杂交清洗系统,通过特有的专利技术实现恒温环境下的三维持续摇摆运动,使杂交盒中的杂交液向各个方向充分流动,芯片上各个样品点与反应液均匀接触,从而使反应的信号增强、结果的一致性提高。
同时,杂交仪还可以实现静止杂交功能。
该仪器稳定可靠,操作简便。
5.2 仪器工作原理其基本原理就是应用核酸分子的变性和复性的性质,使来源不同的DNA(或RNA)片段,按碱基互补关系形成杂交双链分子(heteroduplex)。
杂交双链可以在DNA与DNA链之间,也可在RNA与DNA链之间形成。
5.3 仪器的基本资料5.3.1 芯片数量:配合使用晶芯®基因芯片杂交盒可以一次进行6 张芯片的控温杂交或12 张芯片的控温清洗。
标配3个芯片杂交盒和2 个芯片清洗盒。
5.3.2 芯片尺寸使用BioMixer TMⅡ芯片杂交仪杂交与清洗的芯片应符合以下尺寸条件:厚度 1.00 ± 0.10 mm平整度20 m盖片宽度< 23 mm盖片厚度< 0.8 mm条码宽度< 23 mm5.3.3 配件:芯片杂交盒芯片清洗盒试管架5.3.4 仪器前面板:如下图所示包括杂交室门。
只有指定规格的芯片可放入芯片杂交仪;在芯片杂交仪工作时,不要打开杂交室门;芯片杂交时置于杂交盒内,清洗时置于清洗盒内。
5.3.5 LED (发光二极管) 指示灯:LED包括3个灯。
1号灯显示芯片杂交仪供电电源已打开,但还没有进行杂交或清洗工作; 2号灯显示芯片杂交仪正在工作中;3号灯显示芯片杂交仪已经关机。

Bio-Rad QX200微滴式数字PCR 实验操作指南地区:中国版本:1.0QX200实验流程简介样本核酸提取制备微滴PCR扩增检测微滴数据分析目录流程简介 .................................................................................................. 错误!未定义书签。
第一章实验耗材清单............................................................................... 错误!未定义书签。
第二章QX200实验流程.......................................................................... 错误!未定义书签。
第三章QX200软件操作. (10)第一章实验耗材信息1.耗材列表:a)DG8 Catridges(货号:1864008)b)DG8 Gaskets(货号:1863009)c)Droplet Generator oil for probes (货号:1863005)d)ddPCR Droplet Reader oil(货号:1863004)e)ddPCR Supermix for probes (货号:1863026)f)Pierceable Foil Heat Seal(货号:1814040)g)PCR Plate 96,semi-skirted(货号:12001925)h)8连管i) 1.5ml Tubesj)96-well plate holderk)Eppendorf tube rackl)Rainning multiple-channel Pipettes&tips (50ul,100ul)m)pipettor and tips - 10uln)pipettor and tips - 100ulo)pipettor and tips - 200ulp)Generator oil 储液槽q)无酶水2.设备列表:r)Droplet generator(货号:1864002)s)Dropler reader(货号:1864003)t)PX1 PCR plate sealer(货号:1814000)u)Thermal cycler(T100 货号:1861096 OR C1000 Touch 货号:1851196) v)小型离心机(1.5ml tube and 8连管适配器)w)漩涡震荡仪第二章QX200实验流程一、标本的提取1.组织样本DNA的提取组织样本DNA的提取使用QIAamp DNA FFPE Tissue Kit,详细方法参照试剂盒说明书2.血浆样本的ctDNA提取cfDNA的提取使用QIAamp DNA blood mini kit (Qiagen, Hilden, Germany),采用SpinProcedure方案。

Gentier 48E简易操作流程
1. 打开电脑与仪器并检查连接完好,拉起上盖,将配制好的试剂放入样品块上,闭合上盖。
2. 双击进入“MED-TL”软件,点击“新建文件”,按照小框提示进行编辑实验名称→点击完成。
3. 点击下面的虚框中的加号,根据说明书中的运行实验程序进行编辑→,选择“预变性”、“2步法扩增”或“3步法扩增”→按照说明书中的温度及时间进行更改→保存运行模版输入项目名称,以便下次实验直接调用运行模版。
4. 点击→选中样本放置位置→“样本名称”→“样本类型”→“选择荧光素及输入目标基因”→“输入标准品浓度”→“唯一性编码”→设置完成。
5. 点击→右下角运行界面→点击设备的IP 地址(选中状态变蓝色)→实验开始运行,在其他三个界面中可以实时观察“荧光曲线”、“热学循环”、“样本设置”等信息。
6.点击→对实验结果进行分析,看单个样本曲线需点击,使用其他分析方法→根据所需方法新建分析结果。
7.科研报告:“工具”→“科研报告”→根据需求选择设置→保存→打印。
8.其他报告:点击LIS →选择导出TL Lis(xlsx)→保存→使用天隆报告软件系统进行数据分析。
实时荧光定量Gentier 48E实验操作步骤
→
进行编辑“温度循环”
“样本设置”并保存
检查设备与电脑连接情况
并启动运行程序
查看结果分析并处理扩增产物
检查实时荧光仪器
设备及其连接
打开软件文件→“新建实验”
打开上盖放置配制好的样本。

矿产资源开发利用方案编写内容要求及审查大纲
矿产资源开发利用方案编写内容要求及《矿产资源开发利用方案》审查大纲一、概述
㈠矿区位置、隶属关系和企业性质。
如为改扩建矿山, 应说明矿山现状、
特点及存在的主要问题。
㈡编制依据
(1简述项目前期工作进展情况及与有关方面对项目的意向性协议情况。
(2 列出开发利用方案编制所依据的主要基础性资料的名称。
如经储量管理部门认定的矿区地质勘探报告、选矿试验报告、加工利用试验报告、工程地质初评资料、矿区水文资料和供水资料等。
对改、扩建矿山应有生产实际资料, 如矿山总平面现状图、矿床开拓系统图、采场现状图和主要采选设备清单等。
二、矿产品需求现状和预测
㈠该矿产在国内需求情况和市场供应情况
1、矿产品现状及加工利用趋向。
2、国内近、远期的需求量及主要销向预测。
㈡产品价格分析
1、国内矿产品价格现状。
2、矿产品价格稳定性及变化趋势。
三、矿产资源概况
㈠矿区总体概况
1、矿区总体规划情况。
2、矿区矿产资源概况。
3、该设计与矿区总体开发的关系。
㈡该设计项目的资源概况
1、矿床地质及构造特征。
2、矿床开采技术条件及水文地质条件。

UltraPlex ® 1-Step ToughMix ® ROX™ (4X)DescriptionUltraPlex 1-Step ToughMix ROX is a ready-to-use, 4X-concentrated master mix for reverse transcription quantitative PCR (RT-qPCR) of RNA templates using hybridization probe detection chemistries such as TaqMan ® 5’-hydrolysis probes on real-time PCR systems that utilize ROX passive reference with 490 nM excitation. First-strand cDNA synthesis and PCR amplification are carried out in the same tube without opening between procedures. It is ideal for highly sensitive quantification of RNA viruses or low abundance RNA targets in uni- or multiplexed RT-qPCR applications as well as high throughput gene-expression studies. The system has been optimized to deliver maximum RT-qPCR efficiency, sensitivity, and specificity in reduced reaction volumes and fast cycle times. UltraPlex 1-Step ToughMix ROX contains all required components for RT-qPCR except RNA template and probe. It is compatible with all dual-labeled probe chemistries.qScript ® XLT is an engineered M-MLV reverse transcriptase (RT) with reduced RNase H activity for improved activity and stability at higher temperatures. The use of higher temperatures (50 to 54°C) for the first-strand step of one-step RT-qPCR provides higher specificity for primer annealing and disruption of RNA secondary structure that can interfere with cDNA synthesis. These beneficial properties of qScript XLT RT are further enhanced by a hot-start mechanism for the reverse transcription step. Minimizing off-target extension by the RT during reaction assembly provides highly reproducible low copy quantification as well as extended room temperature stability of fully assembled reactions for high throughput operations.UltraPlex 1-Step ToughMix ROX is highly resistant to PCR inhibitors. A key component of the ToughMix is an ultra pure, highly processive thermostable DNA polymerase that is combined with high avidity monoclonal antibodies. This provides an extremely stringent automatic hot-start that minimizes the potential for primer-dimer and other non-specific PCR artifacts.Instrument CompatibilityDifferent real-time PCR systems employ different strategies for the normalization of fluorescent signals and correction of well-to-well optical variations. It is critical to match the appropriate qPCR reagent to your specific instrument. UltraPlex 1-Step ToughMix ROX provides seamless integration on the Applied Biosystems 7000, 7300, 7700, 7900HT StepOne™, or StepOnePlus™. Please visit our web site at to find an optimized kit for your instrument platform(s).ComponentsReagent DescriptionUltraPlex 1-Step ToughMix ROX (4X) 4X reaction buffer containing dATP, dCTP, dGTP, dTTP, magnesium, qScript XLT reversetranscriptase, RNase inhibitor protein, hot-start DNA polymerase , stabilizers, and ROX Reference DyeStorage and Stability Store components in a constant temperature freezer at -25°C to -15°C protected from light upon receipt. For lot specific expiry date, refer to package label, Certificate of Analysis or Product Specification Form.Guidelines for One-Step RT-qPCRThe design of highly specific primers and probes is a critical parameter for successful one-step RT-qPCR. The use of computer aided primerdesign programs is encouraged in order to minimize the potential for internal secondary structure and complementation at 3’-ends within each primer, the primer pair, and primer/probe combinations. Regions of strong RNA secondary structure should be avoided as this can interfere with primer hybridization and/or impede procession of the reverse transcriptase. For best results, amplicon size should be between 70 and 150 bp. Optimal results may require titration of primer concentration between 400 and 900 nM. A final concentration of 450 nM each primer and 100 to 150 nM probe is effective for most applications. The efficacy and efficiency of any primer/probe set should be validated under fast cycling and/or rapid ramp rate protocols before use in qPCR studies.Cat No. 95167-100 Size: 100 x 20 µL reactions (500 µL)Store at -25ºC to -15°C protected from lightGuidelines for One-Step RT-qPCR continued:▪If frozen, thaw UltraPlex 1-Step ToughMix ROX on ice. Thoroughly mix by vortexing, and then centrifuge to collect contents to the bottom of the tube. Retain on ice before use.▪First-strand synthesis can be carried out between 42°C and 60°C. Optimal results are generally obtained with a 10-minute incubation at 50°C.Longer incubation times for first-strand synthesis (up to 20 min) may be used. Incubation at temperatures over 54°C may result in delayed Cqs for assays that are optimal at 48 - 50°C.▪We recommend a minimum of 30s incubation at 95°C to inactivate the RT and activate the hot-start polymerase prior to PCR cycling. Longer activation times (2 – 10 minutes) will not adversely affect product performance and may reduce early cycle background noise experienced with some hydrolysis probe chemistries.▪The kit is compatible with both fast or standard qPCR cycling protocols. Annealing and or extension temperatures may need to be optimized for a given primer/probe design or fluorogenic probe chemistry. Use the suggested protocol as a starting point. Multiplexed RT-qPCR may benefit froma slightly longer extension time (60 to 90s). Use of a slower ramp rate (~2.5°C/s) between the denaturation step and annealing/extension step mayimprove performance for some assays.▪To maximize specificity, reactions should be assembled and retained on ice before transfer to the qPCR instrument.▪Preparation of a reaction cocktail is recommended to reduce pipetting errors and maximize assay precision. Assemble the reaction cocktail with all required components except RNA template and mix thoroughly by vortexing. Then, dispense equal aliquots into each reaction tube. Add RNA to each reaction as the final step. Addition of sample as 2 to 5-µL volumes will improve assay precision.▪Suggested input quantities of template are: 1 pg to 100 ng total RNA; 10 fg to 10 ng poly A(+) RNA; 10 to 1x108 copies viral RNA.▪After sealing each reaction, mix contents by vortexing, and then centrifuge briefly to collect components at the bottom of the reaction tube. Reaction AssemblyComponent Volume for 20-μL rxn. Final ConcentrationUltraPlex 1-Step ToughMix ROX (4X) 5 µL 1XForward primer(s) variable 300 – 900 nMReverse primer(s) variable 300 – 900 nMProbe(s) variable 50-200 nMNuclease-free water variableRNA template 2 to 10 µL variableFinal Volume (μL) 20 µLNote: For smaller, or larger, reaction volumes scale all components proportionally.RT-qPCR Cycling ProtocolIncubate complete reaction mix in a real-time PCR detection system as follows:cDNA Synthesis 50°C, 10 minInitial denaturation 95°C, 3 minPCR cycling (30 - 45 cycles) 95°C, 3s to 10s60°C, 30s to 90s (data collection step)‡‡Note: The use of longer extension times (90s at 60°C), or a 3-step cycling protocol with an extension step of 60s at 72°C can help mitigate suppression of a low copy amplicon when co-amplified with a high copy target sequence.Quality ControlKit components are free of contaminating DNase and RNase. UltraPlex 1-Step ToughMix ROX is functionally tested in duplexed RT-qPCR. Kinetic analysis must demonstrate linear resolution over six orders of dynamic range (r2≥ 0.990) and a PCR efficiency ≥ 90% for the primary GOI with constant detection for the limiting exogenous positive control assay.Limited Label LicensesUse of this product signifies the agreement of any purchaser or user of the product to the following terms:1.The product may be used solely in accordance with the protocols provided with the product and this manual and for use with components contained in the kitonly. Quantabio, LLC. grants no license under any of its intellectual property to use or incorporate the enclosed components of this kit with any components not included within this kit except as described in the protocols provided with the product, this manual, and additional protocols available at . Some of these additional protocols have been provided by Quantabio product users. These protocols have not been thoroughly tested or optimized by Quantabio, LLC. Quantabio, LLC. neither guarantees them nor warrants that they do not infringe the rights of third-parties.2.Other than expressly stated licenses, Quantabio, LLC. makes no warranty that this kit and/or its use(s) do not infringe the rights of third-parties.3.This kit and its components are licensed for one-time use and may not be reused, refurbished, or resold.4.Quantabio, LLC. specifically disclaims any other licenses, expressed or implied other than those expressly stated.5.The purchaser and user of the kit agree not to take or permit anyone else to take any steps that could lead to or facilitate any acts prohibited above. Quantabio, LLC.may enforce the prohibitions of this Limited License Agreement in any Court, and shall recover all its investigative and Court costs, including attorney fees, in any action to enforce this Limited License Agreement or any of its intellectual property rights relating to the kit and/or its components.©2021 Quantabio, LLC. 100 Cummings Center Suite 407J Beverly, MA 01915; Telephone number: 1-888-959-5165.Quantabio products are manufactured in Beverly, Massachusetts, Frederick, Maryland and Hilden, Germany.Intended for molecular biology applications. This product is not intended for the diagnosis, prevention or treatment of a disease.UltraPlex, ToughMix and qScript are registered trademarks of Quantabio, LLC. TaqMan is a registered trademark of Roche Molecular Systems, Inc.. ROX is a trademark of Life Technologies Corporation.。

c16全自动核酸提取仪操作说明仪器构造简介:1 触控屏HID-Pro 3202 仪器前门3 样品盘适配器4 LED指示灯5 触摸感应器1 带滤芯的枪头2 收集管3 枪头、洗脱管放置架4 压板5 样品槽(可直接放置试剂板或加装适配器后放置试剂条)6 传送轨道7 试剂条适配器样品架穿孔工具1 InnuPure接口2 网络接口3 USB接口简易操作说明:1.插上仪器电源线,打开仪器背部的电源开关,仪器前面LED指示灯显示红色,此时仪器为待机状态。
用手指轻触触摸感应器圆形键一秒钟(注意,不要使劲按),手指拿开后LED指示灯由红色变为绿色,此时仪器为工作状态。
开机后界面如下图所示:Menu:菜单Select protocol:选择程序Tools:工具Settings:一般设置2.样品准备和纯化1)样品盘放入样品架,打开夹板。
2)将试剂板放入样品槽。
试剂板上的红线要与样品槽上的红点对齐。
3)将条管适配器放入样品槽。
同样适配器上的红点要与样品槽上的红点对齐。
4)将试剂条放入样品槽。
注意:有“AJ”字样的一头朝向样品盘上刻字的一边。
5)试剂条管放完后,放下压板。
6)放入枪头和收集管。
注意:没放试剂条的位置不要放。
7)将试剂条管第一和第三孔刺穿,可以使用穿孔工具也可以使用干净的枪头。
(“AJ”字样边为第一孔)8)将裂解后的样品加入第一孔。
(具体操作详见试剂盒说明)9)准备好的样品盘放入仪器内,轻推,样品盘会自动进入。
10)根据起始样本选择合适的程序,点击开始即可。
11)纯化完成后样品盘会自动退出,盖子盖好纯化产物放冰箱保存待用。
3.软件功能介绍①菜单:设置新用户、设置开机密码等②日期时间:设置日期时间③选择程序:开始、导入、删除程序④工具:自动校正、仪器初始化、磁力测试等仪器自检功能⑤一般设置:用户管理、语言、更新等⑥版本信息。

实时荧光定量PCR仪器操作规程
1.打开电脑,打开Bio-Rad iQ5电源和光路电源;
2.点击Bio-Rad iQ5图标,启动软件;
3.预热30分钟;
4.点击PROTOCAL键,点edit编辑,保存;
5.点击plate键,点edit编辑,保存;
6.根据所编辑的plate,将待扩增物放入样品槽中;
7.点击run set ,在界面右上角点击run;
8.点击begin run,运行;
9.待扩增完成后,点击result,分析数据;
10.关闭Bio-Rad iQ5软件;
11.关闭电脑及Bio-Rad iQ5电源;
注意事项
1.关闭仪器前应先关闭iQ5软禁啊,否则仪器会认为是因为故障停电而导致仪器
关闭,下次开机时,仪器会有自我保护功能;
2.实验前后都要填写仪器使用登记表,实验完成后填写仪器使用记录;
3.正确操作仪器,使用过程中如发现仪器出现异常情况,应及时与实验室管理人
员联系;
4.要求用托盘盛放药品,避免药品直接放在实验台上,实验结束及时清洁实验台,
带走所有实验试剂及样品;
5.仪器使用人员有义务打扫实验室卫生;
6.仪器使用人员有义务和责任负责实验室内实验仪器的安全;
7.仪器使用人员有义务和责任负责实验室内水、电安全;。
核酸预制胶说明书保存:4℃,2个月。
操作步骤:非变性预制胶:1.准备样品:将样品和loading buffer(5×)按照4:1混合均匀。
2.准备1×running buffer:取200ml的5×TBE buffer,加入去离子水至1L,制备成1×TBE buffer。
3.将预制胶装入兼容的电泳槽中,加入电泳缓冲液,再缓慢地将梳子拔出。
4.上样:在梳孔内加入适当浓度和体积的DNA样品。
5.电泳条件:150V,50~75min(电泳时间取决于凝胶浓度)6.电泳结束,取出凝胶。
尿素变性预制胶:1.准备样品:将样品和UREA-TBE loading buffer(5×)按照4:1混合均匀,100℃下加热3-5min。
2.准备1×running buffer:取200ml的5×TBE buffer,加入去离子水至1L,制备成1×TBE buffer。
3.将预制胶装入兼容的电泳槽中,加入电泳缓冲液,再缓慢地将梳子拔出。
4.上样:用1×TBE buffer反复冲洗梳孔以清除梳孔内的尿素,在梳孔内加入适当浓度和体积的DNA样品。
5.电泳条件:150V,50~75min(电泳时间取决于凝胶浓度)6.电泳结束,取出凝胶。
预制胶兼容的电泳槽:Precast-EZgel可以兼容大部分的mini SDS-PAGE电泳槽,包括∙Bio-Rad Mini-PROTEAN(II/3/Tetra System)∙Hoefer Mighty Small(SE250/SE260/SE280)∙Life Technology Novex Mini-Cell(请与特制挡板配合使用)预制胶在Bio-Rad电泳槽的应用a.将Bio-Rad电泳槽中的U型密封条(如图绿色部分)拉出,注意这时的密封条两端是有突起的,突起的这面为正面,无突起的为反面。
b.将密封条旋转180度(正面朝里,反面朝外),重新装回电泳槽中,注意把密封圈周边压实,防止发生漏液。
一系统基本操作在本练习中,我们将学习如何开启设备、正确放置实验所需的试剂和缓冲液、放取芯片和试管架、冲洗流路系统以及响应信号归一化处理。
当系统更换缓冲液或者芯片后,必须运行Prime程序。
Prime时缓冲液会冲洗整个流路系统,为下一步的实验做好准备。
1.1开机操作1) 打开Biacore T200系统和电脑的电源开关。
Biacore T200的电源开关位于系统背面的左下角。
开机后,需等待检测单元的温度达到预设温度(用户自设,通常为25℃)。
待温度稳定后,面板上的温度指示灯(黄色)会停止闪烁,该过程可能需要30-60分钟。
2) 打开Biacore T200 控制软件(Strart→Program→Biacore→Biacore T200 Control Software),运行后软件程序会自动和主机系统建立连接。
3) 准备运行缓冲液。
本练习采用10X HBE-EP+缓冲液(已经0.22μm膜过滤):量取50ml 10XHBS-EP+缓冲液、450ml去离子水,混匀后放入500ml缓冲液瓶。
1.2缓冲液的放置1) 将已经配制好的缓冲液放在T200系统左侧的缓冲液托架上,换上黑色的单孔盖。
2) 将缓冲液进液管A(注意软管上的蓝色标签)插入至缓冲液瓶底部。
其余三根进液管(B、C和D)放在左侧的舱门后。
在实验中,Biacore T200最多支持4种缓冲液;只使用一种缓冲液时,进液管默认为A管。
3) 将2L的废液瓶放置在T200系统右侧的缓冲液托架上,连接上专用的盖子。
4) 取500ml去离子水装入500ml缓冲液瓶,放置在右侧缓冲液托架上,并将进液管A插至瓶底。
瓶中的水将用于清洗进样针。
1.3 芯片的放置1) 点击工具条中的按钮或选择Tools菜单中的Insert Chip选项,打开芯片舱门。
2) 如果已经有芯片在芯片舱内,点击工具条中的按钮或选择Tools菜单中的Eject Chip选项。
点击Eject Chip,流动池中的液体将被排空,随后芯片舱门将自动打开。
Bio-Plex核酸偶联操作手册1,原理介绍Bio-plex液相悬浮芯片系统提供了多达100种的羧基微球,用户可以自己偶联探针或蛋白,用于检测目标核酸或蛋白。
本手册介绍的是核酸偶联的操作步骤。
核酸的偶联需要首先合成2种寡聚核苷酸片断,分别是1,捕获探针:一段带有5’氨基修饰的C6或C12手臂的寡核苷酸。
这段序列需要与待检测的序列反向互补。
Note:关于究竟用C12 还是用C6作为手臂,有人认为C12 会提供更好的结果,但是也有人认为两者之间并没有什么差异;但是没有人认为C12 会得到不好的结果,所以用C12做手臂是一种比较安全的选择。
2,验证探针:一段带有5’生物素标记的片断,序列与捕获探针反向互补。
这段序列用于验证偶联的效率。
如下图所示,列出了捕获探针和验证片断的例子:表1,捕获探针和验证探针示意(序列按照5’-3’的方向)下图是整个偶联过程的示意图:2.0材料2.1寡核苷酸,如Biosearch Technologies或者Integrated DNA Technologies;2.2 微球偶联所需试剂√试剂供应商/货号数量1.5ml离心管USA Scientific 1415-2500 多个EDC(1-乙基-(3-二甲基氨基丙基)碳化二亚胺盐酸盐)Pierce 77149 2X10mg (一次偶联所需)血球计数器VWR 15170-172 1MES (2-吗啉乙磺酸)Sigma M2933 25g5N NaOH Fischer SS256-500 500mlTween 20 Sigma P9416 50mlSDS,10% 溶液Sigma L4522 100mlTE buffer,pH8.0,100X Sigma T9285 100ml2.3 杂交检测所需试剂√试剂供应商/货号数量Sarkosyl(N-月桂酰肌氨酸)Sigma L9150 50g5M TMAC(四甲基氯化铵)Sigma T3411 500ml链霉亲和素-藻红素(SA-PE),1mg/ml Prozyme PJ31S 1mg3.0 微球偶联以下是微球偶联过程中所需的试剂,按照下表列出的数量,然后加分子生物学级的水至250ml的体积。
0.1M MES pH4.5 (偶联缓冲液),250ml试剂目录号终浓度每250ml所需量MES Sigma M2933 0.1 M 4.88g 调整pH至4.5,0.22um的膜过滤除菌,4度保存。
0.02% Tween 20 (清洗缓冲液I),250ml试剂目录号终浓度每250ml所需量Tween 20 Sigma P9416 0.02% 50ul0.22um的膜过滤除菌,室温保存0.1% SDS (清洗缓冲液II),250ml试剂目录号终浓度每250ml所需量SDS,10% Sigma L4522 0.1% 2.5ml0.22um的膜过滤除菌,室温保存TE,pH8.0 (样品稀释液),250ml试剂目录号终浓度每250ml所需量TE buffer,pH8.0,100X Sigma T9285 1X 2.5ml0.22um的膜过滤除菌,室温保存4.0 杂交验证以下试剂用于验证杂交效率。
按照所列出的体积混合所需试剂,在需要水的地方,请使用分子生物学级的水。
20% Sarkosyl,250ml试剂目录号终浓度每250ml所需量Sarkosyl Sigma L9150 20% 50gH2O 250ml终体积0.22um的膜过滤除菌,室温保存1.5XTMAC杂交缓冲液(稀释微球),500ml试剂目录号终浓度每500ml所需量5M TMAC Sigma T3411 4.5 M 450ml20% Sarkosyl Sigma L9150 0.15% 3.76ml1M Tris-HCl,pH8.0 Sigma T3038 75mM 37.5ml0.5M EDTA,pH8.0 Gibco 15575-038 6mM 6mlH2O 2.74ml室温保存1XTMAC杂交缓冲液(杂交液),250ml用83ml水稀释167 ml 1.5X TMAC试剂来源每500ml所需量1.5XTMAC 见上167mlH2O 83ml室温保存5.0 微球偶联步骤5.1 重要提示1,不要用TE悬浮氨基偶联的微球。
2,EDC非常易于吸潮,而且一旦与水接触后很快失去活性。
所以一定要防止EDC变潮。
瓶子刚从冰箱拿出来,没有恢复到室温的时候不要打开。
3,偶联的过程反应迅速,所以每一管加了EDC之后要立刻混匀。
4,微球应该尽可能得避光。
在孵育或者每个步骤之间放置的过程中,尽量避光以防止荧光淬灭。
可以用锡箔纸。
5,为了防止破坏沉淀,尽量不要用顶端太锐利的枪头,或者用凝胶点样专用枪头。
6,5.2 准备1,按照0.1nmol/l的浓度用水稀释探针。
2,确保已经准备好了第二页所列出来的全部材料;3,确保已经准备好了第三页中所列出的全部试剂;4,按照下表确定反应体积。
表2,反应体积微球体积样品数量100ul ~200200ul ~400400ul ~8001ml ~2,0005ml ~10,00010ml ~20,0002,如果使用的体积不是表中标出的部分,请根据表格中的数据调整体积。
3,在实验开始前,取2份新鲜的EDC粉末,使之恢复到室温。
(大概需要30-60分钟)5.3 微球偶联步骤1,蜗旋仪上以最高转速悬浮微球,然后在超声清洗仪上超声微球30s;2,取400ul微球到USA scientific离心管中。
3,1-2分钟最高转速离心;4,弃上清,用45ul 0.1M,pH4.5 的MES重悬微球,彻底蜗旋,使微球分散。
5,加入2ul探针。
6,制备新鲜的EDC溶液(10mg/ml),加入2.5ul EDC至微球中,彻底混匀。
7,室温黑暗推荐下孵育30min。
8,制备新鲜的EDC溶液(10mg/ml),加入2.5ulEDC至微球中,彻底混匀。
9,室温黑暗条件下孵育30min。
10,加入1ml 0.02% Tween 20,稍微蜗旋混匀。
11,最高转速离心1-2min。
12,弃上清,用0.1%SDS重悬沉淀40s。
13,最高转速离心1-2min。
14,用80ul pH8.0 TE buffer 重悬沉淀。
15,蜗旋仪上最高转速蜗旋30s。
16,4℃ 黑暗条件下储存。
表3,以上步骤所需的体积。
根据微球的体积确定每部所需的体积微球体积pH4.5 0.1M MES 探针(0.1nmol/ul)EDC(10mg/ml)TE100ul 8.5ul 1ul 0.5ul 20ul200ul 22ul 2ul 1.25ul 40ul400ul 45ul 2ul 2.5ul 80ul1,000ul 37ul 10 ul 2.5ul 200ul5ml 22ul 25ul 2.5ul 1,000ul10ml 44ul 40ul 5ul 2,000ul5.4 微球计数通过计算微球的数量来估算微球的回收率。
每孔至少要有40,000个微球。
通过血球计数器来计算偶联微球的数量。
1,在蜗旋仪上重悬微球,40s。
2,用双蒸水按照1:100的比例稀释微球。
3,彻底混匀。
4,取10ul转移到血球计数器中(图2)。
5,数位于血球计数器4个角落里的微球的数量。
6,微球的浓度(微球/ul)=(4个角落里微球的数量之和)X2.5X100。
图1,血球计数器的示意图图2,加样到细胞计数器中。
图3,细胞计数器的4个角落示意图。
6.0 检测杂交效率偶联好了之后就需要检测偶联的效率。
如果偶联反应做的比较好,加入验证探针与否会对MFI值带来很大的变化。
6.1 准备工作微球混合物A.准备第5页中的缓冲液。
B.按照下面的描述准备工作微球混合物(够30个孔所需)。
选择合适的寡核苷酸偶联的微球。
在蜗旋仪上混匀每种微球(40s,6-7级的转速),然后制备工作微球混合物:1.加入660ul 1.5XTMAC杂交缓冲液(稀释微球);2.加入约150,000个偶联好了的微球到混合物中。
按照以下公式进行:B=微球的浓度;V=每种微球混合物所需的体积150,000/B=V3.加入双蒸水至终体积为990ul。
4. 冰上放置。
(不要使之结冰)。
C. 制备验证探针在实验当天准备新鲜稀释的验证探针。
最后每个寡核苷酸片断的浓度应该为10fmol/ul。
1,起始液的浓度为1nmol/ul (1mM)。
2,按照表4所列,取4ul寡核苷酸片断加入到pH 8.0 的TE中(即按照1:250稀释)。
这时得到的浓度为4pmol/ul。
3,充分混匀。
4,取2.5ul该溶液到997ul pH8.0 的TE中(1:400稀释)。
这时的浓度为10fmol/ul。
5,彻底蜗旋混匀。
6,放置在冰上。
表4 :所需TE的体积6.2 检测杂交效率1. 在蜗旋仪上混合工作微球30s。
2. 在标准PCR管中,加入33ul微球的混合物。
3. 在每个管中分别加入验证探针和TE到每一行的前3个孔中(按照12页所示的方式做板)。
4.反复吸打每个管,充分混匀。
5.盖上板子,以防止蒸发,95-100度加热5分钟。
用类似于Microseal B粘贴膜。
6.在脱色摇床上孵育杂交液15分钟(第一次用比所计算的退伙温度低5-10度)。
7.准备新鲜制备的用1XTMAC溶解的3ug/ml的SA-PE。
a.3ml 1XTMACb.9ul 1mg/ml 的Strep-R-PENote:需要事先把滤板预热到退火温度。
8.把样品加入预热了的滤板中。
用真空抽滤装置快速得把杂交缓冲液抽走。
(25-50 mm Hg)9.加入100ul报告激光混合物到每个孔中。
反复吸打混匀。
10.室温孵育5min。
11.根据Bioplex使用说明书分析荧光值。
(做板以及设定标准品)做板以及设定标准品)主要的软件分析步骤(7.主要的软件分析步骤在运行之前按照下列描述做板。
7.1 做板7.2 设置标准品按照下图设置标准品的信息7.3 运行后修改板子的模式和标准品的信息如果在设置的时候忘记了设定标准品的信息或者板子的设定出现了错误可以在运行之后再做修改。
运行之后修改板子的模式:运行之后修改标准品的信息7.4 显示结果设定标准曲线的目的不是为了产生一个标曲,而是为了分析饱和浓度,评价背景,以及分析样品的浓度。
按照以下描述进行设定A.回归方式:Point to point (Semi-log)B.X Y轴的转换:线对线8. 对结果的解释仔细分析结果以去认以下几点:1,空白中的信号要相对很低。
2,在空白对照和含有探针的孔之间的信号区别要非常大。
3,在较低的不同稀释浓度下的信号要有较好的线性。
4,在较高的浓度时会看到饱和。
5,记录下较低标准的浓度,作为以后偶联的基准。
表5 列出了标准杂交反映的试验结果。