细菌基因组测序
- 格式:pdf
- 大小:3.59 MB
- 文档页数:6

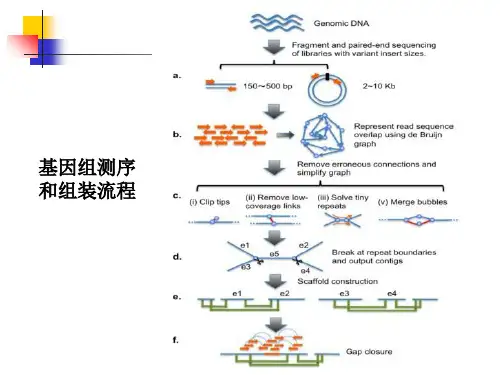

细菌基因组测序方法
细菌基因组测序方法主要有以下几种:
1. 整体基因组测序(Whole Genome Sequencing,WGS):将
细菌的整个基因组进行测序,并通过比对参考基因组或组装建立一个完整的基因组序列。
2. 目标区域测序(Targeted Sequencing,TS):针对特定基因、基因组区域或有意义的变异位点进行测序。
3. 细胞单体测序(Single-cell Sequencing,SCS):将细胞单
体进行测序,通过技术手段将单个细胞的DNA扩增到足够浓
度后进行测序。
4. 转录组测序(RNA Sequencing,RNA-Seq):将细菌转录产物测序,包括mRNA、ncRNA等,可以了解细菌的转录水平
和转录后调控。
5. 甲基化测序(Methylation Sequencing,Methyl-Seq):对细
菌基因组进行甲基化修饰位点的检测以获得表观遗传信息。
6. 大肠杆菌的测序方法还包括平滑野百合花青素基因组测序(PacBio)和人工合成DNA 和4D核磁共振测序。

细菌鉴定测序-概述说明以及解释1.引言1.1 概述细菌鉴定是一项关键的实验室技术,它能够帮助科学家们确定细菌的物种、种群和亚种信息。
通过细菌鉴定,我们可以了解到细菌的特性、生活习性以及与人类健康和环境的关系。
在过去的几十年中,细菌鉴定技术得到了快速的发展和进步,其中细菌鉴定测序技术的突破尤为显著。
细菌鉴定测序是利用DNA测序技术对细菌的基因组进行分析和比对,以确定细菌的物种和相关信息。
相比传统的细菌鉴定方法,如形态学观察或生化检测,测序技术的优势在于其高度准确性、高通量和高效率。
它能够迅速鉴定大量的细菌样本,并且对不同物种之间的差异性进行全面的分析。
目前,常用的细菌鉴定测序方法主要包括16S rRNA测序和全基因组测序两种。
16S rRNA测序是一种常用的DNA测序技术,通过分析细菌的16S rRNA基因序列来确定其物种和亲缘关系。
全基因组测序则是对细菌的整个基因组进行测序和分析,从而获得更加全面和详细的细菌信息。
细菌鉴定测序技术在许多领域都得到了广泛的应用,包括医学、农业、环境科学等。
在医学领域,细菌鉴定测序可以帮助医生准确诊断细菌感染病例,指导治疗和药物选择。
在农业领域,细菌鉴定测序可以帮助农民和科研人员了解农作物病害的病原菌,从而采取相应的防治措施。
在环境科学领域,细菌鉴定测序可以帮助我们了解细菌在自然环境中的分布和功能,从而指导环境保护和污染治理工作。
随着测序技术的不断发展和突破,细菌鉴定测序技术也将呈现出更加广阔的应用前景。
未来我们可以预见,细菌鉴定测序技术将在医学诊断、食品安全、环境监测等领域发挥更大的作用。
同时,随着测序技术的成本不断降低和分析方法的不断改进,细菌鉴定测序技术将变得更加便捷、高效和经济。
综上所述,细菌鉴定测序技术在现代生物学和医学领域具有重要的意义和应用价值。
随着技术的不断进步,我们相信细菌鉴定测序技术将进一步推动细菌学研究的发展,并为人类的健康和环境保护作出更大的贡献。


细菌基因序列研究报告细菌基因序列研究报告细菌基因序列研究报告是利用高通量测序技术对细菌基因组进行测序和分析的报告。
本报告以大肠杆菌(Escherichia coli)为研究对象,通过对其基因组序列的测定和分析,揭示了细菌基因组的结构、功能和演化等方面的信息。
一、研究目的本研究的目的是通过对大肠杆菌基因组的测序和分析,探索细菌基因组的特征和功能,为进一步的细菌基因研究提供参考和依据。
二、实验方法1. 样本处理:从培养的大肠杆菌中提取基因组DNA,并通过PCR扩增得到足够数量的DNA样本。
2. 基因组测序:采用Illumina高通量测序平台对样本进行测序,得到海量的短读序列。
3. 数据处理和拼接:使用适当的软件对测序数据进行预处理、质控和拼接,得到完整的基因组序列。
4. 基因组注释和分析:将得到的基因组序列与已知数据库进行比对和注释,如基因预测、基因功能注释、基因家族分类等。
5. 演化分析:通过多序列比对和系统发育树构建等方法,分析大肠杆菌与其他相关物种之间的演化关系。
三、结果与讨论1. 基因组结构:通过测序和拼接,我们得到了大肠杆菌的完整基因组序列,并发现其具有单个的圆形染色体。
基因组大小为4.6兆碱基对,含有大约4000个基因。
2. 基因功能和注释:通过对基因组进行注释和功能预测,我们发现其中包含多个致病性因子和抗生素抗性基因。
此外,还发现了许多调控基因和代谢酶基因,这些基因对大肠杆菌的生长和适应环境起着重要作用。
3. 演化关系:通过与其他相关物种进行比较和分析,我们发现大肠杆菌与其他肠道细菌存在较高的相似性,这可能说明它们具有共同的起源和进化历史。
综上所述,本研究通过细菌基因序列的测定和分析,揭示了大肠杆菌基因组的结构、功能和演化等方面的信息。
这对于进一步理解细菌基因组的特征和功能具有重要的意义,也为生物医学研究和药物开发提供了新的线索与依据。

“Iontorrent微生物(细菌)全基因组重测序文库构建实验方案”清晨的阳光透过实验室的窗户,洒在略显拥挤的操作台上,我的思绪开始像电流一样流转,10年的方案写作经验让我对每一个细节都了如指掌。
就让我们一起探索这个关于Iontorrent微生物(细菌)全基因组重测序文库构建的实验方案吧。
我们要明确实验的目的:通过Iontorrent平台,对微生物(细菌)进行全基因组重测序,以获取更准确、更全面的基因组信息。
那么,就是具体的步骤了。
一、实验材料与仪器1.材料:微生物(细菌)样本、DNA提取试剂盒、磁珠、PCR试剂、测序试剂盒等。
2.仪器:Iontorrent测序仪、离心机、PCR仪、磁珠分离器、凝胶成像仪等。
二、实验步骤1.样本处理:将微生物(细菌)样本进行适当的预处理,如离心、洗涤等,确保样品的纯净度。
2.DNA提取:使用DNA提取试剂盒,按照说明书操作,提取微生物(细菌)的基因组DNA。
3.DNA定量:利用纳米滴技术或紫外可见分光光度计,对提取的DNA进行定量,以确保DNA的浓度和纯度。
4.DNA片段化:将提取的DNA进行片段化处理,使其成为适合测序的片段大小。
5.文库构建:将片段化的DNA与测序适配器连接,通过PCR扩增,构建测序文库。
6.文库质检:利用凝胶成像仪,对构建的文库进行质检,确保文库的质量。
7.测序:将质检合格的文库上机测序,使用Iontorrent测序仪进行测序。
8.数据分析:将测序得到的原始数据进行质控、比对、组装等分析,得到微生物(细菌)的全基因组序列。
9.结果验证:对测序结果进行验证,如通过PCR、一代测序等方法,确保测序结果的准确性。
三、注意事项1.实验过程中,要注意无菌操作,避免样本污染。
2.操作过程中,要严格按照说明书进行,确保实验的准确性。
3.测序数据的质量控制和分析是关键步骤,要密切关注每个环节,确保结果的可靠性。
4.实验过程中,要随时记录实验数据和操作过程,以便后续的数据整理和分析。

微生物基因组测序技术及其应用随着科技进步,微生物基因组测序技术在医学、环境、农业等领域受到广泛关注和应用。
本文将简要介绍微生物基因组测序技术的基本原理和应用场景,以及未来的发展方向。
一、微生物基因组测序技术的基本原理微生物基因组测序技术是指将微生物DNA分子逐一排列,从而得到一条由ATCG四种碱基构成的“基因序列”。
这种技术的基本原理是将DNA从细胞中分离出来,通过PCR扩增等方法得到大量的DNA片段,然后用高通量测序仪对这些DNA片段进行测序,最后将这些片段拼接得到完整的基因组序列。
目前,微生物基因组测序技术主要分为三种方法:Sanger测序技术、454逆转录聚合酶链反应测序技术和Illumina测序技术。
其中,Illumina测序技术是目前最常用的基因组测序方法之一。
二、微生物基因组测序技术的应用场景1.医学应用微生物基因组测序技术被广泛应用于临床诊断中。
如何对感染病原体进行准确快速的鉴定,是临床医生面临的一项困难。
传统的菌落培养法不仅时间长,而且不能鉴定细菌的种系,因此不能满足对临床诊断的要求。
微生物基因组测序技术可以直接从感染部位分离出细菌DNA,进行基因组测序后,通过对基因组序列的比对,快速高效地鉴定病原菌种类以及其耐药性。
同时,该技术还被应用于研究小肠细菌群的变化,对于小肠细菌感染和肠道菌群失调的诱因和机制的研究有着重要的作用。
而在抗生素的研究和开发中,微生物基因组测序技术也发挥着越来越重要的作用。
2.环境应用微生物基因组测序技术的应用不仅局限于医疗领域,也被广泛应用于环境监测领域。
通过微生物基因组测序技术,可以对环境中微生物丰度、多样性和功能进行高通量测定,揭示微生物群落结构和功能特征。
例如,饮用水中的微生物群落结构和数量分布对水质安全和人体健康有着至关重要的作用。
通过微生物基因组测序技术,可以对水中的细菌、病毒和病原真菌等微生物进行定量和定性分析,为水质监测提供有效的手段。
3.农业应用微生物基因组测序技术在农业领域的应用也越来越广泛。

Ion torrent微生物(细菌)全基因组重测序文库构建实验方案微生物(细菌)全基因组重测序文库构建实验方案一、重测序原理全基因组重测序是对已知基因组序列的物种进行不同个体的基因组测序,并在此基础上对个体或群体进行差异性分析。
二、技术路线培养至对数期的单一菌落↓基因组DNA提取细菌DNA(纯化)↓超声波打断 DNA片段化↓ 文库构建↓Ion OneTouch 乳液PCR、ES↓Ion PGM、Ion Proton 上机测序↓生物信息学分析DNA片段化↓ 末端修复↓纯化接头连接、缺口修复↓纯化文库片段筛选(E-Gel 法)↓ 文库片段扩增↓纯化 Agilent Test、Qubit定量↓ Ion OneTouch System 重测序三、实验方案 1.细菌总DNA的提取液氮速冻、干冰保存的细菌菌液:若本实验室可以提供该细菌生长的条件,则对菌液进行活化,培养至对数期时,对该细菌进行DNA提取;若本实验室不能提供该细菌的生长条件,则应要求客户提供尽可能多的样本,以保证需要的DNA量。
细菌DNA采用试剂盒提取法(如TianGen细菌基因组提取试剂盒)。
取对数生长期的菌液,按照细菌DNA提取试剂盒操作步骤进行操作。
提取完成后,对基因组DNA进行纯度和浓度的检测。
通过测定OD260/280,范围在1.8-2.0之间则DNA较纯,使用Qubit对提取的DNA进行定量,确定提取的DNA浓度达到文库构建的量。
2.DNA片段化采用Covaris System超声波打断仪(Covaris M220),将待测DNA打断步骤:1)对待打断的DNA进行定量,将含量控制在100ng或者1μg2)打开Covaris M220安全盖,将Covaris AFA-grade Water充入水浴容器内,至液面到最高刻度线(约15mL),软件界面显示为绿色3)将待打断DNA装入Ep LoBind管中,其中DNA为100ng或1μg,加入Low TE至总体积为50mL4)将稀释的DNA转移至旋钮盖的Covaris管中(200bp规格),转移过程中不能将气泡带入,完成后旋紧盖子5)选择Ion_Torrent_200bp_50μL_ScrewCap_microTube,将对应的小管放入卡口,关上安全盖,点击软件界面“RUN”6)打断结束后,将混合液转移至一支新的1.5mL离心管中3.末端修复及接头连接 3.1 末端修复使用Ion Plus Fragment Kit进行,以100ng DNA量为例,各组分使用前瞬时离心2s 步骤:1)加入核酸酶free水至装有DNA片段的1.5mL离心管中,至总体积为79μL 2)向体系中加入20μL 5×末端修复buffer,1μL末端修复酶,总体积为100μL 3)室温放置20min3.2 片段纯化片段纯化使用Agencourt AMpure XP Kit进行步骤:1)加入180μL Agencourt AMpure XP Reagent beads于经过末端修复的1.5mL离心管中,充分混匀,室温放置5min2)瞬时离心后,将离心管放置于磁力架(如DynaMag-2磁力架)3min,至溶液澄清,小心去除上清,离心管保持放置在磁力架上3)离心管放置于磁力架上不移动,配置新的500μL 70%乙醇,加入,30s后,盖上盖子颠倒混匀2次,使磁珠悬浮,放回磁力架至溶液澄清后,小心除去上清 4)重复第三步5)用20μL墙头小心吸去多余的液体6)将离心管留在磁力架上,室温风干不超过5min7)取下离心管,加入25μL Low TE缓冲液,盖上盖子,上下颠倒混匀5次,旋窝震荡10s,充分混合8)瞬时离心后,将离心管放于磁力架上至少1min,溶液澄清后,将上清移入一支新的1.5mL EP管中(不可吸到磁珠) 9)纯化的DNA片段用于下一步的接头连接4.接头连接、缺口修复和纯化片段两端的接头分别为barcode接头和P1接头 4.1 接头连接在0.2mL体系中加入(以100ngDNA为例)DNA 25μL10×Ligase Buffer 10μL Ion P1 Adaptor 2μL Ion XpressBarc ode X+ 2μL dNTP Mix 2μL Nuclease-free water49μL DNA Ligase 2μL Nick pair polymerase 8μL Total 100μL将体系配置好后,放入PCR仪按照下面的温度进行1个循环扩增25℃ 15min72℃ 5min 4℃ hold 循环×1立即转移至一个新的1.5mL离心管中 4.2 纯化片段纯化使用Agencourt AMpure XP Kit进行步骤:1)加入140μL Agencourt AMpure XP Reagent beads于经过末端修复的1.5mL离心管中,充分混匀,室温放置5min2)瞬时离心后,将离心管放置于磁力架(如DynaMag-2磁力架)3min,至溶液澄清,小心去除上清,离心管保持放置在磁力架上3)离心管放置于磁力架上不移动,配置新的500μL 70%乙醇,加入,30s后,盖上盖子颠倒混匀2次,使磁珠悬浮,放回磁力架至溶液澄清后,小心除去上清 4)重复第三步5)瞬时离心,用20μL墙头小心吸去多余的液体 6)将离心管留在磁力架上,室温风干不超过5min7)取下离心管,加入20μL Low TE缓冲液,盖上盖子,上下颠倒混匀5次,旋窝震荡10s,充分混合8)瞬时离心后,将离心管放于磁力架上至少1min,溶液澄清后,将上清移入一支新的1.5mL EP管中(不可吸到磁珠)5.文库片段筛选方案1E-Gel Agarose System采用E-Gel SizeSelect 2% Agarose Gel,在E-Gel电泳、成像系统中按照操作说明进行。

细菌传代测序
细菌传代测序是指对细菌进行多代传代培养,并在每代传代后对其进行基因组测序的过程。
这个过程通常用于研究细菌在不同生长条件下的遗传变化和进化过程,以及分析其遗传适应性和表型变化。
在细菌传代测序实验中,一般包括以下步骤:
1.细菌培养和传代:选择目标细菌菌株,进行预培养,然后进行第一代传代培养。
在每一代培养结束后,取部分细菌进行下一代的培养,以此类推,直至达到所需的传代次数。
2.DNA提取:在每代传代结束后,收集细菌样品,提取其总DNA。
DNA提取可以采用各种商业化的DNA提取试剂盒或自制提取方法。
3.建库和测序:对提取得到的DNA样品进行文库构建,一般采用Illumina、PacBio等常见的测序平台。
根据实验目的,可以选择全基因组测序、16S rRNA基因测序等不同的测序方法。
4.数据分析:对测序得到的数据进行生物信息学分析,包括序列质控、序列比对、变异检测、基因组组装等步骤。
通过比较不同代次的细菌测序数据,可以分析其在遗传水平上的变化。
5.功能分析:进一步对不同代次的细菌测序数据进行功能分析,包括基因功能注释、基因组结构分析、代谢通路预测等,从而揭示细菌在传代过程中可能发生的遗传适应性变化和表型特征。
细菌传代测序实验可以帮助科研人员深入了解细菌在不同生长条件下的遗传变化和进化机制,为解析微生物的适应性进化和资源利用提供重要线索。
1 / 1。

细菌宏基因组学和代谢组学一、细菌宏基因组学1. 基因组测序技术细菌宏基因组学主要利用基因组测序技术对环境中的细菌进行大规模的基因测序。
测序技术包括第二代测序技术和第三代测序技术,如Illumina、PacBio等。
这些技术可以产生大量的序列数据,为后续的基因组组装和分析提供基础。
2. 基因组组装与注释在得到大量测序数据后,需要对这些数据进行基因组组装,将测序读段组装成完整的细菌基因组。
同时,还需要对基因组进行注释,识别出基因的编码区和调控序列,从而揭示细菌的遗传特征和功能。
3. 基因功能与进化分析通过对细菌基因组的注释和功能分析,可以揭示细菌的生理功能、代谢途径以及进化关系。
这对于理解细菌在环境中的作用、传播机制以及与宿主之间的相互作用具有重要意义。
4. 宏基因组学在环境中的应用宏基因组学可以用于研究环境中的微生物群落结构、功能和进化特征。
通过对环境样本进行宏基因组测序和分析,可以了解环境中细菌的多样性、丰度和分布情况,为环境保护和生态修复提供科学依据。
二、细菌代谢组学1. 代谢产物的提取与分离代谢组学主要研究细胞内小分子代谢物的种类、含量及其变化规律。
首先需要对细菌样本进行代谢产物的提取和分离,常用的方法包括有机溶剂萃取、色谱分离等。
2. 代谢产物的鉴定与分类提取得到的代谢产物需要进行鉴定和分类,常用的方法包括质谱分析、核磁共振分析等。
通过这些方法可以确定代谢产物的化学结构、分子量和类别。
3. 代谢产物的定量分析除了鉴定代谢产物的种类外,还需要对代谢产物进行定量分析,以了解其在不同条件下的含量变化。
常用的定量方法包括色谱-质谱联用技术、荧光光谱分析等。
这些方法可以提供定性和定量信息,有助于深入了解细菌的代谢过程和调控机制。
4. 代谢组学在疾病诊断和治疗中的应用代谢组学在疾病诊断和治疗方面具有广泛应用。
通过对细菌代谢产物的分析,可以了解细菌在感染过程中的代谢变化,为疾病诊断提供依据。
同时,通过对代谢产物的调控和干预,可以开发新的治疗策略和方法,提高疾病的治疗效果。
一、实验背景随着生物技术的发展,细菌基因组研究已成为微生物学领域的重要研究方向。
通过对细菌基因组进行深入研究,我们可以了解细菌的遗传信息、生物学特性以及与人类健康的关系。
本实验旨在通过提取细菌基因组DNA,进行PCR扩增、测序和生物信息学分析,探讨细菌的基因组结构和功能。
二、实验过程1. 细菌基因组DNA提取实验中,我们选取了常见的细菌菌株作为研究对象,采用酚-氯仿法提取细菌基因组DNA。
具体步骤如下:(1)将细菌培养至对数生长期,收集菌液。
(2)加入适量的SDS和蛋白酶K,充分混匀,进行细胞裂解。
(3)加入酚-氯仿,混匀,离心分离。
(4)取上清液,加入等体积的异丙醇,混匀,离心沉淀。
(5)用75%乙醇洗涤沉淀,干燥,溶解于TE缓冲液中。
2. PCR扩增为了获得细菌基因组中的特定基因片段,我们采用PCR技术进行扩增。
具体步骤如下:(1)设计特异性引物,用于扩增目标基因。
(2)配制PCR反应体系,包括模板DNA、引物、dNTPs、Taq酶等。
(3)进行PCR扩增,包括预变性、变性、退火、延伸等步骤。
(4)扩增产物进行琼脂糖凝胶电泳检测。
3. 基因组测序将PCR扩增产物进行纯化,然后进行测序。
测序结果经过比对和组装,得到细菌基因组的完整序列。
4. 生物信息学分析利用生物信息学软件对测序结果进行注释、比较和分析,了解细菌基因组的结构和功能。
三、实验心得1. 实验过程中,我们充分认识到细菌基因组研究的重要性。
通过对细菌基因组的深入研究,我们可以揭示细菌的生物学特性、进化关系以及与人类健康的关系。
2. 在实验操作中,我们学会了酚-氯仿法提取细菌基因组DNA、PCR扩增、测序和生物信息学分析等关键技术。
这些技术为今后的研究奠定了基础。
3. 实验过程中,我们遇到了一些问题,如DNA提取效率低、PCR扩增产物不稳定等。
通过查阅文献、请教老师和同学,我们找到了解决问题的方法,提高了实验成功率。
4. 实验过程中,我们深刻体会到团队协作的重要性。
菌株全基因组测序测目的随着科技的飞速发展,基因组测序技术也得到了热烈关注。
全基因组测序作为一种高通量测序技术,被广泛应用于生物学研究中。
对于微生物,全基因组测序技术在分子生物学、生态学、农业以及医学等领域都有着广泛的应用。
本文将介绍菌株全基因组测序的目的、意义及运用等方面的相关信息。
一、菌株全基因组测序的目的菌株全基因组测序的主要目的涉及以下三个方面:1.识别基因组中的基因和功能。
首先,菌株全基因组测序可以帮助我们准确、快速地识别菌株中的基因和功能。
通过测序,我们可以确定菌株整个基因组的序列信息,并通过生物信息学方法将其转化为可识别的DNA序列。
基于这些序列信息,我们可以确定菌株基因组中的基因和其对应的功能,从而揭示菌株的生物学特征。
2.深入了解菌株的演化历史和遗传变异。
其次,菌株全基因组测序可以帮助我们深入了解菌株的演化历史和遗传变异。
对于微生物而言,基因组具有广泛的可塑性和适应性。
通过测序和比较不同菌株基因组序列,我们可以了解菌株之间存在的遗传差异和基因演化历史,为后续科学研究提供重要的参考数据。
3.为微生物学研究提供基础数据和资源。
最后,菌株全基因组测序不仅可以在直接应用方面得到应用,同时也为研究社区提供了重要的数据和资源。
这些数据和资源可以用于虚拟基因组计划、菌群组学和转录组学等领域,可以为微生物学、生态学、医学等各个领域的研究提供强有力的支持。
二、菌株全基因组测序的意义1.准确分析菌株的生物学特征。
菌株全基因组测序可以帮助我们准确分析菌株的生物学特征。
在微生物学领域,全基因组测序成为了研究微生物多样性、环境适应性和致病性等方面的强有力工具。
通过对菌株基因组序列的分析,我们可以了解其生态环境、代谢途径、转录调控等方面的生物学特征。
2.揭示微生物的进化关系。
全基因组测序可以揭示微生物之间的进化关系。
通过比较不同菌株的基因组序列,我们可以了解它们之间的遗传关系和进化轨迹。
同时,全基因组测序还可以检测相关基因的变异和表达水平的变化,对了解微生物的丰富性和多样性提供了重要的支持。
菌株基因组全序列测序及其多样性分析引言随着现代生命科学技术的不断发展,基因组学在生物学研究中越来越重要。
其中,菌株基因组测序技术是一种重要手段,可以帮助我们深入了解菌株的生长、发育和适应环境的机制。
本文将重点介绍菌株基因组全序列测序技术及其在多样性分析中的应用。
术语解释在探讨菌株基因组全序列测序技术及其多样性分析应用之前,我们先了解一些术语的定义:1.基因组(genome):指一个生物体所有基因组成的遗传信息的总和。
2.基因(gene):指DNA序列中一段可以编码蛋白质或非编码RNA的区域。
3.序列(sequence):指DNA或RNA的一连串碱基的排列顺序。
4.全序列(whole genome sequencing):指对一个生物体的所有基因组进行测序。
5.测序(sequencing):指将DNA或RNA样本进行分离、扩增、纯化、切割、测序等一系列步骤,最终得到一连串碱基的排列顺序的过程。
6.多样性(diversity):指同一物种或同一系统中个体之间在形态、遗传、生理、生态等方面的差异。
基因组全序列测序技术菌株基因组全序列测序技术是指对一个菌株的所有基因组进行测序。
具体的测序方法有很多种,包括Sanger测序、454测序、Illumina测序、Ion Torrent测序、PacBio测序等。
选择何种方法进行测序,需要根据实际情况而定。
例如,Sanger测序虽然可以对单个DNA分子进行分析,但是速度相对较慢、成本较高,适合测序较小的区域;而Illumina测序虽然速度较快、成本较低,但是对特定的GC含量和重复序列存在局限性。
因此,实验者需要根据实验对象的大小、GC含量、复杂度和所需深度等,选择最适合的测序方法。
基因组全序列测序技术的应用1.挖掘独特基因通过对菌株基因组全序列测序,可以挖掘出该菌株独特的基因。
独特基因的存在,可能是某个生物体适应特定环境、对特定营养物的吸收、抵御重金属离子等环境压力所需。
【⾼通量测序服务】细菌基因组测序随着细菌基因组研究的迅速发展,全基因组测序已逐步成为微⽣物基础研究的重要⼿段之⼀。
截⾄2010年11⽉,全世界已发布了1748个细菌基因组、61个真菌基因组和103个古细菌基因组序列。
新⼀代⾼通量测序技术⼤⼤减少了基因组测序的成本和时间,让更多实验室可以开展微⽣物基因组测序项⽬。
根据不同的研究⽬的和测序要求,细菌基因组测序可分为四种类型:细菌基因组Scanning、细菌基因组框架图、细菌基因组精细图和细菌基因组完成图。
美吉⽣物灵活应⽤Roche 454长⽚段测序、Illumina Solexa短⽚段⾼覆盖测序,结合454双末端测序(Paired-End Sequencing)及Solexa末端配对测序(Mate-Pair Sequencing,MP)技术,针对不同的基因组测序类型,提供专业、合理、⾼性价⽐的测序服务。
美吉优势1.拥有⾃主实验室和⾼通量测序平台,可以根据合作伙伴要求灵活安排实验,实验周期短,质量可靠。
2.拥有ABI3730xl常规测序平台、Roche 454⾼通量测序平台和Illumina Solexa⾼通量测序平台,三种平台联合应⽤,优势互补,在微⽣物基因组测序中取得1+1+1>3的效果。
3.技术⼈员经验丰富,每年完成数百个BAC全长测序、⼏⼗个微⽣物基因组测序的Gap Closing⼯作。
4.拥有专业的⽣物信息团队和⼤型计算机平台,可以为合作伙伴提供个性化的⽣物信息分析服务。
⼀.细菌基因组Scanning细菌基因组Scannning采⽤Illumina测序技术,对基因组进⾏50×低深度测序,根据序列组装结果,以获取基因组序列的基本信息。
⼆.细菌基因组框架图细菌基因组框架图采⽤Illumina短⽚段测序技术和末端配对测序(MP)技术,对基因组进⾏100×深度测序。
MP测序数据⽤来确定Contigs之间的排列关系,结合100×测序深度,可以得到较佳的基因组拼接结果和较⾼的基因区覆盖度,为随后的基因组信息挖掘提供良好基础。
BMKONT细菌基因组测序服务合同1. 服务概述BMKONT提供细菌基因组测序服务,包括DNA样品提取、测序、数据分析和报告撰写等方面的服务,以满足客户对于细菌基因组测序的需求。
2. 服务内容2.1 样品处理客户需提供符合标准的样品,BMKONT将负责对样品进行质量检测、提取DNA 等处理工作。
2.2 测序BMKONT采用高通量测序技术对样品进行测序,包括Illumina HiSeq测序,以确保样品的高质量测序结果。
2.3 数据分析BMKONT将对测序数据进行多重分析,包括基因组组装、基因注释、基因功能注释、基因组比较等方面的分析,以生成详细的基因组测序数据报告。
2.4 报告撰写BMKONT将根据客户需要,编写符合标准的基因组测序数据报告,包括基因组特征、基因组组装质量评估、基因表达水平分析等。
3. 服务价格BMKONT细菌基因组测序服务价格如下:服务项目价格(元/个)样品处理500测序1200数据分析1500报告撰写800以上价格仅供参考,具体价格根据客户需求而定。
4. 服务周期BMKONT细菌基因组测序服务周期如下:服务项目服务周期样品处理3-5个工作日测序5-7个工作日数据分析7-10个工作日报告撰写1个工作日(初稿)以上周期仅供参考,实际周期根据样品的质量和复杂度而定。
5. 质量保证BMKONT将以最高质量标准确保服务结果的准确和可靠性。
对于非人为因素导致的数据质量问题,将提供免费重测和修改服务。
6. 保密协议所有客户提供的样品和数据将严格保密,不得泄露。
7. 版权声明BMKONT将尽最大努力确保在无侵犯版权的前提下,提供高质量的细菌基因组测序服务报告。
8. 免责声明由于基因组测序可能受到多种因素的影响,如样品质量、分析算法等,因此分析结果可能存在误差。
BMKONT不对分析结果的准确性做出任何明示或暗示的保证或担保,同时BMKONT对于因客户使用基因组测序服务报告而引起的任何直接或间接损失概不负责。