人类全基因组测序
- 格式:pdf
- 大小:1.71 MB
- 文档页数:4

人类基因组测序人类基因组(英语:Human genome)又译人类基因体。
是人类(Homo sapiens)的基因组。
共组成23对染色体,分别是22对体染色体和性染色体X染色体与Y染色体。
含有约31.6亿个DNA碱基对。
碱基对是以氢键相结合的两个含氮碱基,以A、T、C、G四种碱基排列成碱基序列。
其中一部分的碱基对组成了大约20000到25000个基因。
1990年~1998年,人类基因组序列已完成和正在测序的共计约330Mb,占人基因组的11%左右;已识别出人类疾病相关的基因200个左右。
此外,细菌、古细菌、支原体和酵母等17种生物的全基因组的测序已经完成。
值得一提的是,企业与研究部门的携手,将大大地促进测序工作的完成。
美国的基因组研究所(The Institute of Genome Research, TIGR)与PE(Perkin-Elmar)公司合作建立新公司,三年内投资2亿美元,预计于2002年完成全序列的测定。
这一进度将比美国政府资助的HGP的预定目标提前三年。
美国加州的一家遗传学数据公司(Incyte)宣布(1998年〕,两年内测定基因组中的蛋白质编码序列以及密码子中的单核苷酸的多态性,最后将绘制一幅人的10万个基因的定位图。
与Incyte公司合作的HGS(Human Genome Science)公司的负责人宣称,截止1998年8月,该公司已鉴定出10万多个基因(人体基因约为12万个),并且得到了95%以上基因的EST (expressed sequence tag)或其部分序列。
1998年9月14日美国国家人类基因组计划研究所(NHGRI)和美国能源部基因组研究计划的负责人在一次咨询会议上宣布,美国政府资助的人类基因组计划将于2001年完成大部分蛋白质编码区的测序,约占基因组的三分之一,测序的差错率不超过万分之一。
同时还要完成一幅“工作草图”,至少覆盖基因组的90%,差错率为百分之一。

全基因组测序技术的应用和意义随着科技的不断发展,全基因组测序技术(Whole genome sequencing,WGS)的应用越来越广泛。
它作为一种高通量测序技术,能够对任何一个生物体的基因组进行全面的分析和解读,为生物学、医学的研究和应用提供重要支撑和帮助。
一、生物学研究上的意义全基因组测序技术可以通过充分研究生物体的基因组结构、特点、变异等多方面信息,精确掌握其遗传演化规律、适应环境的形成原因以及种群间的差异等方面的问题,并对生命的进化、种群遗传多样性、生物体养殖、育种等领域的研究产生深远的影响。
同时,人们在这一技术的应用下,已经解析了众多生物的基因组,包括人、小鼠、花生、水稻、玉米等模式生物和重要作物,建立了全球基因组测序数据库,为后续研究和应用提供了丰富的资源和信息。
二、医学研究上的意义全基因组测序技术在医学领域中的应用是其最受瞩目的一面。
医学领域中,基因与多种疾病之间有着紧密的联系。
通过全基因组的高通量测序,可以揭示基因在某些疾病发生、发展、预后等方面的相关信息,帮助医生预测患者的疾病风险,更好地为患者制定个性化的诊疗方案。
比如,对于遗传性疾病,全基因组测序技术能够发现患者的致病突变,阐明不同家系中的遗传模式和表型特点,为家系中其他患者以及携带者提供区分和辨识。
同时,全基因组测序技术还能够为新药研发提供更有针对性的方案,提高新药研发的成功率。
三、全基因组测序技术在可持续发展中的应用全基因组测序技术在可持续发展中也有非常重要的应用。
人们利用全基因组测序技术解析各类微生物,提供了丰富的基因组资源,数十万种微生物的基因组结构和特点得到了深入研究,并在医药、工业、环境等方面产生了广泛应用。
比如,人们利用全基因组测序技术开展了对地下水微生物群落的变化规律研究,为环境污染治理和地下水利用提供了可靠的科学依据和技术支持。
综上所述,全基因组测序技术的应用广泛,具有极其重要的意义。
人们在应用全基因组测序技术的过程中,不断坚持科学、严谨、可控的原则,严格保护广大群众的权益和隐私,让科学技术发挥最大的价值,推动人类发展和进步。

全基因组重测序技术的应用从20世纪末开始,以人类基因组计划的启动为标志,全球范围内的生物技术领域展开了快速发展,其中一项重要技术就是全基因组重测序技术。
全基因组重测序技术是将高通量测序技术应用于全基因组水平的DNA测序,能够获取生物体的全部基因组序列信息。
正是由于这项技术的出现,推动了基因组学、遗传学、生命科学等领域的发展,使之成为当前生物信息学领域的重要分支。
全基因组重测序技术的应用领域非常广泛。
从基础科学到实际应用,都具有很大的潜力。
在这里,我们主要从以下几个方面来介绍它的应用。
一、基因组分析和生物学研究。
全基因组重测序技术为基因组分析和研究提供了重要的工具。
对于不同物种的基因组重测序,可以在全基因组水平上观察基因及其调控因子的数量、位置、序列、变异等信息。
对于某些对研究有意义的基因进行全基因组重测序,可以获取这些基因的精确序列,包括外显子和内含子等,从而对它们的功能进行更深入的探究。
对于复杂性状的研究,全基因组重测序技术可以帮助鉴定和分析与疾病相关的基因和位点,从而为疾病的预防和治疗提供更准确的依据。
二、药物开发和个体化医疗。
全基因组重测序技术对药物开发和个体化医疗也有很大启示意义。
首先,它可以帮助科学家了解药物的作用机制和药物代谢途径,从而更好地指导药物开发过程。
其次,在基因组水平上观察药物的作用,人们可以了解哪些基因和变异与药物反应有关。
如果药物在不同的人身上展现出不同的疗效,全基因组重测序技术能够说明其中的遗传学机制,并更好地指导个体化药物治疗下一步的发展。
三、种质资源保护和遗传改良。
全基因组重测序技术在种质资源保护和遗传改良中也具有重要的应用价值。
它可以快速准确地鉴定基因型、基因组结构等信息,为选择优良材料、育种和遗传改良提供重要的依据。
同时,全基因组重测序技术还能延长物种的遗传多样性,帮助保护珍稀物种和资源,以及保障农业生产、食品安全等方面的发展。
四、环境保护和实用科研。
全基因组重测序技术还可以应用到环境保护和实用科研领域。

全基因组测序流程
1 全基因组测序
全基因组测序的全称为Whole Genome Sequencing(WGS),是一
种通过分析基因组中每一条核酸序列,来研究物种及其相关基因的技术。
WGS技术可以揭示产生个体的全部遗传信息,对理解复杂疾病、遗传研究、肿瘤细胞分离等有重要意义。
2 WGS流程
WGS流程由四大步骤:基因组制备、测序生成、数据分析、解析准备。
1)基因组制备:在做全基因组测序之前,需要首先准备新鲜收集
的细胞样本、微生物或动物组织样本,过程中常使用DNA抽提技术和
微流技术进行处理,以获得样品中的有效DNA。
2)测序生成:将经过DNA抽提的样本,或捐赠的自然样本,都会
经过一定的过程进行萃取后,分别进入测序和组装。
除了采用Sanger
测序和pyro sequencing等常规技术外,近年来,产生测序技术出现
较大进展,基本上都采用了massive parallel sequencing(MPS)技术,可以获取数以百万计的序列数据,节省了大量的时间和费用。
3)数据分析:这一步需要将生成的原始序列比对到参考基因组中,以及将低质量的序列移除,通常会采用bioinformatics、Python、R
等计算工具进行分析。
4)解析准备:最后可以解析样本的基因组,发现影响疾病遗传机制及基因变异的差异,以作进一步的分析。
3 总结
全基因组测序是一种研究物种及其相关基因的技术,它需要经历四步技术。
第一步要生成可用于测序的有效DNA;第二步要采用MPS技术,生成数量庞大的序列数据;第三步采用计算工具进行数据分析;最后进行解析准备,发现潜在的基因变异进而对其进行分析。

全基因组测序技术在遗传变异研究中的应用近年来,随着科技的发展,全基因组测序技术越来越广泛地应用于医学研究领域。
全基因组测序技术的出现,让我们有了更全面和深入的了解人类基因组的机会。
现在,全基因组测序技术在遗传变异研究中的应用备受瞩目,成为了人们研究遗传变异的有力工具。
遗传变异是指一种基因或多种基因在人群中存在不同表现的现象,包括了单核苷酸多态性(SNP)、插入缺失等多种类型。
不同的遗传变异类型可能与人类疾病的发生以及药物代谢有关,因此,对于研究这些变异及其与疾病的关系,全基因组测序技术具有不可替代的重要性。
具体来说,全基因组测序技术可以帮助我们解决以下三个问题。
第一个问题,全基因组测序技术可以更全面、更精确地鉴定遗传变异。
相对于传统的遗传变异鉴定技术,例如SNP芯片技术等,全基因组测序技术覆盖面更广,可以检测到更多的变异类型。
此外,全基因组测序技术测序深度也更高,使得我们能够更准确地检测到低频变异。
这些优势使得全基因组测序技术检测的变异更为全面和精确,有利于深入研究变异对人类健康的影响。
第二个问题,全基因组测序技术可以探索人类基因组的序列多样性。
不同种族、不同人群之间的基因组序列存在着巨大的多样性,这种多样性是直接影响疾病发生的重要因素之一。
全基因组测序技术可以将这种多样性展现得淋漓尽致,可以比较不同人群基因组序列相似性和差异性,为跨种族和跨地区的研究提供了有力的工具。
第三个问题,全基因组测序技术还可以挖掘罕见变异对人类健康的影响。
相对于常见变异,罕见变异出现的频率更低,因此缺乏统计学的分析能力,不容易被察觉。
而全基因组测序技术可以有效地检测到罕见变异,有助于发现罕见变异与健康状况之间的联系。
这对于药物开发和疾病预防具有重要的意义。
总之,全基因组测序技术的出现加速了我们对遗传变异的研究,对于疾病预防、精准医学、生命科学研究等方面都有着重要的帮助。
与此同时,全基因组测序技术也在不断更新和完善中,未来还有着更广阔的应用前景。

全基因组测序与拓跋1.引言1.1 概述全基因组测序是一种高效且广泛应用的基因组研究技术,它可以对个体的整个基因组进行测序和分析。
在过去的几十年里,随着测序技术的发展和成本的不断降低,全基因组测序已经成为基因组研究的主要手段之一。
全基因组测序的概念就是对某一生物体的全部DNA进行测序,并将所得数据进行分析和解读。
相比其他测序方法,全基因组测序具有高通量、高分辨率和全面性的优点。
通过全基因组测序技术,我们可以了解整个基因组的组成、结构和功能,从而深入理解生物体的遗传特征和调控机制。
全基因组测序在许多领域都具有广泛的应用价值。
首先,它为人类和动植物基因组的研究提供了有力的工具。
通过全基因组测序,我们可以揭示人类基因组的组成和变异情况,进一步研究与疾病相关的基因突变和遗传变异,为疾病的预防和治疗提供重要依据。
此外,全基因组测序还可以用于揭示不同生物种群间的遗传关系和进化历史,探索物种多样性和进化机制。
全基因组测序技术的发展也为生物科学和医学领域带来了革命性的变化。
通过对个体基因组的高通量测序,我们可以进行个性化医疗和药物治疗,根据个体的遗传信息进行精准诊断和治疗。
此外,全基因组测序还可以应用于农业领域,通过研究作物和家畜的基因组,提高农作物的抗病能力和产量,改良家畜的育种选择。
综上所述,全基因组测序作为一种高效、全面的基因组研究技术,在生物科学、医学和农业领域都具有重要的应用和意义。
随着测序技术的不断革新和发展,全基因组测序将继续发挥重要作用,并为我们揭示基因组的奥秘和推动科学进步做出更大的贡献。
1.2文章结构文章结构部分的内容可以如下所示:文章结构:本文主要分为引言、正文和结论三个部分。
其中,在引言部分,我们将对全基因组测序和拓跋进行一个概述,并介绍文章的目的。
接下来,在正文部分,我们将详细探讨全基因组测序的原理和应用。
具体来说,我们将解释全基因组测序的原理,包括二代测序和三代测序技术的工作原理;同时,我们还将介绍全基因组测序在疾病诊断、基因研究、群体遗传学等方面的应用。

全基因组测序技术是富人专属吗?随着人类基因组计划的完成和高通量生物芯片、高通量基因测序等一系列检测技术的发展,产前诊断技术进展迅猛,进入了全基因组时代。
高通量测序成主流随着测序技术的发展,高通量测序以准确性高、通量高的优势成为测序技术的主流。
伴随着测序的成本大幅下降和大样本人类基因组数据快速累积,高通量测序技术在遗传病检测领域得到了广泛应用,也给产前的应用带来了新的机遇和挑战。
目标基因测序、全外显子测序(WES)和全基因组测序(WGS)等不同的测序策略也使得产前遗传病的检测平台发生了从单一到多样化转变。
全基因组低深度测序结合后续复杂的生物信息学分析, 能对孕妇外周血中游离DNA进行测序分析,实现了胎儿非整倍体的无创产前检测。
与此同时, 基于高通量测序的低深度全基因组拷贝数测序(CNV-seq)技术在产前诊断中的应用逐步深入,由于其高通量、低成本的特点,最近被建议可作为一线产前诊断技术对可能存在胎儿染色体异常的孕妇进行产前诊断,以解决现阶段产前诊断服务供给严重不足的矛盾。
全基因组测序(WGS)理论上可以同时检测单核苷酸变异、结构变异(含拷贝数变异)及线粒体变异等,有望进一步提升临床遗传检测的效能。
但由于目前受限于人们对基因组内含子/调节区域序列变异的解析能力,WGS仅仅作为替代全外显子组(WES)更好的替代方式,以解决WES测序在基因组GC富集区的覆盖度问题。
WES测序极具应用前景由于基因组的外显子仅占基因组DNA的1.5%,且与孟德尔遗传病有关的多数基因涉及外显子,包含约80%~85%的已知致病变异。
相比WGS,WES测序费用较低,成为产前结构异常胎儿的另一个有前景的技术。
近期两项前瞻的队列研究表明,在超声检测到结构异常并排除染色体异常和CNVs的病例,利用WES可以增加8.5%和10.3%的致病性基因变异检出率,且其致病性基因突变具有高比例的新发变异(61.5%~63%)。
这些研究提示WES在产前诊断中的应用能够提高胎儿发育异常或畸形的诊断率。

全基因组测序和四代测序的关联随着科技的不断发展,基因组学领域的研究也日新月异。
全基因组测序和四代测序作为基因组学研究中的重要方法,一直备受关注。
在这篇文章中,我将以从简到繁、由浅入深的方式,探讨全基因组测序和四代测序的关联,希望能为您带来更深层次的理解。
## 什么是全基因组测序和四代测序?让我们简单回顾一下全基因组测序和四代测序的概念。
全基因组测序是指对一个个体的全部基因组进行测序,包括其DNA序列和所有基因。
这种测序方式可以帮助科学家更好地理解个体的遗传特征,发现疾病的基因变异,并为个性化医疗和精准医学提供数据支持。
四代测序则是指第四代测序技术,相较于传统的Sanger测序和二代测序技术,四代测序技术具有高通量、低成本、快速测序等特点。
通过四代测序技术,科学家们可以更快速、更准确地获得基因组的信息,为基因组学研究和临床诊断提供更多可能性。
## 全基因组测序和四代测序的关联在基因组学研究中,全基因组测序和四代测序具有紧密的关联。
四代测序技术的发展为全基因组测序提供了更好的技术支持。
通过四代测序技术,科学家们可以更快速地、更准确地对个体的全部基因组进行测序,大大提高了测序效率,降低了成本,使全基因组测序更加可行。
全基因组测序为四代测序技术的发展提供了更多的数据支持。
通过对不同个体的全基因组数据进行分析,科学家们可以更好地优化四代测序技术,提高测序的准确度和覆盖度,推动了四代测序技术的不断发展和优化。
全基因组测序和四代测序在基因组学研究和临床应用中也相互促进。
全基因组测序可以为四代测序技术的优化和改进提供数据支持,而四代测序技术的发展也为全基因组测序的应用提供了更好的技术保障,为基因组学研究和临床诊断带来了更多的可能性。
## 个人观点和理解在我看来,全基因组测序和四代测序的关联不仅体现在技术上的互相促进,更重要的是它们共同推动了基因组学研究和临床诊断的进步。
随着两种技术的不断发展和完善,我们相信在未来的基因组学研究和临床应用中,将会有更多的突破和创新。
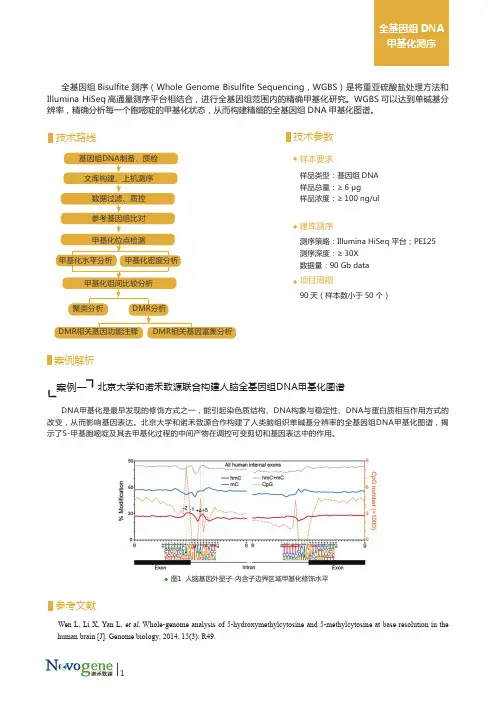

全基因组文献解读全基因组重测序是一种高通量的技术,可以对生物体的整个基因组进行深度测序,从而提供关于基因组结构、变异和表达的全面信息。
以下是一篇关于全基因组重测序的文献解读:一、研究背景随着人类基因组计划的完成,全基因组重测序已成为研究人类基因组变异和基因功能的重要手段。
这项技术可以检测到单核苷酸变异(SNV)、插入和缺失(INDEL)、拷贝数变异(CNV)等多种类型的基因组变异,对于理解遗传疾病、药物反应和生物进化等方面具有重要意义。
二、研究方法本研究采用全基因组重测序技术对一组具有特定表型特征的个体进行基因组测序,然后利用生物信息学方法对这些数据进行深入分析,以寻找与表型特征相关的基因组变异。
具体而言,研究人员首先对样本进行DNA提取和文库构建,然后利用高通量测序平台进行测序,获取原始数据。
接着,研究人员利用生物信息学软件对原始数据进行质量控制、序列比对和变异检测,最后对这些变异进行基因注释和功能分析,以揭示它们与特定表型特征之间的关联。
三、研究结果通过对全基因组重测序数据的深入分析,研究人员发现了一些与表型特征显著相关的基因组变异。
其中一些变异位于已知与特定表型特征相关的基因内或其邻近区域,而另一些变异则位于以前未被认为与该表型特征相关的基因内。
此外,研究人员还发现了一些新的基因组变异,这些变异可能是导致特定表型特征的关键因素。
四、结论本研究通过全基因组重测序技术深入分析了特定表型特征相关的基因组变异,为理解遗传疾病、药物反应和生物进化等方面提供了重要信息。
未来,随着全基因组重测序技术的不断发展和完善,我们有望更深入地了解人类基因组的奥秘,为人类健康和疾病防治提供更多有益的信息。
请注意,上述解读仅是一篇文献的概述,具体的研究内容和结果可能更加复杂和深入。
如需了解更多信息,建议直接阅读相关文献或咨询专业人士。

人类基因组的测序和解读随着科技的不断进步,人类基因组的测序和解读已成为热门话题。
人类基因组指的是人体内所有基因的总和,是人类的遗传信息库。
人类基因组的测序和解读有助于深入了解人类的遗传信息,有助于治疗疾病、推动医学科技的发展,因此备受关注。
一、什么是人类基因组测序和解读?人类基因组测序和解读是指对人类基因组的测序,包括获取人体基因的DNA序列和对DNA序列的信息分析。
通过对DNA序列分析,人们可以了解基因类型、基因变异和基因功能等。
过去,需要耗费大量的时间和人力物力来完成基因组的测序。
但现在,随着技术的进步,测序时间和成本大大降低。
通过高通量测序技术,科学家可以快速获取高质量的基因组数据,进而进行全面的基因组分析。
二、人类基因组的应用人类基因组的应用非常广泛。
它可以用于研究人类群体的遗传信息,对疾病的发病机理进行深入了解,可以协助医生从遗传角度对病人进行诊断和治疗,也可以通过基因编辑等技术进行基因治疗,为疾病治疗带来新的思路和可能。
1、基于人类基因组的疾病研究人类基因组测序和解读对于疾病研究有着重要的作用。
通过对基因组数据的分析,可以发现和疾病有关的基因或基因变异。
人们还可以利用人类基因组数据研究不同种族和不同地域间基因变异的差异,以便更好地了解人类进化历程和人类健康的差异。
最近,人类基因组研究帮助人们深入了解痴呆、癌症等多种疾病的基础机制。
科学家可以利用这些知识和数据研发更有效的治疗方法。
2、基于人类基因组的医疗应用人类基因组测序和解读不仅对疾病的研究有帮助,同时也可以为医生进行个性化诊疗提供帮助。
通过分析基因组数据,医生可以了解病人的基因特征、风险因素等,根据数据调整药物和剂量、制定更有针对性的治疗方案和预防措施。
通过基因编辑技术,科学家正在探索利用人类基因组数据进行基因修复、基因治疗的可能,这将可以为疾病治疗和疾病预防带来更多的希望。
三、挑战和风险人类基因组测序和解读尽管带来了许多好处,但同时也面临了挑战和风险。
全基因组扫描在抗癌基因筛选中的研究随着肿瘤学的快速发展以及基因测序技术的日益完善,现代医学已经进入了基于基因的个性化抗癌治疗时代。
而全基因组扫描就是其中一个重要的手段之一。
本文主要分为以下几个方面来探讨全基因组扫描在抗癌基因筛选中的研究。
一、什么是全基因组扫描全基因组扫描是指对人类全基因组进行测序,并对其进行全面的基因组分析的方法。
这种技术主要分为两种方法:第一种是短读长度测序技术,主要是指Illumina、SOLiD、454等;第二种为长读长度测序技术,例如PacBio和Oxford Nanopore等。
目前大多数全基因组测序都采用了第一种方法。
这种方法可以在较短的时间内测序整个基因组,并产生大量的序列数据。
二、全基因组扫描在癌症基因筛选中的应用现在,全基因组扫描已成为癌症基因筛选和新型抗癌药物开发的重要手段。
因为随着人们对基因的研究逐渐深入,越来越多的人发现癌症是以基因突变为基础的。
全基因组扫描是一种技术和方法,可以帮助医学工作者更全面地理解人类基因组,从而推动下一步基于基因的治疗手段的开发。
三、全基因组扫描的应用流程1.样本采集和总DNA提取2.质控3.高通量测序4.数据处理和分析全基因组扫描应用流程中一般会先对样本进行采集和总DNA 提取。
在这一步中,需要从人体中获取DNA样本,并将其提取至高纯度的样本。
接下来就是质控环节。
质控环节主要用来除去一些低质量的样本来确保后续流程的准确性和可靠性,一般包括三方面:1.测序质量控制:光学密度和光碟恒温度。
2.检测序列的碱基质量3.剔除掉low quality的reads经过了质控部分,就可以正式进入到高通量测序环节。
全基因组扫描通常采用高通量测序技术,如Illumina和SOLiD等。
最后,数据处理和分析是流程中的一个重要环节。
数据处理和分析主要包括两个方面,一个是基因组序列的比对,一个是突变筛选。
最后确定是否选择这个基因作为我们后续抗癌基因的筛选对象。
【科普】什么是全外显⼦组测序与全基因组测序?DNA测序即通过特定的⽅法确定某个体的DNA遗传密码组件(核苷酸)的顺序。
如今,DNA测序技术不仅推进了遗传学研究,也被⽤于各种遗传疾病的检查。
特别是利⽤全外显⼦组测序(whole exomesequencing,WES)与全基因组测序(wholegenome sequencing,WGS)两种⽅法发现基因变异,被越来越多的应⽤于科学研究与临床诊断。
这两种⽅法都基于⼀种能够快速且⼤量对进⾏DNA测序的新技术,被称作⼆代测序技术(next-generationsequencing,NGS,或称“下⼀代测序”技术)。
“Sanger法”是科学家们最早应⽤的测序技术(以纪念它的发明者,英国⽣物化学家Frederick Sanger)。
这是⼀项具有突破意义的发明,⾃此,科学家们可以读出DNA携带的遗传密码,但Sanger法耗时⽽且昂贵。
⽬前Sanger法仍被应⽤于科研与临床研究,并且已经可以由专门的仪器更快速地完成。
然⽽这种⽅法只能⽤于短⼩DNA⽚段的测序,如果想测出⼈类DNA的全长序列(即⼈基因组),Sanger法可能要花费数年的时间。
⽽⼆代测序技术使得完整的⼈基因组的测序可以在数周甚⾄数天内完成,同时花费更少。
什么是全外显⼦组测序与全基因组测序?利⽤⼆代测序技术,对⼤量DNA⽚段进⾏测序就完全可⾏了。
例如,⼀部分DNA⽚段含有蛋⽩质合成的密码“指令”,这部分⽚段被称作“外显⼦”(exon)。
⽬前认为,外显⼦只占到⼈类基因组的⼤约1%,基因组中所有的外显⼦被统称为“外显⼦组”(exome),对这部分序列的测序就被称为“全外显⼦组测序”。
这种⽅法能够检测出所有基因的蛋⽩质编码区域的变异,⽽不仅仅是被选择的有限的若⼲基因。
由于已知的⼤多数导致疾病的突变均发⽣在外显⼦中,全外显⼦组测序从⽽被认为是⼀种⾼效的识别可能致病的突变的⽅法。
但是,近年来研究⼈员发现,外显⼦区域以外的DNA序列也可以影响基因活性,继⽽影响蛋⽩质的表达,导致疾病发⽣。
全基因组重测序名词解释
全基因组重测序是一种高通量测序技术,它涉及对一个个体的
全部基因组进行多次测序。
这项技术可以提供个体基因组的完整信息,包括基因组中的所有DNA序列。
全基因组重测序通常用于研究
个体的遗传变异,包括单核苷酸多态性(SNP)、插入缺失(Indels)和结构变异等。
通过对同一基因组进行多次测序,可以提高测序数
据的准确性和覆盖度,有助于发现更多的变异类型。
全基因组重测序的过程包括DNA提取、文库构建、高通量测序、数据分析和解读。
在数据分析阶段,科研人员会利用生物信息学工
具对测序数据进行比对、变异检测和功能注释,以识别个体基因组
的变异信息。
全基因组重测序在医学研究和临床诊断中具有重要意义。
它可
以帮助科学家们理解遗传疾病的发病机制,发现新的致病基因,并
为个性化医学提供基因组水平的信息。
在临床诊断中,全基因组重
测序可以帮助医生们进行遗传病风险评估、疾病诊断和治疗方案选择。
总的来说,全基因组重测序是一种强大的基因组学技术,它为
我们提供了深入了解个体遗传信息的途径,对基础科学研究和临床医学都具有重要意义。
全基因测序原理随着基因组学的发展,全基因测序已经成为了研究基因组和遗传变异的重要方法。
全基因测序,也被称为全基因组测序或次代测序,是指对一个生物个体的所有基因组进行高通量测序的方法。
它能够提供一个全面的基因组信息,包括基因的序列、基因的变异以及基因组的结构。
全基因测序的原理基本上是通过将DNA样本分解成小片段,并使用高通量测序技术对这些小片段进行测序。
这些小片段的长度通常在100到300个碱基对之间,可以通过不同的测序平台进行测序。
常用的测序技术包括Illumina的高通量测序技术、Ion Torrent的半导体测序技术以及Pacific Biosciences的长读长测序技术等。
全基因测序的过程可以分为以下几个步骤:1. DNA样本制备:首先需要从生物体中提取DNA样本,并对提取出的DNA样本进行纯化和质量检测。
这是保证后续测序结果准确可靠的关键步骤。
2. DNA片段化:将DNA样本分解成小片段,通常采用酶切或超声波方法。
这些小片段的长度可以根据实验需求进行调整,一般在100到300个碱基对之间。
3. 连接测序适配体:为了将DNA片段固定在测序平台上,需要将测序适配体连接到DNA片段的两端。
测序适配体是一种短的DNA片段,能够与测序平台上的引物结合,从而进行测序反应。
4. PCR扩增:为了增加测序适配体的数量,需要进行PCR扩增。
PCR反应会在DNA片段的两端引入引物序列,以便后续的测序反应。
5. 测序反应:将PCR扩增后的DNA片段固定在测序平台上,并进行测序反应。
根据不同的测序技术,测序反应可以使用不同的方法进行,例如Illumina的测序反应是通过逐个碱基对测序的,而Ion Torrent的测序反应则是通过电信号测序的。
6. 数据分析:测序反应完成后,得到的数据会经过一系列的数据处理和分析步骤,包括质控、序列比对、变异检测等。
最终可以得到一个个体的全基因组序列及其变异信息。
全基因测序技术的出现和发展,极大地推动了基因组研究的进展。
1
技术优势
全基因组测序(Whole Genome Sequencing,WGS)是利用高通量测序平台对人类不同个体或群体进行全基因组测序,并在个体或群体水平上进行生物信息分析。
可全面挖掘DNA 水平的遗传变异,为筛选疾病的致病及易感基因,研究发病及遗传机制提供重要信息。
全基因组测序
平台优势
HiSeq X 测序平台
读长:PE150 通量:1.8T/run 测序周期:3
天
专为人全基因组测序准备、测序周期短、通量高
生物信息分析
技术路线
技术参数
样品要求
样本类型:DNA 样品
样本总量:≥1.0 μg DNA (提取自新鲜及冻存样本) ≥1.5 μg DNA (提取自FFPE 样本)样品浓度:≥ 20 ng/µl 测序平台及策略HiSeq X PE150
测序深度
肿瘤:癌组织(50X),癌旁组织/血液样本(30X)遗传病:30~50 X 项目周期37天
3
案例解析
该研究选取3个家系中6个患者和1个正常个体,首先使用基因芯片寻找纯合突变位点,然后对其中无亲缘关系的2例患者采用全基因组测序研究,在2例患者非编码区域均发现相同的变异,10号染色体PTF1A 末端发生一个点突变(chr10:23508437 A>G),且变异在患病人群和细胞试验中均得到了验证。
研究解释了生长发育启动子隐性变异是罕见孟德尔遗传病的常见致病原因,同时说明许多疾病的致病突变也可能位于非编码区。
图1 检出的变异信息
智力障碍是影响新生儿心智发育的一类疾病。
这项研究选取50个经过基因芯片和全外显子测序未确诊致病因子的trio 家系,全基因组测序检出84个de novo SNVs 和8个de novo CNVs,及一些结构变异(如VPS13B、STAG1、IQSEC2-TENM3),检出率为42%。
揭示编码区的de novo SNVs 和de novo CNVs 是导致智力障碍的主要因素,全基因组测序可以作为可靠的遗传性检测应用工具。
案例一 单基因病研究——全基因组测序鉴定PTF1A末端增强子常染色体隐性突变导致胰腺 发育不全[1]
案例二 复杂疾病研究——全基因组测序解析智力障碍的主要致病因素[2]
图2 PTF1A 的家系图谱
参考文献
案例三 癌症研究——全基因组测序揭示卵巢癌化疗抗性特征[3]
选取92个浆液性卵巢癌(HGSCs)患者原发实体瘤、腹水样本或尸检样本共114个样本及正常对照进行WGS研究,重点关注前期化疗有效,后期产生化疗抗性的患病个体。
在产生化疗抗性的个体中频繁检测到CCNE1基因突变,通过检测不同治疗阶段癌细胞的变异情况,发现基因断裂导致抑癌基因失活是产生化疗抗性的重要原因。
此项研究揭示了在化疗选择压力下HGSCs患者基因组的异质性和适应性,在选择化疗方案作为HGSC治疗手段时,需要采用必要的策略避免产生化疗抗性。
图4 产生化疗抗性的个体样本中体突变的数量及频率
[1] Weedon M N, Cebola I, Patch A M, et al. Recessive mutations in a distal PTF1A enhancer cause isolated pancreatic
agenesis [J].Nature Genetics, 2013, 46(1): 61-64.
[2] Christian G, Hehir-Kwa J Y, Djie Tjwan T, et al. Genome sequencing identifies major causes of severe intellectual
disability [J]. Nature, 2014, 511(7509): 344-347.
[3] Patch A-M, Christie E L, Etemadmoghadam D, et al. Whole genome characterization of chemoresistant ovarian cancer [J].
Nature, 2015, 521: 489-494.
图3 智力障碍患者中检出 IQSEC2基因突变
4。