条斑星鲽和圆斑星鲽线粒体12S rRNA与16S rRNA基因序列及其系统分析
- 格式:docx
- 大小:37.38 KB
- 文档页数:2

利用线粒体16S rRNA 基因全序列分析直翅目主要类群的系统发生关系崔爱明;黄原【期刊名称】《遗传》【年(卷),期】2012(34)5【摘要】In order to reconstruct a robust phylogenetic relationship among major groups of Orthoptera and to explore the phylogenetic utility and performance of 16S Ribosomal RNA gene, complete sequences of 16S Ribosomal RNA were sequenced from 18 species in 9 families and 4 superfamilies of Orthoptera, and analyzed with other 40 species that have been completely sequenced. The result showed that the average length of 16S Ribosomal RNA was 1 310 bp. The positions of Tridactuloidea and Gryllotalpidae in Orthoptera were uncertain based on the 16S rRNA data, and the phylogenetic relationships of other major groups in Orthoptera were rather robust. Except for Oedipodidae and Gomphoceridae, Acrididae, Catantopidae, and Arcypteridae in Xia's taxonomic system were not monophyletic groups, and the genetic distances among the five groups were small. This indicates that the five families should be combined into one family. The genetic distances among Pamphagidae, Chrotogonidae, and Pyigomorphidae were also small. The loops of 16S rRNA gene could provide more information than stems when they were used for phylogenetic analysis. Complete sequence of 16S rRNA gene can be usedto reconstruct robust phylogenetic relationship at the taxonomic category of species, genera, and suborder in Orthop-tera, but lack of resolution at family and superfamily levels.%为了构建稳健的直翅目主要类群间的系统发生关系并探讨16S rRNA 基因序列在构建直翅目昆虫不同分类阶元系统发生关系时的可行性、功效以及性能,文章测定了直翅目4 总科9 科18 种昆虫的16S rRNA 基因全序列,联合已知该基因全序列的其他40 种昆虫,构建了直翅目主要类群之间的系统发生关系,并分析了16S rRNA 基因全序列的系统发生性能和功效.结果表明,直翅目昆虫的16S rRNA 基因全长平均为1 310 bp; 除生活方式特化的蚤蝼总科和蝼蛄总科的地位无法确定外,直翅目其他主要类群系统发生关系比较稳定; 蝗总科下除了斑翅蝗科和槌角蝗科外,剑角蝗科、斑腿蝗科、网翅蝗科都不是单系群,且用不同的方法构建的系统发生树中聚类情况完全一致,各科间遗传距离差异不大,建议将其合为一科; 锥头蝗科、瘤锥蝗科和癞蝗科间的遗传距离差异也不大; 在构建系统发生树时,16S rRNA 基因环区的信息量要比茎区的大; 16S rRNA 基因可以构建可靠的直翅目属与种水平和目与亚目高级阶元的系统发生关系,但对科和总科阶元缺乏足够的分辨力.【总页数】12页(P597-608)【作者】崔爱明;黄原【作者单位】陕西师范大学生命科学学院,西安,710062;陕西师范大学生命科学学院,西安,710062【正文语种】中文【相关文献】1.基于线粒体16S rRNA基因探讨蜘蛛几个重要类群的系统发生关系 [J], 毕可然;张利民2.基于线粒体16S rRNA基因探讨鹿科动物系统发生关系 [J], 刘忠权3.基于线粒体16S rRNA和COI基因序列探讨对虾属(Penaeus)物种系统发生关系[J], 刘帅;李墨非;叶嘉;王亚;王春琳4.基于线粒体COⅠ基因序列分析宝贝科主要类群的系统发生关系 [J], 王莹;苏成勇;潘鸿春;郝家胜5.基于18S rRNA基因序列分析唇口目苔藓动物主要类群的系统发生关系 [J], 焦晓霞;杨群;赵华斌;石庆会;郝家胜因版权原因,仅展示原文概要,查看原文内容请购买。



5个品种(系)罗非鱼线粒体12S rRNA基因序列及其RFLP分析李娴; 杨玲; 王成武; 杨德光; 张志山; 张延华; 付佩胜【期刊名称】《《长江大学学报(自然版)理工卷》》【年(卷),期】2008(005)004【总页数】5页(P57-60,66)【关键词】尼罗罗非鱼(Oreochromis niloticus); 奥利亚罗非鱼(O.aureus); 线粒体DNA; 12S rRNA; RFLP【作者】李娴; 杨玲; 王成武; 杨德光; 张志山; 张延华; 付佩胜【作者单位】山东省淡水水产研究所山东济南 250117【正文语种】中文【中图分类】Q75罗非鱼属鲈形目(Perciformes)丽鱼科(Percoidei)的热带鱼类,原产于非洲,现已成为世界性的鱼类养殖对象之一,是联合国粮农组织向全世界推广的优良品种[1,2]。
目前国内罗非鱼的主要养殖品种有尼罗罗非鱼、奥利亚罗非鱼、奥尼鱼(尼罗罗非鱼♀×奥利亚罗非鱼♂)、红罗非鱼等[3]。
线粒体DNA(mitochondrial DNA,mt DNA)是动物体内唯一存在的核外遗传物质,具有母性遗传的特点,它的拷贝数多、突变率高,进化速度比核基因组快5~10倍,突变固定后所形成的多态性位点可反映出群体遗传特征、种群分化和种属关系等,是系统进化和种群遗传研究的理想标记[4~6]。
mtDNA通常由13个蛋白编码基因、2个rRNA基因,22个tRNA基因、控制区和轻链复制起始区组成[7],其中rRNA基因进化最慢,是线粒体中最保守的一类基因[8]。
多种动物和鱼类的12S rRNA基因已用于系统发育和遗传多样性的研究,如鲤鱼、鲫鱼[7]、星鲽[9]、沙塘鳢属鱼类[10]、尖塘鳢属鱼类[5]等,但对罗非鱼的相关研究还未见报道。
限制性片段长度多态性(RFLP)是从分子水平上研究动物遗传变异的一种比较经济有效的手段,在鱼类的遗传研究中已经得到较广泛的应用[11,12],应用PCR技术对特定目的片段进行扩增,扩增产物利用限制性核酸内切酶处理,得到的限制性片段长度多态具有相对的稳定性,并能反映一定的变异,利用酶切片段数目和大小的差异鉴别种内遗传组成和群体间细微差异,也可作为鉴别群体和种属的分子遗传标记[13~15]。


基于16S rRNA部分序列探讨部分鳚亚目鱼类的分子系统进化关系张源真;王伟;姜志强;张钊【期刊名称】《海洋科学》【年(卷),期】2012(000)009【摘要】通过PCR扩增获得了3科5属6种中国黄渤海海域的鳚亚目(Blennioidei)鱼类的线粒体16S rRNA基因序列片段约669bp碱基,结合来自GenBank的5种鳚亚目其他科鱼类的相应基因片段,并以眼斑雪冰鱼(Chionodraco rastrospinosus)为外群,生成供系统发育分析的序列矩阵,利用MEGA 4.0软件分析序列的碱基组成、差异百分比、转换/颠换值等,应用最大简约法(MP)和邻接法(NJ)构建系统发育树.结果显示:在鳚亚目鱼类的16S rRNA 片段生成的序列矩阵中发现有碱基的插入缺失现象,共有207 bp变异位点,转换/颠换值为0.8,碱基平均差异为3.36;支持绵鳚(Enchelyopus elongates)归于鳚亚目绵鳚科(Zoarcidae),鳚(Azuma emmnion)归于鳚亚目线鳚科(Stichaeidae);方氏云鳚(Enedrias fangi)和云鳚(Enedrias nebulosus)种间遗传距离只有0.01,亲缘关系最近.【总页数】7页(P89-95)【作者】张源真;王伟;姜志强;张钊【作者单位】大连海洋大学生命与水产学院农业部海水增养殖学重点开放实验室, 辽宁大连 116023;大连海洋大学生命与水产学院农业部海水增养殖学重点开放实验室, 辽宁大连 116023;大连海洋大学生命与水产学院农业部海水增养殖学重点开放实验室, 辽宁大连 116023;大连海洋大学生命与水产学院农业部海水增养殖学重点开放实验室, 辽宁大连 116023【正文语种】中文【中图分类】Q959【相关文献】1.基于ITS-1序列的部分鳚亚目鱼类的分子系统进化关系研究 [J], 张源真;王伟;姜志强2.基于16S rDNA部分序列探讨中国近海30种石斑鱼类的分子系统进化关系 [J], 丁少雄;王颖汇;王军;庄轩;苏永全;尤颖哲;李祺福3.基于线粒体16S rRNA基因部分序列探讨贵州9种菊头蝠(翼手目:菊头蝠科)的分子系统进化关系 [J], 宋华;王会;陈学平;谷晓明4.基于16SrRNA部分序列探讨中国近海十三种石首鱼类的分子系统进化关系 [J], 张永;马春艳;马凌波;倪勇;沈盎绿5.基于16S rRNA部分序列探讨12种鲹科鱼类的分子系统进化关系 [J], 郑文娟;朱世华;邹记兴;杨迎春;沈锡权因版权原因,仅展示原文概要,查看原文内容请购买。

3种星虫线粒体16S rRNA、COⅠ和Cyt b基因片段的序列比较陈子安;杜晓东;王庆恒;邓岳文【摘要】对裸体方格星虫(Sipunculus nudus)、可口革囊星虫(Phascolosoma esculenta)和澳洲管体星虫(Siphonosoma australe)的线粒体16S rRNA、COⅠ和细胞色素b(Cyt b)基因片段序列进行比较,并对其系统发生进行了初步探讨.采用PCR方法得到总长度分别为531~544 bp(16S)、652~675 bp(COⅠ)和406~453 bp(Cyt b)的线粒体片段.片段碱基A+T比例较高(16S rRNA基因58.3%,COⅠ基因56.9%,Cyt b基因59.5%). 16S rRNA片段存在169个碱基变异位点(其中包括167个简约信息位点)和44个碱基插入/缺失,种内个体间变异较小;COⅠ片段有512个碱基(333个简约信息位点)存在变异,79个碱基插入/缺失;Cyt b片段存在347个碱基(318个简约信息位点)变异位点,16个碱基插入/缺失.数据分析结果支持3种星虫和环节动物的分类地位较近,与软体动物较远的分类观点.此外,裸体方格星虫与澳洲管体星虫之间亲缘关系较近(D=0.3159、03156、0.2361).认为3种星虫线粒体16S rRNA、COⅠ和Cyt b基因在种间存在明显的多态性,证实了三种基因序列均普遍适用于星虫种及以上阶元的系统学分析.【期刊名称】《广东海洋大学学报》【年(卷),期】2007(027)004【总页数】8页(P3-10)【关键词】星虫;线粒体;16SrRNA基因;COⅠ基因;Cyt b基因;基因序列【作者】陈子安;杜晓东;王庆恒;邓岳文【作者单位】广东海洋大学水产学院,广东,湛江,524088;广东海洋大学水产学院,广东,湛江,524088;广东海洋大学水产学院,广东,湛江,524088;广东海洋大学水产学院,广东,湛江,524088【正文语种】中文【中图分类】Q19星虫营穴居生活,主要分布于河口、港湾及沿岸地表径流较丰富的泥沙或沙泥滩涂,我国沿海均有分布[1-2]。

基于线粒体16S rRNA、COX1和Cytb基因探讨11种小丑鱼的系统发育关系何丽斌;陈芳;朱志煌;马敏宁;张河长;吴孙龙;周宸【摘要】采用线粒体16S rRNA、COX1和Cytb基因测定11种小丑鱼的序列,分析其基因序列差异和遗传距离关系,并用邻接法、最大似然法和贝叶斯法构建系统进化树,探讨了11种小丑鱼的系统发育关系.序列分析结果显示,经比对处理后的3个基因总长度4388 bp,其中多态位点1028个,简约信息位点616个,转换与颠换比为2.12,A+T的平均含量为54.7%,高于C+G的平均含量45.3%.系统发育关系表明:(1)双锯鱼亚科为单系发生,但双锯鱼属不为单系发生,建议对棘颊雀鲷属重新定位;在双锯鱼属中,公子小丑鱼Amphiprion ocellaris和黑边公子小丑鱼Amphiprion percula聚为一支,为双锯鱼亚科分子亲缘关系基础;(2)透红小丑鱼Premnas biaculeatus在双锯鱼属的定位较为模糊,贝叶斯树显示透红小丑鱼P.biaculeatus 与公子小丑鱼A.ocellaris和黑边公子小丑鱼A.percula聚成一支并位于该支的底端,而NJ树和ML树中透红小丑鱼却没能和公子小丑鱼A.ocellaris和黑边公子小丑鱼A.percula聚为一支,而是和其他小丑鱼汇聚成一支;(3)透红小丑鱼P.biaculeatus与金透红小丑鱼Premnas epigrammata是同一种.%Sequences of mitochondrial 16S rRNA,COX1 and Cytb genes were determined to establish the phylogenetic relationship among 11 species of anemonefish using the neighbor joining (NJ),maximum-likelihood (ML) and Bayesian methods with the divergence and genetic distances determined aswell.The 3 genes had a combined length of 4388 bp that consisted of 1028 polymorphic and 616 parsimonious information sites.The ratio of transition to transversion was 2.12,and the average A+T content was 54.7%,whichwas higher than 45.3% for C+G in the genes.It was found that (1) the subfamily Amphiprioninae was monophyly,but the genus Am phiprion was not monophyletic,implying Premnas should be reclassified;(2) the phylogenetic trees grouped the percula complex into a clade that formed the ancestral taxon of anemonefish,but the phylogenetic relationship was uncertain in regard to Premnas biaculeatus;and,while the Bayesian tree placed the percula complex and P.biaculeatus in a same clade,NJ and ML tree classified P.biaculeatus in a clade with subgeneraAmphiprion,Paramphiprion and Phalerebus;and,(3) the genetic distance between species suggested a possibility of P.biaculeatus and Premnas epigrammata being identical.【期刊名称】《福建农业学报》【年(卷),期】2018(033)003【总页数】6页(P230-235)【关键词】16S rRNA基因;COX1基因;Cytb基因;小丑鱼;遗传差异;系统进化【作者】何丽斌;陈芳;朱志煌;马敏宁;张河长;吴孙龙;周宸【作者单位】福建省水产研究所,福建厦门 361013;福建省水产研究所,福建厦门361013;福建省水产研究所,福建厦门 361013;福建珊瑚海生态科技有限公司,福建福州 350102;福建珊瑚海生态科技有限公司,福建福州 350102;福建珊瑚海生态科技有限公司,福建福州 350102;福建省水产研究所,福建厦门 361013【正文语种】中文【中图分类】S917.4小丑鱼隶属于雀鲷科Pomacentridae,双锯鱼亚科Amphiprioninae。
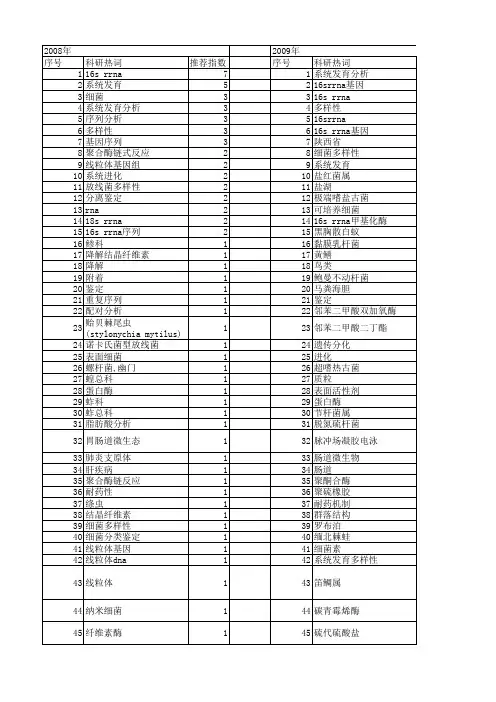
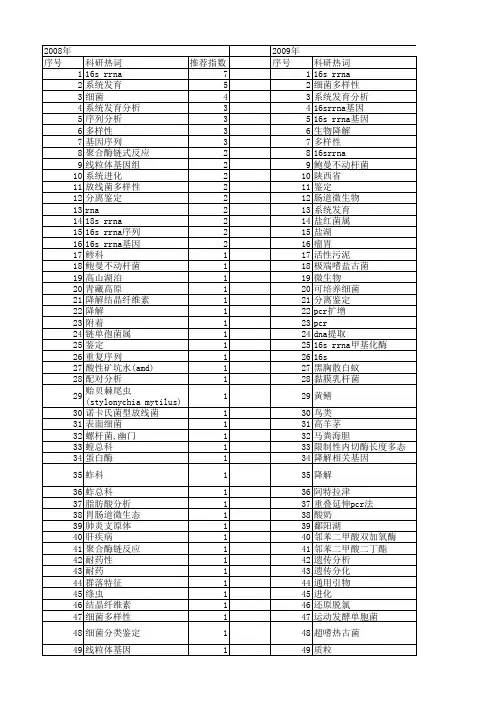

6种笛鲷属鱼类线粒体16SrRNA基因片段的序列比较周发林;江世贵;苏天凤;吕俊霖【期刊名称】《中国水产科学》【年(卷),期】2004(011)002【摘要】对笛鲷属(Lutjanus)的紫红笛鲷(Lutjanus argentimaculatus)、白星笛鲷(Lutjanus stellatus)、千年笛鲷(Lutjanus sebae)、勒氏笛鲷(Lutjanus russellii)、红鳍笛鲷(Lutjanus erythropterus)线粒体DNA 16S rRNA 基因片段进行了PCR 扩增和测序,得到长度约418 bp的序列.结合GenBank中斜带笛鲷(Lutjanus decussatus)该区段的16S rRNA序列,用Clustal_X排序软件进行16S rRNA序列的对位排列.通过Mega 2.1软件对所得线粒体16S rRNA片段序列进行比较,共检测53个碱基存在变异,其中包括21个简约信息位点,并用"Pairwise distance"计算了各属间的相对遗传距离,结果表明,其序列差异(转换+颠换)在0.027~0.083,其中勒氏笛鲷与斜带笛鲷的序列差异最小, 红鳍笛鲷与勒氏笛鲷的序列差异最大.以高体四长棘鲷(Argyrops spinifer)为外类群,采用Mega 2.1软件中的"Neighbore-Joining"法得到唯一1个分子系统树,系统树各分支的置信度由"Bootstrap"1000 循环检验.结果表明,6种笛鲷鱼类聚成明显的3个分支,第1个分支,包括勒氏笛鲷、斜带笛鲷和白星笛鲷;第2个分支,包括紫红笛鲷;第3个分支,包括红鳍笛鲷和千年笛鲷.【总页数】5页(P99-103)【作者】周发林;江世贵;苏天凤;吕俊霖【作者单位】中国水产科学研究院,南海水产研究所,广东,广州,510300;中国水产科学研究院,南海水产研究所,广东,广州,510300;中国水产科学研究院,南海水产研究所,广东,广州,510300;中国水产科学研究院,南海水产研究所,广东,广州,510300【正文语种】中文【中图分类】Q959.483【相关文献】1.6种笛鲷属鱼类Cyt b基因片段序列的比较 [J], 周发林;江世贵;苏天凤;吕俊霖2.紫红笛鲷线粒体全序列与笛鲷属鱼类分化年代的贝叶斯分析 [J], 廖杰;王中铎;郭昱嵩;刘楚吾3.笛鲷属(Lutjanus)鱼类线粒体16S rRNA基因序列比较及系统学分析 [J], 刘楚吾;徐田军;刘丽;郭昱嵩;董秋芬4.笛鲷属线粒体基因组编码序列的系统进化分析能力评估 [J], 王中铎;谭围;郭昱嵩;刘丽;刘楚吾;刘筠5.两种鲷属鱼类线粒体COI基因片段序列的比较 [J], 张凤英;马凌波;施兆鸿;夏连军;马春艳因版权原因,仅展示原文概要,查看原文内容请购买。
16SrRNA和COI基因条形码在12种观赏鱼种类鉴定中的应用陈信忠;郭书林;龚艳清;袁芳;许奕宸;杨俊萍【期刊名称】《福建农业学报》【年(卷),期】2016(031)012【摘要】应用通用引物测定观赏鱼市场上价值较高的红尾金龙鱼、黄尾龙鱼、青龙鱼、珍珠龙鱼、银龙鱼、神仙鱼、非洲珠宝鱼、接吻鱼、珍珠毛足鲈、蓝钻石孔雀、孔雀鱼、锦鲤等12种观赏鱼的16 S rRNA基因和COI基因部分序列。
应用DNA序列分析软件,对这些基因进行同源性比较和系统发育分析。
通过GenBank中的核酸BLAST和BOLD SYSTEMS数据库对这些基因序列进行比对,结果表明16 S rRNA基因和COI基因可以在物种水平作为基因条码对亚洲龙鱼、珍珠龙鱼、银龙鱼和神仙鱼等观赏鱼进行准确的鉴别,而且应用COI 基因构建的系统发育树比16 S rRNA基因更加接近现有的鱼类分类体系。
但对形态十分相似的慈鲷等观赏鱼,以及红尾金龙鱼、黄尾龙鱼、青龙鱼等同一个物种不同品系的观赏鱼依靠16 S rRNA 基因或COI 基因都无法准确鉴定,需要研究进化速度更快的DNA基因条码。
【总页数】6页(P1267-1272)【作者】陈信忠;郭书林;龚艳清;袁芳;许奕宸;杨俊萍【作者单位】厦门出入境检验检疫局,福建厦门361026;厦门出入境检验检疫局,福建厦门 361026;厦门出入境检验检疫局,福建厦门 361026;厦门出入境检验检疫局,福建厦门 361026;厦门出入境检验检疫局,福建厦门 361026;厦门出入境检验检疫局,福建厦门 361026【正文语种】中文【中图分类】Q179;Q523.8【相关文献】1.128条形码技术在艺术招生管理系统中的应用 [J], 任传成;徐凤生;孙志卓2.条形码与RFID的较量 RFID和二维条形码两种技术在应用中各有优势 [J], Christiane Hilsmann3.四种增色剂在观赏鱼中应用的研究 [J], 王锐;费小红4.DNA条形码在篮子鱼科鱼类种类鉴定和系统进化分析中的应用 [J], 闫亚利;张楠;郭华阳;郭梁;朱克诚;刘宝锁;张殿昌5.DNA条形码在篮子鱼科鱼类种类鉴定和系统进化分析中的应用 [J], 闫亚利;张楠;郭华阳;郭梁;朱克诚;刘宝锁;张殿昌;;;;因版权原因,仅展示原文概要,查看原文内容请购买。
两种鯻科鱼类线粒体16S rRNA基因片段序列分析张龙岗;孟庆磊;安丽;董学飒;付佩胜【摘要】利用PCR技术成功扩增了高体革鯻(Scortum barcoo)和厚唇弱棘喇(Hephaestus fuliginosus)的线粒体16S rRNA基因序列.通过序列测定,得到791 bp的基因片段,碱基A、T、G、C平均含量分别为31.4%、21.2%、20.5%和26.9%,序列中的A+T含量明显高于G+C的含量,这与其他鱼类的16S rRNA基因片段研究结果相一致.通过序列分析发现,高体革鯻和厚唇弱棘喇两序列共存在23处碱基变异,其中碱基转换位点20个,碱基颠换位点3个,转换与颠换比率为6.7,没有发现碱基插入与缺失.与从GenBank中查到的3种蜊科鱼类的同源序列比对,邻接法(neighbor-joining)构建亲缘关系树,结果显示,5种蜊科鱼类聚在一起,分为独立的2支,匀蜊和单色匀鯻及花身蜊聚成1支,高体革鯻和厚唇弱棘鯻聚为1支,说明高体革鯻与厚唇弱棘蜊的亲缘关系比较近,而与其他另外3种鯻科鱼类亲缘关系比较远.【期刊名称】《生物技术通报》【年(卷),期】2011(000)008【总页数】5页(P148-152)【关键词】鯻科;线粒体;16S;rRNA;序列分析【作者】张龙岗;孟庆磊;安丽;董学飒;付佩胜【作者单位】山东省淡水水产研究所山东省淡水水产遗传育种重点实验室,济南250117;山东省淡水水产研究所山东省淡水水产遗传育种重点实验室,济南250117;山东省淡水水产研究所山东省淡水水产遗传育种重点实验室,济南250117;山东省淡水水产研究所山东省淡水水产遗传育种重点实验室,济南250117;山东省淡水水产研究所山东省淡水水产遗传育种重点实验室,济南250117【正文语种】中文鱼类线粒体DNA(简称mtDNA)与核DNA相比,具有分子小、编码效率高、拷贝数多、结构简单、进化速度快、不同区域进化速度存在差异以及母性遗传等特点,是一个相对独立的复制单位。
第41卷 第5期 渔 业 科 学 进 展Vol.41, No.5 2020年10月Oct., 2020* 国家重点研发计划(2016YFF0201805)资助 [This work was supported by the National Key Research and Development Plan of China (2016YFF0201805)]. 陆键萍,E-mail:**************************① 通讯作者:王联珠,研究员,E-mail:***************.cn ;许加超,教授,E-mail:*************.cn 收稿日期: 2019-07-03, 收修改稿日期: 2019-07-26DOI: 10.19663/j.issn2095-9869.20190703002 /陆键萍, 姚琳, 信红梅, 曲梦, 江艳华, 李风铃, 郭莹莹, 王联珠, 许加超. 线粒体CO Ⅰ、Cyt b 和16S rRNA 基因在6种金枪鱼鉴定中的适用性分析. 渔业科学进展, 2020, 41(5): 73–81Lu JP, Yao L, Xin HM, Qu M, Jiang YH, Li FL, Guo YY, Wang LZ, Xu JC. Applicability analysis of mitochondrial CO Ⅰ, Cyt b and 16S rRNA genes in identification of six tuna species. Progress in Fishery Sciences, 2020, 41(5): 73–81线粒体CO Ⅰ、Cyt b 和16S rRNA 基因在6种金枪鱼鉴定中的适用性分析*陆键萍1,2 姚 琳2 信红梅2,3 曲 梦2 江艳华2李风铃2 郭莹莹2 王联珠2① 许加超1①(1. 中国海洋大学食品科学与工程学院 青岛 266000;2. 中国水产科学研究院黄海水产研究所 农业农村部水产品质量安全检测与评价重点实验室 青岛 266071;3. 大连工业大学 大连 116000)摘要 探讨细胞色素氧化酶亚基Ⅰ基因(CO Ⅰ)、细胞色素b 基因(Cyt b)及16S rRNA 基因对主要渔获区的蓝鳍金枪鱼(Thunnus thynnus )、马苏金枪鱼(Thunnus maccoyii )、黄鳍金枪鱼(Thunnus albacares )、大目金枪鱼(Thunnus obesus )、长鳍金枪鱼(Thunnus alalunga )和正鲣(Katsuwonus pelamis ) 6种重要的生食金枪鱼及其易混品种的物种鉴定和进化分析的适用性。
两种近方蟹线粒体16S rRNA基因序列分析徐敬明【摘要】测定了绒螯近方蟹和肉球近方蟹线粒体16S rRNA基因部分片段的序列,其长度分别为529bp和525bp.二者的核苷酸序列A、T、G、C的含量相似,分别为36.3%、37.2%、15.9%、10.6%,35.2%、37.5%、17.1%、10.2%;不包括4处插入/缺失位点,两序列间有31个变异位点,核苷酸差异率为5.91%,其中转换18个、颠换13个,转换与颠换比约为1.4.对国外3种近方蟹的16S rRNA基因序列与本文的2种近方蟹序列一起比对获得512bp的同源序列(含插入/缺失位点)进行分析,A+T的平均含量为73.5%,明显高于G+C的平均含量,共检测到变异位点67个,简约信息位点19个.基于已知的弓蟹科43种蟹类的16S rRNA基因502bp同源序列获得的系统发生树的拓扑结构表明,Helicana、Eriocheir、Helice、Cyrtograpsus、Metaplax、Brachynotus属的蟹类各自聚为一支,且与形态学分类结果一致,表明其分别为一单系;但Hemigrapsus、Gaetice、Cyclograpsus属的蟹类却没有各自聚为一支,分子数据不支持它们分别为一单系.【期刊名称】《海洋通报》【年(卷),期】2010(029)006【总页数】5页(P638-642)【关键词】绒螯近方蟹;肉球近方蟹;16S rRNA;序列;系统发生【作者】徐敬明【作者单位】重庆文理学院生命科学与技术学院,重庆,402168【正文语种】中文【中图分类】Q178.53;Q953近方蟹(Hemigrapsus)隶属甲壳纲(Crustacea)、十足目(Decapoda)、方蟹科(Grapsidae)、弓蟹亚科(Varuninae),其中绒螯近方蟹(H. penicillatus)和肉球近方蟹(H. sanguineus)不仅外形相似,而且生活环境也基本相同,均栖息于海边岩石下或石缝中;数量较大,是制作蟹稣、蟹酱的原料,具有一定的经济价值[1]。
条斑星鲽和圆斑星鲽线粒体12S rRNA与16S rRNA基因序
列及其系统分析
丛林林;宋广军;木云雷;陈姝君;葛陇利;高祥刚;赫崇波
【期刊名称】《水产科学》
【年(卷),期】2007(26)7
【摘要】采用PCR产物直接测序法分别测定了条斑星鲽和圆斑星鲽线粒体基因组序列,并对12S rRNA和16S rRNA基因核苷酸全序列进行分析, 条斑星鲽和圆斑星鲽线粒体12S rRNA的核苷酸序列长度分别为948 bp和949 bp,16S rRNA均为1716 bp.试验结果表明,由12S rRNA和16S rRNA基因所构建的两个系统树的结构基本一致,21种鱼主要分为3个大的分支:鲤形目、鲶形目聚为1支,鲑形目独立聚为1支,鲈形目和鲽形目聚为1大支.鲆鲽类与鲈形目的鲹科、鲷科鱼类聚类在一支,且与鲹科的日本竹荚鱼、大西洋竹荚鱼构成姊妹群,支持鲆、鲽类是从鲈形目分化出来的观点, 而同属于鲽形目的鳎科鱼类塞内加尔鳎在12S rRNA 树中成为一个独立的分支,在16S rRNA树中聚到鲈形目和鲽形目的分支中,并且与鲈形目关系更近些,鳎类是1个特殊类群,其分类地位有待进一步探讨.线粒体基因组序列已提交到GenBank中,序列号为:DQ403797和EF025506.
【总页数】4页(P390-393)
【作者】丛林林;宋广军;木云雷;陈姝君;葛陇利;高祥刚;赫崇波
【作者单位】辽宁师范大学,生命科学学院,辽宁,大连,116029;辽宁省海洋水产科学研究院,辽宁省应用海洋生物技术开放实验室,辽宁省海洋水产分子生物学重点实验室,辽宁,大连,116023;辽宁省海洋水产科学研究院,辽宁省应用海洋生物技术开放实
验室,辽宁省海洋水产分子生物学重点实验室,辽宁,大连,116023;辽宁省海洋水产科学研究院,辽宁省应用海洋生物技术开放实验室,辽宁省海洋水产分子生物学重点实验室,辽宁,大连,116023;辽宁师范大学,生命科学学院,辽宁,大连,116029;辽宁省海洋水产科学研究院,辽宁省应用海洋生物技术开放实验室,辽宁省海洋水产分子生物学重点实验室,辽宁,大连,116023;辽宁省海洋水产科学研究院,辽宁省应用海洋生物技术开放实验室,辽宁省海洋水产分子生物学重点实验室,辽宁,大连,116023;大连水产学院,生命科学与技术学院,辽宁,大连,116023;辽宁省海洋水产科学研究院,辽宁省应用海洋生物技术开放实验室,辽宁省海洋水产分子生物学重点实验室,辽宁,大连,116023;辽宁省海洋水产科学研究院,辽宁省应用海洋生物技术开放实验室,辽宁省海洋水产分子生物学重点实验室,辽宁,大连,116023
【正文语种】中文
【中图分类】Q344.11
【相关文献】
1.两种野生经济鱼类圆斑星鲽和石鲽的最新形态观测 [J], 刘曼红;于洪贤;马玉堃;高天翔;田艳丽
2.RAPD对圆斑星鲽和条斑星鲽养殖群体的遗传多样性研究 [J], 杨奔;张培军;尤锋;李军;徐冬冬;吴志昊;于道德;倪静;徐世宏;徐永立
3.圆斑星鲽及相关种类线粒体DNA控制区结构分析 [J], 赫崇波;曹洁;刘卫东;周遵春;葛陇利;高祥刚;王效敏
4.圆斑星鲽线粒体基因组全序列结构及其进化 [J], 赫崇波;高祥刚;王效敏;刘卫东;周遵春;木云雷;葛陇利
5.圆斑星鲽piwil2基因的克隆与表达分析 [J], 杨珍珍; 边力; 张岩; 常青; 陈四清; 刘长琳; 葛建龙; 胡建成; 张盛农
因版权原因,仅展示原文概要,查看原文内容请购买。