分子生物学实验技术 郭秋平
- 格式:ppt
- 大小:4.55 MB
- 文档页数:40

摘要:在进行基因分离、克隆和核苷酸序列分析等生物过程中,通常会运用到聚合酶链反应技术,即PCR技术。
荧光PCR是近年来科学家和学者常用的手段,本文就对荧光PCR的原理、常用探针的优缺点以及多重荧光PCR技术的进展进行了综述。
关键词:荧光PCR;探针;多重荧光PCR聚合酶链反应( PCR) 技术自1985年问世以来,以其灵敏性高、特异性强和速度快在分子生物学等科研领域得到了广泛应用[1]。
但是,利用传统的PCR 技术进行检测鉴定时,需要进行扩增反应后的电泳分离及染色处理,且不能准确定量,使其应用受到限制[2]。
荧光PCR 技术拥有特异性强、灵敏度高、重复性好、定量准确、速度快、全封闭反应等优点,已成为了分子生物学研究的重要工具[1]。
1、荧光PCR原理荧光PCR的原理是以荧光共振能量转移原理为基础。
荧光共振能量转移原理是当一个荧光分子(供体分子)的荧光光谱与另一个荧光分子(受体分子)的激发光谱重叠时,供体荧光分子自身的荧光强度衰减,受体荧光分子的荧光强度增强。
Ct值是指每个反应管内的荧光信号到达设定的阈值时所经历的循环数。
Ct值是实时荧光PCR 中一个很关键的因素,C 代表循环(Cycle),t代表阈值(Threshold)。
每个模板的Ct 值与该模板的起始拷贝数的对数存在线性关系,起始拷贝数越多,Ct 值越小。
利用已知起始拷贝数的标准品可做出标准曲线。
因此,根据荧光探针的发光基团所发出的荧光强度与PCR 产物的数量呈对应关系,只要对荧光信号进行检测并获得未知样品的Ct值,即可从标准曲线上计算出该样品的起始拷贝数。
与普通PCR相比,荧光PCR可以利用荧光信号实时监测PCR反应过程中每一个循环扩增产物的变化,可以对初始模板量进行定量分析。
概括地说,荧光PCR的基本原理就是样本核酸扩增呈指数增长,在反应体系和条件完全一致的情况下,样本DNA含量与扩增产物的对数成正比,由于反应体系中的荧光染料或荧光标记物(荧光探针)与扩增产物结合发光,其荧光量与扩增产物量成正比,因此通过荧光量的检测就可以测定样本核酸量[2]。
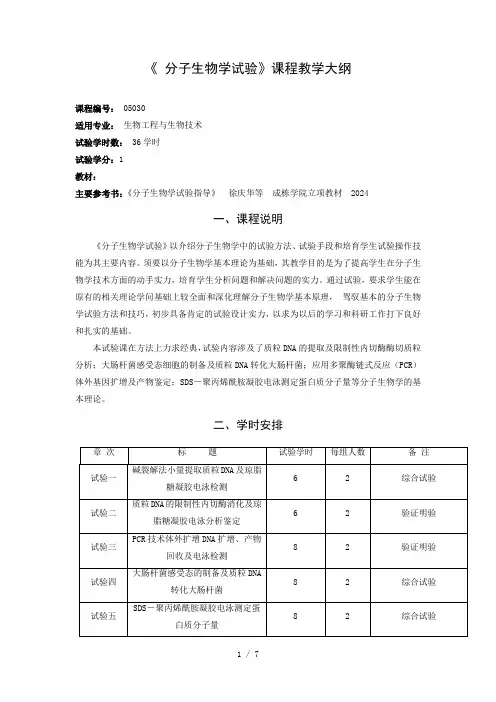
《分子生物学试验》课程教学大纲课程编号: 05030适用专业:生物工程与生物技术试验学时数:36学时试验学分:1教材:主要参考书:《分子生物学试验指导》徐庆华等成栋学院立项教材 2024一、课程说明《分子生物学试验》以介绍分子生物学中的试验方法、试验手段和培育学生试验操作技能为其主要内容。
须要以分子生物学基本理论为基础,其教学目的是为了提高学生在分子生物学技术方面的动手实力,培育学生分析问题和解决问题的实力。
通过试验,要求学生能在原有的相关理论学问基础上较全面和深化理解分子生物学基本原理,驾驭基本的分子生物学试验方法和技巧,初步具备肯定的试验设计实力,以求为以后的学习和科研工作打下良好和扎实的基础。
本试验课在方法上力求经典,试验内容涉及了质粒DNA的提取及限制性内切酶酶切质粒分析;大肠杆菌感受态细胞的制备及质粒DNA转化大肠杆菌;应用多聚酶链式反应(PCR)体外基因扩增及产物鉴定;SDS-聚丙烯酰胺凝胶电泳测定蛋白质分子量等分子生物学的基本理论。
二、学时安排三、教学内容及教学基本要求试验一碱裂解法小量提取质粒DNA及琼脂糖凝胶电泳检测一、试验特点试验类型:综合试验类别:专业基础安排学时:6 每组人数:2二、试验目的1、驾驭碱裂解法小量提取质粒DNA和试剂盒提取质粒DNA的原理和方法。
2、驾驭琼脂糖凝胶的制备及外源DNA的检测原理。
3、了解质粒DNA的粗略定量方法。
三、试验内容提要1、将单菌落接种于3m1含相应抗生素的LB培育基中,37℃摇菌过夜;2、12,000rpm离心30sec,收集菌体;3、加200u1溶液I(含RNaseA 100ug/ml ),振荡悬浮菌体;4、加200ul新配制的溶液II,颠倒混匀;5、溶液澄清后马上加入预冷的200u1溶液III混匀后冰上放置5~10 min;6、4℃、12,000rpm离心15min,取上清,加0.7倍异丙醇混匀,室温静置5min;7、12,000rpm离心10min,70%乙醇洗涤沉淀,抽干后溶于适量水或TE,-20℃保存备用。

可编辑修改精选全文完整版分子生物学实验方法与步骤表达蛋白的SDS-聚丙烯酰胺凝胶电泳分析一、原理细菌体中含有大量蛋白质,具有不同的电荷和分子量。
强阴离子去污剂SDS与某一还原剂并用,通过加热使蛋白质解离,大量的SDS 结合蛋白质,使其带相同密度的负电荷,在聚丙烯酰胺凝胶电泳(PAGE)上,不同蛋白质的迁移率仅取决于分子量。
采用考马斯亮兰快速染色,可及时观察电泳分离效果。
因而根据预计表达蛋白的分子量,可筛选阳性表达的重组体。
二、试剂准备1、30%储备胶溶液:丙烯酰胺(Acr)29.0g,亚甲双丙烯酰胺(Bis)1.0g,混匀后加ddH2O,37O C溶解,定容至100ml, 棕色瓶存于室温。
2、1.5M Tris-HCl(pH 8.0:Tris 18.17g加ddH2O溶解, 浓盐酸调pH至8.0,定容至100ml。
3、1M Tris-HCl(pH 6.8:Tris 12.11 g加ddH2O溶解, 浓盐酸调pH至6.8,定容至100ml。
4、10% SDS:电泳级SDS 10.0 g加ddH2O 68℃助溶,浓盐酸调至pH 7.2,定容至100ml。
5、10电泳缓冲液(pH 8.3:Tris 3.02 g,甘氨酸18.8 g,10% SDS 10ml加ddH2O溶解, 定容至100ml。
6、10%过硫酸铵(AP): 1gAP加ddH2O至10ml。
7、2SDS电泳上样缓冲液:1M Tris-HCl (pH 6.82.5ml,-巯基乙醇1.0ml,SDS 0.6 g,甘油2.0ml,0.1%溴酚兰1.0ml,ddH2O 3.5ml。
8、考马斯亮兰染色液:考马斯亮兰0.25 g,甲醇225ml,冰醋酸 46ml,ddH2O 225ml。
9、脱色液:甲醇、冰醋酸、ddH2O以3∶1∶6配制而成。
二、操作步骤采用垂直式电泳槽装置(一)聚丙烯酰胺凝胶的配制1、分离胶(10%的配制:ddH2O 4.0ml30%储备胶 3.3ml1.5M Tris-HCl2.5ml10% SDS 0.1ml10% AP 0.1ml取1ml上述混合液,加TEMED(N,N,N’,N’-四甲基乙二胺10μl 封底,余加TEMED4μl ,混匀后灌入玻璃板间,以水封顶,注意使液面平。

分子生物学技术在新型脱氮工艺微生物学研究中的应用与进展
全部作者:
郭劲松秦宇方芳杨国红
第1作者单位:
重庆大学3峡库区生态环境教育部重点实验室
论文摘要:
分子生物学技术作为1门现代生物学,弥补了常规微生物学试验的不足,能对环境样品中的微生物进行原位观察,有效、快速、准确地反映环境样品中的微生物信息。
回顾分子生物学技术在SHARON、ANAMMOX、CANON以及OLAND等新型脱氮工艺中微生物学研究方面的应用与进展,该技术为其机理探讨提供了可靠的科学依据,作出了重大贡献。
同时介绍了应用于环境领域中最常用的几种分子生物学技术,并对其在该领域的发展进行了展望。
关键词:
分子生物学,微生物,新型脱氮工艺 (浏览全文)
发表日期:
2006年10月09日
同行评议:
(暂时没有)
综合评价:
(暂时没有)
修改稿:。


医学分子生物学实验技术课程教学大纲
课程负责人:苑辉卿张利宁
课程名称:医学分子生物学实验技术(Experimental Protocols in Medical Molecular Biology)
开课学期:
课程学时:72学时(3学分)
课程教学目标:通过对现代分子生物学实验基本原理和技术的学习,从医学生物学角度培养研究生从事科学研究的能力。
课程内容:
基因克隆:构建一个载体
基因表达:mRNA水平
蛋白质水平
真核基因组分析技术
教学要求:
通过实验原理的讲解、实验示教和实验操作,掌握现代分子生物学实验的基本原理、操作技能和实际应用。
考核方式:实验设计
参考书目:
1. 孙汶生等编.《基因工程学》
2. 金冬雁等译.《分子克隆实验指南》.科学出版社
3. 吴乃虎编.《基因工程原理》
4. 子颖等译.《精编分子生物学实验指南》.科学出版社
5. 卢圣栋等编译.《现代分子生物学实验指南》。



基于免标记发夹型探针和核酸外切酶Ⅲ的荧光信号放大DNA检测郭秋平;赵下雨;谢琴;王柯敏;万俊;袁宝银;谭誉宇【摘要】A novel label-free fluorescence signal amplifying method for DNA detection was developed with high specificity and sensitivity based on a long arm hairpin nucleic acid probe and exonuclease Ⅲ( Exo Ⅲ) . Without the target DNA, the SYBR Green Ⅰ dye could be embedded into the stem of hairpin nucleic acid probe to generate strong fluorescence. While in the presence of target DNA, Exo Ⅲ could catalyze the step-wise removal of mononucleotides from 3’-OH termini of double-stranded DNA with the degradation of the hair-pin probe. After that, the target DNA was released, triggering the next cycle of exonuclease digestion reac-tion. Then the SYBR Green Ⅰ was continuously released, resulting in fluorescence intensity decreased. It rea-lized the label-free signal amplifying detection of DNA. The detection limit of this method was as low as 320 fmol/L. It can be expected to provide a novel, simple, label-free and rapid tool for DNA detection.%设计合成了一种长臂发夹型核酸探针,结合核酸外切酶Ⅲ水解反应建立了一种免标记荧光信号放大高灵敏检测DNA的新方法。


BOPPPS教学模式在分子生物学教学中的探索与实践目录一、内容描述 (2)1.1 研究背景 (3)1.2 研究意义 (5)1.3 文献综述 (6)二、BOPPPS教学模式概述 (7)2.1 BOPPPS教学模式的定义 (8)2.2 BOPPPS教学模式的核心要素 (9)2.3 BOPPPS教学模式的特点 (10)三、BOPPPS教学模式在分子生物学教学中的理论基础 (11)3.1 基于建构主义的学习理论 (13)3.2 基于认知主义的学习理论 (14)3.3 基于掌握学习的学习理论 (15)3.4 基于人本主义的学习理论 (17)四、BOPPPS教学模式在分子生物学教学中的探索与实践 (19)4.1 教学设计 (20)4.1.1 明确教学目标 (21)4.1.2 设计教学策略 (22)4.1.3 选择教学资源 (24)4.1.4 评估教学效果 (25)4.2 教学实施 (27)4.2.1 情境创设 (28)4.2.2 问题引导 (29)4.2.3 小组讨论 (30)4.2.4 实验操作 (31)4.2.5 反思评价 (32)4.3 教学效果评估 (33)4.3.1 学生学习成果评价 (35)4.3.2 教师教学效果评价 (36)4.3.3 课程体系评价 (38)五、结论与展望 (39)5.1 研究结论 (40)5.2 研究不足 (41)5.3 研究展望 (42)一、内容描述BOPPPS教学模式是一种以学生学习为中心的教学方法,广泛应用于各种学科的教学过程。
分子生物学作为生命科学的核心学科,其教学要求高,知识点复杂,对学生的学习能力和理解能力要求严格。
将BOPPPS教学模式引入分子生物学教学中,有助于提升教学质量,促进学生深度学习和全面发展。
本文档主要探讨并实践了BOPPPS教学模式在分子生物学教学中的应用。
分子生物学是生命科学领域的重要分支,涉及生命现象分子层面的研究。
随着科技的进步,分子生物学的研究内容和技术手段日新月异,使得分子生物学的教学面临着巨大的挑战。
学习指南分子生物学实验课程是在分子生物学及基因工程等理论课的基础上,开设的一门综合性兼设计性实验课程。
该的教学内容主要突出实验技术的基础性和实用性,把目前最基本的分子生物学实验技术融入具体的系列实验中形成综合与设计性大实验。
要求学生掌握基因工程的实验技术与方法、实验的设计原理、结果分析方法和分子生物学的基本原理和基本实验操作技术,进一步培养学生的实践能力、独立分析问题、解决问题、动手操作的能力。
该课程126学时,3学分。
教材:魏群主编,《分子生物学实验指导》(第二版),高等教育出版社,2007年。
主要参考书:魏群等译,基因克隆和DNA分析,第五版,高等教育出版社,2007。
吴乃虎编著,《基因工程原理》,第二版,科学出版社,2001年。
黄培堂等译,《分子克隆实验指南》,第三版,科学出版社,2002年。
梁国栋主编,《最新分子生物学实验技术》,科学出版社,2001年。
Molecular biology (5th edition,by Robert F. Weaver)Molecular cloning: a laboratory manual (4th edition, by Michael R. Green and Joseph Sambrook)教学资源:北京师范大学分子生物学实验精品课程教学网络平台。
教学策略与方法建议:本课程采用传统的实验教学方法,结合多媒体、讲座、小组讨论与集体讨论、实验设计、网络教学、教学录像、分子生物学小知识和小技巧介绍等多种教学手段,帮助学生通过本课程的学习全面地掌握分子生物学的基本实验方法与技能以及实验设计的思路,以提高学生从事科学研究的综合素质和实践能力。
要求学生先学习普通化学及实验;有机化学及实验、微生物学等课程。
上课期间注意听讲,做好记录,多动手,善于观察,与组员协作好,合理安排时间。
分子生物学实验教学改革初探盖颖;史玲玲;侯佳音;汪晓峰【摘要】The aim of molecular biology experiments is to train students to research ability, combined with the frontier to help students understand the basic skills. This article explored the teaching reform of molecular biology experiment by rearranging teaching contents, teaching and evaluation methods to improve the students’ learning interest and ability, and achieved good teaching effect.%分子生物学实验是为了配合分子生物学理论课教学、培养学生的实践动手能力和科研能力,并结合学科前沿帮助学生了解实验的基本过程与掌握实验的基本技能。
通过重新安排教学内容、丰富教学手段、完善考核方式三方面的教学改革,有效提高学生的学习兴趣和能力,取得了良好的教学效果。
【期刊名称】《中国现代教育装备》【年(卷),期】2015(000)009【总页数】2页(P46-47)【关键词】分子生物学实验;内容重排;教学方式【作者】盖颖;史玲玲;侯佳音;汪晓峰【作者单位】北京林业大学生物科学与技术学院北京 100083;北京林业大学生物科学与技术学院北京 100083;北京林业大学生物科学与技术学院北京 100083;北京林业大学生物科学与技术学院北京 100083【正文语种】中文分子生物学实验教学是学生理解和掌握分子生物学基本原理的必需环节。
分子生物学技术具有发展快、种类多的特点,本科学生不可能实际操作所有实验。
分子生物学技术实验手册高雄医学院生物学系游仲逸、张永福编着目录实验一:Genomic DNA的制备实验二:Total RNA的制备实验三:聚合脢连锁反应实验四:DNA选殖实验五:基因转型实验六:质体DNA的少量制备和限制脢切割分析实验七:DNA定序分析实验八:北方点墨分析实验九:在大肠杆菌内大量表现重组蛋白实验一:Genomic DNA的制备实验目的纯化genomic DNA是一系列DNA分析,如南方点墨法(Southern blotting),聚合脢连锁反应(polymerase chain reaction, PCR),基因组库(genomic library)之建构及筛选等的前置步骤。
本实验将从你所选定之动物组织或细胞中抽取genomic DNA。
你将从本实验中学习如何自细胞或组织中萃取并纯化genomic DNA。
实验原理本实验是以动物组织或细胞作为genomic DNA的来源。
先以阴离子性清洁剂如sodium dodecyl sulfate (SDS)将细胞打破,并使蛋白质变性,以抑制DNase的活性,也让DNA上的蛋白质解离。
同时加入高浓度的ethylenediaminetetraacetic acid (EDTA)亦可抑制DNase的活性。
另外以RNase除去RNA。
而蛋白质则不以有机溶剂如phenol来萃取掉,而是以盐类沉淀方法去除。
如此可避免使用具毒性之有机溶剂,而DNA亦较不会断裂。
萃取到的genomic DNA以酒精沉淀法浓缩,并回溶于TE缓冲溶液中保留。
最后,测波长260 nm与280 nm之吸光值,以计算所抽出DNA之浓度与纯度。
纯DNA的A260/A280比值约为1.8,比值太低时,表示蛋白质残留太多。
DNA浓度计算方式为OD260=1时,DNA浓度约为50 g/ml。
实验材料∙RBC lysis solution:ammonium chloride, EDTA, sodium bicarbonate∙Cell lysis solution:10 mM Tris-HCl (pH 8.0), 100 mM EDTA, 0.5% SDS∙RNase A solution:4 mg/ml RNase A∙Protein precipitation solution:10 M ammonium acetate∙Isopropanol∙70% ethanol∙TE缓冲溶液:10 mM Tris-HCl, pH 8.0, 1 mM EDTA实验步骤1. 首先要将细胞溶解,不同材料之处理方式略有差别。
作者: 维洞
出版物刊名: 天水师范学院学报
页码: 38-38页
主题词: 分子生物学;实验技术;生物化学;出版社;教程;聚合酶链式反应技术;DNA重组技术;
分离纯化技术;分子杂交技术
摘要:由我校生命科学与化学学院教师安建平、王廷璞合著的《生物化学与分子生物学实验技术教程》一书,已由兰州大学出版社正式出版。
该书分为三篇,第一篇为生物化学和分子生物学实验技术基本理论,包括生物大分子制备技术、分光光度技术、电泳技术、离心技术、层析技术、核酸的分离纯化技术、DNA重组技术、分子杂交技术、聚合酶链式反应技术、DNA序列测定技术、生物芯片技术等十一章。