第六讲 基因芯片数据质量
- 格式:ppt
- 大小:10.77 MB
- 文档页数:94

基因表达谱芯片的数据分析基因芯片数据分析就是对从基因芯片高密度杂交点阵图中提取的杂交点荧光强度信号进行的定量分析,通过有效数据的筛选和相关基因表达谱的聚类,最终整合杂交点的生物学信息,发现基因的表达谱与功能可能存在的联系。
然而每次实验都产生海量数据,如何解读芯片上成千上万个基因点的杂交信息,将无机的信息数据与有机的生命活动联系起来,阐释生命特征和规律以及基因的功能,是生物信息学研究的重要课题[1]。
基因芯片的数据分析方法从机器学习的角度可分为监督分析和非监督分析,假如分类还没有形成,非监督分析和聚类方法是恰当的分析方法;假如分类已经存在,则监督分析和判别方法就比非监督分析和聚类方法更有效率。
根据研究目的的不同[2,3],我们对基因芯片数据分析方法分类如下。
(1)差异基因表达分析:基因芯片可用于监测基因在不同组织样品中的表达差异,例如在正常细胞和肿瘤细胞中;(2)聚类分析:分析基因或样本之间的相互关系,使用的统计方法主要是聚类分析;(3)判别分析:以某些在不同样品中表达差异显著的基因作为模版,通过判别分析就可建立有效的疾病诊断方法。
1 差异基因表达分析(difference expression, DE)对于使用参照实验设计进行的重复实验,可以对2样本的基因表达数据进行差异基因表达分析,具体方法包括倍数分析、t检验、方差分析等。
1.1倍数变化(fold change, FC)倍数分析是最早应用于基因芯片数据分析的方法[4],该方法是通过对基因芯片的ratio值从大到小排序,ratio 是cy3/cy5的比值,又称R/G值。
一般0.5-2.0范围内的基因不存在显著表达差异,该范围之外则认为基因的表达出现显著改变。
由于实验条件的不同,此阈值范围会根据可信区间应有所调整[5,6]。
处理后得到的信息再根据不同要求以各种形式输出,如柱形图、饼形图、点图等。
该方法的优点是需要的芯片少,节约研究成本;缺点是结论过于简单,很难发现更高层次功能的线索;除了有非常显著的倍数变化的基因外,其它变化小的基因的可靠性就值得怀疑了;这种方法对于预实验或实验初筛是可行的[7]。

生物信息学讲义——基因芯片数据分析生物信息学是指运用计算机技术和统计学方法来解析和理解生物领域的大规模生物数据的学科。
基因芯片数据分析是生物信息学研究的一个重要方向,通过对基因芯片数据进行分析,可以揭示基因在生物过程中的功能和调节机制。
本讲义将介绍基因芯片数据的分析方法和应用。
一、基因芯片数据的获取与处理基因芯片是一种用于检测和测量基因表达水平的高通量技术,可以同时检测上千个基因的表达情况。
获取基因芯片数据的第一步是进行基因芯片实验,如DNA芯片实验或RNA芯片实验。
实验得到的数据一般为原始强度值或信号强度值。
接下来,需要对这些原始数据进行预处理,包括背景校正、归一化和过滤噪声等步骤,以消除实验误差和提高数据质量。
二、基因表达分析基因芯片数据的最主要应用之一是进行基因表达分析。
基因表达分析可以揭示在不同条件下基因的表达模式和差异表达基因。
常用的基因表达分析方法包括差异表达分析、聚类分析和差异共表达网络分析等。
差异表达分析常用来寻找在不同条件下表达差异显著的基因,如差异表达基因的筛选和注释;聚类分析可以将表达模式相似的基因分为一组,如聚类分析可以将不同样本中的基因按照表达模式进行分类;差异共表达网络分析可以找到一组在差异表达样本中共同表达的基因,揭示潜在的功能模块。
三、功能富集分析对差异表达基因进行功能富集分析可以帮助我们理解这些基因的生物学功能和参与的生物过程。
功能富集分析可以通过对差异表达基因进行GO(Gene Ontology)注释,找到在特定条件下富集的生物学过程、分子功能和细胞组分等。
另外,功能富集分析还可以进行KEGG(Kyoto Encyclopedia of Genes and Genomes)富集分析,找到差异表达基因在代谢通路和信号传导通路中的富集情况。
四、基因调控网络分析基因调控网络分析可以帮助我们揭示基因间的调控关系和寻找关键调控基因。
基因调控网络是基于差异表达数据构建的,它可以包括转录因子-靶基因调控网络和miRNA-mRNA调控网络等。



基因芯⽚(Affymetrix)分析1:芯⽚质量分析TAIR,NASCarray 和 EBI 都有⼀些公开的免费芯⽚数据可以下载。
本专题使⽤的数据来⾃NASCarray(Exp350),也可以⽤FTP直接下载。
下载其中的CEL⽂件即可(.CEL.gz),下载后解压缩到同⼀⽂件夹内。
该实验有1个对照和3个处理,各有2个重复,共8张芯⽚(8个CEL⽂件)。
为什么要进⾏芯⽚质量分析?不是每个⼈做了实验都会得到⾼质量的数据,花了钱不⼀定就有回报,这道理⼤家都懂。
芯⽚实验有可能失败,失败的原因可能是技术上的(包括⽚⼦本⾝的质量),也可能是实验设计⽅⾯的。
芯⽚质量分析主要检测前者。
1 R软件包安装使⽤到两个软件包:affy,simpleaffy:library(BiocInstaller)biocLite(c("affy", "simpleaffy"))另外还需要两个辅助软件包:tcltk和scales。
tcltk⼀般R基础安装包都已经装有。
install.packages(c("tcltk", "scales"))2 读取CEL⽂件载⼊affy软件包:library(affy)library(tcltk)选取CEL⽂件。
以下两种⽅法任选⼀种即可。
第⼀种⽅法是通过选取⽬录获得某个⽬录内(包括⼦⽬录)的所有cel⽂件:# ⽤choose.dir函数选择⽂件夹dir <- tk_choose.dir(caption = "Select folder")# 列出CEL⽂件,保存到变量cel.files <- list.files(path = dir, pattern = ".+\\.cel$", ignore.case = TRUE,s = TRUE, recursive = TRUE)# 查看⽂件名basename(cel.files)第⼆种⽅法是通过⽂件选取选择⽬录内部分或全部cel⽂件:# 建⽴⽂件过滤器filters <- matrix(c("CEL file", ".[Cc][Ee][Ll]", "All", ".*"), ncol = 2, byrow = T)# 使⽤tk_choose.files函数选择⽂件cel.files <- tk_choose.files(caption = "Select CELs", multi = TRUE, filters = filters,index = 1)# 注意:较⽼版本的tk函数有bug,列表的第⼀个⽂件名可能是错的basename(cel.files)## [1] "NRID9780_Zarka_2-1_MT-0HCA(SOIL)_Rep1_ATH1.CEL"## [2] "NRID9781_Zarka_2-2_MT-0HCB(SOIL)_Rep2_ATH1.CEL"## [3] "NRID9782_Zarka_2-3_MT-1HCA(SOIL)_Rep1_ATH1.CEL"## [4] "NRID9783_Zarka_2-4_MT-1HCB(SOIL)_Rep2_ATH1.CEL"## [5] "NRID9784_Zarka_2-5_MT-24HCA(SOIL)_Rep1_ATH1.CEL"## [6] "NRID9785_Zarka_2-6_MT-24HCB(SOIL)_Rep2_ATH1.CEL"## [7] "NRID9786_Zarka_2-7_MT-7DCA(SOIL)_Rep1_ATH1.CEL"## [8] "NRID9787_Zarka_2-8_MT-7DCB(SOIL)_Rep2_ATH1.CEL"读取CEL⽂件数据使⽤ReadAffy函数,它的参数为:# Not run. 函数说明,请不要运⾏下⾯代码ReadAffy(..., filenames = character(0), widget = getOption("BioC")$affy$use.widgets,compress = getOption("BioC")$affy$compress.cel, celfile.path = NULL, sampleNames = NULL,phenoData = NULL, description = NULL, notes = "", rm.mask = FALSE, rm.outliers = FALSE,rm.extra = FALSE, verbose = FALSE, sd = FALSE, cdfname = NULL)除⽂件名外我们使⽤函数的默认参数读取CEL⽂件:data.raw <- ReadAffy(filenames = cel.files)读⼊芯⽚的默认样品名称是⽂件名,⽤sampleNames函数查看或修改:sampleNames(data.raw)## [1] "NRID9780_Zarka_2-1_MT-0HCA(SOIL)_Rep1_ATH1.CEL"## [2] "NRID9781_Zarka_2-2_MT-0HCB(SOIL)_Rep2_ATH1.CEL"## [3] "NRID9782_Zarka_2-3_MT-1HCA(SOIL)_Rep1_ATH1.CEL"## [4] "NRID9783_Zarka_2-4_MT-1HCB(SOIL)_Rep2_ATH1.CEL"## [5] "NRID9784_Zarka_2-5_MT-24HCA(SOIL)_Rep1_ATH1.CEL"## [6] "NRID9785_Zarka_2-6_MT-24HCB(SOIL)_Rep2_ATH1.CEL"## [7] "NRID9786_Zarka_2-7_MT-7DCA(SOIL)_Rep1_ATH1.CEL"## [8] "NRID9787_Zarka_2-8_MT-7DCB(SOIL)_Rep2_ATH1.CEL"sampleNames(data.raw) <- paste("CHIP", 1:length(cel.files), sep = "-")sampleNames(data.raw)## [1] "CHIP-1" "CHIP-2" "CHIP-3" "CHIP-4" "CHIP-5" "CHIP-6" "CHIP-7" "CHIP-8"3 查看芯⽚的基本信息Phenotypic data数据可能有⽤,可以修改成你需要的内容,⽤pData函数查看和修改:pData(data.raw)## sample## CHIP-1 1## CHIP-2 2## CHIP-3 3## CHIP-4 4## CHIP-5 5## CHIP-6 6## CHIP-7 7## CHIP-8 8pData(data.raw)$Treatment <- gl(2, 1, length = length(cel.files), labels = c("CK","T"))pData(data.raw)## sample Treatment## CHIP-1 1 CK## CHIP-2 2 T## CHIP-3 3 CK## CHIP-4 4 T## CHIP-5 5 CK## CHIP-6 6 T## CHIP-7 7 CK## CHIP-8 8 TPM和MM查看:# Perfect-match probespm.data <- pm(data.raw)head(pm.data)## CHIP-1 CHIP-2 CHIP-3 CHIP-4 CHIP-5 CHIP-6 CHIP-7 CHIP-8 ## 501131 127.0 166.3 112.0 139.8 111.3 85.5 126.3 102.8## 251604 118.5 105.0 82.0 101.5 94.0 81.3 103.8 103.0## 261891 117.0 90.5 113.0 101.8 99.3 107.0 85.3 85.3## 230387 140.5 113.5 94.8 137.5 117.3 112.5 124.3 114.0## 217334 227.3 192.5 174.0 192.8 162.3 163.3 235.0 195.8## 451116 135.0 122.0 86.8 93.3 83.8 87.3 97.3 83.5# Mis-match probesmm.data <- mm(data.raw)head(mm.data)## CHIP-1 CHIP-2 CHIP-3 CHIP-4 CHIP-5 CHIP-6 CHIP-7 CHIP-8 ## 501843 89.0 88.0 80.5 91.0 77.0 75.0 79.0 72.0## 252316 134.3 77.3 77.0 107.8 98.5 75.0 99.5 71.3## 262603 119.3 90.5 82.0 86.3 93.0 89.3 94.5 83.8## 231099 123.5 94.5 76.5 95.0 89.3 87.8 95.5 91.5## 218046 110.3 93.0 74.8 100.5 86.0 89.5 104.5 102.3## 451828 127.5 77.0 80.3 94.5 72.3 79.0 86.3 67.84 显⽰芯⽚扫描图像(灰度)# 芯⽚数量n.cel <- length(cel.files)par(mfrow = c(ceiling(n.cel/2), 2))par(mar = c(0.5, 0.5, 2, 0.5))# 设置调⾊板颜⾊为灰度pallette.gray <- c(rep(gray(0:10/10), times = seq(1, 41, by = 4)))# 通过for循环逐个作图for (i in 1:n.cel) image(data.raw[, i], col = pallette.gray)如果芯⽚图像有斑块现象就很可能是坏⽚。

基因芯片数据处理流程与分析介绍关键词:基因芯片数据处理当人类基因体定序计划的重要里程碑完成之后,生命科学正式迈入了一个后基因体时代,基因芯片(microarray) 的出现让研究人员得以宏观的视野来探讨分子机转。
不过分析是相当复杂的学问,正因为基因芯片成千上万的信息使得分析数据量庞大,更需要应用到生物统计与生物信息相关软件的协助。
要取得一完整的数据结果,除了前端的实验设计与操作的无暇外,如何以精确的分析取得可信数据,运筹帷幄于方寸之间,更是画龙点睛的关键。
基因芯片的应用基因芯片可以同时针对生物体内数以千计的基因进行表现量分析,对于科学研究者而言,不论是细胞的生命周期、生化调控路径、蛋白质交互作用关系等等研究,或是药物研发中对于药物作用目标基因的筛选,到临床的疾病诊断预测,都为基因芯片可以发挥功用的范畴。
基因表现图谱抓取了时间点当下所有的动态基因表现情形,将所有的探针所代表的基因与荧光强度转换成基本数据(raw data) 后,仿如尚未解密前的达文西密码,隐藏的奥秘由丝丝的线索串联绵延,有待专家抽丝剥茧,如剥洋葱般从外而内层层解析出数千数万数据下的隐晦含义。
要获得有意义的分析结果,恐怕不能如泼墨画般洒脱随兴所致。
从raw data 取得后,需要一连贯的分析流程(图一),经过许多统计方法,才能条清理明的将raw data 整理出一初步的分析数据,当处理到取得实验组除以对照组的对数值后(log2 ratio),大约完成初步的统计工作,可进展到下一步的进阶分析阶段。
图一、整体分析流程。
基本上raw data 取得后,将经过从最上到下的一连串分析流程。
(1) Rosetta 软件会透过统计的model,给予不同的权重来评估数据的可信度,譬如一些实验操作的误差或是样品制备与处理上的瑕疵等,可已经过Rosetta error model 的修正而提高数据的可信值;(2) 移除重复出现的探针数据;(3) 移除flagged 数据,并以中位数对荧光强度的数据进行标准化(Normalized) 的校正;(4) Pearson correlation coefficient (得到R 值) 目的在比较技术性重复下的相似性,R 值越高表示两芯片结果越近似。

生物信息学讲义——基因芯片数据分析资料基因芯片是一种高通量的技术,可以用于同时检测和量化数以千计的基因在一个样本中的表达水平。
通过分析基因芯片数据,我们可以获得大量的基因表达信息,并进一步了解基因在不同条件和疾病状态下的调控和功能。
下面是一份关于基因芯片数据分析的讲义。
一、基因芯片数据的处理与预处理1.数据获取与质控-从基因芯片实验中获取原始数据(CEL文件)。
-进行质控,包括检查芯片质量、样本质量和数据质量。
2.数据预处理-背景校正:去除背景信号,减小非特异性杂音。
-样本标准化:对样本间进行标准化处理,消除技术变异和样本间差异。
-基因过滤:去除低表达和不变的基因,减少多重检验问题。
二、差异基因分析1.统计分析-基于统计学的差异表达分析方法,如t检验、方差分析(ANOVA)等。
-根据差异分析结果,获取差异表达的基因列表。
2.功能注释与生物学解释-对差异表达的基因进行功能注释,包括富集分析、通路分析和基因功能类别分析等。
-通过生物学数据库查询和文献阅读,解释差异表达基因的生物学意义和可能的调控机制。
三、基因共表达网络分析1.相关性分析-计算基因间的相关系数,筛选出相关性较高的基因对。
-构建基因共表达网络,通过网络可视化方式展示基因间的关系。
2.模块发现和功能注释-使用聚类算法将基因分组成不同的模块,每个模块表示一组具有相似表达模式的基因。
-对每个模块进行功能注释,了解模块内基因的共同功能或通路。
四、基因云图和热图分析1.基因云图-使用基因注释信息和基因表达水平,绘制基因表达的云图。
-通过颜色和大小表示基因的表达水平、功能注释等信息。
2.热图分析-根据基因表达水平计算基因间的相似性,将相似性转换为颜色,绘制热图。
-热图可用于显示基因表达模式的相似性和差异。
五、整合分析与生物信息学工具1.基因集富集分析-将差异表达的基因列表输入基因富集分析工具,寻找与特定通路、功能或疾病相关的基因集。
2.数据可视化工具- 使用生物信息学工具和软件,如R、Bioconductor、Cytoscape等,进行数据可视化和交互式分析。



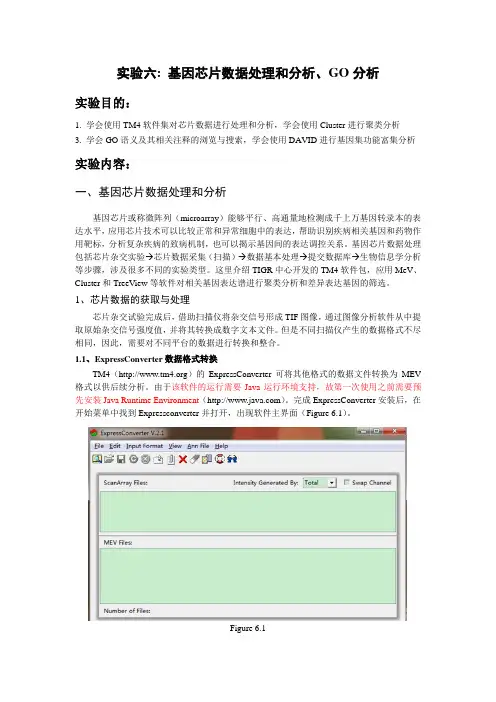
实验6基因芯片数据处理分析与GO分析实验背景:基因芯片技术是通过检测靶基因在不同样本中的表达量差异,并分析其生物信息学特性,来揭示基因调控网络与疾病发生发展的过程的一种高通量技术。
基因芯片数据处理和分析是基因芯片研究的关键步骤之一、通过对基因芯片数据进行预处理、差异分析、聚类分析等,可以获得与研究目标相关的基因列表,并进一步进行GO(Gene Ontology)的功能富集分析,揭示差异表达基因的功能特性。
实验目的:通过基因芯片数据处理分析和GO功能富集分析,获得与研究目标相关的差异表达基因,并揭示其在生物学功能、分子过程和细胞组分方面的富集情况,为后续的生物学实验和机制研究提供理论依据。
实验步骤:1.基因芯片数据的预处理:包括数据导入、数据清洗、标准化和基因注释等。
首先,将基因芯片数据导入到数据分析软件中,然后针对数据质量进行清洗,剔除异常值和低质量的基因。
接下来,对基因表达谱数据进行归一化处理,保证不同芯片之间的数据可比性。
最后,对基因进行注释,将基因名与其对应的功能注释进行关联。
2.差异分析:通过比较不同组别之间的基因表达差异,筛选出差异表达基因。
差异分析方法包括t检验、方差分析等。
根据统计学中的显著性水平,设定p值的阈值,将差异表达基因筛选出来。
3.聚类分析:将差异表达基因按照其表达谱进行聚类分析,可将具有相似表达模式的基因聚集在一起。
常用的聚类方法包括层次聚类和K均值聚类等。
实验结果与分析:通过基因芯片数据处理和分析,我们得到了与研究目标相关的差异表达基因。
结合GO分析的结果,我们可以进一步了解这些差异表达基因在生物学功能、分子过程和细胞组分方面的富集情况。
例如,在生物学过程方面,我们可以得知这些基因是否与细胞增殖、凋亡、信号传导等生物学过程相关;在分子功能方面,我们可以了解这些基因是否具有催化活性、结合能力等分子功能特性;在细胞组分方面,我们可以了解这些基因在细胞核、细胞质、细胞膜等细胞组分的分布情况。
基因芯片及其数据分析基因芯片(gene chip)是一种高通量的基因表达分析工具,也被称为基因表达芯片或基因表达板。
它可以同时检测和分析数以万计的基因,以了解基因在细胞或组织中的表达情况。
基因芯片的制备过程包括两个主要步骤:生物实验和芯片制造。
首先,采集感兴趣的生物样本,例如人体组织或细胞。
然后,从这些样本中提取RNA或DNA,将其转录为互补DNA(cDNA),并进行标记。
接着,将这些标记的cDNA片段加入芯片上的特定位置,称为探针。
这些探针是经过设计和合成的特定序列,可以与目标基因或RNA分子特异性结合。
在数据分析方面,基因芯片的分析流程包括数据预处理、差异分析和功能注释等步骤。
数据预处理主要是对原始芯片数据进行质量控制、标准化和归一化等处理,以消除技术偏差和样本间的差异。
差异分析是通过比较不同处理组的表达谱,找到差异表达的基因或通路,从而揭示不同条件下基因表达的变化。
功能注释是将识别出的差异基因进行生物学功能描述,包括基因本体论(Gene Ontology)、通路富集分析等,从而理解这些基因的生物学意义和参与的生物过程。
基因芯片的应用非常广泛。
在生物医学研究中,它常被用于筛选差异表达的基因,发现与特定疾病相关的生物标志物,探寻病理生理过程中的致病机制等。
例如,通过对癌症患者和正常人组织样本的基因芯片分析,可以发现不同癌症类型的分子标记物,用于早期诊断和治疗监测。
此外,基因芯片还被广泛应用于农业、食品安全、环境监测等领域,用于研究植物生长发育、种子品质、环境胁迫等相关问题。
然而,基因芯片的数据分析也面临一些挑战。
首先,由于芯片技术的快速发展,数据量急剧增加。
如何高效地处理和存储这些庞大的数据成为一个问题。
其次,芯片技术本身存在一定的误差和噪音,如何准确地分析和解释数据结果也是一个难题。
此外,芯片分析常常需要结合其他实验验证结果,以确认差异表达基因的生物学意义。
总的来说,基因芯片及其数据分析是现代生物学和医学研究中的重要工具。