生物信息学及其软件平台
- 格式:ppt
- 大小:1.03 MB
- 文档页数:48


生物信息学软件的使用教程与数据分析生物信息学是一门结合生物学和计算机科学的学科,通过利用计算机科学和统计学的方法来研究生物学中的大规模生物分子数据。
在生物研究中,大量的生物信息数据被产生,如基因组测序数据、蛋白质结构数据、转录组数据等,这些数据的分析对于理解生物过程和疾病发生机制至关重要。
生物信息学软件是专门用于处理和分析这些生物信息数据的工具。
本文将介绍一些常见的生物信息学软件的使用教程和数据分析方法。
1. BLAST(Basic Local Alignment Search Tool):BLAST是最常用的序列比对工具之一,用于在数据库中寻找类似序列或通过序列相似性比对两个或多个序列。
BLAST可以用于查找一个给定的序列是否存在于一个已知的数据库中,也可用于快速比较两个序列的相似性,并寻找具有高度相似性的区域。
在使用BLAST时,首先需要选择合适的数据库,然后输入待比对的序列,设置相似性阈值和其他参数,最后运行BLAST程序并分析结果。
2. NCBI(National Center for Biotechnology Information)工具:NCBI提供了许多生物信息学工具,如BLAST、Entrez等。
Entrez是一个可检索多种生物信息学数据库的工具,包括GenBank(存储核酸序列)、PubMed(存储科学文献摘要与索引)、Protein(蛋白质序列数据库)等。
通过使用NCBI提供的工具,可以比对和分析大量的生物序列和相关的生物信息。
使用NCBI工具时,可以通过访问NCBI网站或使用命令行工具来查询和分析数据。
3. R和Bioconductor:R是一种用于统计计算和数据可视化的自由软件环境,而Bioconductor是一个在R环境中为生物学研究提供的开源生物信息学软件包。
R和Bioconductor提供了丰富的统计和生物信息学分析方法,可用于分析基因表达数据、基因组测序数据、蛋白质结构数据等。

生物大数据分析的软件和工具随着生物技术的迅速发展,生物大数据的产生呈现出爆炸式增长的趋势。
然而,要从这些浩瀚的数据中提取有效的信息并加以解读,需要大量的计算和分析工作。
这就需要生物大数据分析的软件和工具来对数据进行处理和分析。
本文将介绍一些主流的生物大数据分析软件和工具,以便选择出最适合自己实验室的软件和工具。
1. BLASTBLAST(Basic Local Alignment Search Tool)是一种能够在数据库中搜索和比对序列的工具,是生物大数据分析中最为基础和常见的软件之一。
该软件通过比较存储在NCBI数据库中十分庞大的蛋白质或核酸序列数据库,查找出目标序列在数据库中的位置,并将它们按相似性排列。
BLAST算法拥有高度的适应性以及灵活性,不仅可以比对蛋白质序列,还可以比对基因组序列、转录组数据、蛋白质结构等。
其使用简单且运行速度快,是生物学领域的所有人在研究中必备的分析工具之一。
2. BowtieBowtie是一种基于快速算法的序列比对工具,能够高效地比对大规模的、二代测序数据。
如今,像Illumina和Solexa等技术,都可以生成大量的测序数据。
在这种情况下,Bowtie通过使用索引和FM索引的算法,实现了高速比对操作。
它可以用来定位基因组中的SNP、RNA编码区、结构变异等,具有很强的通用性,是生物信息学领域中的重要工具之一。
3. CufflinksCufflinks是一款常用于基因表达分析的工具,主要用于定量RNA测序的数据分析。
它是用来识别甲基化基因包、识别单基因外显子模式以及补全未知转录本等诸多生物信息学任务。
而且它在RNA测序方面使用了一种非常独特的分析策略,因此也被称为“近似最大似然”方法。
这种技术可以明确地表达不同基因内RNA 的转录变体和各种表达模式,能够快速、准确地解析表观转录组问题。
Cufflinks功能丰富、使用灵活且易于学习,是RNA测序数据分析的一种主流工具。

生物信息学网站分子生物学数据库综合目录1. SRS序列查询系统(分子生物学数据库网络浏览器) http://www.embl-heidelberg.ed/srs5/2. 分子生物学数据库及服务器概览/people/pkarp/mimbd/rsmith.html3. BioMedNet图书馆4. DBGET数据库链接http://www.genome.ad.jp/dbget/dbget.links.html5. 哈佛基因组研究数据库与精选服务器6. 约翰. 霍普金斯大学(Johns Hopkins University) OWL网络服器/Dan/proteins/owl.html7. 生物网络服务器索引,USCS /network/science/biology/index.html8. 分子生物学数据库列表(LiMB) gopher:///11/molbio/other9. 病毒学的WWW服务器,UW-Madison /Welcome.html10. UK MRC 人类基组图谱计划研究中心/11. 生物学家和生物化学家的WWW资源http://www.yk.rim.pr.jp/~aisoai/index.html12. 其他生物网络服务器的链接/biolinks.html13. 分子模型服务器与数据库/lap/rsccom/dab/ind006links.html14. EMBO实际结构数据库http://xray.bmc.uu.se/embo/structdb/links.html15. 蛋白质科学家的网络资源/protein/ProSciDocs/WWWResources.html16. ExPASy分子生物学服务器http://expasy.hcuge.ch/cgi-bin/listdoc17. 抗体研究网页18. 生物信息网址http://biochem.kaist.ac.kr/bioinformatics.html19. 乔治.梅森大学(George Mason University)的生物信息学与计算分子生物学专业/~michaels/Bioinformatics/20. INFOBIOGEN数据库目录biogen.fr/services/dbcat/21. 国家生物技术信息研究室/data/data.html22. 人类基因组计划情报/TechResources/Human_Genome23. 生物学软件及数据库档案/Dan/software/biol-links.html24. 蛋白质组研究:功能基因组学的新前沿(著作目录) http://expasy.hcuge.ch/ch2d/LivreTOC.html序列与结构数据库一.主要的公共序列数据库1. EMBL WWW服务器http://www.EMBL-heidelberg.ed/Services/index.html2. Genbank 数据库查询形式(得到Genbank的一个记录) /genbank/query_form.html3. 蛋白质结构数据库WWW服务器(得到一PDB结构) 4. 欧洲生物信息学研究中心(EBI) /5. EBI产业支持/6. SWISS-PROT(蛋白质序列库) http://www.expasy.ch/sprot/sprot-top.html7. 大分子结构数据库/cgi-bin/membersl/shwtoc.pl?J:mms8. Molecules R Us(搜索及观察一蛋白质分子) /modeling/net_services.html9. PIR国际蛋白质序列数据库/Dan/proteins/pir.html10. SCOP(蛋白质的结构分类),MRC /scop/data/scop.l.html11. 洛斯阿拉莫斯的HIV分子免疫数据库/immuno/index.html12. TIGR数据库/tdb/tdb.html13. NCBI WWW Entrez浏览器/Entrez/index.html14. 剑桥结构数据库(小分子有机的及有机金属的结晶结构) 15. 基因本体论坛/GO/二. 专业数据库1. ANU生物信息学超媒体服务(病毒数据库、分类及病毒的命名法) .au/2. O-GL YCBASE(O联糖基化蛋白质的修订数据库) http://www.cbs.dtu.dk/OGLYCBASE/cbsoglycbase.html3. 基因组序列数据序(GSDB)(已注释的DNA序列的关系数据序) 4. EBI蛋白质拓扑图/tops/Serverintermed.html5. 酶及新陈代谢途径数据库(EMP) /6. 大肠杆菌数据库收集(ECDC)(大肠杆菌K12的DNA序列汇编) http://susi.bio.uni-giessen.de/ecdc.html7. EcoCyc(大肠杆菌基因及其新陈代谢的百科全书) /ecocyc/ecocyc.html8. Eddy实验室的snoRNA数据库/snoRNAdb/9. GenproEc(大肠杆菌基因及蛋白质) /html/ecoli.html10. NRSub(枯草芽胞杆菌的非冗余数据库) http://pbil.univ-lyonl.fr/nrsub/nrsub.html11. YPD(酿酒酵母蛋白质) /YPDhome.html12. 酵母基因组数据库/Saccharomyces/13. LISTA、LISTA-HOP及LISTA-HON(酵母同源数据库汇编) /14. MPDB(分子探针数据库) http://www.biotech.est.unige.it/interlab/mpdb.html15. tRNA序列及tRNA基因序列汇编http://www.uni-bayreuth.de/departments/biochemie/trna/index/html16. 贝勒医学院(Baylor College of Medicine)的小RNA数据库/dbs/SRPDB/SRPDB.html17. SRPDB(信号识别粒子数据库) /dbs/SRPDB/SRPDB.html18. RDP(核糖体数据库计划) /19. 小核糖体亚蛋白RNA结构http://rrna.uia.ac.be/ssu/index.html20. 大核糖体亚蛋白RNA结构http://rrna.uia.ac.be/lsu/index.html21. RNA修饰数据库/RNAmods/22. 16SMDB及23SMDB(16S和23S核糖体RNA突变数据库)/Departments/Biology/Databases/RNA.html23. SWISS-2DPAGE(二维凝胶电泳数据库) http://expasy.hcuge.ch/ch2d/ch2d-top.html24. PRINTS /bsm/dbbrowser/PRINTS/PRINTS.html25. KabatMan(抗体结构及序列信息数据库) /abs26. ALIGN(蛋白质序列比对一览) /bsm/dbbrowser/ALIGN/ALIGN.html27. CATH(蛋白质结构分类系统) /bsm/cath28. ProDom(蛋白质域数据库) http://protein.toulouse.inra.fr/29. Blocks数据库(蛋白质分类系统) /30. HSSP(按同源性导出的蛋白质二级结构数据库) http://www.sander.embl-heidelberg.de/hssp/31. FSSP(基于结构比对的蛋白质折叠分类) /dali/fssp/fssp.html32. SBASE蛋白质域(已注释的蛋白质序列片断) http://www.icgeb.trieste.it/~sbasessrv/33. TransTerm(翻译控制信号数据库) /Transterm.html34. GRBase(参与基因调控的蛋白质的相关信息数据库) /~regulate/trevgrb.html35. REBASE(限制性内切酶和甲基化酶数据库) /rebase/36. RNaseP数据库/RNaseP/home.html37. REGULONDB(大肠杆菌转录调控数据库) http://www.cifn.unam.mx/Computational_Biology/regulondb/38. TRANSFAC(转录因子及其DNA结合位点数据库) http://transfac.gbf.de/39. MHCPEP(MHC结合肽数据库) .au/mhcpep/40. ATCC(美国菌种保藏中心) /41. 高度保守的核蛋白序列的组蛋白序列数据库/Baxevani/HISTONES42. 3Dee(蛋白质结构域定义数据库) /servers/3Dee.html43. InterPro(蛋白质域以及功能位点的完整资源) /interpro/序列相似性搜索1. EBI序列相似性研究网页/searches/searches.html2. NCBI: BLAST注释/BLAST3. EMBL的BLITZ ULTRA快速搜索/searches/blitz_input.html4. EMBL WWW服务器http://www.embl-heidelberg.de/Services/index.html#55. 蛋白质或核苷酸的模式浏览/compbio/PatScan/HTML/patscan.html6. MEME(蛋白质超二级结构模体发现与研究) /meme/website7. CoreSearch(DNA序列保守元件的识别) http://www.gsf.de/biodv/coresearch.html8. PRINTS/PROSIT浏览(搜索motif数据库) /cgi-bin/attwood/SearchprintsForm.pl9. 苏黎世ETH服务器的DARWIN系统http://cbrg.inf.ethz.ch/10. 利用动态规划找出序列相似性的Pima IIhttp://bmerc-www.bu.ede/protein-seq/pimaII-new.html11. 利用与模式库进行哈希码(hashcode)比较找到序列相似性的DashPat /protein-seq/dashPat-new.html12. PROPSEARCH(基于氨基酸组成的搜索) http://www.embl-heidelberg.de/aaa.html13. 序列搜索协议(集成模式搜索) /bsm/dbbrowser/protocol.html14. ProtoMap(SEISS-PROT中所有蛋白质的自动层次分类) http://www.protomap.cs.huji.ac.il/15. GenQuest(利用Fasta、Blast、Smith-Waterman方法在任意数据库中搜索) http://www.gdb.rog/Dan/gq/gq.form.html16. SSearch(对特定数据库的搜索) http://watson.genes.nig.ac.jp/homology/ssearch-e_help.html17. Peer Bork搜索列表(motif/模式序列谱搜索) http://www.embl-heidelberg.de/~bork/pattern.html18. PROSITE数据库搜索(搜索序列的功能位点) /searches/prosite.html19. PROWL(Skirball研究中心的蛋白质信息检索) /index.html序列和结构的两两比对1. 蛋白质两两比对(SIM) http://expasy.hcuge.ch/sprot/sim-prot.html2. LALNVIEW比对可视化观察程序ftp://expasy.hcuge.ch/pub/lalnview3. BCM搜索装置(两两序列比对) /seq-search/alignment.html4. DALI蛋白质三维结构比较/dali/5. DIALIGN(无间隙罚分的比对程序) http://www.gsf.de/biodv/dialign/html多重序列比对及系统进行树1. ClustalW(BCM的多重序列比对) /multi-align/multi-align.html2. PHYLIP(推测系统进行树的程序) /phylip.html3. 其它系统进行树程序,PHYLIP文档的汇编http://expasy.hcuge.ch/info/phylogeny.html4. 系统进行树分析程序(生命树列表) /tree/programs/programs.html5. 遗传分类学软件(Willi hennig协会提供的列表) /education.html6. 用于多重序列比对的BCM搜索装置/multi-align/multi-align.html7. AMAS(分析多重序列比对中的序列) /servers/amas_server.html8. 维也纳RNA二级结构软件包http://www.tbi.univie.ac.at/~ivo/RNA/四. 有代表性的预测服务器1. PHD蛋白质预测服务器,用于二级结构、水溶性以及跨膜片断的预测http://www.embl-heidelberg.de/predictprotein/predictprotein.html2. PhdThreader(利用逆折叠方法预测、识别折叠类) http://www.embl-heidelberg.de/predictprotein/phd_help.html3. PSIpred(蛋白质结构预测服务器) /psipred4. THREADER(戴维. 琼斯) /~jones/threader.html5. TMHMM(跨膜螺旋蛋白的预测) http://www.cbs.dtu.dk/services/TMHMM/6. 蛋白质结构分析,BMERC /protein-seq/protein-struct.html7. 蛋白质域和折叠预测的提交表http://genome.dkfz-heidelberg.de/nnga/def-query.html8. NNSSP(利用最近相邻法预测蛋白质的二级结构) /pss/pss.html9. Swiss-Model(基于知识的蛋白质自动同源建模服务器) http://www.expasy.ch/swissmod/SWISS-MODEL.html10. SSPRED(用多重序列比对进行二级结构预测) /jong/predict/sspred.html11. 法国IBCP的SOPM(自寻优化预测方法、二级结构) http://pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopm.html12. TMAP(蛋白质跨膜片断的预测服务) http://www.embl-heidelberg.de/tmap/tmap_info.html13. TMpred(跨膜区域和方向的预测) /software/TMPRED_form.html14. MultPredict(多重序列比对的序列的二级结构) /zpred.html15. BCM搜索装置(蛋白质二级结构预测) /seq-search/struc-predict.html16. COILS(蛋白质的卷曲螺旋区域预测) /software/coils/COILS_doc.html17. Coiled Coils(卷曲螺旋) /depts/biol/units/coils/coilcoil.html18. Paircoil(氨基酸序列中的卷曲螺旋定位) /bab/webcoil.html19. PREDATOR(由单序列预测蛋白质二级结构) http://www.embl-heidelberg.de/argos/predator/predator_info.html20. EV A(蛋白质结构预测服务器的自动评估) /eva/五. 其他预测服务器1. SignalP (革兰氏阳性菌、革兰氏阴性菌和真核生物蛋白质的信号肽及剪切位点) http://www.cbs.dtu.dk/services/SignalP/2. PEDANT(蛋白质提取、描述及分析工具) http://pedant.mips.biochem.mpg.de/六. 分子生物学软件链接1. 生物信息学可视化工具/alan/VisSupp/2. EBI分子生物学软件档案/software/software.html3. BioCatalog /biocat/e-mail_Server_ANAL YSIS.html4. 生物学软件和数据库档案/Dan/softsearch/biol-links.html5. UC Santa Cruz的序列保守性HMM的SAM软件/research/compbio/sam.html七. 网上博士课程1. 生物计算课程资源列表:课程大纲http://www.techfak.uni-bielefeld.de/bcd/Curric/syllabi.html2. 生物序列分析和蛋白质建模的Ph.D课程http://www.cbs.dtu.dk/phdcourse/programme.html3. 分子科学虚拟学校/vsms/sbdd/4. EMBnet 生物计算指南http://biobase.dk/Embnetut/Universl/embnettu.html5. 蛋白质结构的合作课程/PPS/index.html6. 自然科学GNA虚拟学校http://www.techfak.uni-bielefeld.de/bcd/Vsns/index.html7. 分子生物学算法/education/courses/590bi。


生物信息学软件
生物信息学软件是一类专门用于处理、分析和解释生物学
数据的软件工具。
这些软件通常用于基因组学、蛋白质组学、转录组学和代谢组学研究中。
以下是一些常用的生物
信息学软件:
1. BLAST:用于快速在数据库中搜索相似序列的工具,对
于序列比对和亲缘关系分析非常有用。
2. ClustalW:用于多序列比对的软件,可以比较多个序列
之间的相似性和差异。
3. GROMACS:用于分子动力学模拟和分子力学计算的软件,可以模拟蛋白质、核酸等生物分子的结构和动态行为。
4. PHYLIP:用于构建进化树和系统发育分析的软件,可以根据序列的差异性推断出生物物种之间的进化关系。
5. R:一种统计软件,提供了广泛的生物信息学功能和数据处理方法。
6. Cytoscape:用于网络分析和可视化的软件,可以分析和可视化基因调控网络、蛋白质相互作用网络等。
7. NCBI工具包:由美国国家生物技术信息中心(NCBI)开发的一组工具,包括BLAST、Entrez等,用于生物序列和文献检索。
8. Galaxy:一个基于云计算的生物信息学分析平台,提供了大量的工具和工作流,方便生物学家进行数据分析和可视化。
9. MetaboAnalyst:用于代谢组学数据分析的软件,可以进行代谢物注释、统计分析、通路分析等。
10. Geneious:用于序列分析和比对、系统发育分析、基因预测等多种生物信息学任务的集成软件。
以上只是一小部分常用的生物信息学软件,随着科学研究的进展,新的软件工具不断涌现。


生物信息学分析平台的使用教程与数据挖掘生物信息学是将信息科学和生物学相结合的交叉学科领域,它利用计算机和统计学等工具来管理、解释和分析生物学数据。
生物信息学分析平台是为帮助生物学家处理和分析大规模生物学数据而设计的软件工具。
本文将介绍生物信息学分析平台的使用教程,并探讨如何利用数据挖掘技术在生物学研究中发现新的知识。
一、生物信息学分析平台的基本功能生物信息学分析平台通常提供一系列工具和算法,用于处理和分析生物学数据,包括测序数据、基因表达数据、蛋白质结构数据等。
常见的生物信息学分析平台有NCBI、UCSC、Ensembl等。
1. 数据查询和检索:生物信息学分析平台允许用户通过关键词、ID号或其他属性来查询和检索生物学数据库中的数据。
用户可以根据自己的研究目的来选择合适的数据库,如基因组数据库、蛋白质数据库等。
2. 数据处理和分析:生物信息学分析平台提供各种工具和算法,用于处理和分析生物学数据。
常见的功能包括质量控制、序列比对、基因表达定量、蛋白质互作预测等。
用户可以根据自己的研究问题选择合适的工具和算法进行分析。
3. 数据可视化和结果解释:生物信息学分析平台通常提供数据可视化工具,用于将分析结果以图表或图形的形式展示出来。
这有助于用户理解和解释分析结果,并从中提取有意义的信息。
二、生物信息学分析平台的使用教程以下是一般性的生物信息学分析平台使用教程,具体操作可能因平台而异,仅供参考。
1. 注册账户和登录平台:生物信息学分析平台通常需要用户注册账户后进行登录,以便保存用户的分析结果和设置。
2. 数据查询和检索:在平台的搜索栏中输入关键词、ID号或其他属性,选择合适的数据库,点击搜索按钮进行查询和检索。
3. 数据下载和导入:根据查询结果选择需要的数据,并下载到本地计算机。
下载的文件可能是文本文件、FASTA格式文件等。
将数据导入到生物信息学分析平台中,准备进行后续的数据处理和分析。
4. 数据质量控制:对导入的数据进行质量控制,去除低质量的序列或数据点。
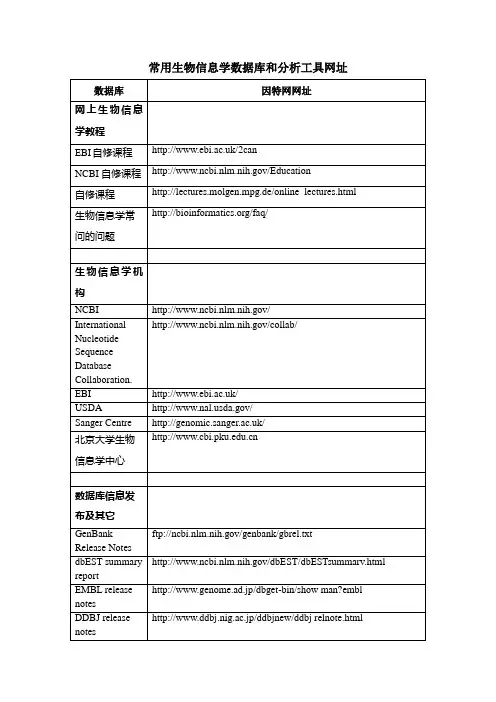

常用生物数据分析软件在生物科学领域中,数据分析是一项重要的任务。
随着技术的进步,生物学研究的数据规模不断扩大,例如基因组测序数据、蛋白质互作数据、表达谱数据等。
为了处理和分析这些大规模的生物学数据,许多生物数据分析软件被开发出来。
本文将介绍一些常用的生物数据分析软件。
1.R:R是一个流行的统计分析和图形化软件,也是生物学家常用的数据分析工具之一、R具有强大的数据分析功能和广泛的统计工具包,适用于各种生物学数据分析任务,例如基因表达分析、蛋白质结构预测、基因组测序等。
2. Python:Python是一种通用的编程语言,也被广泛用于生物数据分析。
Python拥有丰富的生物信息学工具包,例如Biopython,可用于处理和分析蛋白质序列和结构、基因组测序数据等。
Python还具有强大的数据处理和可视化能力,适用于各种生物学数据分析任务。
3. NCBI工具:NCBI(美国国家生物技术信息中心)提供一系列在线工具用于生物数据分析。
NCBI提供的工具包括BLAST用于序列比对、Entrez用于文献检索、GenBank用于基因组测序数据等。
这些工具对于进行一些常见的生物数据分析任务非常有用。
4. Bioconductor:Bioconductor是一个用于生物数据分析的开源软件包集合。
Bioconductor提供了许多R语言工具包,包括用于基因表达分析、蛋白质互作网络分析等。
这些工具包提供了丰富的生物学统计学和机器学习算法,可以帮助研究人员进行高质量的生物数据分析。
5. Cytoscape:Cytoscape是一个用于生物网络分析和可视化的软件。
它可以用来分析和可视化蛋白质互作网络、基因调控网络等。
Cytoscape提供了许多插件和工具,使得生物网络分析更加方便和高效。
6. Galaxy:Galaxy是一个用于生物数据分析的在线平台。
它提供了许多常用的生物数据分析工具,并提供了一个用户友好的界面,使得生物学家可以无需编程就能进行复杂的生物数据分析任务。

生物大数据分析的常用工具和软件介绍生物大数据的快速发展和应用需求推动了生物信息学工具和软件的不断发展。
这些工具和软件提供了一系列功能,如序列分析、基因表达分析、蛋白质结构预测、功能注释等,帮助研究人员从大量的生物数据中提取有意义的信息。
下面将介绍一些常用的生物大数据分析工具和软件。
1. BLAST(Basic Local Alignment Search Tool)BLAST是最常用的序列比对工具之一,用于比对一条查询序列与已知序列数据库中的序列。
通过比对确定序列之间的相似性,从而推断其功能和结构。
BLAST具有快速、准确、用户友好的特点,适用于DNA、RNA和蛋白质序列的比对。
2. GalaxyGalaxy是一个基于Web的开源平台,提供了许多生物信息学工具和软件的集成。
它提供了一个易于使用的界面,使得用户可以通过拖放操作完成复杂的数据分析流程。
Galaxy支持不同类型的数据分析,包括序列比对、组装、注释、表达分析等。
3. R包R是一个功能强大的统计语言和环境,用于数据分析和可视化。
R包提供了许多用于生物数据分析的扩展功能。
例如,"Bioconductor"是一个R软件包,提供了丰富的生物数据分析方法和工具,包括基因表达分析、序列分析、蛋白质分析等。
4. GATK(Genome Analysis Toolkit)GATK是一个用于基因组数据分析的软件包,主要用于研究DNA变异。
它包含了各种工具和算法,用于SNP检测、基因型调用、变异注释等。
GATK还在处理复杂变异(如复杂多态位点)和群体遗传学分析方面具有独特的优势。
5. CytoscapeCytoscape是一个用于生物网络分析和可视化的开源平台。
它可以用于可视化和分析蛋白质-蛋白质相互作用网络、基因共表达网络、代谢网络等。
Cytoscape提供了丰富的插件,使得用户可以根据自己的需要进行网络分析和可视化。
6. DAVID(Database for Annotation, Visualization, and Integrated Discovery)DAVID是一个用于功能注释和富集分析的在线工具。
生物信息学常用数据资源介绍生物信息学是一门将大量数据和信息与生命科学相结合的学科,随着技术的不断发展,越来越多的生物信息学数据资源得到了广泛应用,使得生物信息学研究呈现出爆发式增长的态势。
在接下来的文章中,我将介绍一些常用的生物信息学数据资源。
1. 基因组浏览器基因组浏览器是生物信息学研究中非常常见的一种工具,在基因组浏览器中,用户可以利用多种查询方式快速定位以及查找基因序列、变异位点、基因表达等数据,具体的使用方法可以参考NCBI、UCSC和ENSEMBL等公共数据库。
2. 数据库公共数据库是生物信息学在数据共享和协作方面发挥重要作用的平台之一,NCBI、ENSEMBL、UniProt和GenBank等是生物信息学具有代表性的公共数据库,这些数据库为用户提供了一系列的基因组、转录组、蛋白质、代谢物等多种数据资源,这些数据可以帮助研究者进行基因预测及分析,杂交研究、协同研究等多种生物信息学研究。
3. 软件工具与数据库不同的是,软件工具主要起到数据分析与处理的作用。
对于不同的数据分析任务,不同的软件工具适应程度也不同,因此在生物信息学研究过程中需要不断尝试和探索,比如在转录组分析中,DESeq2和edgeR是非常常用的工具。
4. 人类基因组计划人类基因组计划是一项耗时多年,费用庞大的生命科学研究计划,目的是把人类的基因组解码,并制定新的医学治疗方案等。
在该项目结束后,因为庞大的数据量,成千上万名的研究者可以在其基础上继续开展基因组学研究,这进一步推动了生命科学领域的发展。
5. 元分析数据集随着生物信息学领域的快速发展,元分析数据集作为新工具出现了。
它是由几个相对独立的研究组合而成,旨在研究特定生物过程的数据,比如癌症发病的前因后果,它们包括多个数据来源和测序仪,提供了更全面、多元化的基因数据,为进一步研究确定新的生物标志物和治疗方法提供了更加可靠的基础。
综上,以上我们介绍了一些生物信息学研究中使用频率较高的数据资源,它们共同构成了生物信息学领域的基础设施,在加速科研发展、优化研究流程、减少人力物力成本等方面发挥重要作用,一方面可以帮助科研工作者得到更准确的结果,另一方面又能为更广泛的生命科学研究打开更广的视野。
常用生物信息学网址NCBI 生物信息学研究工具:/Tools/NCBI 生物信息学研究工具网站由美国国家生物技术信息中心支持。
该网站提供了许多程序的链接,内容包括数据挖掘、核酸和蛋白质组分析等。
同时,网站还提供了许多相关链接和资源。
欧洲生物信息学研究所:/欧洲生物信息学研究所是一个非盈利学术机构,是欧洲分子生物学实验室的一部分。
它是生物信息学研究和服务的中心。
它所管理生物数据的数据库包括核酸,蛋白质序列和大分子结构。
它的使命是保证从分子生物学和基因组研究的日益增长的信息向公众公开,并且对科学研究团体提供任何方面的免费使用,以促进科学发展。
欧洲生物信息学研究所Ensembl 基因组浏览器:ttp:///ensembl/index.html欧洲生物信息学研究所Thornton 研究组:/Thornton/index.html欧洲生物信息学研究所多序列联配数据库:/embl/Submission/alignment.html欧洲生物信息学研究所工具箱:/Tools/欧洲生物信息学研究所核酸数据库:/Databases/nucleotide.html欧洲生物信息学研究所计算基因组研究组:/research/CGG/index.html欧洲生物信息学研究所完整基因组数据库:/genomes/欧洲生物信息学研究所序列数据库研究组:/seqdb/index.htmlBrutlag 生物信息学研究组:/Brutlag 生物信息学研究组是斯坦福大学的一个研究团体,主要研究从蛋白质一级结构预测蛋白质结构和功能,其开发了EMOTIF 、EMATRIX 和3MOTIF 软件应用于非鉴定的基因组序列的功能确定,另外还开发了LOCK 和3DSEARCH 软件用于比较蛋白质结构和蛋白质结构数据库的搜索。
生物GBF 信息学小组主页:http://transfac.gbf.de/生物信息学小组主页是德国生物技术研究中心的生物信息组的主页。
常用生物学软件简介1. Oligo 6是目前使用最为广泛的一款引物设计软件,除了可以简单快捷地完成各种引物和探针的设计与分析外,还具有很多其他同类软件所不具有的高级功能: a) 已知一个PCR引物的序列,搜寻和设计另一个引物的序列。
b) 按照不同的物种对MM子的偏好性设计简并引物。
c) 对环型DNA片段,设计反向PCR引物。
d) 设计多重PCR引物。
e) 为LCR反应设计探针,以检测某个突变是否出现。
f) 分析和评价用其他途径设计的引物是否合理。
g) 同源序列查找,并根据同源区设计引物。
h) 增强了的引物/探针搜寻手段。
设计引物过程中,可以“Lock”每个参数,如Tm 值范围和引物3’端的稳定性等。
i) 以多种形式存储结果;支持多用户,每个用户可保存自己的特殊设置。
网址:/2. Vector NTI Suite是一套功能最全,而且界面最美观,最友好的分子生物学应用软件包。
主要包括四个大型软件,它们分别可以对DNA、RNA、蛋白质分子进行各种分析和操作。
Vector⑴ NTI:作为Vector NTI Suite的核心组成部分,它可以在生物研究的全过程中提供数据组织和序列编辑的软件支持。
Vector NTI 是以一种窗口形式,且支持项目组织的数据库来完成这一功能的;通过这个数据库,可以保存和组织大部分的实验数据,比如:基因结构、载体、序列片断、引物、蛋白质、多肽、电泳Markers和限制性内切酶等。
实际上,该数据库还支持对Vector NTI Suite 中各种小型的绘图和结果展示工具的管理。
Vector NTI 可以按照用户要求设计克隆策略。
用户只需提供克隆载体,外源片断序列,明确载体克隆的大致位置或酶切位点,其它工作由软件完成。
设计结果以图文形式输出到屏幕;最后根据客户定制的条件进行模拟电泳。
Vector NTI 还具有强大的设计和评估PCR引物、测序引物和杂交探针功能。
BioPlot⑵:BioPlot是一个对蛋白质和核酸序列进行各种理化特性分析的综合性工具,它是一种方便的桌面程序。
生信分析软件都有哪些?生信分析软件在生物信息学研究中可以帮助研究人员处理、分析和解释生物学数据,从而揭示生物学系统的结构和功能。
如数据处理和格式转换、序列比对和测序数据分析、基因组注释和功能预测、基因表达分析、变异检测和遗传分析、数据可视化等软件功能都可以提高研究效率和数据解读的准确性。
目前生信分析软件有很多种,笔者总结了部分生信分析软件的主要功能及作用,帮助大家更好的选用目标分析软件,排名部分先后:①BioXFinder国内第一个也是一个生物信息数据库,集成了BLAST、生存分析、基因ID转换等生信分析工具。
汇集了核酸、蛋白、蛋白结构、代谢通路和信号通路信息,可高效的搜寻到自己想要的信息(中英双文),并且在无代码的情况下完成生信分析。
举例工具Ⅰ:生存分析图生存分析图功能说明研究某癌症类型中患者的生存情况研究biomarker在癌症中的预后效果研究不同分组之间患者的生存是否存在差异数据输入说明支持txt(制表符分隔)文本文件、csv(逗号分隔)文本文件、以及Excel专用的xlsx格式,以及Excel的xls格式。
输入的数据共三列:第1列(生存时间列):如总生存期、无病生存期、无进展生存期等等,数值为生存天数。
第2列(终点事件列):为二分类变量0或1,1对应终点事件结局(如患者死亡,疾病痊愈等)。
第3列(分组信息列):分类型数据,如男/女、抽烟/不抽烟。
若想要探究的数据为连续型变量,如年龄、基因表达量、风险评分等等,需自行划分分组,如根据中位数将目标基因表达量拆分为高表达、低表达两组,将风险评分拆为高风险、低风险两组,将年龄拆分为幼年、青年、中年、老年等。
参数说明根据自身需求选择是否需要在生存分析主图中显示风险表、删失表、置信区间、P值和中位生存时间线;每个表中的参数可根据需求选择相对应的值。
运行结果说明横轴表示时间轴,纵轴表示生存概率。
不同曲线的颜色,对应相应分组的生存曲线。
经过logrank 检验后发现P 值= 0.0001 < 0.05,表明不同分组的患者生存状况的差异不能用抽样误差来解释,分组因素才是导致两条曲线生存率出现差异的原因。
常用生物信息学软件第一篇:生物信息学软件简介生物信息学软件是指用于分析、处理和组织生物学数据的计算机程序。
在生物信息学领域,一些常用的软件工具是必不可少的。
这些软件包括用于序列比对、蛋白质结构预测、基因注释、基因表达分析和系统生物学建模的工具。
接下来,我们将介绍一些流行的生物信息学软件。
1. BLASTBLAST(Basic Local Alignment Search Tool)是一个用于比较生物序列的软件工具,它可以用来比较DNA序列和蛋白质序列。
BLAST可以在非常短的时间内对大量的生物序列进行比对,它是生物信息学领域中非常流行的软件。
2. ClustalWClustalW是一个多序列比对程序,它可以将多个生物序列进行比对,以便研究它们的相似性。
ClustalW不仅可以比对DNA序列,还可以比对蛋白质序列。
它可以帮助研究人员理解序列之间的关系,进而推断它们的功能。
3. MEGAMEGA(Molecular Evolutionary Genetics Analysis)是一个用于进行分子进化分析的软件。
它可以用来进行系统发育分析、序列比对、基因注释和基因表达分析等工作。
MEGA可以处理多种不同类型的数据,包括DNA、RNA和蛋白质序列。
4. GROMACSGROMACS(GROningen MAchine for ChemicalSimulations)是一个用于分子动力学模拟的软件工具。
它可以模拟原子之间的相互作用,以研究分子的结构和动力学行为。
GROMACS是一个高效的软件,它可以处理复杂的系统,如大型蛋白质和DNA分子。
5. CytoscapeCytoscape是一个用于可视化和分析网络数据的生物信息学软件。
它可以用于存储和处理基因调控网络和代谢通路网络等数据。
Cytoscape还提供了各种不同类型的网络分析工具,如网络布局算法和社区检测工具等。
这些软件工具为生物信息学研究提供了强有力的支持。