宏基因组测序简介(发布版)
- 格式:pptx
- 大小:4.04 MB
- 文档页数:29

完整版)宏基因组测序讲解宏基因组测序的目的是研究藻类物种的分类、与特定环境相关的代谢通路,以及通过不同样品的比较研究微生物内部、微生物与环境以及与宿主的关系。
宏基因组,也称为微生物环境基因组或元基因组,是由Handelsman等于1998年提出的新名词。
它包含了可培养的和未可培养的微生物的基因,主要指环境样品中的细菌和真菌的基因组总和。
宏基因组学是一种以环境样品中的微生物群体基因组为研究对象的微生物研究方法。
它通过功能基因筛选和/或测序分析为研究手段,以微生物多样性、种群结构、进化关系、功能活性、相互协作关系以及与环境之间的关系为研究目的。
一般XXX包括从环境样品中提取基因组DNA,进行高通量测序分析,或克隆DNA到合适的载体,导入宿主菌体,筛选目的转化子等工作。
宏基因组文库是一种重要的研究工具,可以利用转入大肠杆菌中的宏基因组DNA载体,使以前无法研究的不可培养微生物的DNA得到复制、表达,从而进行研究。
所有带有宏基因组DNA载体的模式微生物克隆构成宏基因组文库。
对于宏基因组文库的DNA进行分析,有很多分析方法,主要分为表型功能筛选和序列基因型分析两类。
表型功能筛选是利用模式微生物表型的变化筛选某些目的基因,例如从文库中筛选能表达抗菌物质的克隆。
而序列基因型分析则是对文库中所有或部分的DNA进行测序分析,以应用于生态学研究,例如分析文库中16SrRNA序列,对所研究生态环境的多样性进行评估。
一个典型的宏基因组分析涉及多个轮次,以确保从生态环境标本中分离到目的基因,并尽可能多地分析DNA序列所编码的信息。
XXX是一种以环境样品中的微生物群体基因组为研究对象的新的微生物研究方法。
它主要通过功能基因筛选和测序分析来研究微生物多样性、种群结构、进化关系、功能活性、相互协作关系及与环境之间的关系。
在宏基因组学研究中,样品总DNA的提取及基因或基因组DNA的富集是非常关键的步骤。
提取的样品DNA必须可以代表特定环境中微生物的种类,获得高质量环境样品中的总DNA是宏基因组文库构建的关键之一。


宏基因组测序目的研究藻类物种的分类,研究与特定环境与相关的代谢通路,以及通过不同样品的比较研究微生物内部,微生物与环境,与宿主的关系。
技术简介宏基因组( Metagenome)(也称微生物环境基因组Microbial Environmental Genome, 或元基因组) 。
是由 Handelsman 等 1998 年提出的新名词,其定义为"the genomes of the total microbiota found in nature" , 即生境中全部微小生物遗传物质的总和。
它包含了可培养的和未可培养的微生物的基因,目前主要指环境样品中的细菌和真菌的基因组总和。
而所谓宏基因组学 (或元基因组学, metagenomics) 就是一种以环境样品中的微生物群体基因组为研究对象,以功能基因筛选和/或测序分析为研究手段,以微生物多样性、种群结构、进化关系、功能活性、相互协作关系及与环境之间的关系为研究目的的新的微生物研究方法。
一般包括从环境样品中提取基因组 DNA, 进行高通量测序分析,或克隆DNA到合适的载体,导入宿主菌体,筛选目的转化子等工作。
宏基因组( Metagenome)(也称微生物环境基因组Microbial Environmental Genome, 或元基因组) 。
是由 Handelsman 等 1998 年提出的新名词,其定义为"the genomes of the total microbiota found in nature" , 即生境中全部微小生物遗传物质的总和。
它包含了可培养的和未可培养的微生物的基因,目前主要指环境样品中的细菌和真菌的基因组总和。
而所谓宏基因组学 (或元基因组学, metagenomics) 就是一种以环境样品中的微生物群体基因组为研究对象,以功能基因筛选和/或测序分析为研究手段,以微生物多样性、种群结构、进化关系、功能活性、相互协作关系及与环境之间的关系为研究目的的新的微生物研究方法。

宏基因组测序环境中超过99%的微生物是不可培养的,很多致力于研究微生物多样性的努力由于培养方法的限制而受到制约,为了克服由培养技术所带来的困难和限制,多种以DNA为基础的分子生物学的方法已经被开发。
目前16s rDNA测序可以提供大量关于环境微生物的群落及种类信息,但是在种群中不同微生物的作用以及其携带的基因组信息基本不能体现出来。
相比之下,宏基因组是一种新的,可用于快速分析微生物复杂基因组的方法,它提取环境中的全基因组DNA,构建DNA文库并进行高通量测序。
对数据进行分析,不仅能够获得环境中微生物的组成及丰度信息,还可以通过相关功能及代谢通路注释,获得这些微生物全面的微生物基因组信息,以及在环境中可能的功能。
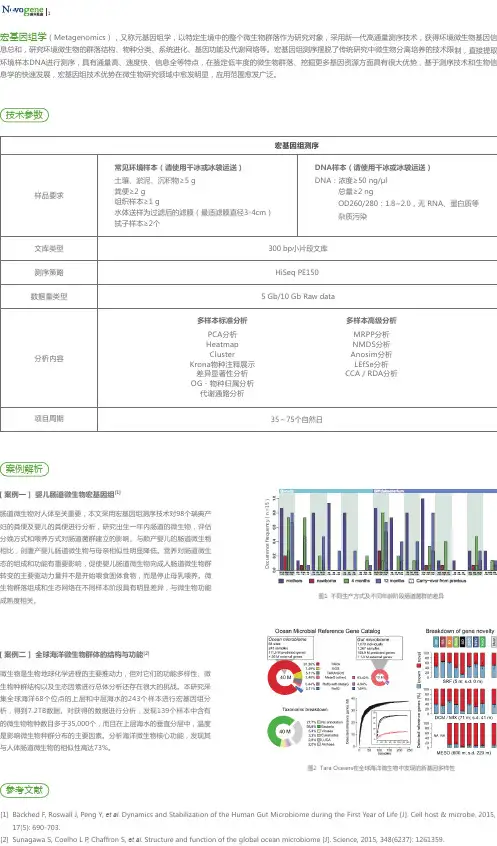
技术参数样品准备测序策略推荐数据周期3ug DNA 300bp DNA文库HiSeq PE150测序一般测序数据量:5Gb clean data大测序数据量:10Gb clean data40个工作日建库方法技术流程技术特点(1)无需分离培养,直接提取样本DNA测序;(2)群落多样性、种群结构、进化关系、功能组成、相互协作关系等多种分析;(3)高效、高通量,一次性获取样本中所有微生物组成等信息。
部分结果展示进化树分析OTU维恩图抗生素类型统计图案例解析排泄物微生物宏基因组可作为结直肠癌标志物为了评估利用排泄物诊断结直肠癌的可行性,作者对来自于中国的74个结直肠癌患者和54个健康人的粪便样本进行宏基因组测序,发现除了已经证实的与结直肠癌相关的具核梭杆菌(Fusobacterium nucleatum)和消化链球菌(Peptostreptococcus stomatis)之外,微小微单胞菌(Parvimonas micra)和口臭致病菌(Solobacterium moorei)也与结直肠癌具有显著相关性。
作者随后选择了20个微生物基因标志物,通过q-PCR发现,来自于具核梭杆菌的丁酰coA脱氢酶和来自于微小微单胞菌的RNA聚合酶亚基β在患者的粪便微生物的基因组中高度表达;利用这两个基因可以准确区分患有结直肠癌的患者和健康人群。

宏基因组测序介绍
宏基因组测序(Metagenomics Sequencing)又称为环境基因组测序,是一种对复杂环境中所有微生物生物群体的遗传信息进行分析的高通量测序技术。
与传统分离培养方式不同,宏基因组测序可以直接提取环境样品中所有微生物的总DNA,并通过测序技术进行分析,从而得到环境样品中所有微生物群体的遗传信息。
宏基因组测序的优点是高通量、快速、高效、无偏差、不需要微生物分离和培养,可以对满足各种环境条件的微生物进行整体性的研究,从而更加真实地反映生物在环境中的生态学特征,为生态学、环境保护等领域的研究提供了数据支持。

宏基因组测序技术检测标准简介:宏基因组测序介绍宏基因组学是以环境样品中的微生物群体基因组为研究对象,通过现代基因组技术手段包括功能基因的筛选和测序分析,对环境中微生物多样性、种群结构、进化关系、功能活性、相互协作关系以及环境之间的关系进行研究的新的微生物研究方法。
随着高通量测序技术的发展,为宏基因组学研究提供了新的理想研究方法。
高通量测序的方法无需分离环境中各种微生物,也无需构建克隆文库就可以直接对环境中所有微生物进行测序。
可以真实客观的反映环境中微生物的多样性、种群结构、进化关系等。
目前又可以分为针对16s DNA/18sDNA/ITS测序和针对宏基因组全序列的测序研究。
下面就是对这两者的具体介绍。
一、16s DNA/18s DNA/ITS测序16sDNA是最常用的微生物物种分子鉴定的标签,,通过对样品中16sDNA测序可以鉴定其中微生物物种的丰度和分布情况。
目前,普遍使用Roche 454平台来对环境样品进行16s DNA测序。
因为16s DNA序列比较相似,读长短的话,难以进行有效的比对,而454平台的平均读长在400bp左右,可以很好的避免此类问题。
二、宏基因组全测序在这种测序方式中,我们可以假定一个环境中的所有微生物就是一个整体,然后对其中所有的微生物进行测序。
这样我们就可以研究样品中的功能基因以及其在环境中所起的作用而不用关心其来自哪个微生物。
可以发现新的基因,可以进行基因的预测,甚至有可能得到某个细菌基因组的全序列。
此外,该项测序不单可以针对DNA水平,也可以针对全RNA进行基因表达水平的研究。
样品处理:宏基因组样品收集主要有口腔,下呼吸道痰液,下呼吸道灌洗液,皮肤和粪便。
样品采集遵照样品采集规范(人)所规定的操作来进行。
尽量留足备份样品。
核酸提取:宏基因组核酸提取主要有两种方法:膜过滤法和直接裂解提取。
对于液体样品如痰液,灌洗液两种方法都适用,对于固体样品如粪便宜采用直接裂解的方法。



送检mNGS宏基因组二代测序报告情况、标本类型、送检保存要求、标本运输要求、测序情况、内容解释、微生物致病概率分级及解读宏基因组二代测序(mNGS) 是基于核酸检测的微生物鉴定技术其非预设性、高通量等优点而得到广泛应用。
下呼吸道感染主要包括社区获得性肺炎、医院获得性肺炎、免疫抑制宿主肺炎、慢性阻塞性肺疾病急性加重、支气管扩张症合并感染等类型,临床表现多样,感染微生物种类复杂,感染和定植鉴别困难,加之mNGS 技术本身存在的局限性,mNGS 诊断效力的发挥有赖于选择恰当患者、采用适宜标本以及进行合理解读。
需送检mNGS情况(1) 免疫抑制宿主疑似发生LRTI 且临床表现提示非CAP 常见病原微生物所致者;(2) LRTI 患者发病初期即出现需要使用血管活性药物的感染性休克、需要有创机械通气的呼吸衰竭、多脏器功能不全等危及生命的状况时;LRTI 经规范经验性抗感染治疗48—72 h 后,感染症状仍持续加重或影像学快速进展者;(3) 聚集性发病疑似具有传染性、但无法明确病原体的LRTI;有特殊病史且经验性治疗无效,病情较为严重的LRTI;临床考虑特殊病原体(感染且病势迅疾或迁延者,常规培养困难或所在医疗机构无法提供可靠的传统检测方案时;(4) 患者有LRTI 症状或影像表现,经规范抗感染治疗后病灶吸收延迟、病程迁延,需鉴别是否由非感染性疾病所致,可以在常规病原微生物检测、感染生物标志物、病理等相关检查同时送检mNGS 以帮助鉴别诊断。
不建议送检 mNGS情况(1) 免疫功能健全宿主罹患LRTI(包括重症肺炎),经过规范的经验性抗感染治疗病情已好转;(2) LRTI 已通过其他方法获得病原学结果,与临床特点相符,或针对性治疗有效;(3) 无法获取优质标本。
mNGS 标本类型在LRTI 的病原微生物诊断中,可用于mNGS 检测的标本包括痰(含诱导痰)、气管吸引物、支气管肺泡灌洗液(BALF)、经支气管肺活检(TBLB)标本、经支气管内超声(EBUS)活检标本、经皮肺穿刺活检标本、血液等。

宏基因组测序原理
宏基因组测序是一种用于研究环境中微生物群落的高通量测序技术。
与传统的基因组测序不同,宏基因组测序可以同时对整个微生物群落中的所有微生物进行测序,从而揭示微生物群落的多样性和功能。
在宏基因组测序中,有几个关键的步骤和原理需要了解。
首先,样品的DNA提取是宏基因组测序的第一步。
在这一步中,需要将微生物群落中的DNA提取出来,并且要尽可能保持DNA的完整性和纯度。
这一步的成功与否直接影响着后续的测序结果。
接下来是DNA文库的构建。
在这一步中,需要将提取出的DNA进行裂解和连接,构建成文库。
文库构建的关键在于选择合适的连接适配体和裂解方法,以确保文库的质量和多样性。
然后是高通量测序。
在这一步中,构建好的DNA文库被送入测序仪进行高通量测序。
高通量测序技术可以同时对数百万个DNA片段进行测序,从而大大提高了测序的效率和覆盖度。
最后是数据分析和生物信息学处理。
通过生物信息学分析,可以将测序得到的海量数据进行拼接、比对、注释和功能预测,从而揭示微生物群落的多样性和功能特征。
总的来说,宏基因组测序的原理是通过高通量测序技术对微生物群落中的DNA进行测序,从而揭示微生物群落的多样性和功能。
这项技术的应用可以帮助我们更好地了解微生物在环境中的分布和作用,对于环境保护和微生物资源开发具有重要意义。
宏基因组测序1宏基因组研究概况宏基因组学(Metagenomics,又称元基因组学)这一概念最早在1998年由威斯康辛大学植物病理学部门的Jo Handelsman等提出,随后伯克利分校的研究人员Kevin Chen和Lior Pachter将宏基因组定义为:应用现代基因组学的技术直接研究自然状态下的微生物的有机群落,而不需要在实验室中分离单一的菌株的科学。
鉴于环境中99%的微生物不可培养,宏基因组无需进行微生物分离操作的技术特点打破了传统微生物学基于纯培养研究的限制,为充分认识全球和人体范围内微生物和开发利用未培养微生物,并从完整的群落水平上研究微生物的活动和发掘潜在功能提供了可能。
传统的宏基因组学研究通过直接从环境样本中提取DNA,构建DNA克隆载体的宏基因组文库,并利用基于核酸序列差异分析、克隆子的特殊代谢活性、底物诱导基因的表达和(或)稳定同位素和荧光原位杂交等技术对宏基因组文库进行筛选和分析,从而有效地利用环境中丰富的微生物资源和挖掘新的特殊功能代谢物。
在宏基因组学研究的发展前沿中,核酸测序技术测序速度和通量的增长引人注目。
在下一代测序技术(NGS,又称第二代高通量测序技术)出现之前,基于宏基因组测序的核酸序列差异分析通常使用Sanger测序(主要基于双脱氧核苷酸链终止反应)方法,对宏基因组克隆文库或功能扩增子进行测序,来研究环境中微生物群落多样性及功能特征。
但Sanger测序方法通量较小、测序周期较长且价格昂贵。
随着新一代测序技术的迅猛发展以及广泛应用,宏基因组测序的方法也发生了翻天覆地的变化,目前科学家们可以对环境中的微生物全部基因组进行测序,在获得海量的数据后,全面地分析微生物群落结构以及基因功能组成,了解微生物如何耐受极端环境,发觉新的生物资源并探索微生物与环境间的相互作用。
下一代测序方法使用了几种不同的高通量平台,最先使用的是基于焦磷酸聚合酶乳液的RocheGS20 454测序仪(又称焦磷酸测序仪)。
小鼠胰腺组织测序宏基因组
摘要:
一、背景介绍
二、实验方法
三、实验结果
四、结果分析
五、结论
正文:
一、背景介绍
近年来,随着生物信息学技术的不断发展,宏基因组学在研究生物系统中微生物群落结构及其功能方面取得了突破性进展。
本文以小鼠胰腺组织为研究对象,通过测序技术对宏基因组进行研究,旨在揭示胰腺组织中微生物群落的组成及其与宿主相互作用的机制。
二、实验方法
1.样本收集:从实验小鼠胰腺组织中提取微生物DNA。
2.宏基因组测序:采用Illumina HiSeq 平台进行微生物DNA 的测序。
3.数据分析:通过生物信息学方法对测序数据进行处理,包括序列拼接、质控、OTU 分析等。
4.功能注释:对差异丰度物种进行功能注释,揭示其在宿主生理功能中的作用。
三、实验结果
1.胰腺组织中检测到丰富的微生物多样性,包括细菌、古菌和真菌等。
2.实验组与对照组小鼠胰腺组织微生物群落结构存在显著差异,表明宿主生理状况对微生物群落有显著影响。
3.差异丰度物种的功能注释结果表明,微生物群落在胰腺组织的生理功能中发挥着重要作用,如能量代谢、免疫调控等。
四、结果分析
通过对小鼠胰腺组织宏基因组的测序和分析,揭示了微生物群落在胰腺组织中的重要作用。
这些发现为进一步研究宿主- 微生物相互作用及其在胰腺疾病中的作用提供了依据。
五、结论
本研究采用测序技术对小鼠胰腺组织宏基因组进行了研究,揭示了微生物群落在胰腺组织中的重要作用。
宏基因组测序(mNGS)是一种在临床中用于诊断新发突发传染病以及常规检验阴性的感染性疾病的方法。
这种方法涉及到的基因组覆盖度是指测序过程中能够覆盖的基因组区域的比例。
在宏基因组测序中,测序产生的数据需要经过比对、筛选、过滤等生物信息学手段处理,以注释微生物物种。
在这个过程中,需要评估测序数据的覆盖度和基因组的覆盖度。
基因组覆盖度是衡量测序数据能够覆盖基因组区域的比例。
高的基因组覆盖度意味着测序数据能够覆盖基因组的更多区域,从而提供更全面的微生物物种信息。
而基因组覆盖度低则意味着还有许多基因组区域未能被测序或未能被充分测序,需要进一步优化测序方案或增加测序深度。
因此,在宏基因组测序中,基因组覆盖度是一个重要的评估指标,能够反映测序方案的优劣和测序数据的质量。
提高基因组覆盖度可以更好地揭示微生物物种的多样性,有助于更准确地诊断和治疗感染性疾病。
小鼠胰腺组织测序宏基因组引言胰腺是人体中一个重要的消化和内分泌腺器官,它在消化系统中起着至关重要的作用。
了解胰腺组织的基因组信息对于研究胰腺相关疾病以及开发相关治疗方法具有重要意义。
本文将探讨小鼠胰腺组织测序宏基因组的相关内容。
小鼠胰腺组织测序的意义1.研究胰腺发育:通过测序小鼠胰腺组织的宏基因组,可以了解胰腺发育的分子机制,进而揭示相关疾病的发生和发展过程。
2.研究胰腺疾病:通过比较正常胰腺组织与疾病胰腺组织的基因组差异,可以发现与胰腺疾病相关的基因,为疾病的早期诊断和治疗提供新的思路和方法。
3.研究胰腺功能:通过测序小鼠胰腺组织的宏基因组,可以了解胰腺在代谢调控、内分泌功能等方面的作用,为相关研究提供基础数据。
测序方法小鼠胰腺组织的宏基因组测序可以采用多种方法,常见的有以下几种:1. 全基因组测序(WGS)全基因组测序是一种测序技术,可以对整个基因组进行测序。
通过WGS,可以获得胰腺组织中所有基因的信息,包括编码蛋白质的基因和非编码RNA的基因等。
这种方法可以全面了解胰腺组织的基因组特征。
2. 转录组测序(RNA-Seq)转录组测序是一种测序技术,可以测定胰腺组织中所有转录的RNA序列。
通过RNA-Seq,可以了解胰腺组织中的基因表达情况,包括哪些基因在胰腺中高表达,哪些基因在胰腺中低表达等。
这种方法可以揭示胰腺组织的功能和调控机制。
3. 甲基化测序甲基化测序是一种测序技术,可以测定胰腺组织中DNA的甲基化状态。
通过甲基化测序,可以了解胰腺组织中DNA甲基化的分布情况,揭示胰腺组织的表观遗传调控机制。
测序数据分析获得小鼠胰腺组织测序宏基因组数据后,需要进行数据分析以获取有意义的结果。
常见的数据分析方法有以下几种:1. 基因表达分析通过转录组测序数据,可以进行基因表达分析,包括差异表达基因的筛选和聚类分析等。
这些分析可以帮助我们了解胰腺组织中哪些基因在不同生理或病理状态下表达差异显著,从而揭示胰腺组织的功能和调控机制。