有参考基因组_转录组ref流程工作手册
- 格式:pdf
- 大小:1.57 MB
- 文档页数:24


转录组ref流程工作手册
一、Reference 流程生物学原理
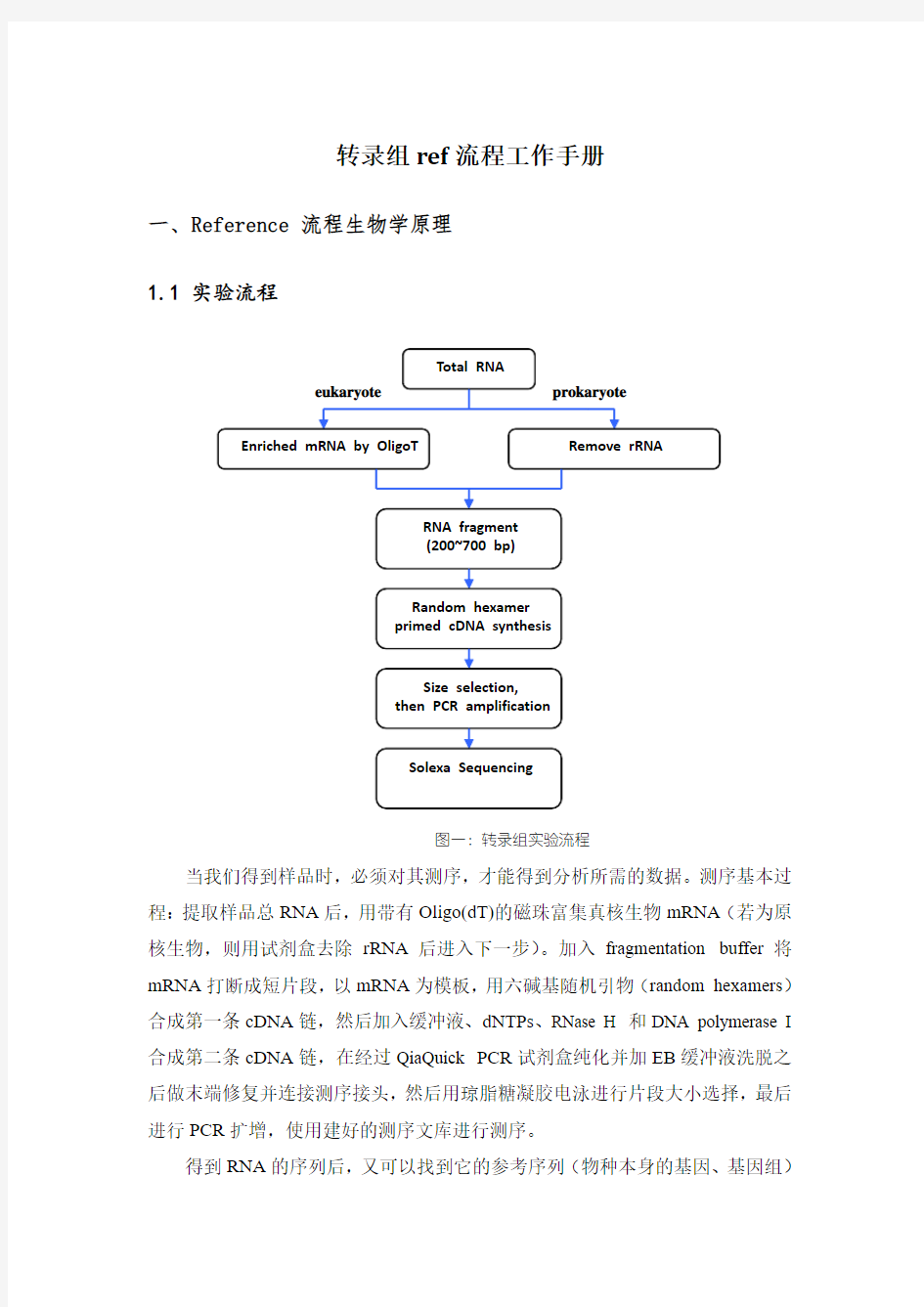
1.1 实验流程
RNA fragment Random hexamer Size selection,prokaryote
eukaryote
Total RNA
Enriched mRNA by OligoT Remove rRNA
(200~700 bp)
primed cDNA synthesis
then PCR amplification
Solexa Sequencing
图一:转录组实验流程
当我们得到样品时,必须对其测序,才能得到分析所需的数据。测序基本过程:提取样品总RNA后,用带有Oligo(dT)的磁珠富集真核生物mRNA(若为原核生物,则用试剂盒去除rRNA后进入下一步)。加入fragmentation buffer将mRNA打断成短片段,以mRNA为模板,用六碱基随机引物(random hexamers)合成第一条cDNA链,然后加入缓冲液、dNTPs、RNase H 和DNA polymerase I 合成第二条cDNA链,在经过QiaQuick PCR试剂盒纯化并加EB缓冲液洗脱之后做末端修复并连接测序接头,然后用琼脂糖凝胶电泳进行片段大小选择,最后进行PCR扩增,使用建好的测序文库进行测序。
得到RNA的序列后,又可以找到它的参考序列(物种本身的基因、基因组)
时,可以用reference流程对数据进行详细的分析。Reference后面所有的流程都是基于参考序列进行的,所以选择正确的参考序列十分重要。
1.2信息分析流程
得到测序序列后,即可利用比对软件,将所测序列比对到参考基因或基因组上,并进行后续分析,信息分析流程图如下:
图二:转录组信息流程
1.2.1原始fq序列简介
测序得到的原始图像数据经base calling转化为序列数据,我们称之为raw data或raw reads,结果以fastq文件格式存储,fastq文件为用户得到的最原始文件,里面存储reads的序列以及reads的测序质量。在fastq格式文件中每个read 由四行描述:
@read ID
TGGCGGAGGGATTTGAACCC
+
bbbbbbbbabbbbbbbbbbb
每个序列共有4行,第1行和第3行是序列名称(有的fq文件为了节省存储空间会省略第三行“+”后面的序列名称),由测序仪产生;第2行是序列;第4行是序列的测序质量,每个字符对应第2行每个碱基,第四行每个字符对应的ASCII值减去64,即为该碱基的测序质量值,比如h 对应的ASCII值为104,那么其对应的碱基质量值是40。碱基质量值范围为0到40。表 1为Solexa测序错误率与测序质量值简明对应关系,具体计算公式如下:
Q phred =-10 log10(e)
表 1 Solexa测序错误率与测序质量值简明对应关系
5% 13 M
1% 20 T
0.1% 30 ^
0.01% 40 h
1.2.2原始fq序列处理
某些原始序列带有adaptor 序列,或含有少量低质量序列。我们首先经过一系列数据处理以去除杂质数据,得到Clean reads。
按如下步骤进行处理:
1.去除含adaptor的reads
2.去除N的比例大于10%的reads
3.去除低质量reads(质量值Q <= 5的碱基数占整个read的50%以上)
4.获得 Clean reads
原始序列数据经过去除杂质后得到的数据称为Clean reads,后续分析都基于Clean reads
1.2.3比对
使用短reads比对软件SOAP2/SOAPaligner{Li, 2009 #155}将clean reads分别比对到参考基因组和参考基因序列(允许两个碱基错配)。
通过这一步骤,我们可以将测序得到的reads对应到基因及基因组上,后续分析都是基于上述比对结果。
1.2.4基本生物信息分析结果
基本信息分析结果包含以下内容:
1 测序数据产量及与Reference 比对结果概述
统计数据量的大小,得到测序数据产量;对soap结果进行处理得到测序数据与Reference序列比对的概况。
2 评价测序随机性
在转录组实验过程中,首先要通过物理或化学方法将转录本打断成短片段,然后上机测序。如果打断随机性差,reads偏向于来自基因特定区域,将会直接影响转录组的各项分析结果。
利用reads在基因上的分布来评价打断随机性。由于不同参考基因有不同长度,我们把reads在基因上的位置标准化到相对位置(reads在基因上的位置与基因长度的比值),然后统计基因的不同位置比对上的reads数。如果打断随机性好,reads在基因各部位应分布得比较均匀。
3 基因覆盖度、测序深度的分布
基因测序覆盖度指每个基因被reads覆盖的百分比,其值等于基因中unique mapping reads覆盖的碱基数跟基因编码区所有碱基数的比值。测序深度指基因被reads 覆盖的次数,其值等于reads覆盖到基因的碱基数与基因编码区所有碱基数的比值。
4 Reads 在参考基因组上的分布
该分析主要是以图形方式概括给出Reads在基因组各个位置的分布情况,以及该位置基因的分布情况。
1.2.5高级生物信息分析结果
高级生物信息分析包含以下结果:
1 对基因结构进行优化
通过比较测序结果和现有基因注释结果,对基因的5'端或3'端进行延长。如图三所示,首先,将reads比对到基因组,提取基因组中被unique mapping reads覆盖的次数大于或等于某阈值(默认为2)且位置连续的区域作为转录活性区(Transcription Active Region, TAR,图中蓝色方块区域);然后通过paired-end reads(图中紫色线条)将不同的TAR连接形成潜在的gene model;最后,通过比较潜在gene model与现有基因注释的差别,对基因的5'端和3'端进行延长(图中表现的仅是基因3’端发生延长的情况)。
图三:基因结构优化
2 鉴定基因的可变剪切
可变剪切使一个基因产生多个mRNA转录本,不同mRNA可能翻译成不同蛋白。因此,通过可变剪切一个基因可能产生多个蛋白,极大地增加了蛋白多样性{Black, 2003 #6}{Stamm, 2005 #21;Lareau, 2004 #22}。虽然已知可变剪切在真核生物中普遍存在,但我们可能仍低估了可变剪切的比例,最近,基于高通量测序的可变剪切研究在人{Pan, 2008 #3} {Wang,