Antheprot蛋白质序列分析软件包ANTHEPROT4.5是位于法
- 格式:doc
- 大小:39.00 KB
- 文档页数:4

常用生物软件(windows)全面介绍一、基因芯片1、基因芯片综合分析软件。
?ArrayVision 一种功能强大的商业版基因芯片分析软件,不仅可以进行图像分析,还可以进行数据处理,方便protocol的管理功能强大,商业版正式版:6900美元。
?Arraypro Media Cybernetics公司的产品,该公司的gelpro, imagepro一直以精确成为同类产品中的佼佼者,相信arraypro也不会差。
?phoretix™ Array Nonlinear Dynamics公司的基因片综合分析软件。
?J-express 挪威Bergen大学编写,是一个用JAVA语言写的应用程序,界面清晰漂亮,用来分析微矩阵(microarray)实验获得的基因表达数据,需要下载安装JAVA运行环境后后,才能运行。
?2、基因芯片阅读图像分析软件?ScanAlyze ,斯坦福的基因芯片基因芯片阅读软件,进行微矩阵荧光图像分析,包括半自动定义格栅与像素点分析。
输出为分隔的文本格式,可很容易地转化为任何数据库。
?3、基因芯片数据分析软件?Cluster 斯坦福的对大量微矩阵数据组进行各种簇(Cluster)分析与其它各种处理的软件。
?SAM Significance Analysis of Microarrays 的缩写,微矩阵显着性分析软件,EXCEL软件的插件,由Stanford大学编制。
?4.基因芯片聚类图形显示?TreeView 斯坦福开发的用来显示Cluster软件分析的图形化结果。
现已和Cluster成为了基因芯片处理的标准软件。
?FreeView 是基于JAVA语言的系统树生成软件,接收Cluster生成的数据,比Treeview增强了某些功能。
?5.基因芯片引物设计?Array Designer DNA微矩阵(microarray)软件,批量设计DNA和寡核苷酸引物工具?二、RNA二级结构RNA Structure RNA Sturcture 根据最小自由能原理,将Zuker的根据RNA一级序列预测RNA二级结构的算法在软件上实现。

分子生物学软件简要功能介绍在进行分子生物学研究时,常需要用到各种计算机软件。
这些软件种类不少,功能大致相同,到底使用哪个,其实可根据各人的使用习惯来选择。
下列的软件,大部分可在网上找到全功能的版本或各种演示版或试用版,只要以软件名为关键词用搜索引擎搜索就可以了。
网上也有此类各种软件的总结。
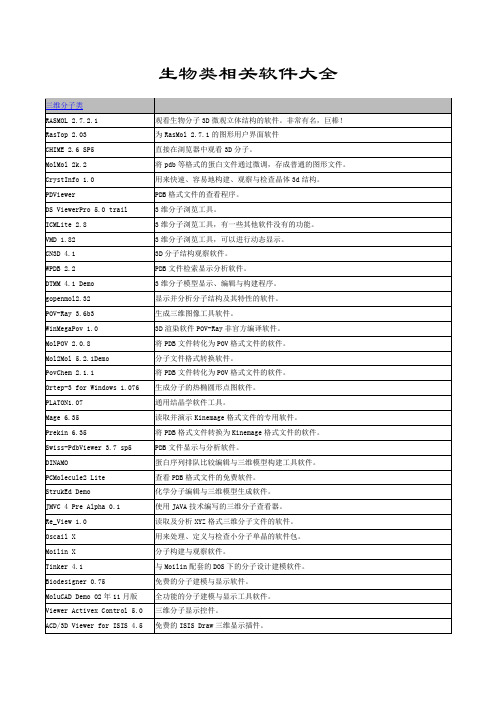
下面为此类软件的简单介绍:1.三维分子类RASMOL:观看生物分子3D微观立体结构RasTop:为RasMol 2.7.1的图形用户界面软件CHIME:直接在浏览器中观看3D分子MolMol:将pdb等格式的蛋白文件通过微调,存成普通的图形文件raswin.exe.gz:rasmol(win)2.7.0.1 rasmol新版本及汉化版本CrystInfo:用来快速、容易地构建、观察与检查晶体3D结构PDViewer:PDB格式文件的查看程序Weblab Viewlite:三维分子浏览工具及大量分子文件例子Weblab ViewerPro:三维分子浏览工具ICMLite:三维分子浏览工具,有一些其他软件没有的功能VMD:三维分子浏览工具,可以进行动态显示CN3D:3D分子结构观察软件WPDB:PDB文件检索显示分析软件DTMM:三维分子模型显示、编辑与构建程序Mole:高性能的大分子三维图形显示计算工具gopenmol:显示并分析分子结构及其特性POV-Rayv:生成三维图像工具软件MolPOV:将PDB文件转化为POV格式文件的软件Mol2Mol:分子文件格式转换软件PovChem:将PDB文件转化为POV格式的文件Ortep-3 for Windows:生成分子的热椭圆形点图PLATON:通用结晶学软件工具Mage:读取并演示Kinemage格式文件的专用软件 Prekin :将PDB格式文件转换为Kinemage格式文件Swiss-Pdb Viewer:PDB文件显示与分析软件DINAMO:蛋白序列排队比较编辑与三维模型构建工具PCMoleeule2 Lite:查看PDB格式文件的免费软件StrukEd :化学分子编辑与三维模型生成软件JMVC:使用JAVA技术编写的三维分子查看器ReView:读取及分析XYZ格式三维分子文件Oscail:用来处理、定义与检查小分子单晶的软件包Moilin:分子构建与观察软件Tinker:与Moilin配套的DOS下的分子设计建模Biodesigner:免费的分子建模与显示软件MoluCAD:全功能的分子建模与显示工具软件 Viewer Activex Control:三维分子显示控件MarvinView:JAVA语言编写的化学分子二维与三维显示程序ACD/3D Viewer for ISIS:免费的ISISDraw三维显示插件Amira:高等三维显示建模系统AmiraMol:Amira 2.3 相应的显示三维分子的增强工具Visualize:分子建模和研究软件包ScientificGL:C++OpenGLAPI三维分子开发工具Sojourner:找出小蛋白的最小能量构形并实时演示的软件2.DNA分析DNAClub:DNA处理软件JaMBW:分子生物学软件包DNATool:功能很全面的DNA序列分析工具包pDRAW:DNA分析与绘图软件,可绘制线性或环形DNA图ANNHYB:用来帮助进行PCR引物设计与基因探针设计的软件RESTRICTION ANALYSIS:限制酶消化工具ABIView:ABI格式文件显示与编辑软件Chromas:ABI格式文件显示与编辑软件Sequence viewer:获取与观察从GSDB获取的DNA序列数据及其关联特性的工具DNAssist:DNA序列分析工具DNAProbe:核苷酸序列设计工具DnaSP:DNA序列种群遗传学分析软件DFW:DNA分析软件Artemis R4:以Java语言写成的序列查看工具’ACT R1:以Java语言写成的序列比较查看器GDA:主要用来进行不连续基因数据的统计分析 RDP:从一组排队比较(Align)的核酸序列中查找潜在的重组体软件Sequencher:装配DNA小片段为大的连续序列或毗连(序列)群"Contig"软件MehCalc:自动计算DNA序列热力学数据的Excel电子表格宏软件基因探索者:中文界面的功能集成、高效、快捷的基因分析软件ConsInspector:DNA蛋白结合位点预测识别软件MatInd与Matlnspector:快速匹配DNA序列与已知共有序列的软件工具GBuilder:JAVA语言编制的用来分析与显示DNA序列的软件GenomePixelizer:帮助理解基因组中的簇基因(C1ustermggene)之间的相互关系的软件LabBook Genomic XML Viewer:图形化显示并处理GenBank序列数据的免费软件Gene Construetion Kit 2 :管理并显示克隆策略中的分子构建过程软件Genalysis:比较基因组或大量基因序列的工具软件3.RNA分析RNAdraw:RNA二级结构分析软件RNAstructure:预测RNA 级结构图RnaViz:RNA二级结构图绘制程序4.蛋白质分析ANTHEPROT:蛋白序列分析软件包pSAAM:蛋白序列分析软件包VHMPT:螺旋状膜蛋白拓扑结构观察与编辑软件aminoXpress:免费的多功能蛋白分析软件包5.生化教学mmp.zip:将生化代谢中的各种途径用图表的形式表示出来linpath.zip:线性酶反应模拟软件protlab:蛋白质纯化仿真软件MOLMED.ZIP:生化基础概念演示教学程序Biochem:生化教学文件photo:光合作用教学程序Adrenalin:肾上腺素在肝糖原代谢中的作用演示Virtual Cell Lab:多媒体细胞生物学教学程序6.生化工程brd.zip:生物反应器(发酵罐)设计软件BioStat:BioStatB发酵罐控制程序PenSimv:青霉素发酵模拟软件BioProSim:发酵实时模拟软件7.序列格式转换vised:序列输入分析和格式转换软件ForCon:多序列文件格式转换软件SeqVerter:序列格式转换软件GeneStudio LE Version:序列格式显示、编辑与转换工具软件FASTA/BLAST SCAN:FASTA与BLAST查询输出文件的处理软件RevComp:序列格式转换软件8.引物分析primer Premier 5.0:引物设计工具Oligo:引物分析著名软件Primer Designer:专门用 pASK-IBA~pPR-IBA表达载体免费的引物设计辅助软件Array Designer:批量设计DNA和寡核苷酸引物工具Beacon Designer:PCR定量分析分子信标(Molecularbeacon)设计软件NetPrimer:基于WEB界面的引物设计程序9.序列综合分析pcgene:分子生物应用软件 MACAW:多序列构建与分析软件Clustal W:用来对核酸与蛋白序列进行多序列比较的软件Clustal X:ClustalWWindows界面程序FASTA:数据库中查找同源序列软件GeneDoc:对序列进行相关分析等操作BLAST与Blastcl3:数据库中查找类似序列的软件及客户端软件SeqPup 0.9:生物分子序列编辑与分析软件K-Estimator 5.5:进化基因学研究软件,评估两条核酸序列核苷酸替代数BioEdit:序列编辑器与分析工具软件DAMBE:综合性序列工具软件LaserGene:综合性序列工具软件SeaView:图形化多序列队列编辑器 Jalview:用Java语言写的多序列队列编辑器DNASIS:序列综合分析工具Genamics Expression:是一个DNA与蛋白序列分析工具Vector NTIViewer:载体查看软件Jellyfish:多功能序列分析软件ProSeq:核酸序列编辑与种群遗传学分析软件Gap4 database viewer:Gap4基因装配数据库读取显示软件SMS:DNA与蛋白序列分析与格式化在线工具集合Omiga:核酸与蛋白序列综合性分析软件Staden:综合序列分析工具软件包Vector NTISuite:综合性蛋白核酸分析工具包INCA:Java脚本语言写成的BLAST服务器客户端程序ISYS:NCGR开发的用JAVA语言写成的数个不同类生物信息软件与数据库的软件集合平台DNA Scriptor:DNA与蛋白序列综合分析软件Sequence Quickie-Calc:非常紧凑的分子生物学工具软件PhyloGrapher:用来显示与研究相类似的基因与蛋白序列之间的进化关系的软件10.进化树分析phylip:进化树分析软件,并可绘制进化树TreeView:进化树处理软件GeneTree:比较基因与种系进化树的程序NDE:用来编辑NEXUS格式文件的程序TreeMap:用来可视化地比较主、从进化树的程序Spectrum:分析进化信息而不用将之转化为进化树的软件Phyltools:计算与处理进化树数据的软件tree-puzzle:核酸序列、蛋白序列相似性分析及进化树构建工具ATV:JAVA语言编写的显示"New Hampshire"与NHX格式的进化树文件软件TREECON:构建和绘制进化树的软件包ProBiosys比较表现型分类法数据和分析计算核酸序列数据距离值的软件。
生物类相关软件大全1. DNASIS for Windows 2.5版是日立软件公司(Hitachi Sofeware Engineering Co.,Ltd.)97年推出的一个功能强大的序列分析软件。
包含有大部分分子生物学软件的常用功能,可进行DNA,RNA,蛋白质序列的编辑和分析,甚至还能进行质粒作图、数据库查询等功能,足可满足一般实验室的要求。
2. DNATools 5.1DNATools设计的用户友好、强壮,以便快速、方便地获取、贮藏和分析序列及数据库查询获得的序列相关信息。
DNATools包容性很好,能把几乎所有文本文件打开作为序列。
当程序不能辨别序列的格式时(通过寻找常用序列格式的特征),会显示这个文件的文本形式,以便你编辑生成正确的蛋白质或DNA序列,编辑后可以再被载入程序。
若你的序列是DNATools格式时(DNA或寡核苷酸序列),程序不加注解的载入序列,程序模式调整成可以接受载入的数据类型(蛋白质、DNA和寡核苷酸引物序列)。
在一个项目中可以加入几千个序列或引物,并在整个项目中分析这些序列及标题。
这个程序的一个特点是给每个序列或引物添加文本标题。
这样就可以用自定义的标题识别序列,而不必通过它们的文件名。
为避免丢失数据-和你的工作-DNATools包括几个挽救丢失数据的功能:一个5层的撤销/重复功能,重新获得原始序列的恢复功能,项目中加入新文件时的安全备份功能,以及在一定的时间间隔作完全备份或备份你的工。
3. DNAClubDNA Club是一个简单的对DNA进行与PCR有关的操作的软件。
它的功能有: 1、输入DNA 序列; 2、查找ORF序列; 3、把DNA翻译成蛋白序列; 4、查找酶切位点; 5、查找PCR 引物序列。
功能虽然都很简单,但非常实用,一般的用户都可以很方便地使用。
4. Premier5.0是由加拿大的Premier公司开发的专业用于PCR或测序引物以及杂交探针的设计和评估的软件,和Plasmid Premier2.02一起是该公司推出的最新的软件产品。

常用生物信息学软件一、基因芯片1、基因芯片综合分析软件。
ArrayVision 7.0 一种功能强大的商业版基因芯片分析软件,不仅可以进行图像分析,还可以进行数据处理,方便protocol的管理功能强大,商业版正式版:6900美元。
Arraypro 4.0 Media Cybernetics公司的产品,该公司的gelpro, imagepro一直以精确成为同类产品中的佼佼者,相信arraypro也不会差。
phoretix™ Array Nonlinear Dynamics公司的基因片综合分析软件。
J-express 挪威Bergen大学编写,是一个用JA V A语言写的应用程序,界面清晰漂亮,用来分析微矩阵(microarray)实验获得的基因表达数据,需要下载安装JA V A运行环境JRE1.2后(5.1M)后,才能运行。
2、基因芯片阅读图像分析软件ScanAlyze 2.44 ,斯坦福的基因芯片基因芯片阅读软件,进行微矩阵荧光图像分析,包括半自动定义格栅与像素点分析。
输出为分隔的文本格式,可很容易地转化为任何数据库。
3、基因芯片数据分析软件Cluster 斯坦福的对大量微矩阵数据组进行各种簇(Cluster)分析与其它各种处理的软件。
SAM Significance Analysis of Microarrays 的缩写,微矩阵显著性分析软件,EXCEL软件的插件,由Stanford大学编制。
4.基因芯片聚类图形显示TreeView 1.5 斯坦福开发的用来显示Cluster软件分析的图形化结果。
现已和Cluster成为了基因芯片处理的标准软件。
FreeView 是基于JA V A语言的系统树生成软件,接收Cluster生成的数据,比Treeview 增强了某些功能。
5.基因芯片引物设计Array Designer 2.00 DNA微矩阵(microarray)软件,批量设计DNA和寡核苷酸引物工具三、序列综合分析V ector NTI Suite 8.0 不喜欢装备各种专业性强的软件,而希望用一个综合性的软件代替的同志可以选择本软件。


ANTHEPROT 5.0简单使用说明生物技术2002 房磊021402144生物的化学,就是蛋白质与核酸的化学。
对蛋白质的研究是生物化学领域一个非常重要的部分。
但是众多蛋白质的复杂序列数据,其分析工作是一个非常困难的工作。
用人工的方法是很难完成如此大量的分析工作的。
运用计算机,利用一定的运算规则,对众多的蛋白质序列数据,进行蛋白序列分析就是最好的解决方法。
蛋白序列分析软件包ANTHEPROT 5.0正是这样的一个程序包。
蛋白序列分析软件包ANTHEPROT 5.0是法国的蛋白质生物与化学研究院(Institute of Biology and Chemistry of Proteins)开发的蛋白质研究软件包。
软件包包括了蛋白质研究领域所包括的大多数内容,功能非常强大。
应用此软件包,使用个人电脑,便能进行各种蛋白序列分析与特性预测。
包括:进行蛋白序列二级结构预测;在蛋白序列中查找符合PROSITES数据库的特征序列;绘制出蛋白序列的所有理化特性曲线;在Internet或本地蛋白序列数据库中查找类似序列;计算蛋白序列分子量,比重与各蛋白残基百分组成;计算蛋白序列滴定曲线与等电点;选定一个片段后,绘制Helical Wheel图;进行点阵图(Dot Plot)分析;计算信号肽潜在的断裂位点等功能。
更重要的是此软件包为免费软件。
软件分为三个版本,分别用于Unix系统,DOS系统与WINDOWS95或98系统,以用于WINDOWS系统的版本为例,介绍一下这个软件的基本用法。
软件包为一个自解压执行文件,文件名Anthe_5_0.exe,大小为2,013,184 字节。
执行此文件,输入解压后存放的目录名,便可将所有文件解压在此目录下。
其文件如下图软件运行后的主程序中按钮是极其简单的相关介绍,基本无任何用处。
但是在软件目录中的自解压压缩文件doc_ant.exe文件,可以解压在另一个目录,是HTML格式的介绍文件,对软件进行了一些较详细说明介绍。
常用分子生物学软件一、基因芯片:1、基因芯片综合分析软件。
ArrayVision 7.0一种功能强大的商业版基因芯片分析软件,不仅可以进行图像分析,还可以进行数据处理,方便protocol的管理功能强大,商业版正式版:6900美元。
Arraypro 4.0Media Cybernetics公司的产品,该公司的gelpro, imagepro一直以精确成为同类产品中的佼佼者,相信arraypro也不会差。
phoretix™Array Nonlinear Dynamics公司的基因片综合分析软件。
J-express挪威Bergen大学编写,是一个用JAVA语言写的应用程序,界面清晰漂亮,用来分析微矩阵(microarray)实验获得的基因表达数据,需要下载安装JAVA运行环境JRE1.2后(5.1M)后,才能运行。
2、基因芯片阅读图像分析软件ScanAlyze 2.44,斯坦福的基因芯片基因芯片阅读软件,进行微矩阵荧光图像分析,包括半自动定义格栅与像素点分析。
输出为分隔的文本格式,可很容易地转化为任何数据库。
3、基因芯片数据分析软件Cluster斯坦福的对大量微矩阵数据组进行各种簇(Cluster)分析与其它各种处理的软件。
SAMSignificance Analysis of Microarrays 的缩写,微矩阵显著性分析软件,EXCEL软件的插件,由Stanford大学编制。
4.基因芯片聚类图形显示TreeView 1.5斯坦福开发的用来显示Cluster软件分析的图形化结果。
现已和Cluster成为了基因芯片处理的标准软件。
FreeView是基于JAVA语言的系统树生成软件,接收Cluster生成的数据,比Treeview增强了某些功能。
5.基因芯片引物设计Array Designer 2.00DNA微矩阵(microarray)软件,批量设计DNA和寡核苷酸引物工具二、RNA二级结构。
RNA Structure 3.5RNA Sturcture 根据最小自由能原理,将Zuker的根据RNA一级序列预测RNA二级结构的算法在软件上实现。
三维分子类RASMOL 2.7.2.1 观看生物分子3D微观立体结构的软件。
非常有名,巨棒!RasTop 2.0 为RasMol 2.7.1的图形用户界面软件CHIME 2.6 SP3 直接在浏览器中观看3D分子。
MolMol 2k.1 将pdb等格式的蛋白文件通过微调,存成普通的图形文件。
CrystInfo 1.0 用来快速、容易地构建、观察与检查晶体3d结构。
PDViewer PDB格式文件的查看程序。
Weblab Viewlite 4.2 3维分子浏览工具及大量分子文件例子。
Weblab ViewerPro 4.2 Demo 3维分子浏览工具。
ICMLite 2.8 3维分子浏览工具,有一些其他软件没有的功能。
VMD 1.72 3维分子浏览工具,可以进行动态显示。
CN3D 3.0 3D分子结构观察软件。
WPDB 2.2 PDB文件检索显示分析软件。
DTMM 4.0 3维分子模型显示、编辑与构建程序。
Mole 1.1.8 Demo 高性能的大分子3维图形显示计算工具。
gopenmol 2.1 显示并分析分子结构及其特性的软件。
POV-Ray 3.5 beta rc5 生成三维图像工具软件。
WinMegaPov 0.7 3D渲染软件POV-Ray非官方编译软件。
MolPOV 2.0.8 将PDB文件转化为POV格式文件的软件。
Mol2Mol Demo 4.1 分子文件格式转换软件。
PovChem 2.1.1 将PDB文件转化为POV格式文件的软件。
Ortep-3 for Windows 1.074 生成分子的热椭圆形点图软件。
PLATON(2002.5.16版) 通用结晶学软件工具。
Mage 6.02 读取并演示Kinemage格式文件的专用软件。
Prekin 6.02 将PDB格式文件转换为Kinemage格式文件的软件。
Swiss-PdbViewer 3.7 PDB文件显示与分析软件。
DINAMO 蛋白序列排队比较编辑与三维模型构建工具软件。
生物的化学,就是蛋白质与核酸的化学。
对蛋白质的研究是生物化学领域一个非常重要的部分。
近年来,通过实施基因组计划,得到了大量的蛋白序列数据,但是,面对如此众多的蛋白质序列数据,其分析工作是一个非常困难的工作。
用人工的方法是不可能完成如此大量的分析工作的。
运用计算机,利用一定的运算规则,进行蛋白序列分析是唯一的方法。
蛋白序列分析软件包ANTHEPROT 4.3正是这样的一个程序。
蛋白序列分析软件包ANTHEPROT 4.3是位于法国的蛋白质生物与化学研究院(Institute of Biology and Chemistry of Proteins)用十多年时间开发出的蛋白质研究软件包。
软件包包括了蛋白质研究领域所包括的大多数内容,功能非常强大。
应用此软件包,使用个人电脑,便能进行各种蛋白序列分析与特性预测。
感谢软件的编写者,他们将此软件包免费提供给此领域的同行使用,这对于我国的科学技术人员来说,实在是一件好事。
因为一个功能再多再好的专业应用软件,如果作为商业软件进行销售的话,往往使大多数需要使用此软件的科技人员因为科研经费的问题,无缘使用,这点在我国尤为突出。
软件分为三个版本,分别用于Unix系统,DOS系统与WINDOWS95或98系统,让我以用于WINDOWS系统的版本为例,介绍一下这个软件的基本用法。
软件包为一个自解压执行文件,文件名Anthe4_3c.exe,大小为1.9兆。
执行此文件,输入解压后存放的目录名,便可将所有文件解压在此目录下。
主程序名为Anthewin,在桌面上建立它的快捷图标,双击快捷图标便打开了ANTHEPROT 4.3主窗口。
通过主程序,我们可以载入蛋白序列,对序列进行编辑、打印、拷贝、改变设定等操作,更重要的是,我们可以在此调用各种所需的分析工具,对蛋白序列进行分析。
一.序列编辑功能ANTHEPROT 4.3的主窗口便是其序列编辑窗口:可以使用按键或菜单中的File/Open命令打开各种序列文件,程序识别的文件类型包括:单序列文件格式:*.SEQ,(ANTHEPROT 3.3支持以下格式的单序列文件:DNA/Strider 格式、EMBL格式、NBRF格式、Pearson/Fasta 格式、PIR 格式与IG/Stanford格式);含有多个蛋白序列的蛋白数据库文件:*.BAS,(以Pearson/Fasta 格式添加序列,上限为30条序列或32K文件大小);ClustalV 或ClustalW 文件:*.ALN 含有ClustalW格式的多队列;含有多队列的Multalin 4.1格式文件:*.MUL;在Prosite 蛋白数据库中查寻出Site结果的文件:*.SIT,蛋白质原子空间结构的PDB格式文件:*.PDB;使用IBCP(http://wwww.ibcp.fr/predict.html)服务器预测蛋白二级结构所获得的结果文件格式:*.CNS;序列可以从ANTHEPROT编辑窗口以键盘输入,或者以*.SEQ 等文件格式载入,也可以从其它文件复制粘贴到编辑窗口。
生物学软件大全(精)分子生物学软件介绍目前,有很多软件可以解决分子生物学研究人员从立项到最后写论文的实际问题。
但由于软件的数目太多,而且各个软件开发环境、运行平台和操作方法都各不相同——即使功能的某些方面是相似的,这就给使用者造成了许多的不便。
赛百盛公司信息部在对相同作用的各类软件进行比较后,推荐一些最实用软件给从事生物研究的工作人员]一、实验准备阶段这时一般要查一些与实验相关的文献以便对自己所要做的课题的最新的进展有一个基本的了解,从而确定自己的实验策略。
推荐使用软件Manager 9.0可以在线通过查找搜索宋体; "Times New Roman"‘>数据库中的专业资料,同时保存查找的资料为本地文件。
资料内容和记录分上下两个屏幕,如有全文或想连接网络时敲键盘就可以到相应的全文文章和。
可以直接在WORD中查找资料,并插入引用。
在文章中对引用的文献可以格式化,引用的参考资料格式有很强的用户自定义功能,可以符合各种杂志对引用格式的要求,引用时不用多窗口切换。
同类软件:Endnote 3.1.2 也是一个在线专业资料查找系统,可以保存查找资料,并在文章中对引用格式化.二、实验实施阶段随着实验的进行,就必须对实验过程中的DNA、RNA和蛋白质的信息进行各种处理,包括限制酶分析、引物设计、同源序列比较、质粒作图、结构域(motif)查找、RNA二级结构预测、蛋白二级结构分析、三维结构显示等方面的内容。
1、综合软件推荐软件:Omiga 2.0实际上,大部分对核酸蛋白的序列分析功能,在Omiga 2.0中都能找到;而且界面非常友好。
Omiga作为强大的蛋白质、核酸分析软件,它还兼有引物设计的功能。
主要功能:编辑、浏览、蛋白质或核酸序列,分析序列组成。
用Clustal. W进行同源序列比较,发现同源区。
实现了核酸序列与其互补链之间的转化,序列的拷贝、删除、粘贴、置换以及转化为RNA链,以不同的读码框、遗传密码标准翻译成蛋白质序列。
Antheprot:蛋白质序列分析软件包ANTHEPROT 4.5是位于法国的蛋白质生物与化学研究院(Institute of Biology and Chemistry of Proteins)用十多年时间开发出的蛋白质研究软件包。
软件包包括了蛋白质研究领域所包括的大多数内容,功能非常强大。
应用此软件包,使用个人电脑,便能进行各种蛋白序列分析与特性预测。
更重要的是该软件能够提供蛋白序列的一些二级结构信息,使用户有可能模拟出未知蛋白的高级结构。
Applied Biosystems Primer Express:这是ABI公司销售附送的软件,可用于设计引物和探针,尤其适用于荧光PCR探针的设计,可以精确计算寡核苷酸与荧光基团鳌合后的Tm 值。
可以预测引物与引物之间与模板之间等的二级结构。
Artemis R5:A DNA sequence viewer and annotation tools,一个DNA序列查看器与注释工具,可以以图形形式查看序列的各种分析结果与特性,程序读取EMBL与GENBANK格式的序列与纯DNA序列。
以Java写成,需要安装JRE1.2。
BioEdit是一个序列编辑器与分析工具软件,功能非常强大,使用十分容易。
功能包括:序列编辑、外挂分析程序、RNA分析、寻找特征序列、支持超过20000个序列的多序列文件、基本序列处理功能、质粒图绘制等等。
BLAST (Basic Local Alignment Search Tool)是一套在蛋白质数据库或DNA数据库中进行相似性比较的分析工具。
BLAST程序能迅速与公开数据库进行相似性序列比较。
BLAST 结果中的得分是对一种对相似性的统计说明。
BLAST对一条或多条序列(可以是任何形式的序列)在一个或多个核酸或蛋白序列库中进行比对。
BLAST还能发现具有缺口的能比对上的序列。
BLAST是基于Altschul等人在J.Mol.Biol上发表的方法(J.Mol.Biol.215:403-410(1990)),在序列数据库中对查询序列进行同源性比对工作。
从最初的BLAST发展到现在NCBI提供的BLAST2.0,已将有缺口的比对序列也考虑在内了。
BLAST可处理任何数量的序列,包括蛋白序列和核算序列;也可选择多个数据库但数据库必须是同一类型的,即要么都是蛋白数据库要么都是核酸数据库。
所查询的序列和调用的数据库则可以是任何形式的组合,既可以是核酸序列到蛋白库中作查询,也可以是蛋白序列到蛋白库中作查询,反之亦然。
1、BLASTP是蛋白序列到蛋白库中的一种查询。
库中存在的每条已知序列将逐一地同每条所查序列作一对一的序列比对。
2、BLASTX是核酸序列到蛋白库中的一种查询。
先将核酸序列翻译成蛋白序列(一条核酸序列会被翻译成可能的六条蛋白),再对每一条作一对一的蛋白序列比对。
3、BLASTN是核酸序列到核酸库中的一种查询。
库中存在的每条已知序列都将同所查序列作一对一地核酸序列比对。
4、TBLASTN是蛋白序列到核酸库中的一种查询。
与BLASTX相反,它是将库中的核酸序列翻译成蛋白序列,再同所查序列作蛋白与蛋白的比对。
5、TBLASTX是核酸序列到核酸库中的一种查询。
此种查询将库中的核酸序列和所查的核酸序列都翻译成蛋白(每条核酸序列会产生6条可能的蛋白序列),这样每次比对会产生36种比对阵列。
Clone:与primer同属一类软件,软件,绿色,无需安装。
可进行酶切图谱分析,质粒描绘。
Clustal X 用来对核酸与蛋白序列进行多序列比较(multiple sequence alignment)的软件。
多序列比较在分子生物学中是一个基本方法,用来发现特征序列,进行蛋白分类,证明序列间的同源性,帮助预测新序列二级结构与三级结构,确定PCR引物,以及在分子进化分析方面均有很大帮助,Clustal X很适合这些方面的要求。
DNAClub:DNA Club是一个简单的对DNA进行与PCR有关的操作的软件。
它的功能有:1、输入DNA序列;2、查找ORF序列;3、把DNA翻译成蛋白序列;4、查找酶切位点;5、查找PCR引物序列。
功能虽然都很简单,但非常实用,一般的用户都可以很方便地使用。
DNAMan:综合分析软件,可进行限制酶分析,引物设计,对排(aligment),翻译,数据库操作,Blast,序列装配(Sequence assembly)。
DNAMend DNAmend是一名德国人编写的用于对DNA序列进行分析,编辑的专业软件,主要的用途是在基因克隆过程中,利用其提供的对DNA序列的限制性酶切分析,开读框搜索,序列转译,末端修剪等功能对DNA序列进行酶切、连接、末端补平等模拟操作,为克隆流程设计及克隆过程监视提供直观的控制工具,便于对克隆中可能出现的问题进行分析,并可将结果输出用于发表文章。
DNAprobe:为了确定蛋白家族新成员基因,根据蛋白序列对比设计核苷酸序列。
DNASIS 2.5。
DNASIS for Windows 2.5版是日立软件公司(Hitachi Sofeware Engineering Co.,Ltd.)97年推出的一个功能强大的序列分析软件。
包含有大部分分子生物学软件的常用功能,可进行DNA,RNA,蛋白质序列的编辑和分析,甚至还能进行质粒作图、数据库查询等功能,足可满足一般实验室的要求。
在DOS时代,DNASIS 7等版本便是流传甚广并曾给过许多人以帮助的分子生物学软件,因此我们有理由期待Win版的DNASIS 会带给我们惊喜。
DNAsisMaxInstall 是DNASIS的完全改版版本,风格转变很大,功能也十分强大,几乎包含所有分子生物学研究所需的功能,当然体积也大了好多好多。
DNAStar:即著名的Lasergene Suite,由EditSeq MegAlign、GeneQuest MapDraw PrimerSelect Protean SeqMan II七个模块组成,该软件的MegAlign模块,可以对多达64000的片段进行拼装。
整个拼装过程即时显示,并提示可能的完成时间。
拼装结果采用序列、策略等方式显示。
DNAstar是哈佛大学医学院是使用的序列分析软件,可见其功能强大。
DNATools 5.1 与Omiga, DNAsis, PCgene等软件属于同一类的综合性软件,操作简单功能多。
DNATools设计的用户友好、强壮,以便快速、方便地获取、贮藏和分析序列及数据库查询获得的序列相关信息。
DNATools包容性很好,能把几乎所有文本文件打开作为序列。
当程序不能辨别序列的格式时(通过寻找常用序列格式的特征),会显示这个文件的文本形式,以便你编辑生成正确的蛋白质或DNA序列,编辑后可以再被载入程序。
若你的序列是DNATools格式时(DNA或寡核苷酸序列),程序不加注解的载入序列,程序模式调整成可以接受载入的数据类型(蛋白质、DNA和寡核苷酸引物序列)。
在一个项目中可以加入几千个序列或引物,并在整个项目中分析这些序列及标题。
这个程序的一个特点是给每个序列或引物添加文本标题。
这样就可以用自定义的标题识别序列,而不必通过它们的文件名。
为避免丢失数据-和你的工作-DNA Tools包括几个挽救丢失数据的功能:一个5层的撤销/重复功能,重新获得原始序列的恢复功能,项目中加入新文件时的安全备份功能,以及在一定的时间间隔作完全备份或备份你的工。
fasta33t07:在Internet上有许多的在线FASTA查找服务,查找某数据库中的同源序列。
也可下载后离线使用。
将一条序列与另一条序列进行比较或在数据库中查找同源序列并输出。
MACAW 2.05多序列构建与分析工作台软件(Multiple Alignment Construction & Analysis Workbench, MACAW)是一个用来构建与分析多序列片段的交互式软件。
MACAW具有几个特点:1. 新的搜索算法查寻类似区,消除了先前技术的许多限制。
2. 应用一个最近发展的数学原理计算block类似性的统计学显著性。
3. 使用各种视图工具,可以评估一个候选block包含在一个多序列中的可能性。
4.可以很容易地编辑每一个block。
在多序列中查找一个类似片段并不是一件简单的事,主要是因为要查找的量极大。
这正式MACAW所要解决的问题。
Mega2.1:分子进化遗传分析软件,可进行进化聚类分析。
该版本对使用界面做了优化,并有多种统计学算法。
NetPrimer:JA V A语言写成的引物设计软件,用IE打开运行,需要安装JA V A Runtime Environment 1.4,11.7M。
WEB界面的引物设计程序,包括JA V A程序和帮助文件,解压后,可在本地直接使用,不须再连到原始网站使用。
Oligo 2.7M,引物分析著名软件,主要应用于核酸序列引物分析设计软件,同时计算核酸序列的杂交温度(Tm)和理论预测序列二级结构。
Omiga 2.0:实际上,大部分对核酸蛋白的序列分析功能,在Omiga 2.0中都能找到;而且界面非常友好。
Omiga作为强大的蛋白质、核酸分析软件,它还兼有引物设计的功能。
主要功能:编辑、浏览、蛋白质或核酸序列,分析序列组成。
用Clustal. W进行同源序列比较,发现同源区。
实现了核酸序列与其互补链之间的转化,序列的拷贝、删除、粘贴、置换以及转化为RNA链,以不同的读码框、遗传密码标准翻译成蛋白质序列。
查找核酸限制性酶切位点、基元(Motif)及开放阅读框(ORF),设计并评估PCR、测序引物。
查找蛋白质解蛋白位点(Proteolytic Sites)、基元、二级结构等。
查寻结果可以以图谱及表格的显示,表格设有多种分类显示形式。
利用Mange快捷键,用户可以向限制性内切酶、蛋白质或核酸基元、开放阅读框及蛋白位点等数据库中添加或移去某些信息。
每一数据库中都设有多种查寻参数,可供选择使用。
用户也可以添加、编辑或自定义某些查寻参数。
可从MacVectorTM、Wisconsin PackageTM等数据库中输入或输出序列。
另外,该软件还提供了一个很有特色的类似于核酸限制酶分析的蛋白分析,对蛋白进行有关的多肽酶处理后产生多肽片段。
Pcgene:在C盘根目录下执行pcgene.exe,按提示安装好(一般回答y),在C:\pcgene 目录下执行pcgene.bat,group name :fudan,password:19730404。
pcgeen的简单说明介绍以及原始网址。
注意:pcgene可以在ms-dos5.0 6.0 6.20 6.22下运行。
在ms-dos7.x&windows4.0 或者说windows9x的dos下运行修改方法,请点击此处。