高通量测序的生物信息学分析报告
- 格式:doc
- 大小:1.17 MB
- 文档页数:10

高通量测序技术的生物信息学分析引言:高通量测序技术作为一种新型基因测序技术,已经被广泛应用于现代生物研究中。
利用高通量测序技术,科学家们可以快速解码基因组序列、转录组序列以及蛋白质组序列。
然而,高通量测序技术不仅仅是一种实验技术,它也需要强大的生物信息学工具来支持数据分析和解读。
本文将介绍高通量测序技术的生物信息学分析,包括原始数据质量控制、序列比对和注释、基因表达分析、基因变异分析等等。
一、原始数据质量控制原始数据质量控制是高通量测序数据分析的第一步,其目的是剔除低质量序列以及包含污染序列的读段,确保下游的数据分析结果准确可靠。
利用FASTQC等软件对原始的FASTQ格式数据进行质量评估,可以得到关于以下几个质量指标的信息:1. GC含量2. Q20和Q30的比例3. 碱基分布的均匀性4. 过度重叠序列的比例基于以上质量指标,可以对数据进行质量控制处理,包括碱基修剪、低质量序列过滤、去除污染序列等等。
二、序列比对和注释序列比对指的是将高通量测序数据基因组参考序列进行比对,得到参考序列上的SNP、InDel的信息,从而对样品进行全面的基因变异检测、注释和分析。
在序列比对和注释的过程中,需要正确选择合适的比对软件和参考序列。
目前较为流行的比对软件包括BWA、Bowtie、STAR等等。
针对RNA-seq数据的注释工具包括Cufflinks、StringTie、Transcriptome Assembly等等。
基于参考序列的比对结果,还可以利用Variant Effect Predictor (VEP)等工具对候选变异位点进行注释。
注释信息包括dbSNP、ClinVar、ExAC等公共数据库的信息,帮助生物学家了解该变异的生物学特性,并识别其潜在的影响。
三、基因表达分析高通量测序技术还可以用于RNA表达谱的分析,以揭示不同组织和不同发育阶段的基因表达差异。
在基因表达分析中,首先将RNA-seq数据进行质量控制和过滤,然后对序列进行比对和注释,获得基因的计数信息。

生物信息学中的高通量测序数据分析研究在生物学领域中,随着人们对生物系统的研究日益深入,高通量测序技术的出现为基因和蛋白质序列的鉴定和分析提供了非常有效的手段。
高通量测序数据分析研究作为生物信息学领域中的一个重要分支,以其高效、快速、准确的特性,不断地吸引着研究者的关注。
高通量测序技术是指一种能够同时测定大量DNA序列的方法,它不仅能够用于基因组组装和注释,还可以通过RNA测序技术来分析基因表达。
这种技术极大地加快了生物学研究的速度和效率,为生物学家提供了大量的测序数据。
然而,这些数据的处理和分析却是非常复杂的。
首先,高通量测序数据的预处理是数据分析的第一步。
当测序数据被生成后,必须对其进行质量控制、过滤低质量序列、去除接头序列等处理,才能得到较为准确的数据。
此外,还有一些其他的预处理过程,例如去重、去序列污染等。
接着,对于各类高通量测序数据进行生物信息学分析非常关键。
生物信息学分析的主要目的是确定基因或蛋白质序列,并了解它们在不同生理状态下的表达和功能。
对于RNA测序数据,其主要方法是将原始数据经过拼接或比对成转录本,然后对得到的转录本进行表达量分析,从而确定不同表达和差异表达基因的信息。
而对于DNA测序数据,则可通过基因组拼装、变异分析、基因预测和功能注释等方法进行深入研究。
近年来,生物信息学领域发展迅速,高通量测序数据分析也成为了该领域中的一个热门研究方向。
许多学者正在研究开发更加准确、更加高效、更加专业的分析工具和方法。
例如,结合机器学习技术的表达量分析方法和差异表达基因分析,能够更加准确地发现差异表达的基因;metagenomics(环境微生物组学)领域,则还需要研究多样性分析、代谢通路分析等更为复杂的问题。
总之,高通量测序数据分析是一个关键的研究领域,它为更深入、全面的生物学研究提供了非常重要的工具和方法。
未来,生物信息学领域需要进一步发展,带来更多的高精度、高效率的数据处理和分析技术,以满足更严格的生物学研究需求。

测序分析报告1. 引言测序分析是一种通过高通量测序技术获得生物样本基因信息的方法,广泛应用于基因组学、转录组学和蛋白质组学等研究领域。
本报告旨在对测序分析的过程和方法进行简要介绍,并展示测序分析的主要应用和发展趋势。
2. 测序分析的基本步骤测序分析一般包括样本准备、测序实验、数据处理和数据分析四个步骤。
2.1 样本准备样本准备是测序分析的第一步,需要从样本中提取RNA或DNA等核酸物质。
具体的方法通常根据研究目的和样本类型的不同而有所差异,常见的样本准备方法包括基因组DNA提取、总RNA提取和单细胞RNA提取等。
2.2 测序实验测序实验是测序分析的核心步骤,主要通过高通量测序技术获取样本中核酸序列的信息。
常用的测序技术包括Sanger测序、454测序、Illumina测序和Ion Torrent测序等。
每种测序技术都有其优势和局限性,研究者需要根据实际需求选择合适的测序平台和方法。
2.3 数据处理数据处理是测序分析的关键步骤,包括原始数据的质量控制、去除低质量序列和适配体序列的修剪,以及将测序数据转化为可供后续分析的格式等。
常用的数据处理工具和软件包括FASTX-Toolkit、Trimmomatic和Cutadapt等。
2.4 数据分析数据分析是测序分析的最后一步,主要针对测序数据进行生物信息学分析和统计学分析。
常见的数据分析内容包括序列比对、基因表达分析、变异检测和功能注释等。
为了完成这些分析,研究者需要使用一系列生物信息学工具和软件包,如Bowtie、STAR、DESeq2和GATK等。
3. 测序分析的主要应用测序分析在生物医学研究、农业科学和环境科学等领域具有广泛的应用价值。
3.1 基因组学研究测序分析在基因组学研究中扮演着重要角色,可以帮助研究者揭示物种的遗传多样性、功能基因组学和进化基因组学等问题。
通过测序分析,研究者可以对各个物种的基因组进行比较和注释,从而深入理解基因组的结构和功能。

生物信息学中的高通量基因测序数据处理与分析随着高通量基因测序技术的发展,大量的基因测序数据得以产生。
这些数据对于生物信息学的研究和应用具有重要意义,因此高通量基因测序数据的处理与分析成为了生物信息学领域的重点之一。
高通量基因测序数据处理是指对原始的测序数据进行加工、清洗和预处理的过程。
首先,需要将测序数据从测序仪中读取出来,得到序列文件。
接着,需要对序列文件进行质量控制,去除低质量序列,过滤掉可能的污染和重复序列。
其次,对于RNA测序数据,需要对序列进行去除adaptor序列、多态性核苷酸等预处理步骤。
最后,对于基因组测序数据,还需要进行比对到参考基因组的工作。
高通量基因测序数据的处理过程中,还需要注意到错误和偏倚的存在。
测序错误可以来源于测序仪的误差,也可以来源于PCR扩增的偏差。
针对这些问题,研究人员可以利用错误纠正算法和统计模型来识别和修复错误的测序数据。
同时,也可以通过样本间和实验间的重复测序来评估和控制测序的偏差。
处理完高通量基因测序数据之后,研究人员就可以进一步进行数据分析。
高通量基因测序数据的分析主要包括基因定量和差异表达分析、基因组注释和变异分析、及关联分析和机器学习等。
基因定量分析可以通过计算读数或转录本的丰度来研究基因的表达模式;差异表达分析可以用于比较不同条件或组织中基因的表达差异,从而找出与特定生物过程或疾病相关的基因。
基因组注释分析可以将基因定位到基因组中的特定位置,并评估基因功能和调控元件的存在。
变异分析可以用于检测和注释基因组中的突变和多态性,研究其与疾病相关性,以及对个体差异的贡献。
关联分析和机器学习可以挖掘大规模测序数据中的相关性和模式,为生物学研究提供新的理解和预测。
为了更好地处理和分析高通量基因测序数据,研究人员还需要掌握一些常用的生物信息学工具和算法。
例如,常用的序列比对算法包括BLAST、Bowtie、BWA等;基因定量和差异表达分析可使用DESeq2、edgeR、limma 等;基因组注释可利用Ensembl、NCBI、UCSC等数据库和工具。

基于高通量测序技术的生物信息学解读高通量测序技术是近年来生命科学和医学领域的重要技术之一,通过对生物样品进行深度测序,能够快速、准确地获取大量生物信息,为基因功能研究、药物研发、疾病诊断和治疗等方面提供了有力支撑。
生物信息学解读是高通量测序技术的重要应用方向之一,涉及到基因组、转录组、蛋白质组等多个层次的分析,具有广泛的研究意义和应用前景。
一、基因组测序基因组测序是高通量测序技术的首要应用方向之一,它能够帮助我们了解生物基因组的组成、结构和功能,为基因功能研究、进化分析等提供数据支持。
与传统方法相比,基因组测序能够在较短时间内对生物基因组进行全面测序,解决了单个基因或单个基因片段测序的限制性问题,提供了更加全面的基因数据。
基因组测序分为宏基因组和微基因组两种。
宏基因组是对各种微生物和大量环境中存在的微生物进行的基因组测序,其目的是揭示微生物种类、多样性、功能等。
微基因组则是对个体或种群的基因组进行的测序,不少研究工作集中在人类基因组的测序上。
基因组测序需要进行序列的拼接、比对、注释等信息学处理才能明确基因组结构和组成。
二、转录组测序转录组是指在细胞内基因转录生成mRNA的总体酶同一时刻产生的所有mRNA分子的总和。
与基因组测序相比,转录组测序可以更加全面地了解生物转录水平的变化。
通过测定生物转录组,可以揭示生物发育、生长、适应环境变化等方面的生物学规律。
在转录组测序中,从样品中提取RNA,然后通过转录组测序技术,对RNA进行深度测序,将结果转化为数字信号,然后进行数据分析和注释,包括基因差异表达分析、聚类分析、功能富集分析等,挖掘生物转录组的生物学意义和作用。
三、甲基化测序在生物体中,DNA甲基化是表观遗传学研究中一个重要的表征,也是人类疾病诊断和治疗的关键因素。
然而,甲基化在不同细胞、组织和环境条件下是动态变化的。
因此,甲基化测序技术可以用来研究DNA甲基化的变化以及与这些变化相关的生物学过程,如基因表达和细胞分化等。

高通量测序生物信息学分析
高通量测序技术产生的DNA序列数据长度较短,而且数据量非常巨大。
分析了高通量测序环境下大数据的挑战和机遇,总结并讨论了数据压缩、宏基因组数据序列拼接、宏基因组数据序列分析方面的算法和工具等研究成果。
最后,展望了高通量测序下DNA短读序列数据研究的发展趋势。
高通量测序分析高通量测序,一次性对几百万到十亿条DNA分子进行并行测序,又称为下一代测序技术,其使得可对一个物种的转录组和基因组进行深入、细致、全貌的分析,所以又被称为深度测序。
主要包括:High-throughput Sequencing,Next Generation Sequencing,Deep Sequencing。
图1 高通量测序流程
高通量测序应用范围广泛:1 DNA测序:全基因组de novo测序,基因组重测序,宏基因组测序,人类外显子组捕获测序。
2 RNA测序:转录组测序,小RNA测序,电子表达谱测序。
3 表观基因组研究:ChIP-Seq,DNA甲基化测序。
基因组测序
基因组测序是对物种的基因组DNA打断后进行高通量测序,根据是否有已知基因组数据主要分为de novo全基因组测序和基因组重测序。
De novo 基因组测序是对未知基因组序列的物种进行基因组从头测序,利用生物信息学分析手段对序列进行拼接、组装,从而获得该物种的基因组图谱。
全基因组重测序是对已知基因组序列的物种进行不同个体的基因组测序,并在此基础上对个体或群体进行差异性分析。
图2 基因组测序策略
图3 Paired-end原理。
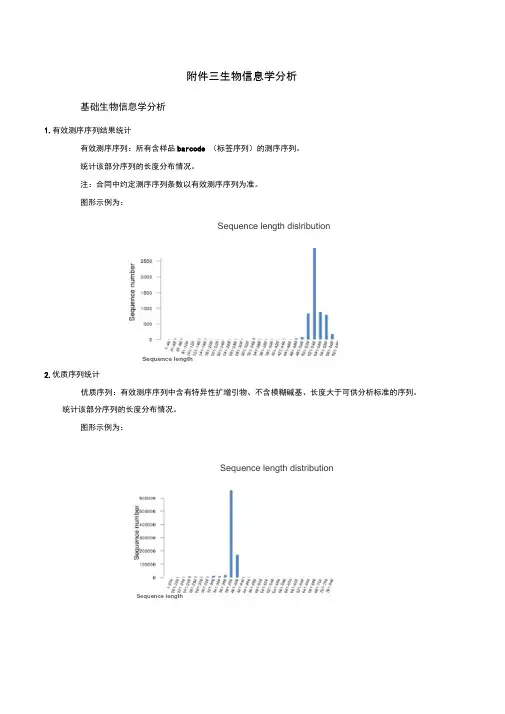
附件三生物信息学分析基础生物信息学分析1.有效测序序列结果统计有效测序序列:所有含样品barcode (标签序列)的测序序列。
统计该部分序列的长度分布情况。
注:合同中约定测序序列条数以有效测序序列为准。
图形示例为:Sequence length dislributionSequence length2.优质序列统计优质序列:有效测序序列中含有特异性扩增引物、不含模糊碱基、长度大于可供分析标准的序列。
统计该部分序列的长度分布情况。
图形示例为:Sequence length distributionSequence length3.各样本序列数目统计:统计各个样本所含有效测序序列和优质序列数目。
结果示例为:4. OTU生成:根据序列的相似性,将序列归为多个OTU(操作分类单元),以便后续分析。
5. 稀释曲线(rarefaction 分析)根据第4条中获得的OTU数据,做出每个样品的Rarefaction 曲线。
本合同默认生成OTU相似水平为0.03 的rarefaction 曲线。
rarefaction 曲线结果示例:Number of R«ads SampladM; 0.036. 指数分析计算各个样品的相关分析指数,包括:丰度指数:ace'chao多样性指数: sha nnon\simps on本合同默认生成OTU 相似水平为0.03的上述指数值。
多样性指数分析结果示例:IDR HM 4H机MOTU■chM|A2W0MO ICNMtlOOO.iOM}S(KS) J O L WCO,UM亠帼血期’ th注:默认分析以上所列指数,如有特殊需要请说明7. Shannon-Wiener 曲线利用各样品的测序量在不同测序深度时的微生物多样性指数构建曲线, 反映各样本在不同测序数量 时的微生物多样性。
当曲线趋向平坦时, 说明测序数据量足够大, 可以反映样品中绝大多数的微生物信 息。
绘制默认水平为:0.03。

基因组测序基础知识㈠De Novo测序也叫从头测序,是首次对一个物种的基因组进行测序,用生物信息学的分析方法对测序所得序列进行组装,从而获得该物种的基因组序列图谱。
目前国际上通用的基因组De Novo测序方法有三种:1. 用Illumina Solexa GA IIx 测序仪直接测序;2. 用Roche GS FLX Titanium直接完成全基因组测序;3. 用ABI 3730 或Roche GS FLX Titanium测序,搭建骨架,再用Illumina Solexa GA IIx进行深度测序,完成基因组拼接。
采用De Novo测序有助于研究者了解未知物种的个体全基因组序列、鉴定新基因组中全部的结构和功能元件,并且将这些信息在基因组水平上进行集成和展示、可以预测新的功能基因及进行比较基因组学研究,为后续的相关研究奠定基础。
实验流程:公司服务内容1.基本服务:DNA样品检测;测序文库构建;高通量测序;数据基本分析(Base calling,去接头,去污染);序列组装达到精细图标准2.定制服务:基因组注释及功能注释;比较基因组及分子进化分析,数据库搭建;基因组信息展示平台搭建1.基因组De Novo测序对DNA样品有什么要求?(1) 对于细菌真菌,样品来源一定要单一菌落无污染,否则会严重影响测序结果的质量。
基因组完整无降解(23 kb以上), OD值在1.8~2.0 之间;样品浓度大于30 ng/μl;每次样品制备需要10 μg样品,如果需要多次制备样品,则需要样品总量=制备样品次数*10 μg。
(2) 对于植物,样品来源要求是黑暗无菌条件下培养的黄化苗或组培样品,最好为纯合或单倍体。
基因组完整无降解(23 kb以上),OD值在1.8~2.0 之间;样品浓度大于30 ng/μl;样品总量不小于500 μg,详细要求参见项目合同附件。
(3) 对于动物,样品来源应选用肌肉,血等脂肪含量少的部位,同一个体取样,最好为纯合。

基于高通量测序技术的37种HPV亚型流行特征鉴定与生物信息学分析高通量测序技术(high-throughput sequencing)是一种快速、准确、高效的基因组学研究方法,被广泛应用于各个领域,包括人类疾病的分子机制研究。
本文将基于高通量测序技术,对37种人类乳头状瘤病毒(HPV)亚型的流行特征进行鉴定,并进行相应的生物信息学分析。
首先,我们需要明确37种HPV亚型的定义和分类。
HPV是一类双链DNA病毒,被广泛认为是导致宫颈癌和其他一些肿瘤的主要原因之一。
根据其基因组序列的相似性,HPV亚型被分为多个不同的类型。
具体的亚型包括HPV16、HPV18、HPV31、HPV33等,共计37种。
接下来,我们将使用高通量测序技术对37种HPV亚型进行测序。
高通量测序技术的优势在于可以高效地测定一次性产生的大量DNA序列,从而揭示基因组的整体特征。
通过测序,我们将获得每种亚型的基因组序列信息,并对其进行初步的分析。
在测序完成后,我们将对这些基因组序列进行生物信息学分析,以鉴定HPV亚型的流行特征。
生物信息学是一门将计算机科学和统计学方法应用于生物学研究的交叉学科,借助它的帮助,我们可以挖掘和分析大规模基因组数据中的有用信息。
在分析过程中,我们可以利用生物信息学工具对这些HPV亚型的基因组序列进行比对和序列标识。
通过比对,我们可以发现亚型之间的相似性和差异性,从而揭示其流行特征。
此外,还可以使用聚类分析方法将HPV亚型分成不同的群组,推断其可能的发生传播路径。
另外,我们还可以进行亚型基因组中功能区域的注释和预测。
通过分析基因组序列中的开放阅读框(open reading frame,ORF),我们可以预测编码蛋白质的基因,并进一步注释这些编码蛋白质的功能。
此外,还可以预测亚型的结构域和功能位点,为后续研究提供有价值的信息。
最后,我们可以利用系统生物学方法研究HPV亚型的调控网络和相互作用网络。
系统生物学是一种研究生物系统中各个组成部分之间相互作用与调控的学科,它可以揭示基因之间的调控关系和信号传导路径。

一、实验目的本实验旨在通过高通量基因测序技术,对某特定样本进行基因检测,分析其基因表达水平、基因突变等信息,为疾病诊断、基因治疗等提供科学依据。
二、实验材料1. 样本:某疾病患者的组织样本2. 试剂:高通量测序试剂盒、DNA提取试剂盒、PCR试剂、荧光定量PCR试剂等3. 仪器:高通量测序仪、PCR仪、凝胶成像系统、离心机、核酸分析仪等三、实验方法1. 样本DNA提取(1)将组织样本加入DNA提取试剂盒,按照说明书进行操作,提取样本DNA。
(2)使用核酸分析仪检测提取的DNA浓度和质量。
2. PCR扩增(1)根据目的基因序列设计特异性引物,进行PCR扩增。
(2)使用荧光定量PCR试剂对PCR产物进行定量分析,确保扩增效率。
3. 高通量测序(1)将PCR产物进行文库构建,包括适配子连接、指数富集等步骤。
(2)将文库进行高通量测序,获得大量测序数据。
4. 数据分析(1)使用生物信息学软件对测序数据进行质控、比对、基因表达水平分析等。
(2)根据分析结果,筛选出与疾病相关的基因突变和差异表达基因。
四、实验结果1. 样本DNA提取提取的DNA浓度为500 ng/μL,A260/A280比值约为1.8,符合实验要求。
2. PCR扩增PCR扩增产物经荧光定量PCR检测,扩增效率达到90%以上。
3. 高通量测序高通量测序获得大量测序数据,测序深度达到100倍。
4. 数据分析(1)基因表达水平分析:与对照组相比,实验组某基因表达水平显著升高。
(2)基因突变分析:在实验组样本中,发现某基因存在突变,与疾病发生密切相关。
五、实验讨论1. 本实验通过高通量基因测序技术,成功检测出与疾病相关的基因突变和差异表达基因,为疾病诊断和基因治疗提供了科学依据。
2. 高通量测序技术具有高通量、高灵敏度、高准确度等优点,在基因检测领域具有广泛应用前景。
3. 本实验中,样本DNA提取、PCR扩增、高通量测序等步骤均严格按照实验操作规范进行,保证了实验结果的可靠性。

生物信息学中的高通量基因测序数据分析与挖掘技术研究随着基因测序技术的发展,大规模的高通量基因测序数据正在迅速增加。
这些海量数据提供了一个宝贵的资源,可以用于了解生物的基因组结构、功能和演化等方面的信息。
为了从这些数据中获取有效的信息,生物信息学中的高通量基因测序数据分析和挖掘技术起到了关键的作用。
高通量基因测序技术是一种高效、高通量的测序方法,可以在较短的时间内获得大量的DNA或RNA序列信息。
这种技术的出现大大加速了生物学研究的进展,也为生物信息学研究提供了大量的数据。
高通量基因测序数据分析的主要目标是从原始的测序数据中提取出有用的信息,包括基因组的组装、基因功能注释、SNP(Single Nucleotide Polymorphism)的检测等。
基于高通量测序技术的数据,研究人员可以更好地理解生物体的基因组结构和功能。
高通量基因测序数据分析的第一步是质量控制。
由于测序过程中存在一定的误差,需要对测序数据进行质量评估和处理。
常用的质量控制方法包括去除低质量的测序数据、去除测序接头和引物等。
在数据质量控制后,研究人员可以进行下一步的数据分析。
第二步是基因组的组装。
基因组组装是将测序数据拼接成较长的连续序列,以还原生物的基因组结构。
对于无参考基因组的组装,采用de novo序列组装方法,通过对大量的短读长序列进行拼接,得到较长的序列。
对于已有参考基因组的组装,采用基于参考序列的对齐方法,通过将测序数据与参考序列进行比对,填充空缺以获得更完整的序列。
基因组组装的主要挑战是解决序列重复和大规模基因组的组装难题。
第三步是基因功能注释。
基因功能注释是将基因组序列和基因之间的功能关联进行分析和注释的过程。
常见的功能注释包括基因的功能类型、基因的表达水平和调控因子等。
通过对测序数据进行基因功能注释,可以帮助研究人员理解基因的功能和相互关系。
第四步是SNP(Single Nucleotide Polymorphism)的检测。
高通量基因测序数据的生物信息学研究高通量基因测序技术近年来在生物学领域得到广泛应用,为基因组学、转录组学和蛋白质组学等领域的研究提供了强有力的工具。
基于高通量测序技术得到的海量数据,生物信息学研究助力于深入挖掘数据内在的生物学信息,为生物学研究和应用提供新的观点和方法。
本文就高通量基因测序数据在生物信息学研究中的应用、算法和软件工具等方面进行探讨。
一、高通量基因测序数据高通量测序技术以短时间、高通量、高准确性、低成本及复杂样品等特点,成为当前基因测序领域的主流技术。
它可以从混合的DNA或RNA中高效、快速地检测出许多序列,并通过计算机分析得到基因组、转录组、蛋白组等多个维度的生物信息。
高通量基因测序过程包括准备样品、建库、测序和数据分析等步骤。
每一步都影响测序质量和数据结果。
二、生物信息学研究在高通量基因测序中的应用高通量基因测序技术生成的数据量通常具有大规模、高维度和高复杂性个特点,需要借助生物信息学来解决这些问题的挑战。
生物信息学研究在高通量基因测序数据中的应用有很多方面,其中最常见的就是:1. 非编码RNA鉴定及功能分析非编码RNA是指不具有编码蛋白的功能的RNA分子,包括长链非编码RNA (lncRNA)、微小RNA(miRNA)以及很多种RNA。
二代测序技术可以深入、高通量地检测出各种类型的非编码RNA,但其功能尚不明确。
生物信息学方法可以通过结合不同的公共数据库和生产软件,预测和鉴定非编码RNA特征,分析其在细胞周期、生长发育等方面调控基因表达的作用及机制。
2. 基因组重测序及变异检测基因组重测序是指在已知的组装序列上重新测序,并将得到的数据与已知的序列进行比较,以检测个体间的遗传变异。
基于高通量基因测序技术,可以大规模地进行基因组重测序,并利用生物信息学方法分析变异位点的发现和功能注释。
这有助于检测基因组重测序对功能区、副本数等的影响,以及识别疾病相关的致病基因或易感位点。
3. 转录组分析转录组数据是高通量基因测序技术的典型应用之一。
高通量测序技术在生物信息学中的应用研究标题:高通量测序技术在生物信息学中的应用研究摘要:随着高通量测序技术的迅速发展,越来越多的研究已经将其应用于生物信息学领域。
本文旨在探讨高通量测序技术在生物信息学中的应用,包括研究问题及背景、研究方案方法、数据分析和结果呈现以及结论与讨论。
通过文献综述和实例分析,本文旨在揭示高通量测序技术在生物信息学中的潜力以及未来发展的趋势。
一、研究问题及背景随着生物领域研究的深入,科学家们面临着越来越复杂的生物信息学问题。
传统的测序方法无法满足高通量测序大规模数据的需求,因此高通量测序技术的出现为生物信息学研究提供了有效的解决方案。
本段介绍了高通量测序技术在生物信息学研究中的应用背景,并阐述了该研究的重要性和意义。
二、研究方案方法本节详细介绍了高通量测序技术在生物信息学研究中的应用方案和方法。
首先,介绍了高通量测序技术的基本原理和常用的测序方法,如Illumina测序、Ion Torrent测序等。
然后,探讨了高通量测序技术在生物信息学研究中的样本准备、测序过程以及数据质控等关键步骤。
最后,介绍了实验设计和数据分析的策略,包括差异表达分析、多组学数据整合以及功能注释等方法。
三、数据分析和结果呈现本节详细介绍了高通量测序技术在生物信息学研究中的数据分析和结果呈现。
首先,介绍了常用的数据分析工具和软件,如Bowtie、TopHat、Cufflinks等。
然后,介绍了数据质量控制和预处理的方法,包括去除低质量序列、过滤噪声和去除冗余等。
接着,详细阐述了差异表达基因的鉴定和功能注释的方法。
最后,通过实际案例展示了高通量测序技术在生物信息学研究中的数据分析流程和结果呈现方法。
四、结论与讨论本节总结了高通量测序技术在生物信息学研究中的应用,并提出了结论和讨论。
首先,总结了高通量测序技术在生物信息学领域的重要性和应用潜力。
然后,讨论了高通量测序技术在解析基因组结构、揭示基因调控机制、发现新的功能基因以及研究复杂疾病等方面的应用前景。
基于高通量测序的生物信息学分析研究生物信息学作为一门新兴的交叉学科,涉及到生物、计算机科学、数学和统计等多个领域。
其中,高通量测序技术是生物信息学发展的重要基础,它是通过自动化、高速、高灵敏度的方法分析DNA或RNA的序列信息,从而深入掌握生命体系的遗传信息。
在这篇文章中,将着重探讨基于高通量测序的生物信息学分析研究。
一、高通量测序技术的基本概念高通量测序技术也被称为次代测序技术,与传统的Sanger测序技术相比,它可以高速、高效、低成本地完成海量基因组、转录组或RNA-Seq等样本的测序。
同时,高通量测序技术也不断发展,并出现了Illumina、Ion Torrent、PacBio等类别的次代测序平台,为生物信息学研究提供了更加多元的数据来源。
二、高通量测序技术的应用领域高通量测序技术的应用领域非常广泛,包括基因组测序、转录组测序、RNA-Seq、染色体捕获、外显子组、甲基化分析、比较基因组学研究等。
这些研究可以用于深入研究生命体系的基因功能、生物进化和疾病发生机制等重要问题。
三、高通量测序的数据分析方法高通量测序技术生成的数据量很大,需要进行一系列的数据分析,以便从生物学的角度理解数据。
这些分析包括序列质量控制、数据清洗、序列比对、差异分析、功能注释、基因富集分析等。
其中,数据清洗和序列比对是高通量测序数据分析的重要步骤,主要是为了保证数据的质量和准确性,并建立样本之间的比较框架。
四、高通量测序在基因功能研究中的应用基于高通量测序的基因功能研究主要包括转录组测序、RNA-Seq和甲基化分析等。
这些手段可以帮助研究人员深入理解基因调控机制、识别基因表达谱和DNA甲基化谱的变化规律。
通过这些研究,研究人员可以发现与疾病发生和进化相关的基因标记,并为新药研发提供支持。
五、高通量测序在精准医学中的应用高通量测序技术在精准医学中的应用正在逐步展开,在癌症诊疗、遗传病筛查和药物敏感性等方面具有广阔的应用前景。
基于高通量测序技术的生物信息学研究引言高通量测序技术是目前生物学领域最重要的技术之一,它的出现在过去几年里不断推动着生物信息学的研究进展。
本文将详细介绍高通量测序技术在生物信息学种的应用。
一、高通量测序技术的原理和基本流程高通量测序技术是指一种可以同步测定基因组大量序列信息的新技术。
其基本原理是将DNA分子随机的分成小片并进行放大和测序。
利用“建库”和测序仪器,对DNA分子进行处理,可以分析DNA信息,并获得DNA序列等信息。
具体的步骤包括DNA样本制备、DNA分子的随机断裂、添加接头引物、片段键合成文库、生产梳形簇、以及高速测序和数据处理等步骤。
二、高通量测序技术在DNA序列组装中的应用DNA序列组装是生物信息学中最基础的应用之一,其应用在测序数据集合中具有重要的意义。
高通量测序技术的出现大大提高了DNA测序的速度和精度,并且可以将DNA分子分解成更小的片段进行测序,从而减少了DNA测序时的杂质干扰。
同时,高通量测序技术可探测多个基因座,不仅有助于对复杂基因组的分析,而且可以发现许多未知性基因并提高基因测序的质量。
三、高通量测序技术在RNA测序中的应用随着对RNA序列结构和功能的研究越来越深入,要求对RNA序列的组装与测序技术提高并快速识别RNA序列和数量。
高通量测序技术可广泛用于RNA序列的测序与识别,可以通过对RNA样本经处理后进行转录及测序,从而测出RNA序列以周期性路径反映转录区的细节和表达方式,给研究人员提供了富有生物信息的RNA数据。
四、高通量测序技术在功能基因组学中的应用高通量测序技术在功能基因组学研究中被广泛应用。
功能基因组学主要关注基因的功能及其编码的蛋白质如何相互作用和调节。
高通量测序技术可以帮助筛选基因及开发新疗法,同时也可以用来分析与人类健康相关的新发现基因,因此在基因诊断、个性化医疗和药物研发等方面具有极大的潜力。
五、高通量测序技术在微生物学中的应用微生物是一类非常重要的生物群落。
高通量基因测序技术的生物信息学分析方法探索随着生物科技的快速发展,高通量基因测序技术已经成为了现代生物学和医学领域中不可或缺的工具。
高通量基因测序技术可以同时对数十万到数百万个DNA 片段进行测序,从而实现了全基因组或全转录组水平的测序和分析。
不过,由于高通量基因测序技术生成的数据量巨大、信息复杂、处理难度大,因此生物信息学分析方法的研发和探索变得尤为重要。
本文将对高通量基因测序技术的生物信息学分析方法进行探究和总结,主要包括数据预处理、DNA序列比对、SNP检测、基因表达分析与差异表达分析以及功能富集分析等几个方面。
一、数据预处理数据预处理是高通量基因测序数据分析的第一步,它包括质量控制、去除低质量序列、建立比对参考基因组等一系列的处理过程。
质控流程通常采用FastQC等相关软件,评估测序数据的总体质量和序列片段的质量。
然后,采用Trimmomatic 或cutadapt等软件去除低质量和重复的序列。
建立比对参考基因组的过程则涉及到基因组序列的建立和比对,采用比对软件如BWA和Bowtie2等可以比对至人类、小鼠等多种物种的基因组,获得准确的序列定位信息。
二、DNA序列比对DNA序列比对是高效地识别DNA序列中的差异和变异的核心步骤。
它运用了基因组学、算法学和统计学等多个领域的知识,根据序列相似性和确定的匹配算法处理大量的DNA序列数据。
主要的DNA序列比对软件有Bowtie2、BWA、Tophat等。
比对结果会输出SAM/BAM格式的文件,其中记录了每个DNA片段的比对位置、匹配质量和变异类型等信息。
三、SNP检测SNP(Single Nucleotide Polymorphism)是指DNA序列中发生了单核苷酸变异的位置。
SNP的检测是高通量基因测序技术中一个重要的应用方向,可用于分析个体间和群体间的遗传结构差异,追踪疾病的遗传基础和变异等。
基因变异的检测方法众多,如质量分数检测、Bayesian检测等。
附件三生物信息学分析
一、基础生物信息学分析
1.有效测序序列结果统计
有效测序序列:所有含样品barcode(标签序列)的测序序列。
统计该部分序列的长度分布情况。
注:合同中约定测序序列条数以有效测序序列为准。
图形示例为:
2.优质序列统计
优质序列:有效测序序列中含有特异性扩增引物、不含模糊碱基、长度大于可供分析标准的序列。
统计该部分序列的长度分布情况。
图形示例为:
3.各样本序列数目统计:
统计各个样本所含有效测序序列和优质序列数目。
结果示例为:
4.OTU生成:
根据序列的相似性,将序列归为多个OTU(操作分类单元),以便后续分析。
5.稀释曲线(rarefaction 分析)
根据第4条中获得的OTU数据,做出每个样品的Rarefaction曲线。
本合同默认生成OTU相似水平为0.03的rarefaction曲线。
rarefaction曲线结果示例:
6.指数分析
计算各个样品的相关分析指数,包括:
•丰度指数:ace\chao
•多样性指数:shannon\simpson
•本合同默认生成OTU相似水平为0.03的上述指数值。
多样性指数分析结果示例:
注:默认分析以上所列指数,如有特殊需要请说明。
7.Shannon-Wiener曲线
利用各样品的测序量在不同测序深度时的微生物多样性指数构建曲线,反映各样本在不同测序数量时的微生物多样性。
当曲线趋向平坦时,说明测序数据量足够大,可以反映样品中绝大多数的微生物信息。
绘制默认水平为:0.03。
例图:
8.Rank_Abuance 曲线
根据各样品的OTU丰度大小排序作丰度分布曲线图。
结果文件默认为PDF格式(其它格式请注明)。
例图:
9.Specaccum物种累积曲线(大于10个样品)
物种累积曲线( species accumulation curves) 用于描述随着抽样量的加大物种增加的状况,是理解调查样地物种组成和预测物种丰富度的有效工具,在生物多样性和群落调查中,被广泛用于抽样量充分性的判断以及物种丰富度( species richness) 的估计。
因此,通过物种累积曲线不仅可以判断抽样量是否充分,在抽样量充分的前提下,运用物种累积曲线还可以对物种丰富度进行预测。
10.样品OTU分布及分类学信息
OTU产生后,统计各个样品含有OTU情况及每个OTU中含有序列的数目。
同时,将所有序列与Silva 库比对,得到序列的分类学信息。
通过寻找最近祖先方法,得到每个OTU的分类学信息。
本合同默认分析相似性水平为0.03的OTU。
结果为一份xls文件,文件容示例为:
第一列为OTU编号,第一行为各个样品名称,中间数字表示该列样品在此行OTU中所占的序列数目,最后一列为该行OTU的种属信息。
二、高级生物信息学分析
11.OTU 分布VENN图
注:选择一组不多于五个样品,分析样品间OTU重合情况,将结果以VENN图形式展示。
结果文件默认为PDF格式(其它格式请注明)。
例图:
12.多样品相似度树状图I (样品无分组)
注:选定需要分析的多个样品作为一组对比分析,使用jest算法,比较该组分析中各样品在OTU
(0.03)水平上的群落结构相似度并作出树状图。
结果文件默认为PDF格式(其它格式请注明)。
例图:
13.多样品相似度树状图II (样品有分组)
注:选定需要分析的多个样品作为一组对比分析,使用jest算法,比较该组分析中各样品在OTU
(0.03)水平上的群落结构相似度并作出树状图。
结果文件默认为PDF格式(其它格式请注明)。
例图:
14.群落结构组组分图(共__N__组分析)
注:选定一个或多个需要分析的样品,选定一个分类学水平,按照相应多样性信息作图,反应各样品的群落结构。
结果文件默认为PDF格式(其它格式请注明)。
可选分类学水平:门、纲、目、科、属;同一组样品选择多个分类学水平为多组分析。
例图:
15.多样品相似度树与柱状图组合分析(默认提供门的水平)
左边是样品间基于群落组成的层次聚类分析,右边是样品的群落结构柱状图。
16.PCA主成分分析Ⅰ(样品无分组)
选取多个样品,进行PCA分析。
结果文件默认为PDF格式(其它格式请注明)。
例图:
17.PCA主成分分析Ⅱ(样品有分组信息)
选取多个样品,进行PCA分析。
结果文件默认为PDF格式(其它格式请注明)。
例图:
18.群落Heatmap图(确定分类学水平及图片颜色)
选择多个样品,作出其在选定的分类学水平上群落结构 Heatmap图。
结果文件默认为PDF格式(其它格式请注明)。
图形颜色默认为彩虹色,如需选黑红色请标明。
可选分类学水平:门、纲、目、科、属、OTU(0.03)
如分析单元数目较多,默认使用序列数较多的前100个种属或OTU作图,如有其它要求请注明。
例图:。