酶切位点识别序列
- 格式:docx
- 大小:15.79 KB
- 文档页数:5
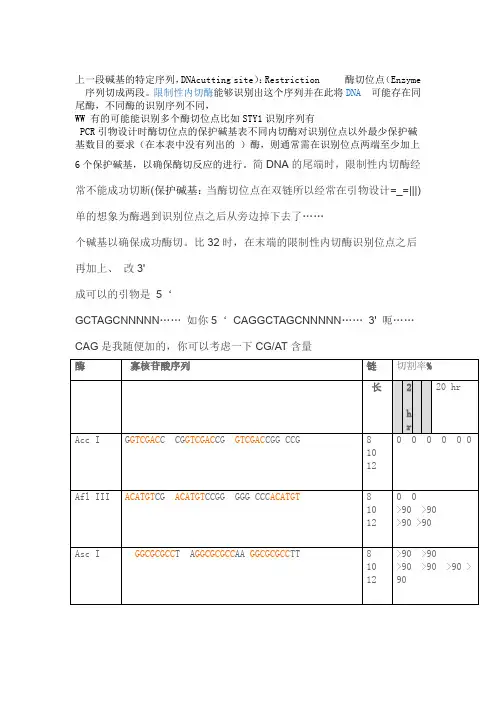
上一段碱基的特定序列,DNAcutting site):Restriction 酶切位点(Enzyme 序列切成两段。
限制性内切酶能够识别出这个序列并在此将DNA可能存在同尾酶,不同酶的识别序列不同,
WW 有的可能能识别多个酶切位点比如STY1识别序列有
PCR引物设计时酶切位点的保护碱基表不同内切酶对识别位点以外最少保护碱
基数目的要求(在本表中没有列出的)酶,则通常需在识别位点两端至少加上
6个保护碱基,以确保酶切反应的进行。
简DNA的尾端时,限制性内切酶经常不能成功切断(保护碱基:当酶切位点在双链所以经常在引物设计=_=|||)单的想象为酶遇到识别位点之后从旁边掉下去了……
个碱基以确保成功酶切。
比32时,在末端的限制性内切酶识别位点之后
再加上、改3'
成可以的引物是5‘
GCTAGCNNNNN……如你5‘CAGGCTAGCNNNNN……3' 呃……CAG是我随便加的,你可以考虑一下CG/AT含量
注释
1.如果要加在序列的5'端,就在酶切位点识别碱基序列(红色)的5'端加上相应的碱基(黑色),如果要在序列的3'端加上保护碱基,就在酶切位点识别碱基序列(红色)的3'端加上相应的碱基(黑色)。
2.切割率:正确识别并切割的效率。
加保护碱基时最好选用切割率高时加的相应碱基。
3.。

takara快切酶酶切位点
Takara快切酶是一种用于分子生物学实验的酶,它能够识别特定的DNA序列并在该序列上进行切割。
酶切位点是指酶在DNA分子上识别并切割的特定序列。
Takara快切酶的切位点取决于具体使用的酶种类,不同的酶有不同的识别序列和切割方式。
从分子生物学角度来看,Takara快切酶的切位点是DNA双链上的特定序列,这些序列通常是4至8个碱基对长,具有特定的碱基配对规律。
酶切位点的选择对于DNA分子的切割和连接至关重要,因为它直接影响着DNA重组、连接和修复的效率和准确性。
在实验操作中,研究人员需要根据所使用的Takara快切酶的特性来选择合适的切位点,以确保实验能够顺利进行。
通常情况下,研究人员会根据酶的说明书或相关文献来确定切位点,然后设计合适的引物或寡核苷酸序列进行实验操作。
除此之外,Takara快切酶的切位点也与基因工程、基因编辑等领域密切相关。
在基因编辑技术中,研究人员经常利用
CRISPR/Cas9等系统来指导Takara快切酶在特定的DNA序列上进行切割,从而实现对基因组的精准编辑。
总的来说,Takara快切酶的切位点是分子生物学领域中非常重要的概念,它涉及到DNA序列的识别和切割,对于基因工程、基因编辑和分子生物学研究具有重要意义。
在实验操作中,科研人员需要根据具体的实验目的和所用酶的特性来选择合适的切位点,并进行相关的实验设计和操作。

酶切位点汇总
酶切位点,又称为限制性内切酶位点,是指DNA分子上特定的序列,这些序列是限制
性内切酶可以识别和切割的地方。
限制性内切酶是一种在细菌和其它生物中广泛存在的酶,能够切割或切除一个或多个DNA碱基对。
这些限制性内切酶在生物技术领域广泛应用,用
于DNA序列分析、DNA重组、基因工程等方面。
以下是常见的几种酶切位点:
1. EcoRI切割位点是5′-GAATTC-3′,这是一种广泛应用的限制性内切酶,通常用于DNA纯化、制备DNA载体等。
2. BamHI切割位点是5′-GGATCC-3′,BamHI能够切割链间,产生具有黏性末端的DNA 序列。
常被用于制备双链DNA的黏性末端。
4. PstI切割位点是5′-CTGCAG-3′,PstI是一种双切酶,可以切割成不同长度的DNA 序列,适用于构建多种不同长度的DNA分子。
总之,酶切位点及其对应的限制性内切酶在现代生物领域有着广泛的应用和重要的作用。
了解不同的酶切位点是有很大帮助的,它可以为实验设计和分子生物学研究提供基础。
同时,也让我们更好地理解限制性内切酶在DNA分子上的作用,帮助我们在生物技术领域
更加熟练地掌握其应用。
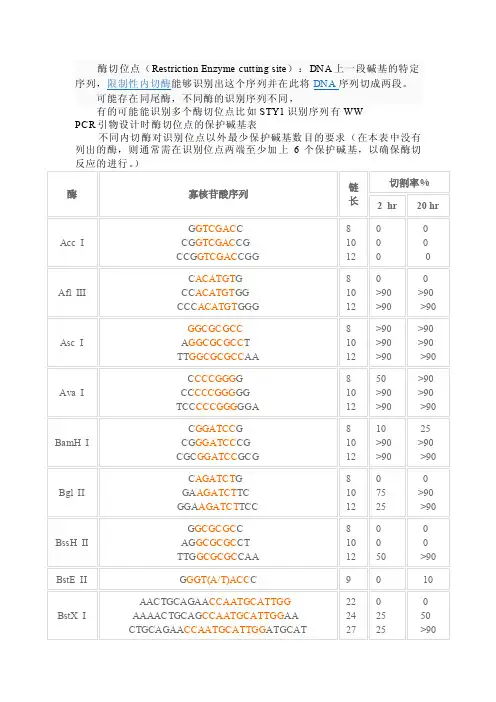
PCR引物设计时酶切位点的保护碱基表
不同内切酶对识别位点以外最少保护碱基数目的要求(在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
)
注释
1.如果要加在序列的5’端,就在酶切位点识别碱基序列(红色)的5’端加上相
应的碱基(黑色),如果要在序列的3’端加上保护碱基,就在酶切位点识别碱基序列(红色)的3’端加上相应的碱基(黑色)。
2.切割率:正确识别并切割的效率。
3.加保护碱基时最好选用切割率高时加的相应碱基。

名词解释【基因工程】:在体外对不同生物的遗传物质(基因)进行剪切、重组、连接,然后插入到载体分子中(细菌质粒、病毒或噬菌体DNA),转入微生物,植物或动物细胞内进行无性繁殖,并表达出基因产物。
【限制性核酸内切酶】:是一类能够识别双链DNA分子中的某种特定核苷酸序列(4-8bp),并由此处切割DNA双链结构的核酸内切酶。
【识别序列】:限制性核酸内切酶在双链DNA上能够识别的特殊核苷酸序列被称为识别序列。
【酶切位点】:DNA在限制性核酸内切酶的作用下,使多聚核苷酸链上磷酸二酯键点开的位置被称为切割位点。
【粘性末端】:是指含有几个核苷酸单链的末端,可通过这种末端的碱基互补,使不同的 DNA片段发生退火。
【平末端】:限制酶在它识别序列的中心轴线处切开时产生的平齐的末端。
【同裂酶】:一些来源不同的但能识别位点的序列相同的限制性内切酶。
【同尾酶】:一些来源不同且识别序列不同,但能产生相同粘性末端的限制性内切酶。
【DNA的甲基化程度】:DNA被甲基化酶甲基化,识别序列中的核苷酸一旦被甲基化,就会影响内切酶的切割效率。
【位点偏爱】:对不同位置的同一个识别序列表现出不同的切割效率的现象【内切酶的star活性】:某种限制性核酸内切酶在特定条件下,可在不是原来的识别序列处切割DNA,这种现象称为star活性。
【末端转移酶】:一种能将脱氧核苷酸三磷酸(dNTP)加到某DNA片段上3’-OH基上的酶。
【DNA连接酶】:借助ATP或NAD水解提供的能量催化DNA双链,DNA片段紧靠在一起的3’-OH末端与5’-PO4末端之间形成磷酸二酯键,使两末端连接【DNA聚合酶】:以DNA为复制模板,使DNA由5'端点开始复制到3'端的酶。
【反转录酶】:与DNA聚合酶作用方式相似:5’→3’聚合,模版是mRNA,合成DNA【碱性磷酸酶】:能够催化核酸分子脱掉5’磷酸基团,从而使DNA(或RNA)片段的5’-P 末端转换成5’-OH末端。

酶切位点识别序列 Document serial number【KKGB-LBS98YT-BS8CB-BSUT-BST108】
酶切位点(Restriction Enzyme cutting site):DNA上一段碱基的特定序列,能够识别出这个序列并在此将序列切成两段。
可能存在同尾酶,不同酶的识别序列不同,
有的可能能识别多个酶切位点比如STY1识别序列有WW
PCR引物设计时酶切位点的保护碱基表
不同内切酶对识别位点以外最少保护碱基数目的要求(在本表中没有列出的
注释
1.如果要加在序列的5’端,就在酶切位点识别碱基序列(红色)的5’端加上相应的
碱基(黑色),如果要在序列的3’端加上保护碱基,就在酶切位点识别碱基序列(红色)的3’端加上相应的碱基(黑色)。
2.切割率:正确识别并切割的效率。
3.加保护碱基时最好选用切割率高时加的相应碱基。

酶切位点的特点
酶切位点是指在DNA或RNA分子中,特定酶能够切割的位置。
以下是酶切位点的一些特点:
1. 序列特异性:每种酶都有特定的序列要求,只能识别并切割特定的核酸序列。
这些序列通常由4种碱基(腺嘌呤、胸腺嘧啶、鸟嘌呤和胞嘧啶)组成,例如常见的EcoRI酶切位点是GAATTC。
2. 对称性:大多数酶切位点是对称的,即从5'到3'方向的顺序与互补链上的同一序列相同。
例如EcoRI酶切位点的互补链也是GAATTC。
3. 切割位置:酶通常在特定的位置切割DNA或RNA分子。
切割位点可以是在切割位点序列内的特定碱基之间,也可以是切割位点序列的边缘。
4. 切割方式:不同的酶可以以不同的方式切割DNA或RNA。
一些酶会切割两条链上的碱基对称地,形成平滑的切割端;而其他酶则会产生不对称的切割,形成突起或粘性末端。
5. 应用广泛:酶切位点在分子生物学和基因工程领域应用广泛。
通过识别和利用酶切位点,可以进行DNA片段的精确切割、连接和重
组,用于构建重组DNA分子、进行基因克隆、检测基因突变等。
总之,酶切位点是特定酶能够识别和切割的DNA或RNA分子中的特定序列,具有序列特异性、对称性、切割位置和切割方式等特点。
这些特点使得酶切位点在分子生物学和基因工程中发挥着重要的作用。

酶切位点的原理酶切位点是指酶在DNA或RNA分子上识别和切割的特定位置。
酶切位点的原理涉及到两个主要方面:酶的识别和酶的催化。
首先,酶能够识别和结合到特定的酶切位点是因为酶和DNA(或RNA)之间存在着特定的相互作用。
这些相互作用可以是静电相互作用、氢键相互作用、范德华力等。
这种相互作用使得酶能够识别出DNA或RNA分子上的特定序列,从而精确地识别并定位到酶切位点。
其次,酶的催化作用使得酶能够切割DNA或RNA分子。
酶通常通过两种主要的方式参与催化反应:核酸酶活性和脱氧核酸酶活性。
核酸酶活性指的是酶能够剪断两个核苷酸之间的磷酸二酯键。
脱氧核酸酶活性指的则是酶能够剪断具有特定结构的DNA分子,如血清蛋白A测序等。
酶切位点的识别和切割原理可以通过以限制性内切酶为例进行解释。
限制性内切酶是一种具有识别和切割特定DNA序列的酶。
它可以在DNA分子中识别一段具有特定序列的碱基对,并在这个特定的序列上切割DNA链。
限制性内切酶的识别过程是通过酶与DNA序列之间的亲和力进行的。
限制性内切酶通常识别的是具有对称性的DNA序列,即两个互补的DNA链具有相同的序列。
例如,EcoRI酶可以识别序列为GAATTC的DNA,在这个序列处切割DNA链。
限制性内切酶的切割过程则是通过酶的催化活性来实现的。
在切割过程中,限制性内切酶会将靠近酶切位点上的磷酸二酯键断裂,产生两个具有黏性末端的DNA片段。
这些黏性末端具有一段未配对的碱基,能够与其他黏性末端互相结合,形成酶切位点原来的DNA分子。
限制性内切酶可根据切割产生的片段末端形式被分类为粘性末端酶和平滑末端酶。
粘性末端酶产生的片段末端具有突出的单链末端,其中一条链长一截,而平滑末端酶产生的片段末端则是完整的双链末端。
酶切位点的原理在实验室中被广泛应用于许多分子生物学技术中,如PCR、DNA 测序、基因克隆等。
通过熟练地选择和使用限制性内切酶,可以精确地切割和定位DNA分子,从而开展基因组研究以及进行DNA分析。

酶切位点识别序列 Last revised by LE LE in 2021
酶切位点(Restriction Enzyme cutting site):DNA上一段碱基的特定序列,能够识别出这个序列并在此将序列切成两段。
可能存在同尾酶,不同酶的识别序列不同,
有的可能能识别多个酶切位点比如STY1识别序列有WW
PCR引物设计时酶切位点的保护碱基表
不同内切酶对识别位点以外最少保护碱基数目的要求(在本表中没有列出的
注释
1.如果要加在序列的5’端,就在酶切位点识别碱基序列(红色)的5’端加上相应的
碱基(黑色),如果要在序列的3’端加上保护碱基,就在酶切位点识别碱基序列(红色)的3’端加上相应的碱基(黑色)。
2.切割率:正确识别并切割的效率。
3.加保护碱基时最好选用切割率高时加的相应碱基。

常见限制性内切酶识别序列(酶切位点)(BamHI、EcoRI、HindIII、NdeI、XhoI等)Time:2009-10-22 PM 15:38Author:bioer Hits: 7681 times在分子克隆实验中,限制性内切酶是必不可少的工具酶。
无论是构建克隆载体还是表达载体,要根据载体选择合适的内切酶(当然,使用T 载就不必考虑了)。
先将引物设计好,然后添加酶切识别序列到引物5' 端。
常用的内切酶比如BamHI、EcoRI、HindIII、NdeI、XhoI等可能你都已经记住了它们的识别序列,不过为了保险起见,还是得查证一下。
下面是一些常用的II型内切酶的识别序列,仅供参考。
先介绍一下什么是II型内切酶吧。
The Type II restriction systems typically contain individual restriction enzymes and modification enzymes encoded by separate genes. The Type II restriction enzymes typically recognize specific DNA sequences and cleave at constant positions at or close to that sequence to produce 5-phosphates and 3-hydroxyls. Usually they require Mg 2+ ions as a cofactor, although some have more exotic requirements. The methyltransferases usually recognize the same sequence although some are more promiscuous. Three types of DNA methyltransferases have been found as part of Type II R-M systems forming either C5-methylcytosine, N4-methylcytosine or N6-methyladenine.酶类型识别序列ApaIType II restrictionenzyme5'GGGCC^C 3'BamHIType II restrictionenzyme5' G^GATCC 3'BglIIType II restrictionenzyme5' A^GATCT 3'EcoRIType II restrictionenzyme5' G^AATTC 3'HindIIIType II restrictionenzyme5' A^AGCTT 3'KpnIType II restrictionenzyme5' GGTAC^C 3'NcoIType II restrictionenzyme5' C^CATGG 3'NdeIType II restrictionenzyme5' CA^TATG 3'NheIType II restrictionenzyme5' G^CTAGC 3'NotIType II restrictionenzyme5' GC^GGCCGC 3'SacIType II restrictionenzyme5' GAGCT^C 3'SalIType II restrictionenzyme5' G^TCGAC 3'SphIType II restrictionenzyme5' GCATG^C 3'XbaIType II restrictionenzyme5' T^CTAGA 3'XhoIType II restrictionenzyme5' C^TCGAG 3'要查找更多内切酶的识别序列,你还可以选择下面几种方法:1. 查你所使用的内切酶的公司的目录或者网站;2. 用软件如:Primer Premier5.0或Bioedit等,这些软件均提供了内切酶识别序列的信息;3. 推荐到NEB的REBASE数据库去查(网址:/rebase/rebase.html)当你设计好引物,添加上了内切酶识别序列,下一步或许是添加保护碱基了,可以参考:NEB公司网站提供的关于设计PCR引物保护碱基参考表下载(也可见图片)双酶切buffer的选择(MBI、罗氏、NEB、Promega、Takara)再给大家推荐一种新的不需要连接反应的分子克隆方法,优点包括:①设计引物不必考虑选择什么酶切位点;②不必考虑保护碱基的问题;③不必每次都选择合适的酶来酶切质粒制备载体;④而且不需要DNA连接酶;⑤假阳性几率低(因为没有连接反应这一步,载体自连的问题没有了)。
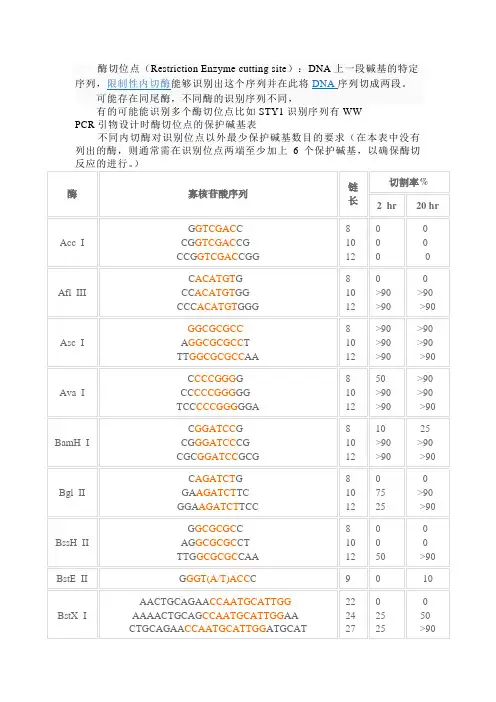
PCR引物设计时酶切位点的保护碱基表
不同内切酶对识别位点以外最少保护碱基数目的要求(在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
)
注释
1.如果要加在序列的5’端,就在酶切位点识别碱基序列(红色)的5’端加上相
应的碱基(黑色),如果要在序列的3’端加上保护碱基,就在酶切位点识别碱基序列(红色)的3’端加上相应的碱基(黑色)。
2.切割率:正确识别并切割的效率。
3.加保护碱基时最好选用切割率高时加的相应碱基。
酶切用的酶简介酶切是分子生物学研究中常用的一种技术手段,用于切割DNA分子。
酶切用的酶是一类特殊的酶,能够识别特定的DNA序列并将其切割成特定的片段。
在基因工程、DNA测序和DNA重组等领域,酶切用的酶发挥着重要的作用。
酶切原理酶切用的酶主要是一类特殊的内切酶,也叫限制性内切酶(Restriction Enzyme)。
它们能够识别DNA分子中的特定核酸序列(酶切位点),并在该位点上进行切割。
酶切位点通常是一个对称的序列,例如5’-GAATTC-3’。
酶切用的酶能够识别并切割这样的序列,产生两个粘性末端或平滑末端的DNA片段。
酶切用的酶具有高度的特异性,即只能识别特定的酶切位点。
它们通过与DNA分子中的特定序列进行互补配对,形成酶-底物复合物,然后在酶切位点上切割DNA链。
酶切用的酶的分类根据酶切位点的序列特征,酶切用的酶可以分为不同的类别。
常见的酶切用的酶包括:1.限制性内切酶I类(Type I):这类酶切用的酶切割DNA时具有两个酶切位点,但切割位点与识别位点并不重合。
它们通常在识别序列附近的随机位置进行切割,产生不规则的切口。
2.限制性内切酶II类(Type II):这是最常用的一类酶切用的酶。
它们能够直接在识别位点进行精确的切割,产生具有平滑末端或粘性末端的DNA片段。
常见的II类酶切用的酶有EcoRI、HindIII等。
3.限制性内切酶III类(Type III):这类酶切用的酶切割DNA时具有两个酶切位点,与Type I类酶相似。
它们也将切割位点与识别位点分开,产生不规则的切口。
与Type I类酶不同的是,Type III类酶需要与其他辅助蛋白一起作用才能发挥酶切功能。
4.限制性内切酶IV类(Type IV):这类酶切用的酶不常见,它们不在识别位点进行切割,而是在较远的位置附近进行切割。
5.限制性内切酶V类(Type V):这类酶切用的酶是一类泛素修饰酶(Methylase)。
它们不能直接切割DNA链,而是在酶切位点上对DNA进行甲基化修饰。
actagt酶切位点酶切位点是指酶在DNA或RNA分子中特定的序列上进行切割的位置。
其中,actagt是一种常见的酶切位点序列,这个序列是由5个核苷酸(A、C、T、G)组成的。
在这篇文章中,我们将探讨actagt酶切位点的相关知识。
1. actagt酶切位点的定义酶切位点是指酶在DNA或RNA分子中特定的序列上进行切割的位置。
actagt酶切位点是其中一种常见的酶切位点序列,它是由5个核苷酸(A、C、T、G)组成的。
在DNA分子中,酶可以通过识别并结合到actagt序列上,然后切割DNA链,从而起到修复、重组或转录等功能。
2. actagt酶切位点的应用actagt酶切位点在分子生物学研究中有着广泛的应用。
首先,它可以用于DNA的限制性酶切。
限制性酶是一类能够识别特定酶切位点并切割DNA的酶,actagt序列正好是一些常用限制性酶的切割位点。
通过限制性酶切反应,可以将DNA分子切割成特定的片段,这对于基因克隆、DNA测序等实验都非常重要。
actagt酶切位点也可以用于引物设计。
在PCR反应中,引物是用于扩增目标DNA片段的短链DNA序列,引物的设计是PCR反应的关键之一。
由于actagt酶切位点的特异性,它可以被用来作为引物的设计依据,以确保引物能够特异性地结合到目标DNA序列上。
3. actagt酶切位点的识别在实验中,识别actagt酶切位点有多种方法。
一种常用的方法是利用限制性酶切酶对DNA进行切割,并通过凝胶电泳检测切割产物的大小来确定酶切位点的位置。
另一种方法是利用生物信息学工具进行预测,通过计算DNA序列中actagt序列的位置来确定酶切位点。
4. actagt酶切位点的变异虽然actagt酶切位点是一种常见的酶切位点序列,但在不同的生物体中也存在一定程度的变异。
这种变异可能是由于基因组演化的结果,也可能是由于突变等原因导致的。
因此,在实际应用中,对于不同物种或个体的DNA样本,需要仔细检查actagt酶切位点的存在与否,以确保实验结果的准确性。
常用酶切位点序列和保护碱基引言在分子生物学和遗传工程领域,酶切位点序列和保护碱基是非常重要的概念。
酶切位点序列指的是DNA或RNA上特定的核苷酸序列,这些序列可以被特定的酶识别并切割。
保护碱基则是指在实验过程中采取措施来保护DNA或RNA上特定的核苷酸,使其不被酶切割。
本文将对常用的酶切位点序列和保护碱基进行详细介绍,包括其定义、常见的酶切位点序列、如何选择合适的保护碱基等内容。
酶切位点序列定义酶切位点序列是指DNA或RNA分子上具有一定规律性、可以被特定的限制性内切酶识别并结合从而发挥催化作用的核苷酸序列。
这些限制性内切酶通常能够识别4-8个核苷酸,并在识别到相应的位点后将DNA或RNA分子切割成片段。
常见的酶切位点序列1.EcoRI: 5’-GAATTC-3’,3’-CTTAAG-5’2.HindIII: 5’-AAGCTT-3’,3’-TTCGAA-5’3.BamHI: 5’-GGATCC-3’,3’-CCTAGG-5’4.XhoI: 5’-CTCGAG-3’,3’-GAGCTC-5’5.NotI: 5’-GCGGCCGC-3’,3’-CGCCGGCG-5’这些酶切位点序列是常用的限制性内切酶的识别序列,它们在分子生物学实验中被广泛应用。
通过将DNA或RNA与特定的限制性内切酶一起反应,可以实现DNA或RNA的特定部位切割。
保护碱基定义保护碱基是指在实验过程中采取措施来保护DNA或RNA上特定的核苷酸,使其不被酶切割。
这种保护通常通过对特定的碱基进行修饰或使用化学试剂来实现。
如何选择合适的保护碱基选择合适的保护碱基需要考虑以下几个因素: 1. 酶切位点序列:首先要了解所使用的限制性内切酶的酶切位点序列,以确定需要保护的碱基。
2. 保护方法:根据实验需求和实验条件选择合适的保护方法。
常见的保护方法包括使用化学修饰剂修饰碱基、使用特殊的核苷酸引物或引入特定的修饰基团等。
3. 保护效果:选择的保护碱基应能够有效地阻止限制性内切酶与目标位点结合并发挥催化作用。
酶切位点的概念酶切位点是指酶在特定的DNA或RNA序列上识别和切割的特定位置。
酶切位点的概念是基于酶与DNA或RNA序列之间的特异性结合性质。
酶切位点的存在使得酶能够在目标序列上识别并结合,并在特定的位点上进行切割,从而实现对DNA或RNA分子的修饰和处理。
酶切位点通常是由几个不连续的核苷酸序列组成的,这些序列在目标分子中具有特定的顺序和排列方式。
这些序列通常与酶的结构和功能密切相关,酶能够通过特异性的相互作用与目标序列结合,并在特定的位点上切割。
酶切位点的概念最早由伊丽莎白·范纳伯根(Elizabeth F. Neufeld)和赫伯特·波因特(Herbert J. Boyer)在1975年提出。
他们通过研究限制性内切酶(restriction endonuclease)对DNA的作用机制,发现酶能够识别和切割具有特定核苷酸序列的DNA分子。
这一发现为分子生物学和基因工程的发展奠定了基础。
限制性内切酶是最常用的酶切位点的研究对象,它们具有非常高的特异性和切割效率。
限制性内切酶一般通过特定的序列识别和结合DNA分子,并在该序列的特定位点上切割DNA链。
不同的限制性内切酶对应着不同的酶切位点,这些位点的序列和排列方式是独特和特异的。
酶切位点的特异性是通过酶的结构和功能来实现的。
限制性内切酶通常由两个或多个亚基组成,其中至少一个亚基与DNA序列特异性结合,而另一个亚基则负责切割DNA链。
通过特异性的相互作用,限制性内切酶能够将特定的酶切位点与其他DNA序列进行区分。
这种特异性识别和结合的机制使得限制性内切酶只作用于具有特定酶切位点的DNA分子,而不对其他DNA序列产生影响。
酶切位点在分子生物学和基因工程中有着重要的应用。
通过利用限制性内切酶对DNA的酶切作用,可以实现对DNA分子的修饰和处理。
通过选择合适的限制性内切酶和切割位点,可以将DNA分子切割为特定的片段,并获得所需的DNA 片段。
⑤酶切位点分析及引物设计酶切位点分析及引物设计是分子生物学中常用的技术手段,用于确定DNA序列中特定的酶切位点,以及设计引物来扩增目标序列。
本文将从酶切位点分析的原理、方法和应用,以及引物设计的原则和方法等方面进行介绍。
一、酶切位点分析的原理和方法酶切位点是指DNA分子中特定的序列,该序列能够被特定酶切割成两个或多个片段。
酶切位点分析的原理是将待分析的DNA序列与特定酶的识别序列进行比对,发现匹配的序列,则认定这里可能存在酶切位点。
常用的酶切酶有限制性内切酶和特异性内切酶。
限制性内切酶是一类能够识别特定DNA序列并在特定位置切割DNA分子的酶,其切割位点是特异性的;特异性内切酶则是能够在DNA序列中找到特定的序列并切割的酶。
酶切位点分析的方法包括PCR-RFLP法、Southern blotting法和测序法等。
PCR-RFLP法是通过PCR扩增DNA片段,然后用特定酶切割PCR产物,并通过凝胶电泳分析切割后的片段大小来确定酶切位点;Southern blotting法则是将DNA分子进行电泳分离,然后经过膜转移,用酶切破坏特定序列,并通过探针的杂交来确定酶切位点;测序法则是通过直接测序DNA序列,发现具有酶切位点的序列。
二、引物设计的原则和方法引物是用于扩增目标DNA序列的短链DNA片段,通常由两个互补的引物组成,分别位于目标序列的两个末端。
引物设计的原则是要确保引物与目标序列的互补度高、无二次结构、无引物间的互相结合等。
引物设计的方法主要有基本方法和计算机辅助方法。
基本方法是根据特定的DNA序列设计引物,考虑引物长度、GC含量以及互补性等因素;计算机辅助方法则是利用计算机软件根据序列的特征自动设计引物。
常用的引物设计软件有Primer3、OligoAnalyzer等,这些软件可以根据用户设定的参数,自动生成合适的引物。
引物设计的时候需要考虑引物的长度、GC含量、互补度、Tm值、引物间的互补性等参数,以确保引物能够特异性地结合到目标序列上,并且能够在PCR反应中起到良好的扩增效果。
常见限制性内切酶识别序列(酶切位点),保护碱基,缓冲液常见限制性内切酶识别序列(酶切位点),保护碱基,缓冲液在分子克隆实验中,限制性内切酶是必不可少的工具酶。
无论是构建克隆载体还是表达载体,要根据载体选择合适的内切酶(当然,使用T载就不必考虑了)。
先将引物设计好,然后添加酶切识别序列到引物5’端。
常用的内切酶比如BamHI、EcoRI、HindIII、NdeI、XhoI等可能你都已经记住了它们的识别序列,不过为了保险起见,还是得查证一下。
下面,我就总结了一些常用的内切酶的识别序列,仅供各位参考。
下面这些内切酶都属于II型内切酶。
先介绍一下什么是II型内切酶吧。
The Type II restriction systems typically contain individual restriction enzymes and modification enzymes encoded by separate genes. The Type II restriction enzymes typically recognize specific DNA sequences and cleave at constant positions at or close to that sequence to produce 5-phosphates and 3-hydroxyls. Usually they require Mg 2+ ions as a cofactor, although some have more exotic requirements. The methyltransferases usually recognize the same sequence although some are more promiscuous. Three types of DNA methyltransferases have been found as part of Type II R-M systems forming either C5-methylcytosine, N4-methylcytosine or N6-methyladenine.ApaI (类型:Type II restriction enzyme )识别序列:5'GGGCC^C 3'BamHI(类型:Type II restriction enzyme )识别序列:5' G^GATCC 3'BglII (类型:Type II restriction enzyme )识别序列:5'A^GATCT 3'EcoRI (类型:Type II restriction enzyme )识别序列:5' G^AATTC 3'HindIII (类型:Type II restriction enzyme )识别序列:5' A^AGCTT 3'KpnI (类型:Type II restriction enzyme )识别序列:5' GGTAC^C 3'NcoI (类型:Type II restriction enzyme )识别序列:5' C^CATGG 3'NdeI (类型:Type II restriction enzyme )识别序列:5' CA^TATG 3'NheI (类型:Type II restriction enzyme )识别序列:5' G^CTAGC 3'NotI (类型:Type II restriction enzyme )识别序列:5' GC^GGCCGC 3'SacI (类型:Type II restriction enzyme )识别序列:5' GAGCT^C 3'SalI (类型:Type II restriction enzyme )识别序列:5' G^TCGAC 3'SphI (类型:Type II restriction enzyme )识别序列:5' GCATG^C 3'XbaI (类型:Type II restriction enzyme )识别序列:5' T^CTAGA 3'XhoI (类型:Type II restriction enzyme )识别序列:5' C^TCGAG 3'当然,上面总结的这些肯定不全,要查找更多内切酶的识别序列,你还可以选择下面几种方法:1. 查你所使用的内切酶的公司的目录或者网站;NEB网站上提供的识别序列图表下载2. 用软件如:Primer Premier5.0或Bioedit等,这些软件均提供了内切酶识别序列的信息;3. 推荐到NEB的REBASE数据库去查(网址:/rebase/rebase.html)当你设计好引物,添加上了内切酶识别序列,下一步或许是添加保护碱基了,可以参考:/html/86.htmlNEB公司网站提供的保护碱基参考表下载/user1/2081/archives/2009/230768.shtml NEB公司网站上关于设计PCR引物保护碱基的参考/userfiles/file/protect.doc双酶切buffer的选择MBI/doubledigest/index.html罗氏http://www.roche-applied-/benchmate/refinder.htmNEB/nebecomm/DoubleDigestCalculator.as pPromega/guides/re_guide/research.asp?se arch=bufferTakara http://catalog.takara-bio.co.jp/en/product/basic_info.asp?unitid=U100005593 这里再给大家推荐一种新的不需要连接反应的分子克隆方法,优点包括:①设计引物不必考虑选择什么酶切位点;②不必考虑保护碱基的问题;③不必每次都选择合适的酶来酶切质粒制备载体;④而且不需要DNA连接酶;⑤假阳性几率低(因为没有连接反应这一步,载体自连的问题没有了)。
酶切位点(Restriction Enzyme cutting site):DNA上一段碱基的特定序列,能够识别出这个序列并在此将序列切成两段。
可能存在同尾酶,不同酶的识别序列不同,
有的可能能识别多个酶切位点比如STY1识别序列有WW
PCR引物设计时酶切位点的保护碱基表
不同内切酶对识别位点以外最少保护碱基数目的要求(在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
)
保护碱基:当酶切位点在双链DNA的尾端时,限制性内切酶经常不能成功切断(简单的想象为酶遇到识别位点之后从旁边掉下去了…… =_=|||)所以经常在引物设计时,在末端的限制性内切酶识别位点之后再加上2、3个碱基以确保成功酶切。
比如你的引物是5‘ GCTAGCNNNNN……3’ 可以改成5‘ CAGGCTAGCNNNNN……3’ 呃……CAG是我随便加的,你可以考虑一下CG/AT含量
注释
1.如果要加在序列的5’端,就在酶切位点识别碱基序列(红色)的5’端加上相应的
碱基(黑色),如果要在序列的3’端加上保护碱基,就在酶切位点识别碱基序列(红色)的3’端加上相应的碱基(黑色)。
2.切割率:正确识别并切割的效率。
3.加保护碱基时最好选用切割率高时加的相应碱基。