分子机制-蛋白与核酸作用检测-FISH
- 格式:pdf
- 大小:122.28 KB
- 文档页数:2

FISH检测:助力精准医疗发展FISH检测,即荧光原位杂交技术,是一种分子生物学检测方法。
它通过使用特定的荧光探针,检测基因、染色体异常以及基因表达水平。
在精准医疗领域,FISH检测为医生提供了强大的工具,帮助他们做出更准确的诊断和治疗决策。
在肿瘤精准医疗中,FISH检测发挥着重要作用。
例如,在非小细胞肺癌中,ALK基因重排是一种常见的分子遗传学改变。
通过FISH检测,医生可以准确地判断ALK基因是否存在重排,从而选择合适的靶向药物治疗。
一项研究表明,使用FISH检测筛选出ALK阳性的非小细胞肺癌患者,接受靶向药物治疗后的无进展生存期显著延长。
另一个例子是乳腺癌患者中的HER2基因扩增。
FISH检测可以帮助医生判断HER2基因是否扩增,从而选择是否使用HER2靶向药物治疗。
研究显示,HER2阳性的乳腺癌患者使用HER2靶向药物治疗后,无进展生存期和总生存期均显著改善。
除了在肿瘤精准医疗中的应用,FISH检测还在遗传性疾病诊断中发挥重要作用。
例如,囊性纤维化是一种常见的遗传性疾病,其发病机制与CFTR基因突变有关。
通过FISH检测,医生可以准确地判断CFTR基因是否存在突变,从而为患者提供合适的治疗方案。
然而,FISH检测在精准医疗中的应用也面临一些挑战。
例如,检测成本较高、操作复杂,且需要专业的技术人员。
FISH检测的标准化和质量控制也是亟待解决的问题。
尽管如此,随着技术的不断发展,FISH检测在精准医疗中的应用将越来越广泛。
FISH检测作为一项重要的分子生物学检测技术,在精准医疗中发挥着重要作用。
通过实际案例可以看出,FISH检测为医生提供了准确的诊断和治疗信息,从而提高了患者的生存率和生活质量。
面对挑战,我们期待未来能有更多的研究和创新,以推动FISH检测在精准医疗中的应用。
重点和难点解析:FISH检测技术在精准医疗中的应用及其价值。
FISH检测作为一种分子生物学检测方法,可以提供关于基因、染色体异常以及基因表达水平的精确信息。

病理学中的分子生物学技术随着分子生物学技术的发展,越来越多的分子生物学技术被引入到病理学中。
这些技术不仅提高了疾病诊断的准确性和精度,还为疾病治疗提供了更多的选择。
本文将探讨病理学中的分子生物学技术及其在疾病诊断和治疗中的应用。
一、PCR技术PCR(聚合酶链式反应)是一种基于DNA复制的技术,可以通过扩增少量的DNA样本来快速检测某些病原体的存在。
PCR技术在病原体检测中应用广泛,如乙肝、艾滋病、结核等疾病的检测都可以使用PCR技术。
二、FISH技术FISH(荧光原位杂交)技术是一种基于DNA分子间的杂交原理,可以在细胞或组织中检测目标DNA序列的存在和位置。
FISH技术在病理学中主要应用于染色体分析和癌症诊断中。
例如,在恶性肿瘤的诊断中,FISH技术可以检测癌细胞中是否存在某些基因的缺失或异常,从而确定治疗方案。
三、免疫组化技术免疫组化技术可以通过特异性抗体与特定蛋白质结合,从而检测细胞或组织中的蛋白质表达情况。
免疫组化技术在病理学中应用广泛,可以用于癌症诊断、炎症检测和免疫功能分析等方面。
四、基因测序技术基因测序技术是指通过对DNA序列进行分析,确定其中的基因序列和突变情况。
基因测序技术在病理学中被广泛应用于癌症诊断和治疗中。
例如,基因测序可以对恶性肿瘤中基因的突变情况进行分析,从而为精准治疗提供基础。
五、蛋白质组学技术蛋白质组学技术是指对细胞或组织中的蛋白质进行分析和研究。
蛋白质组学技术在病理学中可以用于疾病的诊断和治疗。
例如,在肿瘤治疗中,蛋白质组学技术可以帮助识别潜在的治疗靶点。
总结病理学中的分子生物学技术为疾病的诊断和治疗提供了广阔的空间,这些技术的应用可以提高疾病的诊断准确性和精度,为疾病的精准治疗提供基础。
虽然这些技术已经在病理学中应用广泛,但是技术的发展仍然需要不断地努力和探索。

生命科学中的蛋白质与核酸相互作用机制研究生命科学是一门研究生物体及其生命现象的学科,其中的蛋白质与核酸相互作用机制研究属于其中的重要领域。
蛋白质与核酸是生命体系中最为基础和常见的大分子,两者之间的相互作用可谓是生命功能调控的基础。
本文将从以下几个方面进行介绍与探讨。
一、蛋白质与核酸的概念及其结构蛋白质和核酸都是生命体系中最为重要的分子。
蛋白质是由氨基酸组成的多肽,它们在体内担任着各种结构、传递、催化以及调控功能的重任。
而核酸是生命体系中的遗传物质,形成了DNA和RNA两种不同类型的核酸,DNA负责存储遗传信息,而RNA负责将遗传信息转化为具体的功能。
蛋白质与核酸的结构也是二者相互作用的基础。
蛋白质的结构分为四个层次:一级结构指蛋白质中氨基酸的化学序列,二级结构指蛋白质在局部呈现的空间结构,常见的包括a-螺旋和b-片层,三级结构指蛋白质整体的空间结构,包括局部折叠和全局折叠,四级结构指由多个蛋白质组成的复合物。
核酸的结构也具有大的类似性。
DNA分子大部分呈现出螺旋形状,通过镶嵌在螺旋内的氢键和VanderWaals力来保持稳定。
RNA的结构则有更多的变化,可以是线性或环形结构,提供了诸如催化反应和调控遗传信息等功能。
二、蛋白质与核酸的相互作用在生命系统中,蛋白质与核酸之间的相互作用可以体现出多种生物过程,如DNA复制、转录和翻译、RNA修饰、RNA剪切以及蛋白质的折叠和降解等。
其中,DNA复制是生命系统中最为基础和重要的过程之一,它需要依靠DNA聚合酶和其他辅助因子来实现。
在DNA复制过程中,DNA聚合酶能够在模板链上识别特定的配对碱基并合成新的链,一旦出现错配会被修复酶进行纠错。
复制完成后,两个完全相同的双链DNA分子得以产生。
RNA转录也是生命系统中非常重要的过程,它可以从DNA模板中复制一份RNA分子,并且有着诸多的调控机制。
转录过程中,RNA聚合酶沿着DNA模板链滑动,在核酸序列上拼接RNA,以此形成RNA多肽序列。

FISH简介荧光原位杂交(Fluorescence In Situ Hybridization, 简称FISH)由于其直观, 快速, 敏感性⾼和⽅便灵活越来越得到⼴泛应⽤, 尤其是在⾎液学领域中. 因为⽩⾎病标本⽐较容易取得和制备, 不同类型的⽩⾎病⼜往往有其特异的染⾊体异常, FISH在⽩⾎病诊断, 治疗监测, 预后估计和微⼩残留病检测等诸⽅⾯都正成为不可缺少的重要⼿段.FISH的基本原理很简单, 就是标记了荧光的单链DNA(探针)和与其互补的DNA(玻⽚上的标本)退⽕杂交, 通过观察荧光信号在染⾊体上的位置来反映相应基因的情况. FISH探针按标记⽅法可分为直接标记和间接标记: ⽤⽣物素(biotin)或地⾼⾟(digoxingenin)标记称为间接标记, 杂交后需要通过免疫荧光抗体检测⽅能看到荧光信号, 因⽽步骤较多, 操作⿇烦, 其优点是在信号较弱或较⼩时可经抗原抗体反应扩⼤; 直接⽤荧光素标记DNA的⽅法称为直接标记. 由于直接标记的探针杂交后可马上观察到荧光信号, 省去了烦琐的免疫荧光反应, 不再需要购买荧光抗体, 也由于近年来荧光互的亮度和抗淬灭性的不断改进和提⾼, 直接标记的荧光探针越来越成为⾸选, 采⽤多种不同颜⾊的荧光, ⽅便在同⼀标本上同时检测多钟异常. 其荧光强度和信号⼤⼩都易于在普通荧光显微镜下观察, 操作过程中也不需要严格避光, 使FISH过程变得简便⽽易于操作. FISH并不能取代传统的⽩⾎病MCI诊断, 但它却能使MIC分型更为准确和深⼊. , MIC即细胞形态学(M), 免疫学和细胞遗传学(C), 三者结合对⽩⾎病进⾏分型诊断, 对不同类型的⽩⾎病采⽤不同治疗⽅案⼿段. 随着⼈们对⽩⾎病的不断认识, 仅进⾏MIC分型不够全⾯, 还要加上对⽩⾎病的分⼦(M)诊断, 成为MICM分型. FISH就是连接细胞遗传学和分⼦⽣物学的桥梁.FISH流程仪器设备1、医⽤微波炉;2、⽔浴锅;3、OLYMPUS BX51荧光显微镜;4、OLYMPUS DP11数字显微照相机。

分子病理fish技术
分子病理FISH技术是一种重要的分子生物学技术,其通过荧光染色技术检测染色体的异常及其染色体上特定序列的分布情况,从而为疾病的分子诊断提供有力的支持。
FISH技术的原理是利用DNA探针与样本的靶DNA序列的高度互补性结合,形成荧光标记的探针-靶DNA杂交体,在荧光显微镜下进行检测。
根据探针的不同种类和荧光染色物的不同组合,可以检测到染色体的不同缺失、增多、重排和散在分布等染色体异常,也可以对某些特定基因的扩增、突变、融合等变异进行检测。
FISH技术的应用范围非常广泛,涉及到多种疾病的分子诊断和预后评估。
例如,在肿瘤的分子诊断中,FISH技术可以同时检测多种基因的扩增、融合或重排,如HER2、BCL2、BCL6等,在血液病学中,FISH技术可以用来检测染色体异常及其亚克隆性,如慢性髓细胞性白血病、多发性骨髓瘤等。
FISH技术还可以应用于基因组、转录组和蛋白质组的分析。
在基因组水平,FISH技术可以用来确定基因组的大小和组织结构等;在转录组水平,FISH技术可以进行定量分析和空间分布的研究;在蛋白质组水平,FISH技术可以用来研究细胞核和染色体的空间分布和相互作用。
除了以上应用,FISH技术还可以用于辅助生殖医学,如评估胚胎染色体异常和性染色体异常等。
此外,FISH技术还可以用于动植物的基因组研究,如基因定位、染色体序列组装等。
总的来说,FISH技术在现代医学和生命科学中的应用范围非常广泛,可以为疾病的分子诊断、基因组学研究和辅助生殖医学等领域提供有力的支持。
未来,随着技术的不断发展和完善,FISH技术将会在越来越多的领域得到应用,为人类的健康和生命科学的发展贡献更大的力量。

主题:RNA-蛋白Pull-Down概述:此方法是利用脱硫生物素末端标记的RNA 和链霉亲和素磁珠标记的来高效富集RNA 结合蛋白(RBP)。
RNA-蛋白Pull-Down的原理是将RNA与链霉亲和素磁珠结合。
之后在蛋白质-RNA结合缓冲液中平衡RNA结合的磁珠,再加入蛋白裂解液。
随后添加适当的缓冲液、涡旋振荡,并在磁力架上分离,洗涤磁珠。
最后样品可通过非变性的生物素洗脱缓冲液或SDS- PAGE上样缓冲液洗脱,用于下游分析。
目的:寻找与RNA相互作用的蛋白质步骤:(1) RNA 结合到链霉亲和素磁珠1) 加入50 μl 链霉亲和素磁珠到1.5ml 的EP 管,将EP 管放入磁力架,吸去液体;2) 加入50 μl pH7.5 20mM Tris重悬磁珠;3) 重复步骤1,2;4) 将EP 管放入磁力架,吸去液体;5) 加入50 μl 1×RNA Capture Buffer 重悬磁珠;6) 加入50 pmol RNA探针到磁珠中,混匀;7) 室温孵育30min;(2) 蛋白结合到RNA1) 加入50 μl 链霉亲和素磁珠到1.5ml 的EP 管,将EP 管放入磁力架,吸去液体;2) 加入50 μl pH7.5 20mM Tris 重悬磁珠;3) 重复步骤1,2;4) 将EP 管放入磁力架,吸去液体;5) 加入100 μl1×Protein-RNA Binding Buffer 到磁珠,混匀;6)准备Protein-RNA Binding Reaction Master Mix;7) 将EP 管放入磁力架,吸去液体。
加入100 μl Master Mix,混匀;8) 4℃孵育60min;(3) 洗脱RNA-Binding protein1) 将EP 管放入磁力架,吸去液体;2) 加入100 μl 1×wash buffer,混匀,将EP 管放入磁力架,吸去液体;3) 重复步骤1,2;4) 加入50 μl Elution buffer,混匀,37℃孵育30min;5) 将EP 管放入磁力架,收集液体,-20℃保存。

FISH的基本原理及应用1. 引言FISH(Fluorescence In Situ Hybridization)是一种基于分子生物学技术的方法,用于检测染色体或细胞核酸序列的存在和位置。
本文将介绍FISH的基本原理和其在生命科学研究、临床诊断和基因组学等领域的应用。
2. FISH的基本原理FISH的基本原理是将一系列标记有荧光染料或放射性同位素的DNA或RNA探针与待测样品中的互补序列发生靶向杂交反应,从而实现对目标序列的检测和定位。
3. FISH的步骤FISH一般包括以下步骤:•取样:从样品中获取细胞或组织。
•固定:使用适当的方法固定细胞或组织,以保持其形态和结构。
•裂解:通过使用酶解等方法,将细胞或组织中的核酸释放出来。
•杂交:将标记有荧光染料的DNA或RNA探针与待测样品中的互补序列进行杂交,形成稳定的杂交复合体。
•洗涤:通过洗涤的步骤,去除未发生杂交的探针。
•显微镜观察:使用荧光显微镜等设备观察杂交信号,并记录图像。
4. FISH的应用领域FISH在多个领域中广泛应用,下面将重点介绍其在以下几个方面的应用。
4.1 生命科学研究FISH在生命科学研究中发挥重要作用,它可以帮助科学家研究染色体的结构和功能,揭示基因组的复杂性和变异性。
通过对细胞核酸序列的定位,FISH可以帮助研究者探索基因型与表型之间的关系,从而理解基因的功能和表达调控机制。
4.2 临床诊断FISH在临床诊断中应用广泛,尤其在肿瘤诊断中具有重要意义。
通过对肿瘤细胞进行FISH检测,可以鉴定染色体异常和基因突变,帮助确定诊断和预后评估。
例如,FISH可以用于检测BCR-ABL融合基因,在慢性髓系白血病的诊断和治疗方案制定中起到重要作用。
4.3 遗传学研究FISH在遗传学研究中也被广泛应用。
通过FISH技术,可以对染色体进行直接观察,从而帮助研究者了解染色体结构异常和数目异常对个体遗传特征的影响。
此外,FISH还可以帮助研究者进行染色体定位测序,从而进一步揭示基因组的组织和结构。

FISH检测在临床上的应用FISH检测在临床上的应用一、简介- FISH(Fluorescence in situ hybridization)即原位荧光杂交技术,是一种用于研究基因组和染色体结构的分子生物学技术。
- FISH检测在临床应用中可以提供重要的染色体遗传学信息,用于诊断和监测某些疾病。
二、FISH检测的原理- FISH技术基于荧光标记的核酸探针与细胞或组织标本中的特定DNA序列发生互补杂交,通过荧光显微镜观察以检测特定基因组或染色体的异常。
- FISH检测可以提供高度准确的定量和定位信息,特别适用于检测具有微小或亚显性突变的染色体异常。
三、临床应用领域1、适用于癌症诊断和分型- FISH检测可以检测染色体上的特定基因重排,从而辅助癌症的诊断、分型和治疗方案的选择。
- 例如,FISH检测可以用于检测BCR-ABL融合基因以确认慢性髓性白血病的诊断。
2、适用于先天性遗传病的筛选和诊断- FISH检测可用于检测染色体异常,如唐氏综合征(21三体综合征)、爱德华氏综合征(18三体综合征)和智商障碍相关的染色体异常,用于婴儿和先天性遗传病的筛选和诊断。
3、适用于遗传性肿瘤相关基因的检测- FISH检测可以用于检测和定位遗传性肿瘤相关基因的异常,如BRCA1和BRCA2基因异常与乳腺癌和卵巢癌的遗传风险相关。
4、适用于感染性疾病的诊断和追踪- FISH检测可以用于检测和定位细菌、和寄生虫等感染性病原体的核酸序列,辅助感染性疾病的诊断和追踪。
四、技术要点和注意事项1、样本处理- 对于固定的细胞或组织标本,需要进行脱脂、脱水等预处理步骤。
- 不同类型的标本可能需要不同的处理方法,如骨髓涂片、组织切片等。
- 样本处理过程中需注意保持核酸完整性和避免污染。
2、探针设计与标记- 根据要检测的目标基因或染色体序列设计特异性的核酸探针。
- 核酸探针需要选择适当的荧光染料进行标记,以便在荧光显微镜中观察到。
- 标记的探针需要存储在适当的条件下,以保持标记稳定性和活性。

生物化学中核酸和蛋白质的交互作用生物化学中,核酸和蛋白质是两种最基本的生物大分子,它们分别承担着遗传信息的传递和生物化学反应的催化等重要功能。
而核酸与蛋白质之间的相互作用,则是许多生物过程中不可或缺的环节。
一、核酸与蛋白质相互作用的形式和功能核酸与蛋白质之间的相互作用可以分为三种主要形式:一是核酸和蛋白质之间的物理作用,即电荷相互作用、范德华力和疏水作用等;二是核酸和蛋白质之间的结构上的相互作用;三是核酸和蛋白质之间的化学作用,即酶反应。
这些相互作用可以产生许多的生物功能。
例如,某些核酸可以通过与特定蛋白质结合,调节基因转录和翻译过程;另外一些核酸和蛋白质结合可以形成某些酶,在生物化学反应中担任催化剂等。
二、蛋白质识别核酸的基本原理在生物过程中,蛋白质与核酸的相互作用很大程度上依赖于它们之间的空间构象。
蛋白质要识别和结合到核酸上,需要细致的空间匹配。
具体来说,蛋白质通过具有亲和力的氨基酸残基与核酸上的碱基或磷酸基团相互作用,从而实现与核酸的结合。
此外,还有一些重要的氨基酸残基可以在蛋白质-核酸相互作用时起到关键作用。
例如,核酸结合蛋白质中一些亲酸性氨基酸(如精氨酸和赖氨酸)可以通过与核酸上的过氧酰基或磷酸酯键形成离子键或氢键等静电相互作用;而一些碳水化合物结合蛋白质中的赖氨酸残基则可以通过与DNA上的基团形成一个氢键和一个离子键来促进蛋白质与DNA结合。
三、核酸识别蛋白质的基本原理相比蛋白质识别核酸,核酸识别蛋白质非常困难。
不仅如此,在实际的生物过程中,核酸多半不能够独立的关联和结合到蛋白质上。
其中一些较大的核酸分子(如染色质)需要先通过一些特定的辅酶(如组蛋白)形成紧密的团块,才可以识别和组合到蛋白质上。
在核酸识别蛋白质的过程中,DNA倾向于被特定类型的亲酸性氨基酸残基所识别。
这些亲酸性氨基酸残基通常是组成蛋白质大分子的多肽链的一部分。
例如,在基于基序DNA识别的转录因子中,存在着许多亲酸性氨基酸,如精氨酸和赖氨酸,它们通过调整其体内电荷来辅助识别与结合到基序DNA上。

FISH技术的基本原理FISH(Fluorescence In Situ Hybridization)技术是一种在细胞和组织水平上直接可视化和定位特定DNA序列的方法。
它是一种专门用于检测染色体异常、基因缺失或复制数变异、基因重排等的分子生物学技术。
FISH技术的基本原理是利用专门设计的探针与目标DNA序列的互补配对。
这些探针通常是由碱基对序列特异性的荧光分子标记的缩合物组成。
探针的选择性地结合到DNA的目标序列上,并发射出特定颜色的荧光信号,从而实现对该DNA序列的定位和可视化。
首先,需要将探针与荧光标记结合。
这通常通过在探针序列的5'或3'端引入荧光染料或其他标记物实现。
荧光标记的探针可以根据荧光的颜色和光谱特性选择合适的标记染料。
其次,细胞或组织样品需要进行固定,以保持细胞的形态和结构完整。
当然,固定方法的选择会根据具体需求而有所不同。
然后,需要对DNA进行解旋处理。
解旋可以通过加热或化学处理等方式来破坏DNA的双链结构,使探针能够与目标DNA序列发生有效的杂交反应。
随后,探针与DNA目标序列进行杂交。
杂交可以在温度和离子浓度适当的缓冲液中进行,以提高探针与目标DNA序列的互补结合能力。
存在足够长的杂交时间,使得探针能够与目标DNA序列稳定结合,并且特异性高。
最后,样品在显微镜下观察和分析。
荧光显微镜可以通过激发荧光染料,观察到荧光信号的发射和位置。
可以使用多色探针同时进行多个DNA序列的检测和定位。
FISH技术的原理允许在细胞和组织水平上可视化和定位目标DNA序列。
它已广泛应用于医学诊断、研究和遗传学等领域。
例如,在癌症研究中,FISH技术可用于检测癌基因或抑癌基因的缺失、重排或增多,帮助确定肿瘤的类型和分级;在遗传学中,FISH技术可用于检测染色体异常、基因剪接和拷贝数变异;在生殖医学中,FISH技术可用于评估胚胎植入前的染色体异常和遗传病风险。
总之,FISH技术是一种基于探针与目标DNA序列的互补配对,并通过荧光信号标记和显微镜观察,实现对DNA序列的可视化和定位。
实用标准文案常见血液肿瘤FISH检测小册文档大全实用标准文案文档大全一.FISH是什么FISH即荧光原位杂交技术(Fluorescence in situ hybridization),是在细胞遗传学水平上检测染色体及基因数目和结构异常的一种分子病理检测技术。
其基本原理是利用标记了荧光素的核酸作为探针,按照碱基互补原则,与待检样本中与之互补的核酸经过变性-退火而形成杂交双链核酸,然后通过荧光显微镜进行检测和分析。
FISH技术具有直观、快速、敏感性高和方便灵活等特点,目前已经广泛应用于临床的肿瘤遗传学及各种基因相关疾病的分型与个体化治疗等多个领域。
实用标准文案二.血液肿瘤的诊断分型MICM:血液肿瘤诊断的精确分型是临床选择正确治疗方案的前提,目前国际上通用的是结合细胞形态学(Morphology)、免疫学(Immunology)、细胞遗传学(Cytogenetics)和分子生物学(Molecular)的MICM分型。
形态学诊断(Morphology)细胞学:外周血、骨髓涂片,淋巴结穿刺组织学:骨髓、淋巴结活检文档大全实用标准文案●免疫学检查(Immunology)免疫组化(IHC),流式细胞(FCM)●细胞遗传学检查(Cytogenetics)核型分析,荧光原位杂交(FISH)●分子生物学检查(Molecular)PCR,DNA测序三.FISH技术的作用及优势FISH作为一项很重要的分子遗传学检测技术,在血液肿瘤的诊断中有很大的作用,得到了NCCN等国内外各大血液肿瘤诊疗指南的认可和建议。
目前与血液肿瘤相关的FISH探针有接近100种,常用的就有60种左右,包括急慢性白血病、骨髓增生异常综合症(MDS)、多发性骨髓瘤(MM)、淋巴瘤等多种血液肿瘤。
文档大全实用标准文案FISH技术的相对优势:●FISH技术更敏感,可检测出核型分析检测不出的细微缺失或易位,如MDS中的5q缺失综合征;●FISH技术不需要中期分裂相细胞,而核型分析需要培养时间较长且需要较高的操作要求;●FISH技术是在细胞形态的基础上进行结果判读,可有效地降低假阴性或假阳性的风险,而PCR技术经过倍增扩增,不能在形态的基础上判读,对实验要求高,易产生假阴性或假阳性。
fish技术在病理诊断中的应用病理诊断是医学领域中重要的一环,通过观察和分析组织及细胞的形态、结构和功能来确定疾病的类型和特征。
近年来,一种新的分子生物学技术——fish技术(Fluorescence in situ hybridization)逐渐应用于病理诊断中,为病理医师提供了更加准确和有效的诊断手段。
fish技术是一种基于DNA或RNA的分子探针与细胞或组织中特定序列的互补配对而发生的荧光信号的检测技术。
fish技术可以用于检测染色体异常、基因扩增、基因融合等分子水平的变化,从而帮助病理医师确定疾病的诊断和预后。
fish技术在肿瘤病理学中具有广泛的应用。
例如,在肺癌中,fish 技术可以检测EGFR基因突变、ALK基因融合等特定基因的异常,从而为选择靶向治疗提供重要依据。
此外,fish技术还可用于乳腺癌、胃癌、卵巢癌等肿瘤的分子诊断,提高了对肿瘤类型和预后的判断准确性。
fish技术在遗传病的诊断中也有重要作用。
遗传病是由基因突变引起的疾病,fish技术可以帮助病理医师确定染色体异常和基因突变,进而诊断遗传病。
例如,在唐氏综合征的诊断中,fish技术可以检测21号染色体上的三体,确认疾病的存在。
此外,fish技术还可以用于先天性心脏病、遗传性肾病等遗传病的诊断。
fish技术还在微生物学领域有着重要应用。
传统的微生物学诊断方法需要进行细菌培养和鉴定,耗时且存在一定的假阴性结果。
而fish技术可以直接检测细菌、病毒等微生物的核酸序列,提高了微生物学诊断的准确性和迅速性。
例如,在结核病的诊断中,fish技术可以检测结核分枝杆菌的核酸序列,快速确定病原体,缩短了诊断时间。
fish技术还可用于研究染色体结构和功能。
通过fish技术,可以观察和分析染色体的形态和变异,探究染色体异常与疾病发生的关系。
fish技术作为一种新的分子生物学技术,在病理诊断中的应用前景广阔。
它可以帮助病理医师准确定位和诊断疾病的分子变化,提高了病理诊断的准确性和敏感性。
FISH技术的基本原理FISH(Fluorescence In Situ Hybridization)技术是一种基于核酸杂交原理的细胞遗传学技术,主要用于检测和定位细胞或组织中特定的DNA序列。
FISH技术具有高分辨率、高特异性和高灵敏度的特点,在细胞遗传学、生物医学研究和临床诊断中广泛应用。
1.样品制备:首先,需要提取和标记要分析的DNA序列。
样品可以是细胞悬液、组织切片或染色体悬液。
提取样品中的DNA后,将标记的探针与待测DNA杂交,以便检测和定位目标DNA序列。
2.探针标记:在FISH技术中,常用的探针包括DNA或RNA探针。
这些探针通常通过标记荧光染料进行标记,以便在显微镜下进行可视化。
标记可以通过直接标记或间接标记来完成。
直接标记是在DNA或RNA探针上直接连接荧光染料,而间接标记则是在DNA或RNA探针上连接其中一种分子,如生物素或荧光素,然后再使用荧光染料与这些分子发生特异性反应。
3.样品杂交:将标记的探针与样品中的DNA序列进行杂交。
在标记的探针与样品的目标DNA序列碱基互补配对后,形成稳定的DNA双链。
这种DNA探针与目标DNA序列的配对是高度特异性的,因此可以准确地检测和定位目标DNA序列。
4.组织固定:在杂交反应完成后,需要对样品进行固定,以稳定探针与目标DNA序列的结合和形态学结构。
常用的固定方法包括对细胞进行凝胶固定或使用特定的固定剂,如4%的乙醛。
5.洗涤:通过洗涤的步骤去除杂交反应剩余的非特异性杂交产物和杂交探针无法结合的杂质。
洗涤一般使用含有缓冲盐的溶液,如盐溶液或有机溶剂的混合物。
6.显像:使用荧光显微镜观察特定的荧光信号。
标记的探针会发出特定的荧光信号,可以根据不同荧光染料的特点,在不同的通道下观察和记录。
FISH技术通过在细胞或组织样本中直接进行DNA或RNA杂交来检测和定位特定的DNA序列。
通过使用不同颜色的荧光标记,可以同时检测多个目标序列。
此外,FISH技术还可以通过结合核型分析来检测染色体异常和基因突变。
FISH原理范文FISH原理是一种基于荧光原位杂交技术的分子生物学方法,它可以用来检测和定位细胞中的特定核酸序列。
FISH的全称是Fluorescence in situ hybridization,即荧光原位杂交。
该技术的原理是使用标记亲和性探针,与目标核酸序列特异性结合,通过观察荧光信号来确定目标序列在细胞核中的位置。
1.样本处理:将要检测的细胞标本或组织切片固定在载玻片上,并进行预处理步骤,如破碎细胞壁、去除细胞中的蛋白质、使细胞透明等。
2.探针制备:根据目标序列的特点,设计和合成特异性的DNA探针。
探针可以通过化学标记(如荧光染料、生物素等)来识别和检测目标序列。
3.杂交反应:将探针与标本中的DNA序列进行杂交反应,使探针与目标序列特异性结合。
在此过程中,探针的荧光染料或生物素标记可以被目标序列结合并固定在细胞核中。
4.信号检测:通过荧光显微镜或成像系统观察和记录探针结合的荧光信号。
不同的荧光染料或生物素标记可以用来标识不同的目标序列,从而实现多重染色。
FISH技术具有以下特点和优势:1.高空间分辨率:FISH技术可以在细胞核水平上直接观察和定位目标序列,对细胞遗传学和基因组学的研究具有重要意义。
2.高特异性:通过设计和选择特异性的DNA探针,FISH可以检测和定位具有特定序列的DNA或RNA分子。
3.多重染色:通过使用不同的荧光染料或生物素标记,FISH可以同时检测和定位多个目标序列,从而提供更丰富的信息。
FISH技术在生物医学研究和临床诊断中已得到广泛应用。
它可以用于研究基因组结构和功能,发现和定位基因突变,探索染色体异常与疾病之间的关系,以及评估肿瘤细胞中的染色体变异等。
例如,FISH技术在癌症诊断和预后评估中有重要应用。
通过使用特异性的DNA探针,FISH可以在肿瘤组织中检测和定位具有特定重要意义的基因突变。
这些突变可能与肿瘤的发生、发展和治疗反应等相关联。
因此,FISH可以用于协助肿瘤的早期诊断、分子亚型鉴定、预后判断和个体化治疗。
DNA荧光原位杂交(FISH)简要综述DNA荧光原位杂交(FISH)简要综述DNA荧光原位杂交(FISH)技术是70年代末80年代初开始发展起来的一种重要的非放射性原位杂交技术,它的基本原理是将DNA探针用特殊修饰的核苷酸分子标记(如biotin-dUTP或digoxigenin-dUTP),然后将标记的探针直接原位杂交到染色体或DNA纤维切片上,再用与荧光素分子偶联的单克隆抗体与探针分子特性结合来检测DNA序列在染色体或DNA纤维上的定位。
利用FISH进行DNA序列的定位具有实验周期短、灵敏度高、分辨率高、直观可见等优点。
总的说来,FISH技术的发展沿着两条路线前进:一方面是采用不同的探针,从而衍生出许多FISH新技术,如Muticolor-FISH、CGH、GISH、CISS、BAC-FISH、Chromosome Painting、Rreverse Chromosome Painting等等;另一方面则是努力提高FISH技术的分辨率,将靶目标从中期染色体发展到DNA纤维,使其分辨率由1Mb 发展到1kb,这更进一步拓展了FISH技术的应用领域,成为分子细胞遗传学的一项代表技术。
A.不同探针的应用:1.以基因组为探针的GISH技术可以定位外源DNA片段在染色体上的位置、大小、插入点等。
Durnam(1985)首先将GISH应用于体细胞杂种的异源染色体检测。
本实验室采用GISH方法在小麦中成功定位了许多外源染色体、染色体片段以及染色体结构变异。
2.以不同的荧光素标记探针的Muticolor-FISH,可以同时定位不同探针序列的分布。
1990年Nederlof等创建了多色荧光原位杂交技术。
他们用生物素、AAF(氨基乙酰荧光素)和CP三种半抗原对不同探针作单、双、三标记,再用这三种半抗原相应的分别标记了异硫氰酸荧光素(FITC,绿色)、氨甲香豆素乙酸(AMCA,蓝色)和碱性蕊香红(TRITC,红色)的抗体来检测荧光,该技术最多可同时观察7个靶染色体。
(完整版)Fish实验FISH-荧光原位杂交实验(原位杂交)实验目的通过实验了解荧光原位杂交技术的基本原理和在生物学、医学领域的应用。
掌握原位杂交技术的操作方法,熟练掌握荧光显微镜的使用方法。
2. 实验原理荧光原位杂交(Fluorescence in situ hybridization FISH)是一门新兴的分子细胞遗传学技术,是20世纪80年代末期在原有放射性原位杂交技术的基础上发展起来的一种非放射性原位杂交技术。
目前这项技术已经广泛应用于动植物基因组结构研究、染色体精细结构变异分析、病毒感染分析、人类产前诊断、肿瘤遗传学和基因组进化研究等许多领域。
FISH的基本原理是用已知的标记单链核酸为探针,按照碱基互补的原则,与待检材料中未知的单链核酸进行特异性结合,形成可被检测的杂交双链核酸。
由于DNA分子在染色体上是沿着染色体纵轴呈线性排列,因而可以将探针直接与染色体进行杂交从而将特定的基因在染色体上定位。
与传统的放射性标记原位杂交相比,荧光原位杂交具有快速、检测信号强、杂交特性高和可以多重染色等特点,因此在分子细胞遗传学领域受到普遍关注。
杂交所用的探针大致可以分为三类:1)染色体特异重复序列探针,例如a卫星、卫星III类的探针,其杂交靶位常大于1Mb,不含散在重复序列,与靶位结合紧密,杂交信号强,易于检测;2)全染色体或染色体区域特异性探针,其由一条染色体或染色体上某一区段上极端不同的核苷酸片段所组成,可由克隆到噬菌体和质粒中的染色体特异大片段获得;3)特异性位置探针,由一个或几个克隆序列组成。
探针的荧光素标记可以采用直接和间接标记的方法。
间接标记是采用生物系标记的dUTP(biotin-dUTP)经过缺口平移法进行标记,杂交之后用藕联的荧光素的抗生物系的抗体进行检测,同时还可以利用几轮抗生物素蛋白—荧光素、生物素化的抗—抗生物素蛋白、抗生物素蛋白—荧光素的处理,将荧光信号进行放大,从而可以检测500bp的片段。
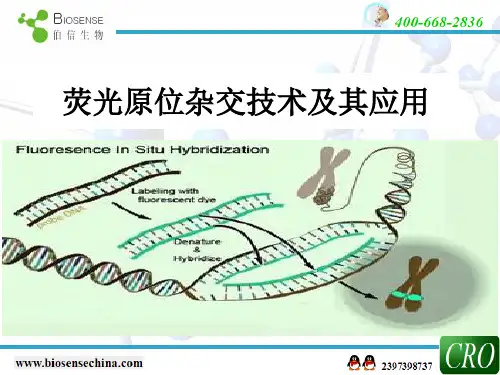
主题:荧光原位杂交技术(Fluorescence in situ hybridization,FISH)
概述:
原位杂交技术是以核酸分子碱基互补配对的原理为基础的实验技术。
通过标记的核酸分子(探针,Probe)与目标核酸分子杂交,再用相应的方法检测探针的存在,从而揭示靶部位目标分子的位置。
最早的原位杂交技术出现于1969年,Gall和Pardue 利用H3标记的核糖体RNA与非洲爪蟾的卵母细胞杂交,揭示了核糖体RNA基因的基因扩增现象(Gall&Pardue,1969)。
早期的原位杂交技术使用放射性标记,存在诸多弊端。
1985年Rayburn等首次将生物素(Biotin)标记的重复序列定位到小麦的中期分裂相上(Rayburn&Gill,1985),开创了非放射性标记的杂交技术。
由于非放射性标记的原位杂交技术具有安全、经济、灵敏度高等优点,因而迅速发展起来。
荧光原位杂交技术(Fluorescence in situ hybridization,FISH)是指利用荧光信号检测探针的原位杂交技术。
广义上讲,FISH靶序列可以是DNA或者RNA,既可以用来进行基因组层面的研究,也可以进行表达层面的研究。
目的:
用于基因定性、定量、整合、表达等方面的研究。
原理:
FISH技术的基本原理是基于核酸分子碱基配对的原则,将染色体或细胞核DNA与探针分别变性后,将探针杂交至靶部位,然后使用荧光显微镜检测杂交信号。
探针可以直接用荧光物质标记(直接法),也可以使用一些抗原物质标记,然后采用荧光标记的抗体与探针上的标记物结合(间接法)。
相对而言,间接法荧光原位杂交相对应用更广。
步骤:
1.FISH样本的制备
2.探针的制备
3.探针标记
4.杂交
5.染色体显带
6.荧光显微镜检测
7.结果分析
流程图:。