MIT数据库详解
- 格式:doc
- 大小:25.50 KB
- 文档页数:5


MIT心律失常数据库包含两个系列的心电数据,第一系列即“100”系列,是在4000个24小时的Holter 记录中随机挑选的,包含23个数据(100~109,111~ 119,121~124);第二系列即“200”系列,是挑选的不太常见但临床上十分重要的心律失常数据,包含25个数据(200~203,205,207~210,212~215,217,2 19~223,228,230~234)。
其中102,104,107,217为Paced beats,207含有部分VF信号,201~203,2 10,217,219,221~222含有AF信号。
每个数据持续30分钟,并都有详细的注释。
MIT心律失常数据库每一个数据记录包括三个文件,“.hea”、“.dat”与“.atr”。
“.hea”为头文件,其由一行或多行ASCII码字符组成。
以10 0.hea为例第一行从左到右分别代表文件名,导联数,采样率,数据点数;第二行从左到右分别代表文件名,存储格式,增益,AD分辨率,ADC零值,导联1第一个值,校验数,数据块大小(0=可以从任意数据块输出,即可以从中间读取任意一段),导联号第三行代表导联2的信息,同第二行以#开始的为注释行,一般说明患者的情况以及用药情况等。
“.dat”为数据文件,MIT-BIH数据库中的数据存储格式有Fo rmat8、Format16、Format80、Format212、Format310等8种,心律失常数据库统一采用212格式进行存储。
“212”格式是针对两个信号的数据库记录,这两个信号的数据交替存储,每三个字节存储两个数据。
这两个数据分别采样自信号0与信号1,信号0的采样数据取自第一字节对(16位)的最低12位,信号1的采样数据由第一字节对的剩余4位(作为组成信号1采样数据的12位的高4位)与下一字节的8位(作为组成信号1采样数据的12位的低8位)共同组成。
以100.da t为例。


mit-bih数据库格式摘要:1.mit-bih 数据库格式简介2.mit-bih 数据库格式的特点3.mit-bih 数据库的应用领域4.mit-bih 数据库的获取与使用5.mit-bih 数据库在我国的研究现状及前景正文:mit-bih 数据库格式是一个广泛应用于生物医学领域的数据库格式,其中包含了大量的心电图数据。
这种格式具有高度的结构化,能够有效地存储和传输心电图数据,因此在医学研究和临床实践中得到了广泛的应用。
mit-bih 数据库格式的特点主要体现在其数据的标准化和规范化上。
所有的数据都严格按照特定的格式进行存储,这不仅有利于数据的读取和解析,也有利于数据的比较和分析。
同时,mit-bih 数据库格式还支持数据的快速检索和精确查询,大大提高了数据的使用效率。
由于其独特的优点,mit-bih 数据库格式在生物医学领域得到了广泛的应用。
除了用于研究正常和异常的心电图模式外,它还被用于开发和测试心电图分析算法,以及评估心电图设备的精度和性能。
此外,mit-bih 数据库格式还被用于心电图的大数据分析,以发现新的疾病规律和治疗策略。
对于研究人员和临床医生来说,获取和使用mit-bih 数据库格式是一个重要的问题。
目前,mit-bih 数据库格式的主要获取途径是通过官方网站下载,也可以通过数据库共享平台获取。
在使用过程中,需要使用特定的软件工具进行数据的读取和解析。
在我国,mit-bih 数据库格式也得到了广泛的研究和应用。
许多研究机构和医疗机构都开展了基于mit-bih 数据库格式的研究项目,并在心电图分析、疾病预测等方面取得了显著的成果。


世界主要心电数据库简介以下内容为小弟个人总结,有不准确的地方敬请指正!目前国际上最重要的,具有权威性的心电数据库有四个:美国麻省理工学院与Beth Israel医院联合建立的MIT-BIH心电数据库;美国心脏学会的AHA心律失常心电数据库;欧盟的CSE心电数据库和欧盟ST-T心电数据库。
除此之外国际上被广泛认可的还有Sudden Cardiac DeathHolter Database,PTB Diagnostic ECG Database,PAF Prediction ChallengeDatabase等心电数据库。
一、数据库介绍美国的MIT-BIH心电数据库美国的MIT-BIH心电数据库是目前在国际上应用最多的数据库,由很多子数据库组成,每个子数据库包含某类特定类型的心电记录,其中应用的最多的是MIT-BIT心律不齐数据库和MIT-BIT QT数据库。
自1999年,在美国国家研究资源中心和国家健康研究院的支持下,他们将该数据库公开到了Internet上,整个MIT-BIT数据库的所有数据都可以免费下载和使用,国内外许多心电方面的研究都是基于该数据库的,使用该数据库作为实验数据的来源和各类识别算法的检测标准。
其包括:MIT-BIH Arrhythmia Database、MIT-BIH Noise Stress Test Database、MIT-BIH Atrial Fibrillation Database、MIT-BIH ECG Compression Test Database、MIT-BIH Long-Term Database、MIT-BIH Malignant Ventricular Arrhythmia Database、MIT-BIH Normal Sinus Rhythm Database、MIT-BIH ST Change Database、MIT-BIH Supraventricular Arrhythmia Database部分子库的参数:MIT-BIH Arrhythmia Database诊断: Arrhythmia采样率:360 Hz分辨率:11 bit导联:2每段数据持续时间:30 min储存格式:Format 212MIT-BIH ST Change Database诊断:Recorded during exercise stress tests and which exhibit transient ST depression采样率:360 Hz分辨率:12 bit导联:2每段数据持续时间:varying lengths储存格式:Format 212MIT-BIH Atrial Fibrillation Database诊断:Atrial fibrillation (mostly paroxysmal)采样率:250 Hz分辨率:12 bit导联:2每段数据持续时间:10 h储存格式:Format 212网址:/AHA心律失常心电数据库由美国国家心肺及血液研究院资助的美国心脏协会(American HeartAssociation,AHA)开发了AHA心律失常心电数据库,该数据库的开发目的是评价室性心律不齐探测器的检测效果。

MIT心律失常数据库数据格式解析MIT心律失常数据库包含两个系列的心电数据,第一系列即“100”系列,是在4000个24小时的Holter记录中随机挑选的,包含23个数据(100~109,111~119,121~124);第二系列即“200”系列,是挑选的不太常见但临床上十分重要的心律失常数据,包含25个数据(200~203,205,207~210,212~215,217,219~223,228,230~234)。
其中102,104,107,217为Paced beats,207含有部分VF信号,201~203,210,217,219,221~222含有AF信号。
每个数据持续30分钟,并都有详细的注释。
MIT心律失常数据库每一个数据记录包括三个文件,“.hea”、“.dat”和“.atr”。
“.hea”为头文件,其由一行或多行ASCII码字符组成。
以100.hea为例第一行从左到右分别代表文件名,导联数,采样率,数据点数;第二行从左到右分别代表文件名,存储格式,增益,AD分辨率,ADC零值,导联1第一个值,校验数,数据块大小(0=可以从任意数据块输出,即可以从中间读取任意一段),导联号第三行代表导联2的信息,同第二行以#开始的为注释行,一般说明患者的情况以及用药情况等。
“.dat”为数据文件,MIT-BIH数据库中的数据存储格式有Format8、Format16、Format80、Format212、Format310等8种,心律失常数据库统一采用212格式进行存储。
“212”格式是针对两个信号的数据库记录,这两个信号的数据交替存储,每三个字节存储两个数据。
这两个数据分别采样自信号0和信号1,信号0的采样数据取自第一字节对(16位)的最低12位,信号1的采样数据由第一字节对的剩余4位(作为组成信号1采样数据的12位的高4位)和下一字节的8位(作为组成信号1采样数据的12位的低8位)共同组成。
以100.dat为例。

MIT心律失常数据库数据格式解析MIT心律失常数据库包含两个系列的心电数据,第一系列即“100”系列,是在4000个24小时的Holter记录中随机挑选的,包含23个数据(100~109,111~119,121~124);第二系列即“200”系列,是挑选的不太常见但临床上十分重要的心律失常数据,包含25个数据(200~203,205,207~210,212~215,217,219~223,228,230~234)。
其中102,104,107,217为Paced beats,207含有部分VF信号,201~203,210,217,219,221~222含有AF信号。
每个数据持续30分钟,并都有详细的注释。
MIT心律失常数据库每一个数据记录包括三个文件,“.hea”、“.dat”和“.atr”。
“.hea”为头文件,其由一行或多行ASCII码字符组成。
以100.hea为例第一行从左到右分别代表文件名,导联数,采样率,数据点数;第二行从左到右分别代表文件名,存储格式,增益,AD分辨率,ADC零值,导联1第一个值,校验数,数据块大小(0=可以从任意数据块输出,即可以从中间读取任意一段),导联号第三行代表导联2的信息,同第二行以#开始的为注释行,一般说明患者的情况以及用药情况等。
“.dat”为数据文件,MIT-BIH数据库中的数据存储格式有Format8、Format16、Format80、Format212、Format310等8种,心律失常数据库统一采用212格式进行存储。
“212”格式是针对两个信号的数据库记录,这两个信号的数据交替存储,每三个字节存储两个数据。
这两个数据分别采样自信号0和信号1,信号0的采样数据取自第一字节对(16位)的最低12位,信号1的采样数据由第一字节对的剩余4位(作为组成信号1采样数据的12位的高4位)和下一字节的8位(作为组成信号1采样数据的12位的低8位)共同组成。
以100.dat 为例。

python database模块详解Python database模块详解数据库是计算机系统中用来存储、管理和查询大量数据的工具。
Python作为一门强大的编程语言,提供了多种数据库模块来帮助开发者连接、操作和管理数据库。
本文将以中括号为主题,一步一步详细介绍Python数据库模块的使用。
一、为何使用数据库模块在开发过程中,我们经常需要与数据库进行交互,数据库模块提供了一种简单而直接的方式来连接和操作数据库。
使用数据库模块可以节约开发时间,提供更高效的数据存储和查询方式,同时也支持多种数据库类型。
二、Python常用的数据库模块1. SQLite3(轻量级数据库)SQLite3是一种嵌入式数据库引擎,它不需要独立的服务器进程,以库的形式被链接到Python程序中。
Python内置了SQLite3模块,可以直接使用。
2. MySQL(开源关系型数据库)MySQL是一种关系型数据库,被广泛应用于Web应用程序开发。
Python提供了多个MySQL驱动模块,如mysql-connector-python、pymysql等。
3. PostgreSQL(高级开源关系型数据库)PostgreSQL是一种功能强大的开源关系型数据库,具有良好的性能和可扩展性。
Python提供了多个PostgreSQL驱动模块,如psycopg2、PyGreSQL等。
4. Oracle(企业级关系型数据库)Oracle是一种功能强大的企业级关系型数据库,广泛应用于企业系统中。
Python 提供了cx_Oracle模块,用于连接和操作Oracle数据库。
5. NoSQL数据库除了传统的关系型数据库外,Python还支持多种NoSQL数据库,如MongoDB、Redis等。
对于这些数据库,可以使用相应的Python模块进行开发。
三、使用数据库模块的基本步骤无论使用哪种数据库模块,一般的连接和操作步骤都类似。
下面以SQLite3为例来介绍基本的步骤。
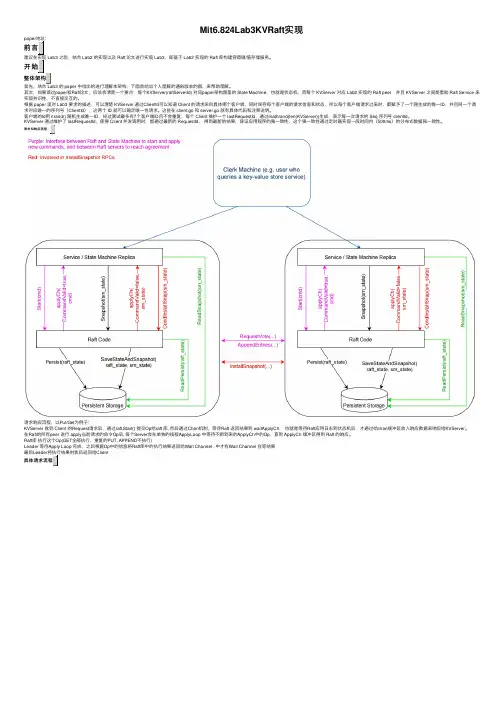
Mit6.824Lab3KVRaft实现paperLab3 之前,结合 Lab2 的实现以及 Raft 论⽂进⾏实现 Lab3,即基于 Lab2 实现的 Raft 库构建容错键/值存储服务。
的 paper 中给出的进⾏理解本架构,下⾯会给出个⼈理解的通俗版本的图,来帮助理解。
其次,如果读过paper和Raft论⽂,应该会清楚⼀个要点:每个KVServer(raftServerId) 对应paper架构图⾥的 State Machine,也就是状态机,⽽每个 KVServer 对应 Lab2 实现的 Raft peer,并且 KVServer 之间是借助 Raft Service 来实现共识性,不直接交互的。
根据 paper ⾥对 Lab3 要求的描述,可以清楚 KVServer 通过ClientId可以知道 Client 的请求来⾃具体哪个客户端,同时保存每个客户端的请求信息和状态,所以每个客户端请求过来时,都赋予了⼀个刚⽣成的唯⼀ID,并且同⼀个请求对应唯⼀的序列号(ClientId),这两个 ID 就可以确定唯⼀性请求。
这些在 client.go 和 server.go 就有具体代码和注释说明。
客户端的Id⽤ nrand() 随机⽣成唯⼀ID,经过测试最多有7个客户端ID且不会重复,每个 Client 维护⼀个 lastRequestId,通过mathrand(len(KVServer))⽣成,表⽰每⼀次请求的 Seq 序列号 clientId。
KVServer lastRequestId,使得 Client 并发调⽤时,能通过最新的 RequestId,得到最新的结果,保证应⽤程序的强⼀致性,这个强⼀致性通过定时器实现⼀段时间内(500ms)的分布式数据强⼀致性。
请求和响应流程请求响应流程,以Put/Get为例⼦:KVServer 收到 Client 的Request请求后,通过raft.Start() 提交Op给raft 库, 然后通过Chan机制,等待Raft 返回结果到 waitApplyCh,也就是等待Raft应⽤⽇志到状态机后,才通过给chan缓冲区放⼊响应数据来响应给KVServer。

美国芝加哥大学出版社 (University of Chicago Press)美国芝加哥大学出版社University of Chicago Press成立于1891年,曾被时任美国芝加哥大学校长William Rainey Harper称为大学的一个“有机部分”,很大程度上扩大了芝加哥大学的学者们在全世界范围的影响力。
如今芝加哥大学拥有50多种期刊和硬皮本文献,为全世界的读者提供来自天文学、生物、医学和植物学领域内顶尖科学家们的最新研究成果。
【使用指南】HighWire Press简介:HighWire出版社是美国斯坦福大学创立。
它自称拥有全球最大的免费全文学术文献库。
通过题名作者/组配/ topic map进行检索。
包含学科:生命科学,医学,物理学,社会科学。
1129种期刊4737282全文1876531篇免费【使用指南】BioMed Central BioMed Central(以下简称BMC)是生物医学领域的一家独立的新型出版社,以出版网络版期刊为主,目前出版170种生物学和医学领域的期刊(不断增加中),少量期刊同时出版印刷版。
BMC出版社基于“开放地获取研究成果可以使科学进程更加快捷有效”的理念,坚持在BMC网站免费为读者提供信息服务,其出版的网络版期刊可供世界各国的读者免费检索、阅读和下载全文,开放存取期刊评审水平较高。
【使用指南】PloS免费期刊简介:公共科学图书馆(PLOS)是一家由众多诺贝尔奖得主和慈善机构支持的非赢利性学术组织,旨在推广世界各地的科学和医学领域的最新研究成果。
PLoS出版了8种生命科学与医学领域的开放获取期刊,可以免费获取全文,比较具有影响力。
包含学科:生物医学(生物医学计算生物遗传学病原体 ONE等)J-STAGE 简介: J-STAGE ( Japan Science and Technology Information Aggregator , Electronic, 日本电子科技信息服务)向全世界即时发布日本科学技术研究的杰出成果和发展,它出版的期刊涉及到各个学科领域。
sqlalchemy用法SQLAlchemy是一个功能强大的Python库,用于在Python应用程序中操作关系型数据库。
它提供了一种高级抽象的方式来管理数据库连接和执行查询,同时允许开发人员使用标准的SQL语句来操作数据库。
本文将详细介绍SQLAlchemy的用法,并提供一步一步的指南,以便读者可以轻松地理解和使用这个强大的工具。
一、安装SQLAlchemy要使用SQLAlchemy,首先需要安装它。
可以通过以下命令使用pip安装SQLAlchemy:pip install sqlalchemy安装完成后,就可以导入SQLAlchemy库并开始使用它了。
import sqlalchemy二、建立数据库连接在使用SQLAlchemy之前,首先需要建立与数据库的连接。
可以使用`create_engine`函数来创建一个连接。
以下是一个示例:pythonfrom sqlalchemy import create_engineengine = create_engine('数据库引擎和连接字符串')这里的'数据库引擎和连接字符串'需要根据你使用的具体数据库类型和配置来进行设置。
比如,对于MySQL数据库,可以使用以下方式来创建连接:pythonengine =create_engine('mysql:username:password@localhost/database_na me')这里的`username`是你的MySQL用户名,`password`是你的密码,`localhost`是你的MySQL服务器地址,`database_name`是你要连接的数据库的名称。
三、定义数据库模型在开始执行数据库操作之前,我们需要定义数据库表格的模型。
SQLAlchemy使用ORM(对象关系映射)的方式,允许我们将数据库表格映射成Python类,并通过操作这些类来执行数据库操作。
% This programm reads ECG data which are saved in format 212.% (e.g., 100.dat from MIT-BIH-DB, cu01.dat from CU-DB,...)% The data are displayed in a figure together with the annotations.% The annotations are saved in the vector ANNOT, the corresponding% times (in seconds) are saved in the vector A TRTIME.% The annotations are saved as numbers, the meaning of the numbers can% be found in the codetable "ecgcodes.h" available at . %% ANNOT only contains the most important information, which is displayed % with the program rdann (available on ) in the 3rd row. % The 4th to 6th row are not saved in ANNOT.%%% created on Feb. 27, 2003 by% Robert Tratnig (V orarlberg University of Applied Sciences)% (email:***************),%% algorithm is based on a program written by% Klaus Rheinberger (University of Innsbruck)% (email:*************************.at)%% -------------------------------------------------------------------------clc; clear all;%------ SPECIFY DA TA ------------------------------------------------------PA TH= 'G:\ecg\mit-cd'; % path, where data are savedHEADERFILE= '100.hea'; % header-file in text formatA TRFILE= '100.atr'; % attributes-file in binary formatDA TAFILE='100.dat'; % data-fileSAMPLES2READ=3000; % number of samples to be read% in case of more than one signal:% 2*SAMPLES2READ samples are read%------ LOAD HEADER DA TA -------------------------------------------------- fprintf(1,'\\n$> WORKING ON %s ...\n', HEADERFILE);signalh= fullfile(PA TH, HEADERFILE);fid1=fopen(signalh,'r');z= fgetl(fid1);A= sscanf(z, '%*s %d %d %d',[1,3]);nosig= A(1); % number of signalssfreq=A(2); % sample rate of dataclear A;for k=1:nosigz= fgetl(fid1);A= sscanf(z, '%*s %d %d %d %d %d',[1,5]);dformat(k)= A(1); % format; here only 212 is allowedgain(k)= A(2); % number of integers per mVbitres(k)= A(3); % bitresolutionzerovalue(k)= A(4); % integer value of ECG zero pointfirstvalue(k)= A(5); % first integer value of signal (to test for errors)end;fclose(fid1);clear A;%------ LOAD BINARY DA TA --------------------------------------------------if dformat~= [212,212], error('this script does not apply binary formats different to 212.'); end; signald= fullfile(PA TH, DA TAFILE); % data in format 212fid2=fopen(signald,'r');A= fread(fid2, [3, SAMPLES2READ], 'uint8')'; % matrix with 3 rows, each 8 bits long, = 2*12bitfclose(fid2);M2H= bitshift(A(:,2), -4); %字节向右移四位,即取字节的高四位M1H= bitand(A(:,2), 15); %取字节的低四位PRL=bitshift(bitand(A(:,2),8),9); % sign-bit 取出字节低四位中最高位,向右移九位PRR=bitshift(bitand(A(:,2),128),5); % sign-bit 取出字节高四位中最高位,向右移五位M( : , 1)= bitshift(M1H,8)+ A(:,1)-PRL;M( : , 2)= bitshift(M2H,8)+ A(:,3)-PRR;if M(1,:) ~= firstvalue, error('inconsistency in the first bit values'); end;switch nosigcase 2M( : , 1)= (M( : , 1)- zerovalue(1))/gain(1);M( : , 2)= (M( : , 2)- zerovalue(2))/gain(2);TIME=(0:(SAMPLES2READ-1))/sfreq;case 1M( : , 1)= (M( : , 1)- zerovalue(1));M( : , 2)= (M( : , 2)- zerovalue(1));M=M';M(1)=[];sM=size(M);sM=sM(2)+1;M(sM)=0;M=M';M=M/gain(1);TIME=(0:2*(SAMPLES2READ)-1)/sfreq;otherwise % this case did not appear up to now!% here M has to be sorteddisp('Sorting algorithm for more than 2 signals not programmed yet!');end;clear A M1H M2H PRR PRL;fprintf(1,'\\n$> LOADING DA TA FINISHED \n');%------ LOAD A TTRIBUTES DA TA ---------------------------------------------- atrd= fullfile(PA TH, A TRFILE); % attribute file with annotation data fid3=fopen(atrd,'r');A= fread(fid3, [2, inf], 'uint8')';fclose(fid3);A TRTIME=[];ANNOT=[];sa=size(A);saa=sa(1);i=1;while i<=saaannoth=bitshift(A(i,2),-2);if annoth==59ANNOT=[ANNOT;bitshift(A(i+3,2),-2)];A TRTIME=[A TRTIME;A(i+2,1)+bitshift(A(i+2,2),8)+...bitshift(A(i+1,1),16)+bitshift(A(i+1,2),24)];i=i+3;elseif annoth==60% nothing to do!elseif annoth==61% nothing to do!elseif annoth==62% nothing to do!elseif annoth==63hilfe=bitshift(bitand(A(i,2),3),8)+A(i,1);hilfe=hilfe+mod(hilfe,2);i=i+hilfe/2;elseA TRTIME=[A TRTIME;bitshift(bitand(A(i,2),3),8)+A(i,1)];ANNOT=[ANNOT;bitshift(A(i,2),-2)];end;i=i+1;end;ANNOT(length(ANNOT))=[]; % last line = EOF (=0)A TRTIME(length(A TRTIME))=[]; % last line = EOFclear A;A TRTIME= (cumsum(A TRTIME))/sfreq;ind= find(A TRTIME <= TIME(end));A TRTIMED= A TRTIME(ind);ANNOT=round(ANNOT);ANNOTD= ANNOT(ind);%------ DISPLA Y DA TA ------------------------------------------------------ figure(1); clf, box on, hold onplot(TIME, M(:,1),'r');if nosig==2plot(TIME, M(:,2),'b');end;for k=1:length(A TRTIMED)text(A TRTIMED(k),0,num2str(ANNOTD(k)));end;xlim([TIME(1), TIME(end)]);xlabel('Time / s'); ylabel('V oltage / mV');string=['ECG signal ',DA TAFILE];title(string);fprintf(1,'\\n$> DISPLAYING DA TA FINISHED \n');% ------------------------------------------------------------------------- fprintf(1,'\\n$> ALL FINISHED \n');。
mimic-iii数据库的申请MIMIC-III数据库的申请随着医疗技术的不断发展,医疗数据的积累和分析变得越来越重要。
为了促进医疗研究和临床实践的进步,MIT(麻省理工学院)开发了MIMIC-III(Medical Information Mart for Intensive Care III)数据库,这是一个开放的、免费的临床数据库,其中包含了数万例ICU(重症监护病房)患者的多种数据。
MIMIC-III数据库的申请过程相对简便,但需要遵守一定的规则和条件。
首先,申请人需要在MIMIC-III官方网站上填写申请表格,并提供一份研究计划或项目描述。
这一步是为了确保申请人对数据库的使用有明确的目的,并且能够正确和合理地运用数据库中的数据。
在填写申请表格时,申请人需要提供一些基本信息,如姓名、联系方式以及所属机构等。
此外,申请人还需要说明自己对医学和数据分析的背景和经验,以及使用MIMIC-III数据库的目的和意义。
这些信息对于评估申请人的资质和研究计划的可行性非常重要。
在提交申请后,申请人需要等待MIMIC-III数据库管理员的审查和批准。
一般来说,审批的时间不会太长,但也有可能需要几周的时间。
审批通过后,申请人将收到一封包含数据库访问链接和密码的确认邮件。
通过确认邮件中的链接和密码,申请人可以登录MIMIC-III数据库的在线平台,开始对数据进行检索和分析。
MIMIC-III数据库包括了大量的临床数据,如患者的基本信息、生理参数、实验室检验结果、用药记录等。
申请人可以根据自己的需求,选择合适的数据进行研究和分析。
在使用MIMIC-III数据库时,申请人需要遵守一些使用规则和道德准则。
首先,申请人需要保护患者的隐私和数据安全,不得将数据库中的个人信息泄露或滥用。
其次,申请人需要遵守数据使用的合法性要求,不得将数据用于非法或违反伦理的目的。
此外,申请人还需要在发表研究成果时,注明数据来源于MIMIC-III数据库,并遵守相关的引用规范。
MIT心律失常数据库包含两个系列的心电数据,第一系列即“100”系列,是在4000个24小时的Holter记录中随机挑选的,包含23个数据(100 ~109,111~119,121~124);第二系列即“200”系列,是挑选的不太常见但临床上十分重要的心律失常数据,包含25个数据(200~203,205,20 7~210,212~215,217,219~223,228,230~234)。
其中102,104,107,217为Paced beats,207含有部分VF信号,201~203,210,217,219,22 1~222含有AF信号。
每个数据持续30分钟,并都有详细的注释。
MIT心律失常数据库每一个数据记录包括三个文件,“.hea”、“.dat”和“.atr”。
“.hea”为头文件,其由一行或多行ASCII码字符组成。
以100.hea为例
第一行从左到右分别代表文件名,导联数,采样率,数据点数;
第二行从左到右分别代表文件名,存储格式,增益,AD分辨率,ADC零值,导联1第一个值,校验数,数据块大小(0=可以从任意数据块输出,即可以从中间读取任意一段),导联号
第三行代表导联2的信息,同第二行
以#开始的为注释行,一般说明患者的情况以及用药情况等。
“.dat”为数据文件,MIT-BIH数据库中的数据存储格式有Format8、Format16、Format80、Forma t212、Format310等8种,心律失常数据库统一采用212格式进行存储。
“212”格式是针对两个信号的数据库记录,这两个信号的数据交替存储,每三个字节存储两个数据。
这两个数据分别采样自信号0和信号1,信号0的采样数据取自第一字节对(16位)的最低12位,信号1的采样数据由第一字节对的剩余4位(作为组成信号1采样数据的12位的高4位)和下一字节的8位(作为组成信号1采样数据的12位的低8位)共同组成。
以100.dat为例。
按照“212”的格式,从第一字节读起,每三个字节(24 位)表示两个值,第一组为“E3 33 F3”,两个值则分别为0x3E3和0x3F3转换为十进制分别为995和1011,代表的信号幅度分别为4.975m v(995/200,值/增益)和5.055mv,这两个值分
别是两个信号的第一采样点,后面依此类推,分别表示了两个信号的采样值。
“.atr”为注释文件,记录了心电专家对相应的心电信号的诊断信息,主要有两种格式:MIT格式和AHA格式。
MIT格式是一种紧凑型格式,每一注释的长度占用偶数个字节空间,多数情况下是占用两个字节,多用于在线的注释文件;而AHA格式的每一注释占用16个字节的空间,多用于交换文件的情况。
这两种格式的在文件中的区分决定于前两个字节的值,若文件的第一字节不为0或第二字节等于“[”(0x5B)或“]”(0x5D),则该文件是以MIT格式存储的,否则是按AHA格式存储的。
心律失常数据库采用的MIT格式。
MIT格式,每一注释单元的前两个字节的第一个字节为最低有效位,16位中的最高6位表示了注释类型代码(具体代码见后表),剩余的10位说明了该注释点的发生时间或辅助信息,若为发生时间,其值为该注释点到前一注释点的间隔(对于第一个注释点为从记录开始到该点的间隔),若为辅助信息则说明了附加信息的长度。
以100.atr为例。
从文件中的第一字节不为0可以判断该文件是以MIT格式存储的。
从第一字节开始按照MIT 格式进行分析,首先读出16位值0x7012,其高6位的值为0x1C(十进制28),低10位的值为0x12,该类型代码为28,代表意义是节律变化,发生时间在0.05秒(18/360Hz);接着读出后面的16位值0xFC03,其高6为的值为0x3F(十进制63),低10位的值为0x03,该类型代码为63,代表的意义是在该16位值后附加了3个(低10位值代表的数)字节的辅助信息,若字节个数为奇数,则再附加一个字节的空值,在本例中就是“28 4E 00 00”;然后再从下一字节读16位值0x043B,其高6位的值为1,低10位的值为0x3B(十进制59),该类型码1代表正常心搏,发生时间为0. 213秒((18+59)/360Hz);依次类推即可读出所有的注释,当读到的16位值为0时,就表示到了文件尾。
另,当高6位为十进制59时,读取之后第3个1 6位的高6位,作为类型代码,读取之后第二个1 6位+第一个16位*2^16,作为发生时间;
高6位为十进制60,61,62时,继续读下一个1 6位。
采用WFDB转换的AHA数据库atr注释,第一个字节为0,其读取方式同MIT格式一致,可采用相同的方式读取。