全基因组关联分析共44页文档
- 格式:ppt
- 大小:4.08 MB
- 文档页数:44


全基因组关联分析(Genome-wide association study;GWAS)是应用基因组中数以百万计的单核苷酸多态性(single nucleotide ploymorphism ,SNP)为分子遗传标记,进行全基因组水平上的对照分析或相关性分析,通过比较发现影响复杂性状的基因变异的一种新策略。
随着基因组学研究以及基因芯片技术的发展,人们已通过GWAS方法发现并鉴定了大量与复杂性状相关联的遗传变异。
近年来,这种方法在农业动物重要经济性状主效基因的筛查和鉴定中得到了应用。
全基因组关联方法首先在人类医学领域的研究中得到了极大的重视和应用,尤其是其在复杂疾病研究领域中的应用,使许多重要的复杂疾病的研究取得了突破性进展,因而,全基因组关联分析研究方法的设计原理得到重视。
人类的疾病分为单基因疾病和复杂性疾病。
单基因疾病是指由于单个基因的突变导致的疾病,通过家系连锁分析的定位克隆方法,人们已发现了囊性纤维化、亨廷顿病等大量单基因疾病的致病基因,这些单基因的突变改变了相应的编码蛋白氨基酸序列或者产量,从而产生了符合孟德尔遗传方式的疾病表型。
复杂性疾病是指由于遗传和环境因素的共同作用引起的疾病。
目前已经鉴定出的与人类复杂性疾病相关联的SNP位点有439 个。
全基因组关联分析技术的重大革新及其应用,极大地推动了基因组医学的发展。
(2005年, Science 杂志首次报道了年龄相关性视网膜黄斑变性GWAS结果,在医学界和遗传学界引起了极大的轰动, 此后一系列GWAS陆续展开。
2006 年, 波士顿大学医学院联合哈佛大学等多个研究机构报道了基于佛明翰心脏研究样本关于肥胖的GWAS结果(Herbert 等. 2006);2007 年, Saxena 等多个研究组联合报道了与2 型糖尿病( T2D ) 关联的多个位点, Samani 等则发表了冠心病GWAS结果( Samani 等. 2007); 2008 年, Barrett 等通过GWAS发现了30 个与克罗恩病( Crohns ' disrease) 相关的易感位点; 2009 年, W e is s 等通过GWAS发现了与具有高度遗传性的神经发育疾病——自闭症关联的染色体区域。

生物大数据技术的全基因组关联分析方法近年来,随着生物大数据技术的快速发展,全基因组关联分析方法已成为生物学、医学研究领域中的重要工具。
全基因组关联分析(GWAS)是一种寻找基因与某一特定性状或疾病之间相互关联的分析方法。
本文将介绍全基因组关联分析的原理和方法,并探讨其在研究中的应用和挑战。
全基因组关联分析的基本原理是将多个个体的基因组数据与其具体的性状或疾病状态进行比较,寻找基因位点与性状或疾病之间的关联。
这种分析方法的关键在于基因型-表型关联的检测。
在全基因组关联分析中,研究对象通常是单核苷酸多态性(SNP)位点,因为SNP是个体基因组中最常见的变异类型。
全基因组关联分析方法通常包括以下几个步骤。
首先,收集研究对象的基因组数据和相关性状或疾病的表型数据。
其次,通过基因组测序技术或芯片技术对个体的基因组进行分析,得到其SNP位点的基因型数据。
然后,通过统计学方法计算基因型与表型之间的关联。
最后,对这些关联进行统计分析,判断是否存在显著的关联信号。
在全基因组关联分析中,常用的统计学方法包括卡方检验、线性回归分析和逻辑回归分析等。
卡方检验适用于疾病的风险和基因型之间的关联分析;线性回归和逻辑回归分析则适用于连续性和二分性表型特征的关联分析。
不同的统计方法适用于不同的研究问题和数据类型。
全基因组关联分析方法在生物学、医学研究中的应用广泛。
它可以揭示基因变异与疾病发生发展之间的关系,有助于发现潜在的疾病风险基因和药物靶标。
全基因组关联分析还可以帮助了解个体在药物代谢、药物反应和药物副作用方面的差异,实现个体化医疗的目标。
此外,全基因组关联分析还可以为遗传病的早期筛查和诊断提供重要依据。
然而,全基因组关联分析也存在一些挑战。
首先,全基因组关联分析需要大样本量来获得可靠的结果,并且需要考虑到样本的种族和人口结构,以避免虚假关联的出现。
其次,全基因组关联分析结果需要进行复制实验来验证其确切性。
此外,全基因组关联分析还需要解决对多个检验进行校正和纠正,以降低虚假关联的发生概率。


全基因组关联分析(Genome-wide association study;GWAS)是应用基因组中数以百万计的单核苷酸多态性(single nucleotide ploymorphism,SNP)为分子遗传标记,进行全基因组水平上的对照分析或相关性分析,通过比较发现影响复杂性状的基因变异的一种新策略。
随着基因组学研究以及基因芯片技术的发展,人们已通过GWAS方法发现并鉴定了大量与复杂性状相关联的遗传变异。
近年来,这种方法在农业动物重要经济性状主效基因的筛查和鉴定中得到了应用。
全基因组关联方法首先在人类医学领域的研究中得到了极大的重视和应用,尤其是其在复杂疾病研究领域中的应用,使许多重要的复杂疾病的研究取得了突破性进展,因而,全基因组关联分析研究方法的设计原理得到重视。
人类的疾病分为单基因疾病和复杂性疾病。
单基因疾病是指由于单个基因的突变导致的疾病,通过家系连锁分析的定位克隆方法,人们已发现了囊性纤维化、亨廷顿病等大量单基因疾病的致病基因,这些单基因的突变改变了相应的编码蛋白氨基酸序列或者产量,从而产生了符合孟德尔遗传方式的疾病表型。
复杂性疾病是指由于遗传和环境因素的共同作用引起的疾病。
目前已经鉴定出的与人类复杂性疾病相关联的SNP位点有439个。
全基因组关联分析技术的重大革新及其应用,极大地推动了基因组医学的发展。
(2005年, Science杂志首次报道了年龄相关性视网膜黄斑变性 GWAS结果,在医学界和遗传学界引起了极大的轰动,此后一系列GWAS陆续展开。
2006年, 波士顿大学医学院联合哈佛大学等多个研究机构报道了基于佛明翰心脏研究样本关于肥胖的 GWAS结果 (Herbert等. 2006);2007年, Saxena等多个研究组联合报道了与 2型糖尿病( T2D )关联的多个位点, Samani等则发表了冠心病 GWAS结果( Samani 等. 2007); 2008年, Barrett等通过 GWAS发现了 30个与克罗恩病( Crohns ' disrease)相关的易感位点; 2009年, W e is s等通过 GWAS发现了与具有高度遗传性的神经发育疾病——自闭症关联的染色体区域。

全基因组关联分析的方法与应用全基因组关联分析(GWAS)是一种采用大样本数量和高密度的基因检测技术,通过寻找基因和表型之间的关联,发现对人类疾病表型贡献的基因变异。
GWAS是人类遗传学和疾病学领域中的一个重大发现,为基因疾病学、基因组医学、以及个性化治疗提供了可靠的理论基础。
GWAS的实验方法是对多个样本进行基因测序,通过对数据进行比对,从数百万个基因中筛选出与表型相关的基因变异。
GWAS的数据处理往往需要使用多个算法,将数据整合,以便得到最准确的结果。
对于GWAS定位到的基因变异,研究人员通常会运用其他实验技术进一步验证其功能和生物学意义,并探究其与特定表型之间的关系。
GWAS的应用领域非常广泛,包括心血管疾病、糖尿病、癌症、眼科疾病、免疫系统疾病和神经系统疾病。
其中,心血管疾病是GWAS最早的应用领域之一。
例如,GWAS研究发现了在心血管疾病中具有风险地位的基因,例如APOE、TCF7L2 和CETP脂蛋白。
目前,疾病治疗中根据基因组数据设计的个性化治疗方案已经被广泛应用。
GWAS研究的终极目标是了解基因变异如何导致疾病,探索更好的治疗方法。
GWAS的发现使得医学迈向了基于基因组的个性化治疗时代,而不是以往的基于症状诊断的治疗方式。
例如,在药物治疗领域,通过GWAS发现在药物代谢途径中的基因多态性,医生可以预测患者对药物的响应和耐受性,并制定更准确的个性化治疗方案,有效提高疗效并降低不良反应的风险。
然而, GWAS也存在一些局限性和挑战。
首先,GWAS需要大量标本和高通量技术、较长时间和高昂经费,因此 GWAS 研究的费用非常昂贵。
其次,许多具有重要生物学意义的基因变异并没有被 GWAS 研究所涵盖,这些基因变异往往具有较低的频率和较小的效应大小,无法被当前的 GWAS 技术所检测。
最后,GWAS所找到的相关位点与表型间的相关并不意味着直接的因果关系,GWAS只能揭示关系,实际具体机制需要进一步研究和探索。



“全基因组关联研究”文件汇整目录一、中国人群食管癌风险预测模型研究及全基因组关联研究数据挖掘二、猪肉质性状的全基因组关联研究三、不同SNP分型技术在猪基因组选择中效果评估及全基因组关联研究四、全基因组关联研究的深度分析策略五、全基因组关联研究现状六、基于全基因组关联研究的中国女性乳腺癌风险预测模型研究中国人群食管癌风险预测模型研究及全基因组关联研究数据挖掘食管癌是全球范围内一种常见的恶性肿瘤,其发病率和死亡率均较高。
中国是食管癌的高发地区之一,因此,针对中国人群开展食管癌风险预测模型研究和全基因组关联研究数据挖掘具有重要意义。
本文旨在探讨中国人群食管癌风险预测模型研究及全基因组关联研究数据挖掘的方法和结果。
本研究采用基于人群的研究方法,选取了中国某地区食管癌高发区和非高发区的居民为研究对象。
通过问卷调查收集人口学、生活习惯、家族史等信息,并采集血液样本进行基因分型。
利用统计模型(如Logistic回归模型)构建食管癌风险预测模型,并利用交叉验证等技术评估模型的预测性能。
本研究利用全基因组关联研究(GWAS)的方法,对大规模的基因组数据进行深入分析,以挖掘与食管癌发病相关的遗传变异。
通过GWAS 数据分析,可以发现与食管癌风险相关的单核苷酸多态性(SNP),并进一步挖掘其生物学机制。
本研究构建的食管癌风险预测模型具有良好的预测性能,其中AUC(曲线下面积)为85,表明该模型能够有效地识别出食管癌高风险人群。
通过该模型,我们可以预测个体患食管癌的风险,并为早期筛查和预防提供依据。
本研究通过GWAS数据分析,发现了多个与食管癌风险相关的SNP。
其中,一些SNP位于基因位点上,这些基因涉及到细胞周期调控、DNA 修复、信号转导等生物学过程。
这些发现为深入探讨食管癌的发病机制提供了重要线索。
本研究通过构建食管癌风险预测模型和全基因组关联研究数据挖掘,取得了以下重要发现:我们成功地构建了一个预测性能良好的食管癌风险预测模型,这为早期识别和预防食管癌提供了有力工具;通过GWAS数据分析,我们发现了一些与食管癌风险相关的SNP,这些SNP 涉及到的生物学过程与食管癌的发生发展密切相关。

“全基因组关联分析”资料合集目录一、全基因组关联分析在作物农艺性状研究中的应用二、玉米12个农艺性状的全基因组关联分析及玉米氮响应相关基因的鉴定三、全基因组关联分析在水稻遗传育种中的应用和研究进展四、支气管哮喘的全基因组关联分析研究进展五、水稻苗期稻瘟病抗性的全基因组关联分析六、全基因组关联分析的进展与反思七、甘蓝型油菜分枝角度和株高全基因组关联分析八、基于SNP芯片和全测序数据的奶牛全基因组关联分析和基因组选择研究九、桃基因组及全基因组关联分析研究进展全基因组关联分析在作物农艺性状研究中的应用一、引言在过去的十年中,随着基因测序技术的飞速发展,全基因组关联分析(Genome-wide Association Study,GWAS)已成为研究作物农艺性状的重要工具。
作物农艺性状是指作物在生长发育过程中表现出的形态、生理和产量等特征,这些性状通常受到多个基因的控制,并且会受到环境因素的影响。
通过GWAS,我们可以识别与特定农艺性状相关的基因变异,进一步理解作物生长发育的规律,并为作物育种提供重要的指导。
二、全基因组关联分析的原理和方法GWAS的基本原理是利用单核苷酸多态性(SNP)作为分子标记,通过比较不同品种或群体中SNP位点的差异,来寻找与特定农艺性状相关的基因变异。
在作物研究中,常用的方法包括基因组重测序和基因组扫描。
基因组重测序是对作物种质资源进行全基因组测序,以获取高精度的基因型信息。
基因组扫描则是利用已发表的SNP数据和农艺性状数据,进行大规模的关联分析。
三、全基因组关联分析在作物农艺性状研究中的应用1、作物产量:通过GWAS,研究者已经识别了许多与作物产量相关的基因变异。
例如,在玉米中,与产量相关的基因变异被发现与植物生长和发育的多个阶段有关,包括叶片大小、节间长度和花粉传播等。
这些发现为提高作物产量提供了重要的理论依据。
2、作物抗病性:GWAS也被广泛应用于研究作物的抗病性。
例如,在小麦中,研究者发现了一种与对白粉病抗性相关的基因变异。
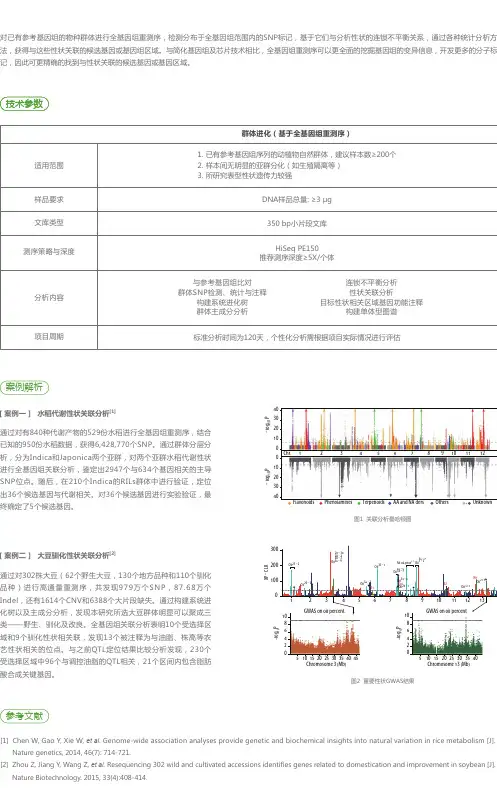
全基因组关联分析(GWAS)在硕士就读期间,就已经做过 GWAS 相关的分析。
当时标记量非常少, windows 系统分析就足够了,作图方面涉及的脚本也基本是蔡师兄帮写的。
后来,随着高通量测序成本的降低,标记数量越来越多,不得不进入linux 和脚本操作的时代,因此我也陆陆续续的学习了R 和 Python等编程语言,但是在编程的世界里,只是一个小菜鸟,大部分的脚本都是“借来的”。
而此次 GWAS方面的相关内容基本取材于百迈客云课堂。
1、基本概念全基因组关联分析(Genome wideassociation study,GWAS)是对多个个体在全基因组范围的遗传变异(标记)多态性进行检测,获得基因型,进而将基因型与可观测的性状,即表型,进行群体水平的统计学分析,根据统计量或显著性 p 值筛选出最有可能影响该性状的遗传变异(标记),挖掘与性状变异相关的基因。
image.png相对于连锁分析的优势· 关联定位的相对优势:· 1)分辨率高(单碱基水平)· 2)研究材料来源广泛,可捕获的变异丰富· 3)节省时间关联分析的基础-连锁不平衡(LD)当位于某一座位的特定等位基因与另一座位的某一等位基因同时出现的概率大于群体中因随机分布的两个等位基因同时出现的概率时,就称这两个座位处于连锁不平衡状态(linkage disequilibrium)image.pngr2 和 D'· r2和D’反映了LD的不同方面。
r2包括了重组和突变,而D’只包括重组史。
D’能更准确地估测重组差异,但样本较小时,低频率等位基因组合可能无法观测到,导致LD强度被高估,所以D’不适合小样本群体研究;· LD衰减作图中通常采用r2来表示群体的LD水平;· Haplotype Block中通常采用D’来定义Block;· 迁移、突变、选择、有限的群体大小以及其他引起等位基因频率改变的因素都会引起LD的改变。
随着基因组学研究以及基因芯片技术的发展,人们已通过GWAS方法发现并鉴定了大量与复杂性状相关联的遗传变异。
近年来,这种方法在农业动物重要经济性状主效基因的筛查和鉴定中得到了应用。
全基因组关联方法首先在人类医学领域的研究中得到了极大的重视和应用,尤其是其在复杂疾病研究领域中的应用,使许多重要的复杂疾病的研究取得了突破性进展,因而,全基因组关联分析研究方法的设计原理得到重视。
人类的疾病分为单基因疾病和复杂性疾病。
单基因疾病是指由于单个基因的突变导致的疾病,通过家系连锁分析的定位克隆方法,人们已发现了囊性纤维化、亨廷顿病等大量单基因疾病的致病基因,这些单基因的突变改变了相应的编码蛋白氨基酸序列或者产量,从而产生了符合孟德尔遗传方式的疾病表型。
复杂性疾病是指由于遗传和环境因素的共同作用引起的疾病。
目前已经鉴定出的与人类复杂性疾病相关联的SNP位点有439个。
全基因组关联分析技术的重大革新及其应用,极大地推动了基因组医学的发展。
(2005年, Science杂志首次报道了年龄相关性视网膜黄斑变性 GWAS结果,在医学界和遗传学界引起了极大的轰动,此后一系列GWAS陆续展开。
2006年, 波士顿大学医学院联合哈佛大学等多个研究机构报道了基于佛明翰心脏研究样本关于肥胖的 GWAS结果 (Herbert等. 2006);2007年, Saxena等多个研究组联合报道了与 2型糖尿病( T2D )关联的多个位点, Samani等则发表了冠心病 GWAS结果( Samani 等. 2007); 2008年, Barrett等通过 GWAS发现了 30个与克罗恩病( Crohns ' disrease)相关的易感位点; 2009年, W e is s等通过 GWAS发现了与具有高度遗传性的神经发育疾病——自闭症关联的染色体区域。
我国学者则通过对 12 000多名汉族系统性红斑狼疮患者以及健康对照者的GWAS发现了 5个红斑狼疮易感基因, 并确定了 4个新的易感位点( Han 等. 2009)。
全基因组关联分析及其在疾病研究中的应用全基因组关联分析(genome-wide association study,GWAS)是一种流行的遗传分析技术,它能够识别与疾病相关的基因序列。
该技术利用DNA芯片、测序等技术,分析人类基因组中数百万个单核苷酸多态性(SNP)位点,从而找出与某种特定疾病有关的基因。
GWAS的基本原理是通过对大样本群体的基因组数据进行多维度分析,找出具有统计显著性的基因变异与疾病的关联,从而探索疾病的遗传机制。
GWAS能够研究不同人群之间在基因结构上的差异,揭示不同人群患病的原因。
它是遗传流行病学领域的重要进展,已经成功地识别出与多种常见疾病有关的基因变异。
在GWAS中,研究者通过比较大量疾病患者和非患者的基因数据,寻找这些患者与非患者之间在特定基因组区域中的常见细微变异。
这些变异,或称作突变,有时与患病有关,或至少能够增加患病的风险。
通过比较患者和非患者的基因序列,研究者可以确定哪些基因可能与特定疾病相关,并且基于这些结果,设计新的治疗方法或药物。
GWAS被广泛应用于多种疾病的研究中,如糖尿病、阿尔茨海默病、癌症等。
其中,目前最成功的应用就体现在研究单倍体遗传病,如囊性纤维化、异常血性状等方面。
GWAS的应用将成为人类遗传学和药物研发的新道路。
除此之外,GWAS也能够用于人类进化史的研究中。
在比较不同民族的基因序列时,我们可以找到一些基因变异,这些变异可能指示了人类进化上的一些关键转折点,包括人类祖先的迁移、人类的种族分化,以及亚洲、欧洲、非洲等地区的历史。
总之,全基因组关联分析是一种现代方法,能够发现基因变异与疾病、特定种族、人类进化等之间的关系。
随着技术的不断发展,GWAS将会成为研究人类遗传学和疾病的重要手段,为研究人类健康提供强有力的支撑。
全基因组关联分析(Genome-wide association study or GWAS)人类基因包含着百万种序列变异,它们对于疾病的形成或者对患者药物的反应程度有直接或间接的影响.全基因组关联分析是指在人类全基因组范围内找出存在的序列变异,即单核苷酸多态性(SNP),从中筛选出与疾病相关的部分。
此项技术能够一次性对疾病进行轮廓性概览,在全基因组层面上,开展多中心、大样本、反复验证基因与疾病的关联研究,全面揭示疾病发生、发展,以及与治疗相关的遗传基因。
随着人类基因组学的大幅度进步和基因测序的飞速进展,这种最新的研究方式开始大规模应用于筛选与人群复杂疾病和药物特异性相关的序列变异。
进行全基因组关联分析研究时,通过采集某类疾病患者与非患者两类人群的DNA,在基因芯片上读出DNA中的序列变异,然后用生物工程技术进行分析比较。
若某些基因变异在患者人群中非常普遍,则该序列变异是与此种疾病‘相关’的。
有了全基因组关联分析,今后从事疾病诊断,患者对药物的反应程度的研究,可以集中于这些与疾病‘相关’的序列变异,从而显著缩短研究时间,提高研究效率。
全基因组关联分析是研究人类复杂疾病的一项重大突破,其优势在于:1 高通量 --- 一个反应监测成百上千个序列变异;2 不只局限于“候选基因”,基因可以是“未知”的;3无需在研究之前构建任何假设。
2005年,Science杂志报道了第一项具有年龄相关性的黄斑变性全基因组关联分析研究,之后陆续出现有关冠心病、肥胖病、II型糖尿病、甘油三酯、精神分裂症以及相关表型的报道。
由此可见,全基因组关联分析研究作为一种全新的疾病研究方式,自人类基因测序大规模展开以来,就被医学界广泛接受和应用。
截止到2010年12月,世界范围内进行了超过1200项针对200多种疾病的全基因组关联分析研究,找到4000多个‘相关’的序列变异。
在全基因组关联分析研究中,SNP基因芯片(SNP array)扮演了非常重要的角色。